hpRNA的制备新方法及其在RNAi载体中的应用
刘晓宁

摘要[目的]验证制备新方法所获得的稳定hpRNA是否可用于RNAi载体的构建并达到基因表达干扰的效果。[方法]利用制备新方法(2 μL DNA与8 μL H2O混匀,95 ℃ 5 min,37 ℃ 15 min)制备hpRNA,后采用REGS方法构建ZmMDAR RNAi载体,通过Grx1roGFP2和荧光实时定量PCR检测基因表达干扰的效果。[结果]利用制备新方法制备50 nt hpRNA。ZmMDAR基因的表达量变化和GrxlroGFP2探针的荧光比值变化表明hpRNA介导的干扰载体引起了原生质体氧化还原状态的变化。[结论]通过制备新方法所制备的hpRNA为RNAi载体所介导的干扰作用奠定基础。
关键词发夹RNA;REGS;ZmMDAR RNAi;Grx1roGFP2
中图分类号Q78文献标识码A文章编号0517-6611(2016)04-175-03
A Prepared Method of hpRNA and Its Application for RNAi Vector
LIU Xiaoning(Medical College,Huanghe S&T College, Zhengzhou, Henan 450063)
Abstract[Objective] The aim was to test a method if it is applied for the RNAi vector and lead to interference effect. [Method] We prepared hpRNA by the new method (2 μL DNA and 8 μL H2O mixed, 95 ℃, 5 min, 37 ℃,15 min), constructed ZmMDAR RNAi vector with REGS method, and tested the interference effect of gene expression by Grx1roGFP2 and realltime PCR. [Result] Using new method for preparing 50nt hpRNA, change of Zm MDAR expression and fluorescence ratio of GrxlroGFP2 indicated that hpRNA mediated interference vector caused the change of redox state of protoplast. [Conclusion] The prepared hpRNA by new method will lay a foundation for interference effect mediated by RNAi vector.
Key wordshpRNA; REGS; ZmMDAR RNAi; Grx1roGFP2
RNAi(RNA interference,RNA干扰)是一种进化上保守的抵御靶向细胞和病毒mRNAs的dsRNA防御机制,在此生物学过程中,小RNA干扰了mRNA转录本水平,从而最终抑制该基因的表达。非编码小RNAs是miRNA和siRNA的dsRNA前体裂解产物,此裂解过程是通过一种Dicer的核酸酶执行完成。这些非编码小RNAs与RISC[1]、AGO蛋白和其他效应子蛋白共同导致了RNA干扰。
RNAi技术日趋成为一种功能基因組研究的重要手段,Sugao等[2]使用cDNA文库构建了RNAi文库;Dietzl等[3]对每个基因进行单独构建UASIR载体,然后形成RNAi文库的方法构建了果蝇RNAi库。此外,RNAi更有利于细胞信号转导通路的鉴定,利用RNAi文库靶向人类基因组中的mRNA,最终鉴定了p53信号通路的5个新调节者。构建RNAi文库是一个相对较繁琐的过程,迄今为止人们相继发展了几种构建RNAi文库的方法:Sen等[5]利用REGS方法构建了鼠胚胎RNAi文库;Luo等[6]利用DNA酶学工程方法构建了小鼠胚胎siRNA文库;针对单个基因进行单独构建,最后形成RNAi文库[3];Nichols等[7]以重组酶为基础构建了能产生随机siRNA的RNAi文库;Wang等[8]构建了滚环复制介导的发卡RNA系统,创建了拟南芥RNAi突变体库[9]和水稻RNAi突变体库[10]。
发夹RNA(hpRNA)在RNAi文库构建中起到了至关重要的作用,研究表明,在一定范围内,短loop有利于hpRNA的形成和稳定,但loop过短会影响反向重复结构DNA分子在细菌内的克隆。当茎环比例超过6∶1时,反向重复序列便不稳定,造成缺失或重排[11]。
hpRNA的特殊结构不仅会在分子内形成茎环状,而且还会在分子间形成二聚体。对于合适序列比例的hpRNA,如何通过合适的条件处理DNA合成公司提供的DNA干粉从而使其形成理想状态的茎环结构,稳定hpRNA的制备条件显得尤为重要。目前,涉及的制备方法迄今鲜见报道。
植物体内ROS(Reactive oxygen species,活性氧)的积累由2个系统控制:ROS产生和ROS清除系统。在ROS清除系统中,有酶类和非酶类抗氧化物质,酶类抗氧化物质包括MDAR、CAT、APX、GR和GPX。GSHASC循环在清除ROS中起到了重要的作用,其中,MDAR是植物体内清除ROS的主要酶类,以MDHA为反应底物,将其还原为ASC。为了验证制备新方法所获得的稳定hpRNA是否可以用于RNAi载体的构建并达到基因表达干扰的效果,笔者利用制备新方法制备hpRNA,采用REGS方法构建ZmMDAR RNAi载体,通过Grx1roGFP2(氧化还原敏感的绿色荧光蛋白探针)和荧光实时定量PCR检测基因表达干扰的效果,以期为RNAi载体所介导的干扰作用奠定基础。
1材料与方法
1.1hpRNA制备方法①配制20%非变性聚丙烯酰胺凝胶(30%聚丙烯酰胺(29∶1)27 mL,5×TBE 8 mL,定容至40 mL,加10%过硫酸铵 250 μL,TEMED 25 μL,室温凝固时间大于1 h),配制退火溶液(10 mmol/L Tris,pH 7.5,1 mmol/L EDTA,pH 8.0);②将DNA合成公司提供的DNA干粉,溶于超纯水定量为500 ng/μL;③制备方法1:2 μL DNA与8 μL H2O混匀煮沸5 min后冰上冷却上样;制备方法2:2 μL DNA与8 μL H2O混匀直接上样;制备方法3:2 μL DNA与8 μL H2O混匀,95 ℃ 5 min,37 ℃ 15 min,后上样。取不同方法制备的10 μL hpRNA,上样。
1.2玉米材料和生长条件 选取饱满的玉米B73种子,用3%H2O2浸泡约24 h,清水冲洗后将其播种在湿润的蛭石中;生长条件:光周期16 h/d,温度22 ℃;21 d后,将幼苗从蛭石中取出,尽量避免伤及根部,用清水将根部附着的蛭石冲洗干净,水中静置1 h以消除创伤刺激。之后取叶片。
1.3ZmMDAR的RNA干扰载体构建 按照制备新方法3制备50 nt hpRNA获得ZmAPX的反向重复片段,采用REGS方法构建ZmMDAR RNAi载体得到9zfPubiZmMDAR RNAiTnos。
1.4原生质体的共转化分离玉米叶肉原生质体,采用Pure YieldTM Plasmid Midiprep System提取质粒9zf()Pubi Grx1roGFP2Tnos和9zfPubiZmMDAR RNAiTnos,浓度约1 μg/μL。将2份质粒等量混合,加入200 μL原生质体,弹匀后加入220 μL 40%PEGCa2+,室温培养15 min,用800 μL W5洗涤2次,后加入1 mL W5重悬,轻轻混匀,平放于培养皿中过夜培养。
1.5Realtime PCR提取玉米叶片或原生质体RNA,利用PrimeScriptTM RT reagent Kit with gDNA Eraser(Perfect Real Time)去除基因组DNA后反转录合成cDNA,当原生质体RNA反转录时,引物更换为oligo d(T)以避免随机扩增造成的基因表达量。在Realtime PCR中,20.0 μL反应体系:8.8 μL cDNA(稀釋20倍),10.0 μL 2× SYBR premix Extaq,0.8 μL primers,0.4 μL ROX。ABI 7500仪器运行程序:95 ℃2 min;95 ℃15 s 和60 ℃34 s,共40个循环;最后进入溶解曲线阶段。以GAPDH为内参基因,未处理的叶片或原生质体为对照样品,采用2-ΔΔCt法计算基因相对表达量。
1.6激光共聚焦显微镜观察以9zf()PubiGrx1roGFP2Tnos和9zfPubiMCSTnos混合质粒转化的原生质体为对照,取适量9zf()PubiGrx1roGFP2Tnos和9zfPubiZmMDAR RNAiTnos原生质体于载玻片上,在405和488 nm 2个通道中进行观察,每个样品中选取10个不同的原生质体细胞进行层扫拍摄。
2结果与分析
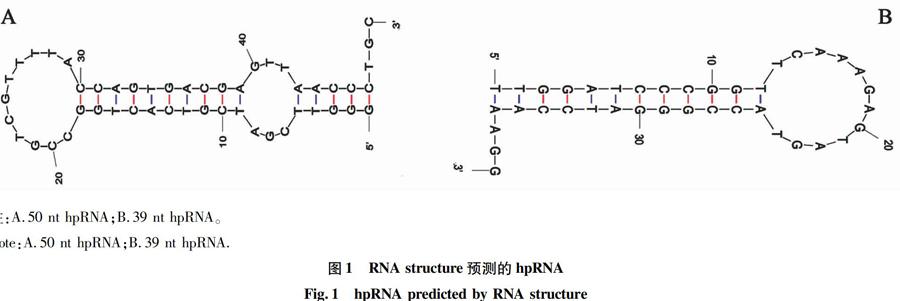
2.1hpRNA制备新方法在hpRNA制备方法中,选择50 nt寡核苷酸(序列:GGGTTCGATCGTCACTGGCCGTCGTTTTACCAGTGACGAGTTAACCCTGC)和39 nt寡核苷酸(序列:TTGGATCCCGGTTCAAAGAGTAGTACCGGGATCCAAAGG)。通过RNA structure 5.7软件预测了50和39 nt寡核苷酸的hpRNA,这表明在合适的制备条件下,寡核苷酸只能在分子内形成预期的结构即hpRNA,约22和18 bp(图1),而不能在分子间形成二聚体,约50和39 bp。为了研究hpRNA的制备条件,首先配制了20%非变性聚丙烯酰胺凝胶,1 μg DNA上样。由图2可知,50和39 nt寡核苷酸在经方法1和方法3处理后,只有22和18 bp的产物,所获得的hpRNA占有几乎100%的比例,表明寡核苷酸经方法1(煮沸5 min后冰上冷却)和方法3(95 ℃ 5 min,37 ℃ 15 min)处理,只能在分子内形成hpRNA,而不能在分子间形成二聚体。因此,在构建RNAi载体时,可以选择方法1和方法3制备出更多比例的hpRNA。
Fig.1hpRNA predicted by RNA structure2.2ZmMDAR家族的基因为了探究ZmMDAR家族和AtMDAR家族的进化关系,将AtMDAR序列号输入到PLAZA2.5软件中,确定了ZmMDAR家族的2个基因(表1)。
2.3hpRNA在ZmMDAR的RNA干扰载体中的应用按照制备新方法3制备50 nt hpRNA,通过REGS方法获得了MDAR1 RNAi和MDAR2 RNAi构建的反向重复片段(图3)。最后,与CIP处理的9zfMCS载体连接,得到了最终的干扰载体,用于瞬时表达系统中内源基因表达变化引起的细胞氧化还原状态改变的分析。
ZmMDAR基因的功能意味着该基因的表达下降必然引起玉米体内氧化还原状态的改变,hpRNA介导的干扰载体是否会引起这种状态改变有待于进一步检测。通过质粒共转化玉米叶肉原生质体,发现玉米原生质体中的ZmMDAR表达量下降(图41),同时也观察到ZmMDAR的表达量下降引起了原生质体内的氧化,相对于对照的荧光比值1.22,Grx1roGFP2探针的荧光比值均不同程度的上升(图42a),MDAR1和MDAR1干扰载体转化的原生质体中的荧光比值大幅度上升,约为2.62和2.60(图42b和42c))。Grx1roGFP2探针的荧光比值变化表明hpRNA介导的干扰载体引起了原生质体氧化还原状态的变化,同时,也表明通过制备方法3所制备的稳定50 nt hpRNA,为基因表达的干扰作用奠定了基础。
1.全式金100 bp ladder;2.玉米APX cDNAs片段被Sfi I酶切后电泳图;3.制备方法3获得的50 nt hpRNA;4.MDAR cDNAs片段与50 nt hpRNA连接后,经phi29聚合酶滾环扩增;5.扩增产物经Asc I酶切,获得单个反向重复片段;6.全式金1 kb ladder。
3结论与讨论
hpRNA在RNAi文库构建中起到了至关重要的作用,该研究利用制备方法3制备50 nt hpRNA,后采用REGS方法构建了ZmMDAR RNAi载体,通过Grx1roGFP
2探针和荧光实时定量PCR检测基因表达干扰的效果。结果表明,通过制备方法3所制备的稳定50 nt hpRNA,为基因表达的干扰作用奠定了基础。
在hpRNA制备方法中,该研究采用95 ℃ 5 min,37 ℃ 15 min可以获得更多比例的预期hpRNA,然而,这些是针对较短序列而言的,对于较长序列,是否具有同样的优化条件,需要进一步研究。
参考文献
[1] REDFERN A D,COLLEY S M,BEVERIDGE D J,et al.RNAinduced silencing complex(RISC)Proteins PACT,TRBP,and Dicer are SRA binding nuclear receptor coregulators[J].Proc Natl Acad Sci USA,2013,110(16):6536-6541.
[2] SUGAO K,HIROSE K.Enzymatic production of RNAi libraries from cDNAs and highthroughput selection of effective shRNA expression constructs[J].Methods Mol Biol,2011,729:123-139.
[3] DIETZL G,CHEN D,SCHNORRER F,et al.A genomewide transgenic RNAi library for conditional gene inactivation in Drosophila[J].Nature,2007,448(7150):151-156.
[4] BERNS K,HIJMANS E M,MULLENDERS J,et al.A largescale RNAi screen in human cells identifies new components of the p53 pathway[J].Nature 2004,428(6981):431-437.
[5] SEN G,WEHRMAN T S,MYERS J W,et al.Restriction enzymegenerated siRNA(REGS)vectors and libraries[J].Nat Genet,2004,36(2):183-189.
[6] LUO B,HEARD A D,LODISH H F.Small interfering RNA production by enzymatic engineering of DNA(SPEED)[J].Proc Natl Acad Sci USA,2004,101(15):5494-5499.
[7] NICHOLS M,STEINMAN R A.A recombinasebased palindrome generator capable of producing randomized shRNA libraries[J].J Biotechnol,2009,143(2):79-84.
[8] WANG L,FAN Y L.Rolling circle amplificationmediated hairpin RNA(RMHR)library construction in plants[J].Nucleic acids research,2008,36(22):149.
[9] WANG L,FAN Y L.Rolling circle amplificationmediated long hairpin RNA library construction in plants[J].Methods molecular biology,2012,894:309-321.
[10] WANG L,ZHENG J,LUO Y Z,et al.Construction of a genomewide RNAi mutant library in rice[J].Plant biotechnology journal,2013,11(8):997-1005.
[11] 李云.hpRNA的茎环比例对RNA介导的病毒抗性产生的影响[D].泰安:山东农业大学,2008.

