基于线粒体控制区部分序列的南海大斑石鲈遗传多样性分析
郜星晨, 章 群, 薛 丹, 宫亚运, 曹 艳, 韩博平
基于线粒体控制区部分序列的南海大斑石鲈遗传多样性分析
郜星晨1, 2, 章 群1, 薛 丹1, 宫亚运1, 曹 艳1, 韩博平1
(1. 暨南大学生态系, 热带亚热带水生态工程教育部工程研究中心, 广州 510632; 2. 中国长江三峡集团公司中华鲟研究所, 湖北宜昌443100)
为了研究南海大斑石鲈()不同地理群体的遗传多样性情况, 作者测定了东兴、乌石、潭门、闸坡4个群体共计61尾大斑石鲈控制区的993 bp序列。检测出变异位点35个, 单倍型47种, 平均单倍型多样性指数为0.9884, 核苷酸多样性指数为0.0076, 总体表现出高单倍型多样性和低核苷酸多样性的特点, 其中潭门群体核苷酸多样性相对较高(0.00879)。中性检验结果显示Fu’s Fs值均为显著负值–5.34(=0.03), 核苷酸不配对分布没有显著偏离群体扩张模型呈现出单峰(SSD值和Rg指数较小), 表明大斑石鲈在历史上经历过种群扩张, 推测扩张时间约4.74万~1.18万年间。群体间(0.0065~0.0089)与群体内遗传距离(0.0066~0.0089)处于同一水平, 总遗传分化指数Fst为–0.0057 (>0.05), 群体间基因流Nm=33.76。不同组群划分方式的分子方差分析均表明群体内遗传差异显著大于群体间, 遗传变异主要来源于群体内部。南海海域大斑石鲈群体遗传多样性匮乏, 群体间不存在显著的遗传分化, 可划归一个管理保护单元, 潭门群体建议优先给予保护。
大斑石鲈(); 控制区; 遗传多样性
大斑石鲈()隶属鲈形目(Perciformes)、石鲈科(Pomadasyidae)、石鲈属(), 俗名猴鲈、海猴, 广泛分布于印度洋和太平洋西部, 在中国分布于南海和东海近海, 主要渔场为北部湾沿岸及涠洲岛附近。大斑石鲈为暖水性中下层中小型鱼类, 肉质鲜美, 生长较快, 有一定经济价值[1]。近年来由于北部湾海区一直处于过度捕捞状态, 鱼类资源持续衰退, 自然海区大斑石鲈鱼类数量减少, 分布区域也大为缩小[2]。目前大斑石鲈研究仅局限于形态、分类和分布, 分子遗传学方面仅在石鲈科系统发育研究中有所涉及, 如朱世华[2]等探讨了大斑石鲈及其他4种石鲈科鱼类亲缘关系; 梁日深[3]等基于S7核糖体蛋白基因序列分析了大斑石鲈在内11种石鲈属鱼类分子系统进化关系; 任岗[4]等选用16S rRNA基因研究石鲈科鱼类种间关系, 尚未见到大斑石鲈遗传多样性及种质资源方面研究报道。
遗传多样性是生物进化和适应复杂环境的物质基础, 是种质资源有效保护和合理利用的依据[5]。线粒体DNA(mtDNA)具有分子结构简单, 严格的母系遗传、无重组和进化速度快等优点, 是群体遗传学研究的理想分子标记[2]。mtDNA中存在一个非编码区, 即控制区, 是整个线粒体DNA序列中变异最大的区域, 能够敏感的反应出海洋鱼类的群体遗传结构, 在鱼类分类、系统发育、群体遗传学等方面已有广泛的应用[5]。本研究通过测定南海4个海域大斑石鲈mtDNA控制区序列, 探讨其遗传多样性和遗传分化情况, 分析其种群遗传结构和历史动态, 旨在促进大斑石鲈有效开发和可持续管理, 为种质资源的保护积累基础材料。
1 材料和方法
1.1 实验材料、DNA提取与PCR扩增测序
2011年~2014年于广东闸坡(ZP)、广东乌石(WS)、海南潭门(TM)和广西东兴(DX)分别采集大斑石鲈(图1), 测得叉长范围9~22 cm, 体质量250~ 400 g。样品用95%的酒精固定后保存, 采用高盐法提取总基因组DNA[6]。采用自行设计的引物DL1 (5′-GTT GGA ATC CTC CCT ACT GCTC-3′), DL2 (5′-TCT TAA CCA CCC TTT ACG CCGA-3′)进行扩增, 扩增条件与检测参照任岗等[4]的方法, PCR产物送至北京六和华大基因有限公司切胶纯化并测序。
1.2 数据处理
使用MEGA 6软件上对手工校对后的测定序列进行对位排列, 统计序列的碱基组成、变异位点、多态简约信息位点、转换或颠换比值等, 根据Kimura双参数模型(Kimura 2-parameter, K2P), 计算遗传距离。使用Dnasp 5.10计算群体的单倍型数、核苷酸酸多样性(Pi)、单倍型多样性(Hd), 分析群体间遗传变异指数(), 1000次重复随机抽样重排后进行显著性检验, 群体间的基因流值()=(1–)/(2), 通过分子方差(AMOVA)分析检验遗传变异分布模式[7]。借助TCS 2.1构建单倍型简约网络图, 分析单倍型分布情况。以Arlequin 3.5软件进行Tajima’s D和Fu’s Fs中性检验分析, 并通过核苷酸不配对分布分析检验种群历史动态; 采用广义非线性最小方差估算扩张参数, 根据=2估算群体历史扩张时间, 其中=2(为所研究的控制区基因的变异速率,表示片段长度),为自扩张以来经历的代数[8]。
2 结果与分析
2.1 大斑石鲈的控制区基因序列特征
序列比对分析后获得控制区前段序列993 bp, A、T、C、G组成的平均值分别是30.7%、29.0%、24.3%、16.0%。A+T平均含量59.6%, G+C平均含量40.4%, 表现出明显的反G偏倚, 与脊椎动物线粒体DNA的特点一致。全部序列共有变异位点35个(占DNA核苷酸总数的3.5%), 简约信息位点30个。转换与颠换比例为17.6, 远大于刘朋朋等[5]提出的期望值0.5, 表明此片段没有饱和, 适合系统发育分析。大斑石鲈遗传多样性参数见表1。其中潭门群体核苷酸多态性指数最高, 乌石群体单倍型多样性和核苷酸多态性均最低。
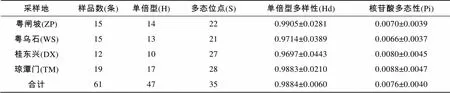
表1 不同群体大斑石鲈的遗传多样性参数
2.2 大斑石鲈的系统发育与遗传分化
在以K2P模型构建的单倍型邻接树和单倍型TCS最大简约网络中(图2)不同来源的单倍型散乱无序分布于各个分支, 不呈现地方群体为单位的家系式分支现象, 没有明显的地理结构和谱系结构[8]。47个单倍型中, 39个为独享单倍型, 仅有4个单倍型为2个以上个体共享, 显示大部分单倍型频率较低。
群体间平均遗传距离为0.0077(0.0065~0.0085), 群体内遗传距离0.0066~0.0089, 二者处于同一水平。群体之间的遗传变异指数值为–0.022~0.003, 总遗传变异指数值为–0.0057, 均未表现出统计学显著性。4个群体基因流值均大于4, 平均基因流值为33.76, 群体间存在较大基因流。通过设定两种AMOVA分析检测大斑石鲈的群体遗传结构: (1)将南海4个群体划分为一个组群以检验群体间是否存在显著的遗传结构; (2)按样品的来源将4个群体划分为2群组检验是否存在显著地理结构: 将琼州海峡东侧潭门、闸坡为一地理区, 华南大陆沿海西部乌石、东兴为另一地理区; 将琼州海峡南部潭门为一地理区, 北部乌石、东兴、闸坡为另一地理区[7]。两种AMOVA分析显示, 4个群体间(=0.659)及不同地理区划分方式(=0.559)均不存在显著的遗传结构, 遗传变异主要集中在群体内个体间(99.08%~100.65%), 只有–0.16%~ 0.01%发生在群体间。
2.3 种群历史动态
Tajima’s D和Fu’s Fs检验结果表明(表2), 分别对4个地理群体及群体整体进行中性分析, 不同群体Tajima’s D多为负值, 整体Tajima’s D= –0.23 (>0.1), 统计学检验不显著; 4个群体Fu’s Fs中性检验均为显著性负值, 整体Fu’s Fs值为–5.34且显著(=0.03), 明显偏离中性突变。由于在相同条件下, Fu’s Fs检验对种群近期扩张事件比较敏感, 故可判断大斑石鲈近期可能经历了快速种群扩张[5]。南海大斑石鲈各个地理群体和整体核苷酸岐点分布呈单峰(图3); 对核苷酸不配对的观测分布和扩张模式下的预期分布一致性进行拟合优度检验显示群体平均方差之和(SSD)和平滑性检验统计值(Raggedness)均较小, 统计检验均不显著, 证实了所观测的核苷酸错配分布与群体扩张模式相符[8], 表明4个群体历史上发生了不同程度的种群扩张。θ1/θ0表明有效群体大小在扩张前后发生很大变化[8], 也提示南海大斑石鲈发生快速扩张, 核苷酸不配对分析与中性进化检验结果基本一致。根据核苷酸错配分析总的τ观察值3.76, 以生殖周期为3 年计, (3%~10%)/百万年的控制区分歧速率, 估算扩张时间约为4.74万年~1.18万年, 处于末次冰期期间。
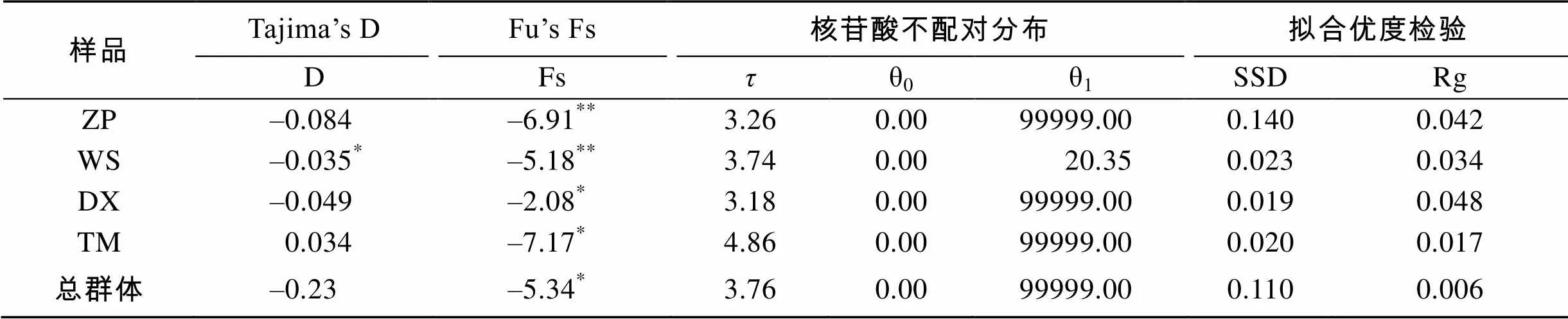
表2 南海近海4个大斑石鲈群体的中性检验和错配分布参数
注: *. 统计显著水平(<0.05), **. 统计极显著水平(<0.01)
3 讨论
3.1 大斑石鲈的种群动态与遗传多样性
大斑石鲈总体表现出高Hd低Pi的特点, 中性检验Fu’s Fs为显著负值, 核苷酸不配对分布为单峰, 表明在末次冰盛期后期(4.74万年~1.18万年)经历了种群扩张。大斑石鲈为近岸繁殖的暖水性鱼类, 栖息在20~110 m的底质海区[1]。在末次冰盛期南海海平面比现在低130~150 m[9], 随着间冰期气温回升海平面不断上升, 生存空间扩大到整个南海大陆架区, 大斑石鲈得以快速扩张。大斑石鲈繁殖周期长(可从1月长达4月份)、产卵场较集中(多在南海北部)等特点[1], 可能利于群体快速增长, 产生高的单倍型多样性, 但尚缺乏足够时间积累核苷酸序列的多样化。类似的高Hd低Pi遗传多样性模式在同海域分布的军曹鱼()[9]、花鲈()[10]等鱼类研究中也被检测到。
群体mtDNA的Pi值是衡量遗传多样性的重要指标, Pi值越大表示群体mtDNA多态程度越高[9]。南海大斑石鲈Hd和Pi平均值分别为0.9884±0.0060和0.0076±0.0040, 核苷酸多样性值较同海域分布鱼类如红鳍笛鲷()[11](Pi=0.031)、黄鳍鲷()[12](Pi=0.026)等低, 较石鲈科其他鱼类如斜带髭鲷()[13](Pi=0.022)、三线矶鲈()[14](Pi=0.0058)等也处于中等偏下水平。海洋鱼类遗传多样性水平受多种因素如有效种群大小、群体扩张等影响, 也与自身栖息地环境相关。大斑石鲈经历过近期种群扩张或“遗传瓶颈”, 群体尚缺乏足够时间在迁移和漂变之间取得平衡, 导致遗传多样性较为匮乏[9]。另外该海域环境污染、过度捕捞等人为因素也可能导致资源的衰退和有效种群的减少, 影响大斑石鲈遗传多样性水平。
3.2 大斑石鲈的遗传分化
TCS网络图中不同来源个体混杂分布, 无明显地理结构。基于样品来源的地理格局, 假设华南沿海与海南岛种群关系较远, 但AMOVA分析显示群体内个体的变异是变异的主要来源, 不同地理区群体间差异不显著, 说明琼州海峡并未起到有效的地理屏障作用。在缺少阻碍扩散的开放海洋环境里, 大斑石鲈的卵、幼体及成体都具有较强的迁移能力, 以致分化水平较低, 群体没有地理结构和谱系结构[14]。遗传分化也与生态习性及生活史密切相关, 大斑石鲈春季繁殖期会洄游向近岸或者河口, 洄游过程中不同群体间个体的混合可能导致群体间产生基因交流。此外, 南海沿海季风漂流、黑潮分支、南海暖流、南海冷水等海流的输送作用加强了大斑石鲈卵和幼体的扩散, 也是遗传分化程度较低的推动原因。
3.3 种质资源的保护
物种的生存适应能力、进化潜力与遗传多样性密切相关, 遗传多样性的降低可导致物种生存抵抗力与生殖力下降、物种退化等[13], 因此遗传多样性受到极大威胁的大斑石鲈野生群体应得到足够保护。本研究4个南海地理群体无明显的遗传差异, 可划归同一个保护单元, 潭门群体野生资源较多, 遗传多样性水平相对较高, 建议优先加以保护。本研究涉及的地理群体量和种群数量有限, 同时控制区单一标记, 可能还不足以准确反映大斑石鲈的遗传结构。为了更好地保护大斑石鲈种质资源, 今后的研究中应增加采样地点和样品量, 并采取多种分子标记分析大斑石鲈遗传变异, 以期为制定科学管理保护措施提供理论依据。
[1] 陈再超, 刘继兴. 南海经济鱼类[M]. 广州: 广东科技出版社, 1982: 189. Chen Zaichao, Liu Jixing. Economic fishes of the South China Sea[M]. Guangzhou: Guangdong Science and Technology Press, 1982: 189.
[2] 朱世华, 郑文娟, 邹记兴, 等. 5种石鲈科鱼类细胞色素b基因序列及分子系统分析[J]. 热带海洋学报, 2006, 4: 41-45.Zhu Shihua, Zheng Wenjuan, Zou Jixing, et al. Molecular phylogenetic analysis of five Pomadasyidae fish based on mitochondrial cytochromeb sequences[J]. Journal of Tropical Oceanography, 2006, 44: 41-45.
[3] 梁日深, 卓孝磊, 龙敏明, 等. 基于S7核糖体蛋白基因部分序列的石鲈属鱼类分子系统进化关系[J]. 海洋学报(中文版), 2013, 35(1): 165-171. Liang Rishen, Zhuo Xiaolei, Long Minming, et al. Molecular phylogenetic relationships of Pomadasys fishes based on partial S7 ribosomal protein gene sequences[J]. Acta Oceanologica Sinica, 2013, 35(1): 165-171.
[4] 任岗, 章群, 钱开诚, 等. 12种石鲈科鱼类线粒体16S rRNA基因的部分序列分析[J]. 热带海洋学报, 2007, 3: 48-52. Ren Gang, Zhang Qun, Qian Kaicheng, et al. Sequence analysis of twelve grunt fishes based on 16S ribosomal RNA gene fragments[J]. Journal of Tropical Oceanography, 2007, 3: 48-52.
[5] 刘朋朋, 钟立强, 潘建林, 等. 基于线粒体D-loop区分析黄颡鱼()5个淡水湖泊群体的遗传多样性[J]. 海洋与湖沼, 2013, 25(3): 728-733. Liu Pengpeng, Zhong Liqiang, Pan Jianlin, et al. Poplulation genetics in mitochondrial DNA control region for five fresh-water yellow catfish[J]. Ocenologia et Limnologia Sinica, 2013, 25(3): 728-733.
[6] Blin N, Stafford D W. A general method for isolation of high molecular weight DNA from eukaryotes[J]. Nucleic Acids Research, 1976, 3: 2303-2308.
[7] Excoffier L, Laval G, Schneider S. Arlequin version 3.01: An integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics, 2005, 1: 47-50.
[8] Fu Y X. Statistical tests of neutrality of mutations against population growth and background selection[J]. Genetics, 1997, 147(2): 915-925.
[9] 阮燕如, 周文漪, 李贵生, 等. 中国近海军曹鱼线粒体细胞色素b基因序列的遗传变异[J]. 海洋渔业, 2014, 2: 97-101. Ruan Yanru, Zhou Wenyi, Li Guisheng, et al. On mtDNA cytochrome b gene sequence variation of Cobia () [J]. Marine Fisheries, 2014, 2: 97-101.
[10] Yokogawa K, Seki S. Morphological and genetic differences between Japanese and Chinese sea bass of the genus[J]. Ichthyol, 1995, 41(4): 437-445.
[11] Zang J B, Cai Z P, Huang L M, et al. Population genetic structure of crimson snapperin East Asia, revealed by analysis of the mitochondrial control region[J]. Journal of Marine Science, 63(4): 693-704.
[12] Xia J H, Huang J H, Gong J B. Significant population genetic structure of yellowfin seabreamin China[J]. Journal of Fish Biology, 2008, 73: 1979-1992.
[13] 陈竹, 钟山, 罗大极, 等. 基于线粒体D-loop区比较分析野生与养殖斜带髭鲷种群的遗传多样性[J]. 水生生物学报, 2011, 5: 761-767. Chen Zhu, Zhong Shan, Luo Daji, et al. Genetic diversity evaluation by the comparative analysis on mitochondrial D-loop area between wild and cultured population of[J]. Acta Hydrobiologica Sinica, 2011, 5: 761-767.
[14] 薛丹, 章群, 郜星晨, 等. 基于线粒体控制区的粤闽三线矶鲈地理群体的遗传变异分析[J]. 海洋渔业, 2014, 6: 496-502. Xue Dan, Zhang Qun, Gao Xingchen, et al. Genetic variations amongin coastal waters of southeast China based on mtDNA control region sequences[J]. Marine Fisheries, 2014, 6: 496-502.
Genetic diversity ofin coastal waters of South China Sea via mtDNA control region partial sequences
GAO Xing-chen1, 2, ZHANG Qun1, XUE Dan1, GONG Ya-yun1, CAO Yan1, HAN Bo-ping1
(1. Engineering Research Center of Tropical and Subtropical Aquatic Ecological Engineering, Ministry of Education, Department of Ecology, Jinan University, Guangzhou 510632, China; 2. Chinese Sturgeon Research Institute, China Three Gorges Corporation, Yichang 443100, China)Received:Nov. 18, 2014
; control region; genetic diversity
This study determined 993bp mtDNA control region sequences of 61 individuals collected in Dongxing, Wushi, Tanmen, and Zhapo where 35 variable sites defined 47 haplotypes. Global haplotype (Hd) and nucleotide diversity (Pi) were 0.9884 and 0.0076, respectively thus indicating a pattern of high haplotype diversity and low nucleotide diversity. A combination of the significant negative neutral test values of Fu’s Fs (−5.34,= 0.03), the mismatch distribution pattern, and the network analysis revealed that a historical population expansion occurred at about 47400–11800 years ago. The genetic distance between populations (0.0065–0.0089) was at a similar level to the genetic distance within populations (0.0066–0.0089); the overall genetic differentiation index (Fst) was −0.0057 (> 0.05), the gene flow coefficient (Nm) among four populations was 33.76, and the analysis of molecular variance showed that most of the genetic variations of the four populations was distributed throughout the populations. In addition, genetic diversity was low and genetic divergence was inconspicuous between populations of. It is thus suggested thatin the coastal waters of the South Sea should be maintained as a single management unit and that the Tanmen population within contains arelatively high genetic diversity and should thus be a priority for protection.
Q958
A
1000-3096(2016)07-0041-05
10.11759//hykx20141118001
2014-11-18;
2015-03-27;
国家自然科学基金项目(41071034); 中央高校基本科研业务费专项资金(21613105, 21611426)
[Foundation: National Natural Science Foundation of China, No. 41071034; Fundamental Scientific Research Funds for the Central Universities No. 21613105, 21611426]
郜星晨(1989-), 男, 安徽淮北人, 硕士研究生, 主要从事鱼类生态学研究, 电话: 15625163028, E-mail: 13039829682@126.com;章群,通信作者, E-mail: zhangqunjnu@gmail.com
(本文编辑: 谭雪静)

