高效聚磷菌的分离鉴定及除磷性能分析
王图锦,潘 瑾,刘雪莲
(重庆交通大学河海学院,重庆 400074)
高效聚磷菌的分离鉴定及除磷性能分析
王图锦,潘瑾,刘雪莲
(重庆交通大学河海学院,重庆400074)
从连续稳定运行的AAO污水处理系统曝气池污泥中分离筛选出4株聚磷菌(编号为P1,P2,P3,P4)进行吸放磷实验。结果表明:4株聚磷菌具有较强的去除污水中磷酸盐的能力,均表现出明显的厌氧释放磷、好氧吸收磷的特征,其中P1细菌的吸放磷特征最为显著,对废水磷的最大去除率可达到75.51%,P2、P3、P4对磷的最大去除率分别为8.77%、47.26%、30.19%。通过对4株细菌的16S rDNA 序列进行测序分析,鉴定出P1、P2、P3、P4分别属于微杆菌属、芽孢杆菌属、塚村氏菌属、塚村氏菌属细菌。
生物除磷;聚磷菌;废水;生物学鉴定
强化生物除磷系统(enhanced biological phosphorous removal,EBPR)广泛用于去除废水中的磷酸盐,采用不同结构不同工艺的生物除磷污水处理厂大多依据经验设计建造,缺乏对生物除磷微生物学机理的深入了解,导致许多污水处理厂设计及运行参数并不符合生物除磷系统最佳条件,出现除磷效果差、污泥膨胀、剩余污泥过多、能耗高等诸多问题[1-4]。因此,加大对生物除磷系统中微生物特别是聚磷菌的研究显得尤为重要,目前这方面也取得了显著的进展。聚磷菌(polyphosphate accumulating organisms,PAOs)具有从废水中超量吸收磷酸盐的能力,从而有效去除废水中的磷酸盐污染物,对污水处理系统生物除磷起着关键性作用,聚磷菌是生物除磷系统中主要的功能菌群之一。目前使用传统的纯培养方法以及非培养方法(如克隆文库,FISH,DNA指纹图谱等)对这类菌群进行了较多研究。被认为属于PAOs类群的微生物有:产碱杆菌属[5-6]、肠杆菌属[7-8]、葡萄球菌属[9]、变形菌属[10]等,在不同的生物除磷系统工艺以及不同的污水成分下,PAOs类群可能由不同的种群组成。对生物除磷机理的深入了解还有待于进一步分离聚磷菌,探讨菌种去除污水中磷的机理。
本研究从一个实验室稳定运行的AAO反应器曝气池污泥中分离筛选出4株聚磷菌(P1、P2、P3、P4),并对菌株去除废水中磷的性能进行了初步探讨,为深入了解生物除磷微生物学机理提供技术支持,同时为高效聚磷菌应用于工程实际打下基础。
1 试验方法
1.1聚磷菌的分离纯化
将5 mL的活性污泥装入灭菌离心管内,加入几粒2~3 mm直径的玻璃珠,在旋涡振荡器上充分振荡,使活性污泥均匀分散在水中,采用涂布法分离纯化菌株。采用的培养基配方为:酵母膏:0.3 g;酪蛋白:1.2 g; CH3COONa·3 H2O:1.0 g;CaCl2·2 H2O:30 mg;KH2PO4:20 mg,MgSO4·7H2O:100 mg;NaCl:100 mg;pH:6.8~6.9;蒸馏水:1 000 mL;微量元素溶液0.6 mL。
微量元素配方(1L):H3BO3:0.15 g;FeCl3·6H2O: 1.5 g;KI:0.03 g;CuSO4·5 H2O:0.03 g;Na2MoO4·2 H2O:0.06 g;ZnSO4·7 H2O:0.12 g;MnCl2·4 H2O:0.12 g;CoCl2·2 H2O:0.15 g。
1.2聚磷菌的筛选与吸放磷实验

图1 厌氧培养装置

吸放磷实验中采用的模拟废水配方如下:
蛋白胨0.1 g,酵母膏:0.01 g;CH3COONa·3 H2O:0.925 g; KH2PO4·3 H2O:65.51 mg;NaCl 0.05 g; MgSO4·7 H2O:153.7 mg;NaHCO3:0.075 g; CaCl2·2 H2O:33.1 mg。
1.3菌种的16S rDNA序列测定与系统发育树分析
PCR简易DNA模板的制取:使用无菌牙签从纯菌落中挑取少许,置于30 μL的无菌水中混匀,使用98℃高温加热5 min,菌液转移到1.5 mL离心管中,于10 000 r/min离心5 min,取上清液备用。
使用16S rDNA引物[11]进行PCR扩增。正向引物为8F序列: AGAGTTTGATCCTGGCTCAG,反向引物为1495R序列:CTACGGCTACCTTGTTACGA。25 μL的PCR反应体系为:简易DNA模板2 μL,10xbuffer(Mg2+)2.5 μL,10 mmol/L dNTP 0.5 μL,10 pmol/L引物各1 μL,2.5 U TaqDNA聚合酶。以无菌的TE缓冲液取代模板作空白对照。
PCR温度设置:95℃预变性5 min;94℃变性1 min;56℃退火1 min,72℃延伸1 min 30s,共35个循环,最后72℃延伸10 min结束反应。PCR产物取5 μL作琼脂糖凝胶电泳检测。
PCR产物16S rDNA测序工作交由上海生工生物工程技术有限公司完成。将测得序列用Blast软件提交GenBank,与GenBank中的已知序列进行同源性分析,选取同源性较高的序列用Clustal x (1.8)软件比对,用MEGAversion 2中的Kimura2-Parameter Distance 模型计算进化距离,用Neighbor-Joining法构建系统发育树。
2 结果与讨论
2.1菌株吸放磷实验结果
通过涂布分离法获得55株纯菌株,对菌株作吸放磷试验得到具有较强除磷能力的4株细菌,其吸放废水中磷能力测定结果如图2所示。

图2 菌株吸放磷能力测定结果


表1 菌株除磷效果
废水的强化生物除磷理论[12-14]指出:在厌氧培养条件下,聚磷菌将胞内存储的多聚磷酸盐分解,以正磷酸盐的形式释放到废水中,同时利用废水中的有机物用于合成聚β羟基丁酸(PHB)贮存能量。在好氧培养条件下,聚磷菌储存的聚β羟基丁酸分解提供能量,用于聚磷菌从废水中超量吸收磷酸盐合成多聚磷酸盐颗粒,在好氧培养阶段聚磷菌吸磷量远高于厌氧释磷量,从而废水中的磷酸盐污染物得以去除。本研究分离纯化获得的4株细菌具有典型的吸放磷特征,可以判定4株细菌属于聚磷菌类群。
2.2菌株的16S rDNA扩增结果
4个菌株P1、P2、P3、P4的16S rDNA扩增结果
如图3所示,结果表明PCR产物均单一明亮,大小约为1 500 bp。

M—1 Kb ladder DNA Marker;Nc—阴性对照图3 16S rDNA扩增结果
2.3菌株16S rDNA的序列测定与系统发育树分析
将P1菌株的16S rDNA核酸序列在GenBank作BLAST比对,结果表明该序列与Microbacteriumoxydans(DQ105974)最为接近,相似度达到99.9%。同时与Microbacteriumsaperdae(Y17236),Microbacteriumluteolum(Y17235),Microbacteriummaritypicum(AJ853910),Microbacteriumparaoxydans(AJ581908)相似度均达到99%以上。选择同源性最高的10个16S rDNA全序列构建系统发育树,结果如图4所示。由图4可见,P1菌株与Microbacteriummaritypicum(AJ853910)与Microbacteriumoxydans(DQ105974)严格聚为一族,根据同源性分析结果将P1菌株鉴定为微杆菌属细菌。
P1菌16S rDNA测序结果如下:
GACGTCGTGATGTGCAGTCGAACGGTGACACGGAGCTTGCTCTGTGGGATCAGTGGCGAACGGG
TGAGTAACACGTGAGCAACCTGCCCCTGACTCTGGGATAAGCGCTGGAAACGGCGTCTAATACTGGAT
ATGTGACGTGACCGCATGGTCTGCGTCTGGAAAGAATTTCGGTTGGGGATGGGCTCGCGGCCTATCAG
CTTGTTGGTGAGGTAATGGCTCACCAAGGCGTCGACGGGTAGCCGGCCTGAGAGGGTGACCGGCCAC
ACTGGGACTGAGACACGGCCCAGACTCCTACGGGAGGCAGCAGTGGGGAATATTGCACAATGGGCG
CAAGCCTGATGCAGCAACGCCGCGTGAGGGACGACGGCCTTCGGGTTGTAAACCTCTTTTAGCAGGG
AAGAAGCGAAAGTGACGGTACCTGCAGAAAAAGCGCCGGCTAACTACGTGCCAGCAGCCGCGGTAA
TACGTAGGGCGCAAGCGTTATCCGGAATTATTGGGCGTAAAGAGCTCGTAGGCGGTTTGTCGCGTCTG
CTGTGAAATCCGGAGGCTCAACCTCCGGCCTGCAGTGGGTACGGGCAGACTAGAGTGCGGTAGGGG
AGATTGGAATTCCTGGTGTAGCGGTGGAATGCGCAGATATCAGGAGGAACACCGATGGCGAAGGCAG
ATCTCTGGGCCGTAACTGACGCTGAGGAGCGAAAGGGTGGGGAGCAAACAGGCTTAGATACCCTGGT
AGTCCACCCCGTAAACGTTGGGAACTAGTTGTGGGGTCCATTCCACGGATTCCGTGACGCAGCTAAC
GCATTAAGTTCCCCGCCTGGGGAGTACGGCCGCAAGGCTAAAACTCAAAGGAATTGACGGGGACCCG
CACAAGCGGCGGAGCATGCGGATTAATTCGATGCAACGCGAAGAACCTTACCAAGGCTTGACATATAC
GAGAACGGGCCAGAAATGGTCAACTCTTTGGACACTCGTAAACAGGTGGTGCATGGTTGTCGTCAGC
TCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTCGTTCTATGTTGCCAGCACGTA
ATGGTGGGAACTCATGGGATACTGCCGGGGTCAACTCGGAGGAAGGTGGGGATGACGTCAAATCATC
ATGCCCCTTATGTCTTGGGCTTCACGCATGCTACAATGGCCGGTACAAAGGGCTGCAATACCGCGAGG
TGGAGCGAATCCCAAAAAGCCGGTCCCAGTTCGGATTGAGGTCTGCAACTCGACCTCATGAAGTCGG
AGTCGCTAGTAATCGCAGATCAGCAACGCTGCGGTGAATACGTTCCCGGGTCTTGTACACACCGCCCG
TCAAGTCATGAAAGTCGGTAACACCTGAAGCCGGTGGCCTAACCCTTGTGGAGGGAGCCGCTAGATA
CGCCCCCA
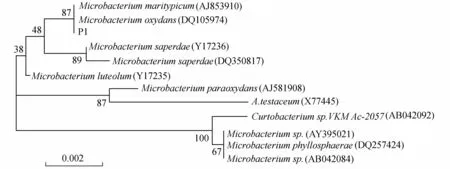
图4 根据P1菌株16S rDNA序列构建的系统发育树
此外,经鉴定菌株P2属于芽孢杆菌属细菌,P3为塚村氏菌属细菌,P4为塚村氏菌属细菌。本实验获得的4株细菌具有聚磷菌明显的厌氧释磷、好氧吸磷特征,在现有文献中还未见报道这几个属的聚磷菌,本研究表明聚磷菌是一个宽泛的微生物类群,在不同地域、不同污水处理系统中存在不同的聚磷菌类群发挥除磷功能。
3 结 论
a. 本研究分离获得的4株聚磷菌具有典型的厌氧释磷、好氧吸磷特征,在厌氧培养条件下,4株聚磷菌向废水中释放磷,使得废水中磷浓度略有升高,转入好氧培养后,4株聚磷菌从废水中超量吸收磷,废水中磷浓度持续下降,约10 h后浓度趋于稳定。
b. P1号聚磷菌除磷效果最为显著,对废水中磷的最大去除率可达到75.51%。P2、P3、P4号聚磷菌最大除磷率分别为8.77%、47.26%、30.19%。
c. 对4株聚磷菌作16S rDNA的测序分析,并将16S rDNA核酸序列在GenBank中作核酸序列鉴定出比对,P1菌株为微杆菌属的细菌,P2为芽孢杆菌属细菌,P3为塚村氏菌属细菌,P4为塚村氏菌属细菌。
[ 1 ] 宋姬晨,王淑莹,杨雄,等. 亚硝酸盐对A2O 系统脱氮除磷的影响[J]. 中国环境科学,2014,34(9):2231-2238.(SONG Jichen, WANG Shuying, YANG Xiong, et al. Effect of nitrite on nutrient removal in A2O system[J]. China Environmental Science, 2014,34(9): 2231-2238. (in Chinese))
[ 2 ] 白雪,张荣兵,顾剑,等. 大型污水处理厂污泥膨胀原因分析及其控制方法[J]. 中国给水排水, 2011, 27(23):31-35.(BAI Xue, ZHANG Rongbing, GU Jian, et al. Cause analysis and control of sludge bulking in large-scale WWTP[J]. China Water & Wastewater, 2011, 27(23):31-35. (in Chinese))
[ 3 ] SEVIOUR R J, MINO T, ONUKI M. The microbiology of biological phosphorus removal inactivated sludge systems[J]. FEMS Microbiology Reviews, 2003, 27(1): 99-127.
[ 4 ] 周可新,许木启,曹宏. 生物除磷活性污泥系统微生物学研究进展[J]. 应用与环境生物学报, 2005, 11(5):638-641.(ZHOU Kexin, XU Muqi, CAO Hong. Advance in microbiological research of activated sludge system for phosphorus removal[J]. Chinese Journal of Applied and Environmental Biology, 2005,11(5): 638-641. (in Chinese))
[ 5 ] FLORES III A, NISOLA G M, CHO E, et al. Bioaugmented sulfur-oxidizing denitrification system withAlcaligenesdefragransB21 for high nitrate containing wastewater treatment[J]. Bioprocess and Biosystems Engineering,2007,30(3): 197-205.
[ 6 ] ZHOU Yan, LIM M, HARJONO S,et al. Nitrous oxide emission by denitrifying phosphorus removal culture using polyhydroxyalkanoates as carbon source[J]. Journal of Environmental Sciences,2012,24 (9):1616-1623.
[ 7 ] 张培玉,孙梦,张晨.一株嗜盐聚磷菌的筛选及除磷性能初探[J].环境科学学报, 2011, 31(11): 2368-2373.(ZHANG Peiyu,SUN Meng,ZHANG Chen. Preliminary study on screening and phosphorus removal characteristics of a halophilic phosphate accumulating bacterial strain[J]. Acta Scientiae Circumstantiae, 2011, 31(11): 2368-2373. (in Chinese))
[ 8 ] NAKAMURA K, HIRAISHI A, YOSHIMI Y, et al.Microlunatusphosphovorusgen. nov., sp. Nov.:a new Gram-positive poly-phosphate accumulating bacterium isolated from activated sludge[J]. International Journal of Systematic and Evolutionary Microbiology, 1995, 45(1):17-22.
[ 9 ] 南亚萍,袁林江,王洋. 金黄色葡萄球菌聚磷特性研究[J].环境科学学报,2013,33(4):1028-1034.(NAN Yaping,YUAN Linjiang,WANG Yang. Characteristic and performance of phosphate accumulation byStaphyloccocusaureus[J]. Acta Scientiae Circumstantiae, 2013, 33(4): 1028-1034. (in Chinese))
[10] NGUYEN H T T, NIELSEN J L, NIELSEN P H.‘CandidatusHalomonas phosphatis’, a novel polyphosphate accumulating organism in full-scale enhanced biological phosphorus removal plants[J]. Environmental Microbiology, 2012, 14(10): 2826-2837.
[11] AMANN R I, LUDWIG W, SCHLEIFER K H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J].Microbiological and Molecular Biology Reviews, 1995, 59(1):143-169.
[12] 郑金伟,冉炜,钟增涛,等. 增强型生物除磷过程中聚磷酸盐积累微生物的研究进展[J].应用生态学报,2004, 15(8):1487-1490.(ZHENG Jinwei, RAN Wei, ZHONG Zengtao, et al. Research advance in polyphosphate-accumulating microorganisms in enhanced biological phosphorus removal process[J]. Chinese Journal of Applied Ecology, 2004, 15(8): 1487-1490. (in Chinese))
[13] WANG Yayi, REN Zhongjia, JIANG Fan, et al. Effect of copper ion on the anaerobic and aerobic metabolism of phosphorus-accumulating organisms linked to intracellular storage compounds[J]. Journal of Hazardous Materials, 2011, 186(1):313-319.
[14] AL-NAJJARA M M, COATSB E R, LOGE F J. The role of the microbial stringent response in excess intracellular accumulation of phosphorous in mixed consortia fed synthetic wastewater[J]. Water Research, 2011, 45(16):5038-5046.
Study on screening of phosphate-accumulating organisms and analysis of phosphorus removal characteristics
WANG Tujin, PAN Jin, LIU Xuelian
(SchoolofRiverandOceanEngineering,ChongqingJiaotongUniversity,Chongqing400074,China)
Four phosphate-accumulating bacterial strains, designated as P1, P2, P3, and P4, were separated from biological sludge in a stably operated AAO system for phosphorus absorption and release experiments. The results show that the four strains had high capabilities of removing phosphate from the waste water, and they exhibited high levels of phosphorus release in the anaerobic stage and phosphorus absorption in the oxic stage. P1 had the most significant phosphorus absorption and release characteristics. The maximum phosphorus removal rates of P1, P2, P3, and P4 were 75.51%, 8.77%, 47.26%, and 30.19%, respectively. Through sequence analysis of 16S rDNA, it was determined that P1 and P2 belonged toMicrobacteriumandBacillus, respectively, and both P3 and P4 belonged toTsukamurella.
biological phosphorus removal; phosphate-accumulating organisms; wastewater; biological identification
10.3880/j.issn.1004-6933.2016.05.013
重庆市基础与前沿研究计划项目(cstc2014jcyjA20011)
王图锦(1981—),男,讲师,博士,主要从事环境微生物方面的研究。E-mail:wangtujin@163.com
X172
A
1004-6933(2016)05-0063-04
2015-12-29编辑:徐娟)

