空气微生物群落解析方法:从培养到非培养
方治国,郝翠梅,姚文冲,欧阳志云
1 浙江工商大学环境科学与工程学院,杭州 310018 2 中国科学院生态环境研究中心城市与区域生态国家重点实验室,北京 100085
空气微生物群落解析方法:从培养到非培养
方治国1,*,郝翠梅1,姚文冲1,欧阳志云2
1 浙江工商大学环境科学与工程学院,杭州3100182 中国科学院生态环境研究中心城市与区域生态国家重点实验室,北京100085
随着世界范围内流行性疾病以及我国空气雾霾事件的不断发生,空气生物性污染的研究开始受到高度重视,其研究方法也随着分子生物学技术的快速发展而不断更新,由早期以生化技术为基础的研究方法转变为以现代分子生物学技术为基础的研究方法。综述了空气微生物群落多样性解析方法从培养到非培养的发展过程,包括培养技术法、BIOLOG技术、生物标记法、基因指纹图谱技术、核酸杂交技术、实时荧光定量PCR、空气微生物宏基因组学及基因芯片技术,阐述了这些技术的基本原理,比较了各种技术的优缺点并重点介绍了它们在空气微生物群落多样性研究中的应用概况,最后展望了空气微生物学研究的发展方向。
空气微生物多样性;生化技术;分子生物学技术;宏基因组学
随着世界范围内流行性疾病以及我国空气雾霾事件的频繁发生,空气生物性污染的研究开始受到高度重视。空气中广泛分布的细菌、真菌孢子、放线菌和病毒等微生物粒子与空气环境质量以及人类健康等密切相关[1-2],小部分具有活性的生物粒子能够导致各种微生物疾病的发生[3]。由生物粒子组成的气溶胶,可间接作为云凝结核和冰核,导致云滴和冰晶的形成,在雨雪的形成过程中发挥着非常重要的功能,并且可通过直接散射或吸收太阳能辐射在全球气候变化中起着至关重要的作用[4-5]。到目前为止,空气微生物的研究工作已有约180年的历史,其研究方法也由传统微生物培养法逐步转化到现代分子生物学法。传统的微生物培养法只能检测空气中可培养微生物,约占空气中微生物总数的0.1%—10%,在营养贫瘠的空气中比例可能更少,因此以可培养微生物的种类和浓度检测空气微生物的种群和数量会严重低估空气中微生物的存在[6]。然而,以PCR为基础的现代分子生物学法能够检测到空气中微生物的全部信息,真实反映空气中微生物的存在情况。文章综述了空气微生物多样性解析方法的发展过程及应用概况,并阐述了各种研究方法的优缺点及发展方向。
1 以生化技术为基础的空气微生物研究方法
以生化技术为基础的方法是空气微生物学研究早期发展起来的,主要包括传统的微生物平板计数与形态学分析方法,以微生物碳源利用能力为基础的解析方法,基于生物标记的解析方法等,这些方法在空气微生物的研究历程中发挥着巨大的作用(表1)。

表1 以生化技术为基础的空气微生物研究方法
1.1基于传统的平板计数与形态学分析技术
平板计数与形态学分析是传统的空气微生物研究方法,一般用自然沉降或采样器把空气中的微生物采集到液体、固体或半固体的采样介质上,再经过培养繁殖生长成菌落后计数、然后进行分离和纯化,利用显微镜对其进行形态学观察,并结合其生理生化特征鉴定种属分类特性[13]。早期空气中微生物的发现也是基于以培养为基础的传统的平板计数与形态学研究方法,并且其在空气微生物学发展过程中起了非常重要的作用。早期Lindemann、Lighthart和Shaffer等利用传统的培养方法做了大量的空气微生物学研究,取得了丰硕的成果[14-23]。Shelton等研究了美国境内室内外空气真菌的群落结构特征,为美国工业卫生学者、变态反应症专科医师和其他公共卫生从业人员提供了可培养空气真菌的比较信息[24]。进入20世纪90年代,我国空气微生物的研究逐渐兴起,军事医学科学院微生物流行病研究所的胡庆轩先生是我国较早研究空气微生物的学者,他所采用的研究方法都是以培养为基础的,为我国学者空气微生物的研究奠定了基础[25-26]。之后,方治国、凌琪等分别研究了我国不同城市室内外空气微生物的污染特征,为空气微生物污染的控制及城市管理部门制定环境政策法规提供了理论指导[27-33]。以上研究表明,传统的平板计数与形态学分析方法对于空气微生物来说是一种较好的研究方法,为世界范围内空气微生物基础数据的储备提供了基础。
1.2基于空气微生物碳源利用差异的解析方法
基于空气微生物碳源利用差异的分析方法主要是根据微生物代谢类型和对不同碳源的利用能力来评价微生物种类多样性,其典型代表方法有BIOLOG技术。BIOLOG微生物鉴定系统是由美国的BIOLOG公司于1989年成功研发的,最初主要应用于纯种微生物鉴定[34]。1991年,Garland 和Mills首次将BIOLOG技术用来描述微生物群落功能多样性特征,并在当时引起了微生物生态学者的广泛关注[34],之后较长时间BIOLOG技术在环境微生物功能多样性评价研究中得到了广泛的应用,并在土壤、沉积物和水体等环境中微生物群落多样性研究中发挥了至关重要的作用[35-37]。将BIOLOG技术用来研究微生物群落功能多样性的理论依据是BIOLOG代谢多样性类型的变化与微生物群落组成变化密切相关[38],其原理是利用以微生物群落水平碳源利用类型为基础的BIOLOG氧化还原技术来表述环境样品微生物群落特征,运用主成分分析或相似类型的多变量统计分析方法展示不同微生物群落产生的不同代谢多样性类型[39]。近年来,BIOLOG技术逐渐应用到空气中微生物碳代谢能力的评价中,并且取得了较好的研究效果。凌琪、龚婵娟和段魏魏等利用BIOLOG技术分别研究了黄山风景区、杭州不同样点及塔克拉玛干沙漠空气中微生物的碳代谢特征[40-42]。BIOLOG技术为空气微生物的研究提供了一种全新的思路,能在空气微生物功能多样性研究方面发挥重要的作用。
1.3基于生物标记的空气微生物解析方法
基于生物标记的空气微生物分析方法主要根据部分生物组分可以作为某些种类微生物的指示标记,以该组分浓度和种类等的变化来指示相关微生物的变化,其主要分为醌指纹法和脂肪酸谱图法。目前,应用在生物气溶胶领域以脂肪酸谱图法为主。该方法主要步骤是通过提取生物气溶胶中某种生化成分作为生物量标记物,利用气相、液相、气质联用等技术手段对该生化成分进行分类鉴定,其组成模式可作为种群组成的标记。在空气微生物研究中,3-羟基脂肪酸(10—18碳)可以指示标记内毒素(脂多糖)和革兰氏阴性菌生物量负荷,支链脂肪酸(15—17碳)可以指示标记革兰氏阳性菌生物量负荷,麦角甾醇可以指示标记真菌生物量负荷,胞壁酸可以指示标记肽聚糖[43]。在基于生物标记的空气微生物解析方法方面,香港理工大学Lau课题组等做了大量的研究工作,开创了利用生物标志法来确定生物气溶胶中特定微生物群落的先河[44-48],他们的研究确定了生物标记法能够为气溶胶中微生物群落提供定量信息。此外,Bauer等通过测定阿拉伯糖醇和甘露醇的含量对空气中真菌孢子进行定量示踪[49];Poole等利用生物标记法的研究结果解释了人体单核细胞和上皮细胞对农业灰尘的炎症反应[50];Filippo等利用麦角甾醇,阿拉伯糖醇和甘露醇等作为示踪剂分析了城市气溶胶的真菌孢子浓度[51]。综上所述,生物标记法是空气中微生物定量研究的又一选择,正逐步用于生物气溶胶群落结构的分型和动态监测,并且研究方法日趋成熟,比传统培养法在取样和物种鉴定方面更简单更快。
2 以现代分子生物学技术为基础的空气微生物研究方法
由于空气环境要素的复杂多变,微生物纯种分离富集培养的方法不但费时费力,而且存在方法学上的缺陷。一是空气中大量微生物的不可培养性,使人类无法培养空气中所有的微生物[6],二是分离富集培养方法具有较强的选择性,使培养得到的空气微生物在种类、数量和功能上都无法反映其微生物群落的真实情况[52]。因此,非常有必要研究不依赖微生物培养来进行空气微生物群落分析的方法,它们主要包括:聚合酶链式反应(PCR)、变性梯度凝胶电泳(Denaturing gradient gel electrophoresis, DGGE)和温度梯度凝胶电泳(Temperature gradient gel electrophoresis, TGGE)、末端限制性酶切片段长度多态性分析(Terminal restriction fragment length polymorphism, T-RFLP)、基因克隆文库分析(Gene cloning library)、实时荧光定量PCR(RT-PCR)、宏基因组学(Metagenomics)、基因芯片(DNA microarray)等[53](表2)。
2.1基于PCR为基础的基因指纹图谱技术
2.1.1DNA 成分多态性图谱分析(DGGE/TGGE)
DGGE是由Fischer 等于1979 年首先提出的用于检测DNA 突变的一种电泳技术,其分辨力高于琼脂糖电泳和聚丙烯酰胺凝胶电泳[64],Muzyer 等首次将DGGE技术应用于分子微生物学研究领域,其在揭示自然界微生物群落遗传多样性和种群差异方面具有明显优越性[65],而TGGE是在DGGE 基础上衍生出的微生物多样性研究的重要手段[66]。DGGE技术是对PCR 扩增微生物 rDNA 产生的DNA 片段混合物的分析,自从DGGE技术首次应用于微生物分子生物学研究以来,该技术迅速被广泛用于土壤[67]、活性污泥[68]、海洋[69]、底泥[54]等样品中微生物多样性分析、微生物鉴定及变异等方面的研究。近年来,DGGE/TGGE已越来越多用于空气中微生物多样性分析的研究。Nehmé 等利用DGGE方法研究了高分娩期猪圈内空气中生物气溶胶粒子的季节分布特征[70],然后利用DGGE和克隆的方法证明了猪圈里存在着高浓度来源于猪粪的产甲烷古生菌,这是首次对生物气溶胶中古生菌的研究[71];Li 等利用DGGE方法比较研究了4种空气微生物取样器的取样效率[72];Hervàs 等利用DGGE方法研究了空气细菌从非洲迁移到欧洲高山湖泊的活力和潜力[73];Lecours 等利用DGGE方法研究了乳牛舍生物气溶胶的特征,结果显示乳牛舍生物气溶胶组分复杂,并且在职业性呼吸系统疾病方面起着非常重要的作用[74]。DGGE/TGGE对于空气中微生物多样性及其群落结构的研究技术已逐步成熟,与其他方法相结合能对空气微生物的结构特征及其物化成分有着更好的阐述。
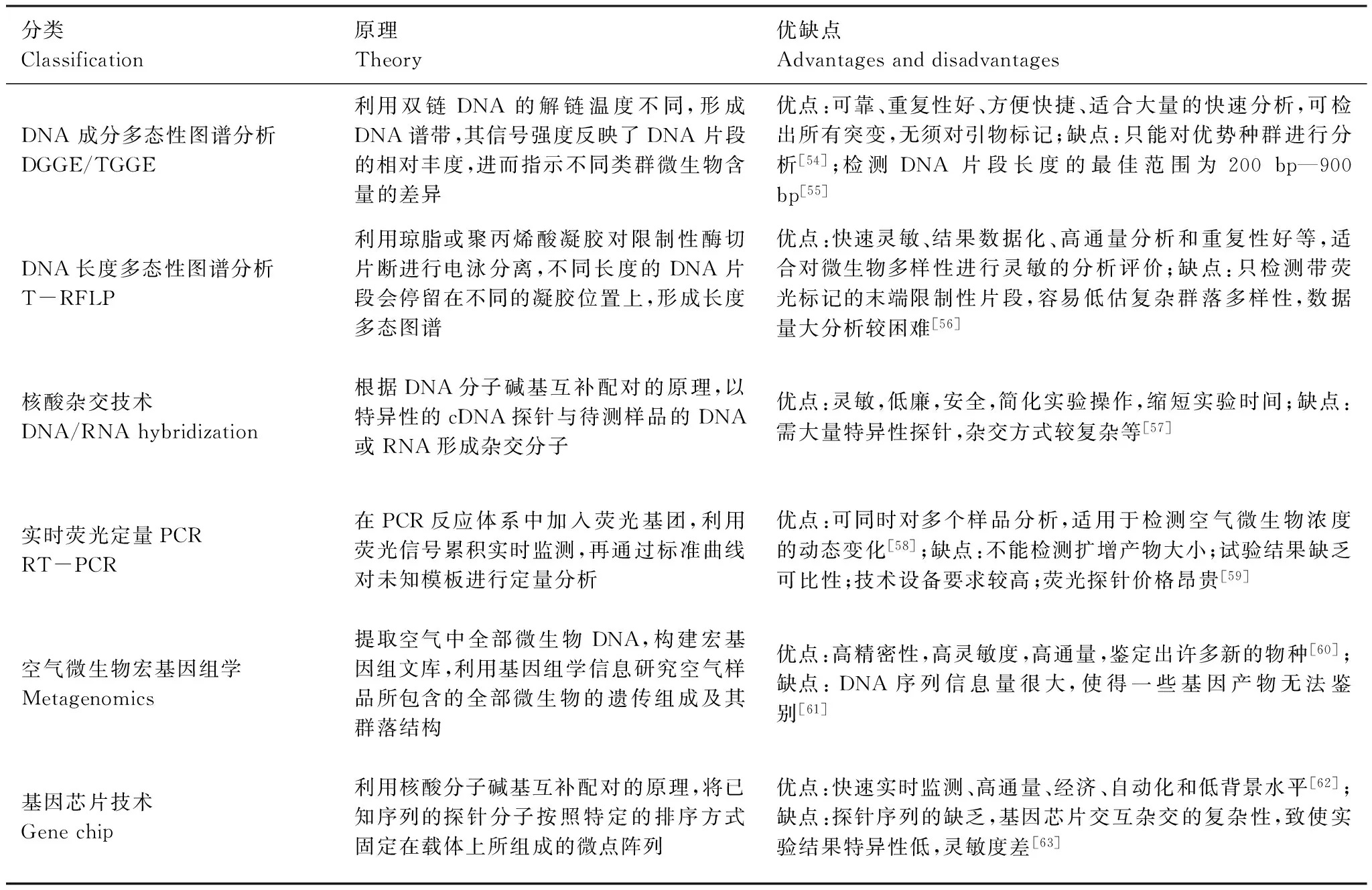
表2 以现代分子生物学技术为基础的空气微生物研究方法
2.1.2DNA长度多态性图谱分析(T-RFLP)
T-RFLP技术是以分子系统学的原理为基础, 综合运用了PCR技术、DNA限制性酶切技术、荧光标记技术和DNA 序列自动分析技术,在DNA水平上通过对特定核酸片段长度多态性的测定来分析比较微生物群落结构和功能[75]。该方法依据微生物的比较基因组学信息,选取一段具有系统进化标记特征的DNA序列为目的分析序列,并据此设计出理想引物,用荧光物质标记出1个或2个引物的5′端(2个引物同时标记时,要用不同的荧光物质标记)。扩增后的PCR 产物用四碱基限制性内切酶进行消化, 荧光标记末端片段可被测序仪识别, 得到不同的末端片段峰, 每一个峰就可至少代表一种类型的微生物, 通常用系统分类操作单元(OTU)来表示。故根据片段峰的大小和数量可以检测和分析空气样品中微生物的末端限制性片段图谱,进而可分析出微生物群体的组成和动态变化等生态信息[76-77]。Lee等利用T-RFLP技术研究了韩国首尔市细菌和真菌的群落结构,并发现其细菌和真菌包括致病菌和过敏原多样性丰富,且不同季节空气细菌和真菌群落结构的多样性和丰富度变化显著[78];Woo等利用T-RFLP技术研究了香港空气微生物群落及与微生物相关过敏原的时间变化特征[79]。Maron等通过基因片段之间的PCR扩增获得长度多态性,其在空气微生物群落研究中起着非常重要的作用[80]。
2.2核酸杂交技术
目前应用最广泛的是近年来发展起来的荧光原位杂交技术(Fluorescence in situ hybridization, FISH),其结合了分子生物学的精确性和显微镜的可视性信息,可以在自然或人工的微生境中监测和鉴定不同的微生物个体,同时对微生物群落进行评价[81-82]。FISH的应用原理是,微生物细胞脱水后,根据DNA碱基配对原则,在不改变其结构和分布格局的情况下与渗透到细胞结构内具有荧光标记的寡核苷酸探针在一定温度下进行定位杂交,只有与探针碱基对完全互补的RNA序列才能与探针发生杂交,经荧光素(FITC)标记的特异性抗体在荧光显微镜下,可清晰地描绘出与DNA靶结合部位,从而使该菌体在荧光显微镜下发出荧光。Lange等运用了FISH技术对暴露在空气中的细菌进行评估,定量识别出生物气溶胶中的绿脓杆菌和大肠杆菌,为生物气溶胶的评估提供了有效的手段[83];Hung等运用核酸杂交技术分析了从空气样品中分离的72个目标菌株和66个非目标参照菌株,其灵敏度和特异度都达到100%[84];Kristiansen等应用荧光标记一般细菌探针和处于分娩期猪圈内气溶胶细胞进行原位杂交,发现厚壁菌门和链球菌为主要的细菌属种,还发现了多样性丰富的真菌群落[85]。FISH技术已被用来研究空气细菌的多样性信息,为空气微生物种类及其分布特征的研究提供了全新的方法。
2.3实时荧光定量PCR(RT-PCR)
RT-PCR技术作为一种核酸定量的手段,能够准确量化环境中微生物群落的组成[86],确定群落的优势类群或具有某种生物学功能的微生物类群的丰度[87],能够检测低浓度空气细菌浓度,其在空气微生物学研究方面具有非常重要的作用。Lee等利用荧光标记的特异性引物对空气中细菌和真菌进行PCR扩增,确定了空气中总悬浮颗粒物中的细菌和真菌的浓度[78];Makino等把炭疽直接加入到空气微生物采样液中,然后用RT-PCR进行了定量分析,结果发现1个炭疽细胞在1 h内就可检出[88];Yamamoto等优化RT-PCR反应条件来定量收集在凝胶过滤器上的空气真菌浓度[89];Rinsoz等对家禽厂和污水处理厂空气中全部数量细菌进行评估,通过将RT-PCR与荧光显微技术及培养方法相比较,证明RT-PCR技术是一种可行有效,精确且简单的方法,能够在复杂多变的环境中评估空气细菌负荷[90-91]。随着RT-PCR技术不断发展和其使用成本的逐渐下降,其在空气微生物学研究中应能得到更广泛的应用。
2.4空气微生物宏基因组学
环境基因组学通过直接从环境样品中提取全部微生物的DNA,构建宏基因组文库,利用基因组学信息研究环境样品所包含的全部微生物的遗传组成及其群落结构,避免了传统微生物学基于纯培养研究的限制,为充分认识和开发利用不可培养微生物,并从完整的群落水平上认识微生物的活性提供了可能[92-93]。Tringe等对城市室内环境的空气样本进行宏基因组研究,发现空气中隐藏着一个独特的社区,可能源于多种生态位,并通过空气环境中的选择性压力发展形成[94];Cao等在雾霾天气利用宏基因组学研究了北京建筑物顶楼顶的空气样品,发现大部分的可吸入性空气微生物宏基因组和土壤宏基因组密切相关,且几种与呼吸道疾病相关的致病菌丰富度随着悬浮性颗粒污染物的浓度增加而增加[95];Oh等运用宏基因组学研究发现汉城的春季空气真菌具有较高多样性,且含有高浓度的真菌过敏原[96];Whon等运用宏基因组学第一次报道了空气中病毒的季节性和其遗传多样性,从而提高在温带地区对病毒生态学的理解[97]。由于空气中微生物的极低的浓度,对16S或18S核糖体基因组的直接分析无法实现,可通过改进采样和扩增的方法来获得足够的DNA,能够运用宏基因组学来分析,从这些样品中产生的宏基因组数据的分析揭示空气微生物群落中的多样性和不同丰度的种属,还可以对细菌的可能来源进行识别[98]。
2.5基因芯片技术
基因芯片又称DNA微阵列,芯片上固定的探针有DNA片段、寡核苷酸、cDNA或来自基因组的基因片段,它们固化于芯片上形成基因探针阵列。经过标记的核苷酸序列与基因芯片特定位点上的探针进行杂交, 然后通过检测杂交信号定性和定量地判断样品中的靶序列分子[99]。目前基因芯片技术已应用于粪便、临床、环境和食品样本等的微生物群落结构研究,主要包括功能基因、特定细菌和病毒的检测及其菌群分析等[100]。近年来,基因芯片技术在空气微生物的研究中得到充分发展,它不仅可以分析和鉴别空气中微生物群落的微生物分布、种类及功能,还能分析空气中环境因素改变对其微生物生态的影响。Hung等使用微阵列技术同时对空气中几种过敏原进行检测和定量测定,鉴定出21个空气真菌物种,且这些真菌都能够导致人们过敏[84];Brodie等使用高密度DNA微阵列监测了美国两个城市的细菌种群,发现这些城市气溶胶中包含至少有1800不同的细菌种类,可能比某些土壤细菌群落更丰富[101]。随着我国空气污染的不断加剧,空气中的过敏原的种类和数量也在发生较大的变化,随着基因芯片技术不断发展,可以同步定量多个过敏原,可为空气质量的控制做出较大贡献[102]。
3 空气微生物学研究的挑战和趋势
(1)不断扩大研究范围以获取更加全面系统的空气微生物研究第一手资料。我国空气中微生物的研究主要集中在城市空气微生物多样性特征的研究,然而从生态学角度对自然风景区和风景旅游区等生态系统以及从公共卫生学角度对地铁和超市等室内公共场所空气微生物的研究报道较少。因此,有必要研究与人类活动密切相关的各类环境空气微生物群落及多样性特征。
(2)不断完善空气微生物多样性的解析技术和方法以获取更多的微生物信息。我国空气微生物学的研究起步较晚,分子生物学技术在空气微生物研究中应用较少。因此,应该利用传统的微生物学与现代分子生物学相结合的方法对不同环境空气微生物多样性信息进行深入的研究。
(3)不断优化空气微生物取样技术和研发新型高流量取样器以保证有足够DNA样本进行分子生物学研究。空气中微生物的种类较少浓度较低,空气微生物基因组DNA的提取比其它样品更加困难。因此,有必要研发高流量取样技术为空气微生物的分子生物学研究提供样品保障。
(4)不断扩大研究尺度以从生物地理学的角度获取空气微生物图谱。目前空气微生物学的研究尺度较小,取样覆盖面较窄。因此,随着高通量和大数据时代的到来,空气微生物的研究有必要扩大至区域尺度甚至全国范围,从生物地理学角度制作全国范围的空气微生物图谱。
(5)不断研究空气微生物与环境因素及污染因子之间的关系以获取空气微生物污染的形成机制。目前城市空气微生物污染的形成机制研究较少。因此,有必要系统研究空气微生物污染与环境因素及污染因子之间的关系,掌握城市空气微生物污染的形成机制,为城市空气中生物性污染的治理提供理论依据。
(6)不断统一空气微生物研究体系完善其综合评价系统以能够对不同环境条件下空气微生物研究指标进行综合评价。采样条件和采样基质对空气微生物检测有重要的影响,需对空气微生物研究的培养基和取样方法制定统一标准,同时加强空气微生物污染与人体健康之间的关系研究,且对空气微生物作为人体健康状况的指标进行综合评价。
[1]Alan M Jones, Roy M Harrison. The effects of meteorological factors on atmospheric bioaerosol concentrations-a review. Science of the Total Environment, 2004, 326(1/3): 151-180.
[2]Li C S, Hsu L Y. Airborne fungus allergen in association with residential characteristics in atopic and control children in a subtropical region. Archives of Environmental Health: An International Journal, 1997, 52(1): 72-79.
[3]Kalogerakis N, Paschali D, Lekaditis V, Pantidou A, Eleftheriadis K, Lazaridis M. Indoor air quality: bioaerosol measurements in domestic and office premises. Journal of Aerosol Science, 2005, 36(5/6): 751-761.
[4]Brent C Christner, Cindy E Morris, Christine M Foreman, Cai R M, David C Sands. Ubiquity of biological ice nucleators in snowfall. Science, 2008, 319(5867): 1214-1214.
[5]Brent C Christner, Cai R M, Cindy E Morris, Kevin S McCarter, Christine M Foreman, Mark L Skidmore, Scott N Montross, David C Sandsg. Geographic, seasonal, and precipitation chemistry influence on the abundance and activity of biological ice nucleators in rain and snow. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(48): 18854-18859.
[6]Amann RI, Ludwig W, Schleifer KH. Phylogenetic identification and in situ detection of individual microbial cells without cultivation. Microbiological Reviews, 1995, 59(1): 143-169.
[7]Fang Z G, Ouyang Z Y, Hu L F, Wang X K, Zheng H, Lin X Q. Culturable airborne fungi in outdoor environments in Beijing, China. Science of the Total Environment, 2005, 350: 47-58.
[8]Fang Z G, Ouyang Z Y, Zheng H, Wang X K, Hu L F. Culturable airborne bacteria in outdoor environments in Beijing, China. Microbial Ecology, 2007, 54(3): 487-496.
[9]郑华, 欧阳志云, 方治国, 赵同谦. BIOLOG在土壤微生物群落功能多样性研究中的应用. 土壤学报, 2004, 41(3): 456-461.
[10]Juliet Preston-Mafham, Lynne Boddy, Peter F Randerson. Analysis of microbial community functional diversity using sole-carbon-source utilisation profiles-a critique. FEMS Microbiology Ecology, 2002, 42(1): 1-14.
[11]车玉伶, 王慧, 胡洪营, 梁威, 郭玉凤. 微生物群落结构和多样性解析技术研究进展. 生态环境, 2005, 14(1): 127-133.
[12]Hill G T, Mitkowski N A, Aldrich-Wolfe L, Emele L R, Jurkonie D D, Ficke A, Maldonado-Ramirez S, Lynch S T, Nelson E B. Methods for assessing the composition and diversity of soil microbial communities. Applied Soil Ecology, 2000, 15(1): 25-36.
[13]Fabian M P, Reponen T, Miller S L, Hernandez M T. Total and culturable airborne bacteria and fungi in Arid region flood-damaged residences. Journal of Aerosol Science, 2000, 31: 35-36.
[14]Julianne Lindemann, Helen A Constantinidou, William R Barchet, Christen D Upper. Plants as sources of airborne bacteria, including ice nucleation-active bacteria. Applied and Environmental Microbiology, 1982, 44(5): 1059-1063.
[15]Lindermann J, Upper C D. Aerial dispersal of epiphytic bacteria over bean plants. Applied and Environmental Microbiology, 1985, 50(5): 1229-1232.
[16]Bruce Lighthart. Survival of airborne bacteria in a high urban concentration of carbon monoxide. Applied and Environmental Microbiology, 1973, 25(1): 86-91.
[17]Bruce Lighthart. Microbial aerosols: estimated contribution of combine harvesting to an airshed. Applied and Environmental Microbiology, 1984, 47(2): 430-432.
[18]Lightart B, Kin J. Simulation of airborne microbial droplet transport. Applied and Environmental Microbiology, 1989, 55(9): 2349-2355.
[19]Bruce Lighthart, Brenda T Shaffer, Balkumar Marthi, Lisa M Ganio. Artificial wind-gust liberation of microbial bioaerosol previously deposited on plants. Aerobiologia, 1993, 9(2/3): 189-196.
[20]Lighthart B, Shaffer B T. Bacterial flux from chaparral into the atmosphere in mid-summer at a high desert location. Atmospheric Environment, 1994, 28(7): 1267-1274.
[21]Lightart B, Shaffer B T. Airborne bacteria in the atmospheric surface layer: Temporal distribution above a grass seed field. Applied and Environmental Microbiology, 1995, 61(4): 1492-1496.
[22]Bruce Lighthart, Brenda T Shaffer. Viable bacterial aerosol particle size distributions in the midsummer atmosphere at an isolated location in the high desert chaparral. Aerobiologia, 1995, 11(1): 19-25.
[23]Shaffer B T, Lighthart B. Survey of culturable airborne bacteria at four diverse locations in Oregon: urban, rural, forest, and coastal. Microbial Ecology, 1997, 34(3): 167-177.
[24]Brian G Shelton, Kimberly H Kirkland, W Dana Flanders, George K Morris. Profiles of airborne fungi in buildings and outdoor environments in the United States. Applied and Environmental Microbiology, 2002, 68(4): 1743-1753.
[25]Hu Q X, Cai Z L, Lu Z X. Preliminary approach on the status of indoor and outdoor airborne fungi particles. Shanghai Environmental Sciences, 1999, 18(10): 454-456.
[26]胡庆轩, 车凤翔, 徐秀芝, 姜黎, 鹿建春, 刘敏霞. 微孔滤膜空气微生物采样器的研制. 卫生研究, 1999, 28 (4): 255-256.
[27]Fang Z G, Ouyang Z Y, Zheng H, Wang X K. Concentration and size distribution of culturable airborne microorganisms in outdoor environments in Beijing, China. Aerosol Science and Technology, 2008, 42(5): 325-334.
[28]凌琪, 王晏平, 王莉, 舒莹, 陶勇, 鲍立宁. 合肥城区空气真菌浓度的时空分布特征. 生物多样性, 2008, 16(2): 175-180.
[29]凌琪, 王晏平, 陶勇, 王莉, 舒莹. 合肥市2004~2006 年空气细菌污染状况调查. 中国公共卫生管理, 2007, 23(1): 67-69.
[30]Fang Z G, Gong C J, Ouyang Z Y, Liu P, Sun L, Wang X Y. Characteristic and concentration distribution of culturable airborne bacteria in residential environments in Beijing, China. Aerosol and Air Quality Research, 2014, 14(3): 943-953.
[31]方治国, 欧阳志云, 刘芃, 孙力, 王小勇. 城市居家环境空气真菌群落结构特征研究. 环境科学, 2013, 34(5): 2031-2037.
[32]方治国, 孙平, 欧阳志云, 刘芃, 孙力, 王小勇. 北京市居家空气微生物粒径及分布特征研究. 环境科学, 2013, 34(7): 2526-2532.
[33]方治国, 欧阳志云, 刘芃, 孙力, 王小勇. 北京市居家空气微生物污染特征. 环境科学学报, 2013, 33(4): 1166-1172.
[34]Jay L Garland, Aaron L Mills. Classification and characterization of heterotrophic microbial communities on the basis of patterns of community-level sole-carbon-source utilization. Applied and Environmental Microbiology, 1991, 57(8): 2351-2359.
[35]Krista L De Fede, Daniel G Panaccione, Alan J Sexstone. Characterization of dilution enrichment cultures obtained from size-fractionated soil bacteria by BIOLOG©community-level physiological profiles and restriction analysis of 16s rRNA genes Soil Biology and Biochemistry, 2001, 33(11): 1555-1562.
[36]Krista L De Fede, Alan J Sexstone. Differential response of size-fractionated soil-fractionated soil bacteria in BIOLOG©microtitre plates. Soil Biology and Biochemistry, 2001, 33(11): 1547-1554.
[37]Kaiser S K, Guckert J B, Gledhill D W. Comparison of activated sludge microbial communities using biologTMmicroplates. Water Science and Technology, 1998, 37(4/5): 57-63.
[38]Haack S K, Garchow H, Klug M J, Forney L J. Analysis of factors affecting the accuracy, reproducibility, and interpretation of microbial community carbon source utilization patterns. Applied and Environmental Microbiology, 1995, 61(4): 1458-1468.
[39]Christine A Hackett, Bryan S Griffiths. Statistical analysis of the time-course of biolog substrate utilization. Journal of Microbiological Methods, 1997, 30(1): 63-69.
[40]凌琪, 包金梅, 李瑞, 陶勇, 鲍立宁, 毛钦焱. Biolog-Eco解析黄山风景区空气微生物碳代谢多样性特征. 应用基础与工程科学学报, 2012, 20(1): 56-63.
[41]龚婵娟, 许晶, 方治国, 楼秀芹, 欧阳志云. 杭州市空气微生物群落碳代谢特征研究. 环境科学, 2014, 35(2): 753-758.
[42]段魏魏, 娄恺, 曾军, 胡蓉, 史应武, 何清, 刘新春, 孙建, 晁群芳. 塔克拉玛干沙尘暴源区空气微生物群落的代谢特征. 环境科学, 2012, 33(1): 26-31.
[43]Aleksandra Sebastian, Lennart Larsson. Characterization of the microbial community in indoor environments: a chemical-analytical approach. Applied and Environmental Microbiology, 2003, 69(6): 3103-3109.
[44]Alex K Y Lee, Chak K Chan, Fang M, Arthur P S Lau. The 3-hydroxy fatty acids as biomarkers for quantification and characterization of endotoxins and Gram-negative bacteria in atmospheric aerosols in Hong Kong. Atmospheric Environment, 2004, 38(37): 6307-6317.
[45]Arthur P S Laua, Alex K Y Lee, Chak K Chan, Fang M. Ergosterol as a biomarker for the quantification of the fungal biomass in atmospheric aerosols. Atmospheric Environment, 2006, 40(2): 249-259.
[46]Alex K Y Lee, Arthur P S Lau, Jessica Y W Cheng, Fang M, Chak K Chan. Source identification analysis for the airborne bacteria and fungi using a biomarker approach. Atmospheric Environment, 2007, 41(13): 2831-2843.
[47]Jessica Y W Cheng, Arhur P S Lau, Fang M. Assessment of the atmospheric fungal prevalence through field ergosterol measurement I—Determination of the specific ergosterol content in common ambient fungal spores and yeast cells. Atmospheric Environment, 2008, 42(22): 5526-5533.
[48]Jessica Y W Cheng, Arthur P S Lau, Fang M. Assessment of the atmospheric fungal prevalence through field ergosterol measurement II: Establishing the conversion factor. Atmospheric Environment, 2008, 42(22): 5534-5541.
[49]Heidi Bauer, Magda Claeys, Reinhilde Vermeylen, Elisabeth Schueller, Gert Weinke, Anna Berger, Hans Puxbaum. Arabitol and mannitol as tracers for the quantification of airborne fungal spores. Atmospheric Environment, 2008, 42(3): 588-593.
[50]Jill A Poole, Gregory P Dooley, Rena Saito, Angela M Burrell, Kristina L Bailey, Debra J Romberger, John Mehaffy, Stephen J Reynolds. Muramicacid, endotoxin, 3-hydroxy fatty acids, and ergosterol content explain monocyte and epithelial cell inflammatory responses to agricultural dusts. Journal of Toxicology and Environment Health, 2010, 73(10): 684-700.
[51]Patrizia Di Filippo, Donatella Pomata, Carmela Riccardi, Francesca Buiarelli, Cinzia Perrino. Fungal contribution to size-segregated aerosol measured through biomarkers. Atmospheric Environment, 2013, 64: 132-140.
[52]Ramon Rosselló-Mora, Rudolf Amann. The species concept for prokaryotes. FEMS Microbiology Reviews, 2001, 25(1): 39-67.
[53]Jordan Peccia, Mark Hernandez. Incorporating polymerase chain reaction-based identification, population characterization, and quantification of microorganisms into aerosol science: a review. Atmospheric Environment, 2006, 40(21): 3941-3961.
[54]Ferris M J, Muyzer G, Ward D M. Denaturing gradient gel electrophoresis profiles of 16S rRNA-defined populations inhabiting a hot spring microbial mat community. Applied and Environmental Microbiology, 1996, 62(2): 340-346.
[55]Fromin N, Hamelin J, Tarnawski S, Roesti D, Jourdain-Miserez K, Forestier N, Teyssier-Cuvelle S, Gillet F, Aragno M, Rossi P. Statistical analysis of denaturing gel electrophoresis (DGE) fingerprinting patterns. Environmental Microbiology, 2002, 4(11): 634-643.
[56]王洪媛, 管华诗, 江晓路. 微生物生态学中分子生物学方法及T-RFLP技术研究. 中国生物工程杂志, 2004, 24(8): 42-47.
[57]胡稳奇, 张志光. 核酸杂交技术在环境微生物检测中的应用. 微生物学通报, 1995, 22(6): 371-374.
[58]Hey Reoun An, Gediminas Mainelis, Lori White. Development and calibration of real-time PCR for quantification of airborne microorganisms in air samples. Atmospheric Environment, 2006, 40(40): 7924-7939.
[59]王曙光, 林先贵, 刁晓君. 环境微生物研究方法与应用. 北京: 化学工业出版社, 2008: 77-80.
[60]Qin J J, Li Y R, Cai Z M, Li S H, Zhu J F, Zhang F, Liang S S, Zhang W W, Guan Y L, Shen D Q, Peng Y Q, Zhang D Y, Jie Z Y, Wu W X, Qin Y W, Xue W B, Li J H, Han L C, Lu D H, Wu P X, Dai Y L, Sun X J, Li Z S, Tang A F, Zhong S L, Li X P, Chen W N, Xu R, Wang M B, Feng Q, Gong M H, Yu J, Zhang Y Y, Zhang M, Torben Hansen, Gaston Sanchez, Jeroen Raes, Gwen Falony, Shujiro Okuda, Mathieu Almeida, Emmanuelle LeChatelier, Pierre Renault, Nicolas Pons, Jean-Michel M Batto, Zhang Z X, Chen H, Yang R F, Zheng W M, Li S G, Yang H M, Wang J, S Dusko Ehrlich, Rasmus Nielsen, Oluf Pedersen, Karsten Kristiansen, Wang J. A metagenome-wide association study of gut microbiota in type 2 diabetes. Nature, 2012, 490(7418): 55-60.
[61]Willy Valdivia-Granda. The next meta-challenge for Bioinformatics. Bioinformation, 2008, 2(8): 358-362.
[62]Shalon D, Smith S J, Brown P O. A DNA microarray system for analyzing complex DNA samples using two-color fluorescent probe hybridization. Genome Research, 1996, 6(4): 639-645.
[63]Zhou J Z, Dorothea K Thompson. Challenges in applying microarrays to environmental studies. Current Opinion in Biotechnology, 2002, 13(3): 204-207.
[64]Fischer S G, Lerman L S. DNA fragments differing by single base-pair substitutions are separated in denaturing gradient gels: correspondence with melting theory. Proceedings of the National Academy of Sciences of the United States of America, 1983, 80(6): 1579-1583.
[65]Muyzer G, de Waal E C, Uitterlinden A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA. Applied and Environmental Microbiology, 1993, 59(3): 695-700.
[66]Gerard Muyzer. DGGE/TGGE a method for identifying genes from natural ecosystems. Current Opinion in Microbiology, 1999, 2(3): 317-322.
[67]Smalla K, Wieland G, Buchner A, Zock A, Parzy J, Kaiser S, Roskot N, Heuer H, Berg G. Bulk and rhizosphere soil bacterial communities studied by denaturing gradient gel electrophoresis: plant-dependent enrichment and seasonal shifts revealed. Applied and Environmental Microbiology, 2001, 67(10): 4742-4751.
[68]Wen-Tso Liua, On-Chim Chan, Herbert H P Fang. Microbial community dynamics during start-up of acidogenic anaerobic reactors. Water Research, 2002, 36(13): 3203-3210.
[69]Andreas Teske, Wawer C, Muyzer G, Ramsing N B. Distribution of sulfate-reducing bacteria in a stratified fjord (Mariager Fjord, Denmark) as evaluated by most-probable-number counts and denaturing gradient gel electrophoresis of PCR-amplified ribosomal DNA fragment. Applied and Environmental Microbiology, 1996, 62(4): 1405-1415.
[70]Benjamin Nehme, Valérie Létourneau, Robert J Forster, Marc Veillette, Caroline Duchaine. Culture-independent approach of the bacterial bioaerosol diversity in the standard swine confinement buildings, and assessment of the seasonal effect. Environmental Microbiology, 2008, 10(3): 665-675.
[71]Benjamin Nehmé, Yan Gilbert, Valérie Létourneau, Robert J Forster, Marc Veillette, Richard Villemur, Caroline Duchaine. Culture-independent characterization of archaeal biodiversity in swine confinement building bioaerosols. Applied and Environmental Microbiology, 2009, 75(17): 5445-5450.
[72]Li K J. Molecular comparison of the sampling efficiency of four types of airborne bacterial samplers. Science of the Total Environment, 2011, 409(24): 5439-5498.
[73]Anna Hervàs, Lluís Camarero, Isabel Reche, Emilio O Casamayor. Viability and potential for immigration of airborne bacteria from Africa that reach high mountain lakes in Europe. Environmental Microbiology, 2009, 11(6): 1612-1623.
[74]Pascale Blais Lecours, Marc Veillette, David Marsolais, Caroline Duchaine. Characterization of bioaerosols from dairy barns: reconstructing the puzzle of occupational respiratory diseases by using molecular approaches. Applied and Environmental Microbiology, 2012, 78(9): 3242-3248.
[75]Liu W T, Marsh T L, Cheng H, Forney L J. Characterization of microbial diversity by determining terminal restriction fragment length polymorphisms of genes encoding 16S rRNA. Applied and Environment Microbiology, 1997, 63(11): 4516-4522.
[76]Terence L Marsh. Terminal restriction fragment length polymorphism (T-RFLP): An emerging method for characterizing diversity among homologous populations of amplification products. Current Opinion in Microbiology, 1999, 2(3): 323-327.
[77]Christopher L Kitts. Terminal restriction fragment patterns: a tool for comparing microbial communities and assessing community dynamics. Current Issues in Intestinal Microbiology, 2001, 2(1): 17-25.
[78]Seung-Hoon Lee, Hyo-Jeong Lee, Se-Jin Kim, Hyung Min Lee, Hojeong Kang, Yong Pyo Kim. Identification of airborne bacterial and fungal community structures in an urban area by T-RFLP analysis and quantitative real-time PCR. Science of the Total Environment, 2010, 408(6): 1349-1357.
[79]Anthony C Woo, Manreetpal S Brar, Yuki Chan, Maggie C Y Lau, Frederick C C Leung, James A Scott, Lilian L P Vrijmoed, Peyman Zawar-Reza, Stephen B Pointing. Temporal variation in airborne microbial populations and microbially-derived allergens in a tropical urban landscape. Atmospheric Environment, 2013, 74: 291-300.
[80]Pierre-Alain Maron, David P H Lejon, Esmeralda Carvalho, Karine Bizet, Philippe Lemanceau, Lionel Ranjard, Christophe Mougela. Assessing genetic structure and diversity of airborne bacterial communities by DNA fingerprinting and 16S rDNA clone library. Atmospheric Environment, 2005, 39(20): 3687-3695.
[81]Ewa Korzeniewska, Monika Harnisz. Culture-dependent and culture-independent methods in evaluation of emission ofEnterobacteriaceaefrom sewage to the air and surface water. Water, Air, & Soil Pollution, 2012, 223(7): 4039-4046.
[82]Weerasekara M L M A W, Noriko Ryuda, Hiroshi Miyamoto, Toru Okumura, Daisuke Ueno, Koichi Inoue, Takashi Someya. Double-color fluorescenceinsituhybridization (FISH) for the detection ofBacillusanthracisspores in environmental samples with a novel permeabilization protocol. Journal of Microbiological Methods, 2013, 93(3): 177-184.
[83]Lange J L, Thorne P S, Lynch N. Application of flow cytometry and fluorescent in situ hybridization for assessment of exposures to airborne bacteria. Applied and Environmental Microbiology, 1997, 63(4): 1557-1563.
[84]Hung W T. Su S L, Shiu L Y, Chang T C. Rapid identification of allergenic and pathogenic molds in environmental air by an oligonucleotide array. BMC Infectious Diseases, 2011, 11(1): 91-91.
[85]Anja Kristiansen, Aaron M Saunders, Aviaja A Hansen, Per H Nielsen, Jeppe L Nielsen. Community structure of bacteria and fungi in aerosols of a pig confinement building. FEMS Microbiology Ecology, 2012, 80(2): 390-401.
[86]Yongju Heo, Jiyeon Park, Sung-il Lim, Hor-gil Hur, Daesung Kim, Kihong Park. Size-resolved culturable airborne bacteria sampled in rice field, sanitary landfill, and waste incineration sites. Journal of Environmental Monitoring, 2010, 12(8): 1619-1624.
[87]Yuan W, Chai T J, Miao Z M. ERIC-PCR identification of the spread of airborneEscherichiacoliin pig houses. Science of the Total Environment, 2010, 408(6): 1446-1450.
[88]Sou-ichi Makino, Hyeng-il Cheun. Application of the real-time PCR for the detection of airborne microbial pathogens in reference to the anthrax spores. Journal of Microbiological Methods, 2003, 53(2): 141-147.
[89]Naomichi Yamamoto, Minoru Kimura, Hideaki Matsuki, Yukio Yanagisawa. Optimization of a real-time PCR assay to quantitate airborne fungi collected on a gelatin filter. Journal of Bioscience and Bioengineering, 2010, 109(1): 83-88.
[90]Thomas Rinsoz, Philippe Duquenne, Guylaine Greff-Mirguet, Anne Oppligera. Application of real-time PCR for total airborne bacterial assessment: comparison with epifluorescence microscopy and culture-dependent methods. Atmospheric Environment, 2008, 42(28): 6767-6774.
[91]Alvarez A J, Buttner M P, Stetzenbach L D. PCR for bioaerosol monitoring: sensitivity and environmental interference. Applied and Environmental Microbiology, 1995, 61(10): 3639-3644.
[92]沈菊培, 张丽梅, 郑袁明, 朱永官, 贺纪正. 土壤宏基因组学技术及其应用. 应用生态学报, 2007, 18(1): 212-218.
[93]贺纪正, 张丽梅, 沈菊培, 朱永官. 宏基因组学(Metagenomics)的研究现状和发展趋势. 环境科学学报, 2008, 28(2): 209-218.
[94]Susannah G Tringe, Zhang T, Liu X G, Yu Y T, Wah Heng Lee, Jennifer Yap, Yao F, Sim Tiow Suan, Seah Keng Ing, Matthew Haynes, Forest Rohwer, Chia Lin Wei, Patrick Tan, James Bristow, Edward M Rubin, Ruan Y J. The Airborne Metagenome in an Indoor Urban Environment. PLoS One, 2008, 3(4): e1862.
[95]Chen C, Jiang W J, Wang B Y, Fang J H, Lang J D, Tian G, Jiang J K, Zhu T F. Inhalable Microorganisms in Beijing′s PM2. 5and PM10Pollutants during a Severe Smog Event. Environmental Science and Technology, 2014, 48(3): 1499-1507.
[96]Seung-Yoon Oh, Jonathan J Fong, Myung Soo Park, Limseok Chang, Young Woon Lim. Identifying airborne fungi in Seoul, Korea using metagenomics. Journal of Microbiology, 2014, 52(6): 465-472.
[97]Tae Woong Whon, Min-Soo Kim, Seong Woon Roh, Na-Ri Shin, Hae-Won Lee, Jin-Woo Bae. Metagenomic characterization of airborne viral DNA diversity in the near-surface atmosphere. Journal of Virology, 2012, 86(15): 8221-8231.
[98]Shibu Yooseph, Cynthia Andrews-Pfannkoch, Aaron Tenney, Jeff McQuaid, Shannon Williamson, Mathangi Thiagarajan, Daniel Brami, Lisa Zeigler Allen, Jeff Hoffman, Johannes B Goll, Douglas Fadrosh, John Glass, Mark D Adams, Robert Friedman, J C Venter. A metagenomic framework for the study of airborne microbial communities. PLoS One, 2013, 8(12): e81862.
[99]Rick W Ye, Wang T, Laura Bedzyk, Kevin M Croker. Applications of DNA microarrays in microbial systems. Journal of Microbiological Methods, 2001, 47(3): 257-272.
[100]Wu Z H, Yoshihiko Tsumura, Göran Blomquist, Wang X R. 18S rRNA gene variation among common airborne fungi, and development of specific oligonucleotide probes for the detection of fungal isolates. Applied and Environmental Microbiology, 2003, 69(9): 5389-5397.
[101]Eoin L Brodie, Todd Z DeSantis, Jordan P Moberg Parker, Ingrid X Zubietta, Yvette M Piceno, Gary L Andersen. Urban aerosols harbor diverse and dynamic bacterial populations. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(1): 299-304.
[102]Concepción De Linares, Idoia Postigo, Jordina Belmonte, Miguel Canela, Jorge Martínez. Optimization of the measurement of outdoor airborne allergens using a protein microarrays platform. Aerobiologia, 2014, 30(3): 217-227.
Culture-dependent and culture-independent approaches for studying airborne microbial diversity
FANG Zhiguo1,*, HAO Cuimei1, YAO Wenchong1, OUYANG Zhiyun2
1SchoolofEnvironmentalScienceandEngineering,ZhejiangGongshangUniversity,Hangzhou310018,China2StateKeyLaboratoryofUrbanandRegionalEcology,ResearchCenterforEco-EnvironmentalSciences,Chinese,Beijing100085,China
With the occurrence of epidemics worldwide and serious haze events in China, studies on airborne biological pollution have attracted considerable attention. The research approach for airborne microorganisms is constantly updated because of the rapid development of molecular biology techniques, and researchers are switching from biochemistry-based technologies to modern molecular biology-based technologies. In this paper, we reviewed the development processes of airborne microbial communities and analyzed their diversity on the basis of culture-dependent and culture-independent approaches, which mainly include culture-dependent technology, BIOLOG technology, biomarkers, gene fingerprinting techniques, nucleic acid hybridization, real-time quantitative polymerase chain reaction, metagenomics of airborne microorganisms, and gene chip technology. In addition, the basic principles of these technologies have been presented in detail in this paper, and the advantages and disadvantages of various technologies have been compared to highlight their applications in the study of airborne microbial communities and their diversity. Finally, the prospects of airborne microbiology have been emphasized.
airborne microbial diversity; biochemical technology; molecular biology techniques; metagenomics
城市与区域生态国家重点实验室开放基金项目(SKLURE2015-2-1);国家自然科学青年基金资助项目(41005085)
2014-11-27; 网络出版日期:2015-10-30
Corresponding author.E-mail: zhgfang77@zjgsu.edu.cn
10.5846/stxb201411272355
方治国,郝翠梅,姚文冲,欧阳志云.空气微生物群落解析方法:从培养到非培养.生态学报,2016,36(14):4244-4253.
Fang Z G, Hao C M, Yao W C, Ouyang Z Y.Culture-dependent and culture-independent approaches for studying airborne microbial diversity.Acta Ecologica Sinica,2016,36(14):4244-4253.

