一种结合SELEX与二代测序技术筛选RNA结合蛋白特异识别序列新方法的建立
张璐,徐晋暤,麻锦彪
一种结合SELEX与二代测序技术筛选RNA结合蛋白特异识别序列新方法的建立
张璐,徐晋暤,麻锦彪
复旦大学 生命科学学院生物化学系 遗传工程国家重点实验室,上海 200438
RNA结合蛋白通过特异识别RNA底物发挥重要的生物学作用。指数富集的配体系统进化 (Systematic evolution of ligands by exponential enrichment,SELEX) 技术是一种体外筛选核酸底物的基本方法,SELEX技术通过重复多轮筛选从随机核酸序列库中筛选出特异性与靶物质高度亲和的核酸底物,本研究将利用该技术与二代高通量测序 (NGS) 相结合,体外合成含有20个随机碱基的RNA文库,将所要研究的蛋白构建到带有可被链亲和酶素磁珠捕获的SBP标记的载体上去,显著提高筛选效率,仅需1轮筛选即可获得所需RNA底物motif。通过该方法获得了人的hnRNP A1的UP1结构域特异识别AGG和AG二种RNA序列,并通过EMSA实验证实其可以与获得的RNA motif结合。这一方法的建立对于研究RNA结合蛋白识别底物的序列特异性,并进一步了解其在生物体内的调控机制有重要意义。
SELEX,高通量测序技术,hnRNP A1,RNA motif
SELEX技术是Systematic Evolution of Ligands by Exponential Enrichment的缩写,即指数富集的配体系统进化技术。该技术根据DNA/RNA结合蛋白的能力,从随机的DNA/RNA文库中找到高亲和力的DNA/RNA[1-3]。1990年,Tuerk等[4]应用该技术从人工构建的随机寡核苷酸文库中筛选到能特异性结合噬菌体T4 DNA聚合酶的寡核苷酸配体。这项技术自1990年发现至今,已经应用于多种不同靶物质的筛选[5-6]。传统的SELEX技术的主要技术流程是:体外化学合成一个寡核苷酸文库,再与靶蛋白混合,形成蛋白-核酸复合物,洗去未与靶蛋白结合的核酸,分离与靶蛋白结合的核酸并扩增,重复多轮的筛选,最后构建克隆文库,挑取50–100个克隆测序,分析数据才能获得最适核酸配体。而二代测序技术的出现,因其测序数据通量远远高于一代测序[7],将其应用于SELEX筛选核酸配体的新方法对蛋白-核酸底物的识别提供了更加深入的认识[8-10]。
本研究主要是基于SELEX筛选方法,结合高通量测序技术,研究人的hnRNP A1的RRMs结构域特异识别的RNA motif,hnRNP A1是一种核糖核蛋白,重要的转录后调控因子[11]。其N端含有两个RRM结构域,RRM1和RRM2,也被称为UP1 (1-196) 结构域,通过UP1参与底物RNA的特异性识别[12],hnRNP A1参与调控不依赖于mRNA的5′帽子结构的翻译过程[13],microRNA的形成[14]以及可变剪切位点的选择[15]。已有研究利用SELEX筛选技术找出hnRNP A1的高亲和力结合位点UAGGGA/U,且该位点类似于脊椎动物5′和3′的剪切位点[16]。我们将以这一研究成果作为对照,评价SELEX与高通量测序技术相结合发现RNA结合蛋白特异性识别的RNA motif的可行性。
我们在体外合成了带有20个随机碱基的RNA待筛选文库,将UP1蛋白构建到带有可筛选标记的载体中,构建了pET28a-SUMO-SBP-UP1的重组质粒,蛋白的纯化过程中切除His-SUMO标签,纯化得到带有SBP-tag的蛋白,SBP-tag可以特异结合链亲和酶素,它含有38个氨基酸,且其亲和力强于strep-tag[17-18]。通过带有链亲和酶素的磁珠来筛选特异结合SBP-UP1的RNA文库,具有结合UP1能力的RNA被筛选出来并通过反转录成cDNA,cDNA经10–11轮的PCR扩增,在PCR的过程中将测序的adapter和barcode加在cDNA的两端,这样构建出可以用于高通量测序的文库。传统SELEX方法需要通过重复多轮的筛选,最后得到的只是亲和力最高的motif,而且在PCR的过程中,会引入一些偏好性,而通过高通量测序方法,可以省去重复筛选的过程,并且通过数据分析,我们可以得到更多关于转录后调控因子靶位点的信息,而不仅仅是一段motif[8-9]。相信这一方法的建立对于今后发现新的转录调控因子的靶作用位点会有重要的意义。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
大肠杆菌DH5α和BL21 (DE3) 由本实验室保存。pET28a-SUMO-SBP载体由本实验室构建。
1.1.2 主要试剂和耗材
限制性内切酶、T4 DNA连接酶和ssRNA ladder购自New England Biolabs。DNA凝胶回收试剂盒、PCR清洁试剂盒购自Axygen公司。T7 RNA polymerase由本实验室保存。5 mL HisTrap镍柱、5 mL Heparin肝素亲和层析柱、Superdex75 16/60凝胶过滤层析柱购自GE。蛋白分子量marker购自Thermo Scientific。DNA marker购自TaKaRa公司。链霉亲和素偶联的Dynabeads和反转录酶购自Invitrogen。超滤管购自Millipore。透析袋购自Spectrum。引物由上海生工生物工程技术服务有限公司合成。
1.2 重组质粒的构建和蛋白的表达纯化
1.2.1 质粒构建
以pET28a-SUMO-UP1 (1-196) 重组质粒为模板,设计引物UP1 F和UP1 R,引物两端分别加RⅠ和Ⅰ酶切位点,扩增UP1 (1-196) 基因,引物序列见表1。
表1 PCR引物序列
Table 1 Primer Sequences

*Restriction enzyme cutting site was underlined.
将UP1 (1-196) 亚克隆到pET28a-SUMO-SBP质粒,室温连接3 h。将pET28a-SUMO-SBP-UP1 (1-196) 重组质粒转化DH5α,挑选阳性克隆送上海睿迪生物公司测序。
1.2.2 蛋白的表达纯化
将测序正确的表达质粒转化BL21 (DE3),挑取克隆在5 mL LB培养基中,37 ℃培养6–8 h,再将菌液转移至1 L LB培养基中,于37 ℃、200 r/min振荡培养。当菌液600达到0.6–0.8,降温至18 ℃,加入终浓度0.2 mmol/L IPTG,18 ℃、200 r/min诱导16 h后收菌。
菌液重悬于buffer 1 (20 mmol/L Tris,pH 8.0,500 mmol/L NaCl,25 mmol/L 咪唑),经高压破菌,17 000 r/min离心1 h,留上清,上样至预先用buffer 1平衡的HisTrap柱,用buffer 2 (20 mmol/L Tris,pH 8.0,500 mmol/L NaCl, 500 mmol/L咪唑) 以梯度洗脱,收集目的峰,加入Ulp1酶切SUMO-tag,并在20 mmol/L Tris,pH 8.0,500 mmol/L NaCl的溶液中透析3 h,去除咪唑,再将样品上样至预先用buffer 1平衡的HisTrap柱,收集流穿液,Ulp1和6×His-SUMO-tag则结合在HisTrap柱上。将蛋白液的盐浓度稀释至100 mmol/L NaCl上样到预先用buffer A (20 mmol/L Tris,pH 8.0,100 mmol/L NaCl) 平衡的Heparin柱,用buffer B (20 mmol/L Tris,pH 8.0,1 mol/L NaCl) 梯度洗脱,收集目的蛋白峰。目的蛋白经超滤浓缩后上样至预平衡的Superdex 75 16/60,对纯化获得的蛋白峰样品取样进行SDS-PAGE检测。平衡Superdex 75 16/60采用的缓冲液是10 mmol/L Tris,pH 7.5, 100 mmol/L NaCl,1 mmol/L DTT。
1.3 T7转录RNA template
将含有20个随机序列的T7 template和T7 promoter (表2) 均稀释至100 μmol/L,取等体积混匀,95 ℃退火5 min,参照冷泉港的T7转录体系[19],37 ℃转录12 h,转录结束后加入DNase I消化DNA模板。利用16%的尿素PAGE胶纯化RNA转录产物。最终获得的随机RNA文库浓度用Nanodrop2000定量。
1.4 SBP-UP1-RRM特异识别RNA文库的构建
取250 μL的结合缓冲液 (25 mmol/L Tris,pH 7.5,150 mmol/L KCl,3 mmol/L MgCl2,0.01% Tween20,1 mmol/L DTT,30 μg/mL polyI/C) 将SBP-UP1稀释至250 nmol/L,冰上孵育30 min。然后加入终浓度为1 μmol/L的RNA随机文库和40 U RNase Inhibitor,冰上孵育1.5 h。同时,吸取100 μL Dynabeads用清洗缓冲液 (25 mmol/L Tris,pH 7.5,150 mmol/L KCl,0.5 mmol/L EDTA,0.01% Tween20) 清洗3次,每次200 μL,之后将Dynabeads平衡于200 μL的结合缓冲液中。将SBP-UP1和RNA文库的混合物,与Dynabeads在室温充分混合45 min,再加入1 mL的清洗缓冲液,洗去未结合SBP-UP1的RNA,然后将Dynabeads溶于洗脱缓冲液 (10 mmol/L Tris,pH 7.0,1 mmol/L EDTA,1% SDS),70 ℃,10 min。洗脱下来的SBP-UP1和RNA复合物,通过苯酚/氯仿/异戊醇 (25∶24∶1) 抽提和乙醇沉淀,去除蛋白,得到被筛选出来RNA的文库。特异识别UP1蛋白的RNA经体外转录和11轮的PCR扩增,最终获得可用于二代测序的文库。为了文库最终可用于二代测序,PCR引物含有可用于二代测序的adapter和barcode。PCR产物利用8% Native PAGE胶纯化。整体实验流程如图1所示,RT-PCR的引物序列见表2。

图1 SELEX实验流程图
表2 SELEX文库构建核酸序列
Table2 Oligonucleotides used for SELEX
*The barcode was underlined.
1.5 二代测序及数据分析
二代测序文库送至上海晶能生物公司,在Illumina HiSeq2500上测序,预计每个文库测序得到2 000万条序列,参照Streaming kmer Analysis (SKA) 的方法分析数据[8],该方法基于多轮读出序列数据,持续更新相对识别参数,将每一个序列概率分配识别到一个特定kmer motif上,k=5–8。最终将每一个kmer的权重排序,得到优先识别的RNA-motif。相对于传统的寻找motif的方法MEME,SKA更适合运用到SELEX的数据分析中,因为SKA估算的是含有每一个motif的序列数,而不是在一组序列中显著富集的motif。在本实验中,考虑到UP1含有2个RRM domain,因此计算k=8的优先识别的RNA-motif。
1.6 SBP-UP1的体外结合试验
1.6.1 RNA oligo的化学合成
以3’-亚磷酰胺及2’-叔丁基二甲基硅 (TBDMS) 保护的核糖核酸为单体,以CPG为固相合成载体,在ABI 394 DNA/RNA synthesizer化学合成经SELEX筛选得到的最优RNA motif,合成的RNA oligo经氨解和脱保护基,利用16%的尿素PAGE胶纯化,最后将纯化得到的RNA oligo用Nanodrop2000定量。
1.6.2 凝胶迁移实验 (EMSA)
在10 μL的RNA oligo和SBP-UP1反应体系中,底物RNA的终浓度固定为10 μmol/L,RNA和UP1以1∶0,1∶0.5,1∶1,1∶2及1∶4的比例混合,4 ℃反应30 min,反应buffer采用平衡superdex75 16/60的buffer。反应结束后加入1 μL 100%甘油,进行Native PAGE电泳。
2 结果与分析
2.1 SBP-UP1 (1-196) 的表达纯化
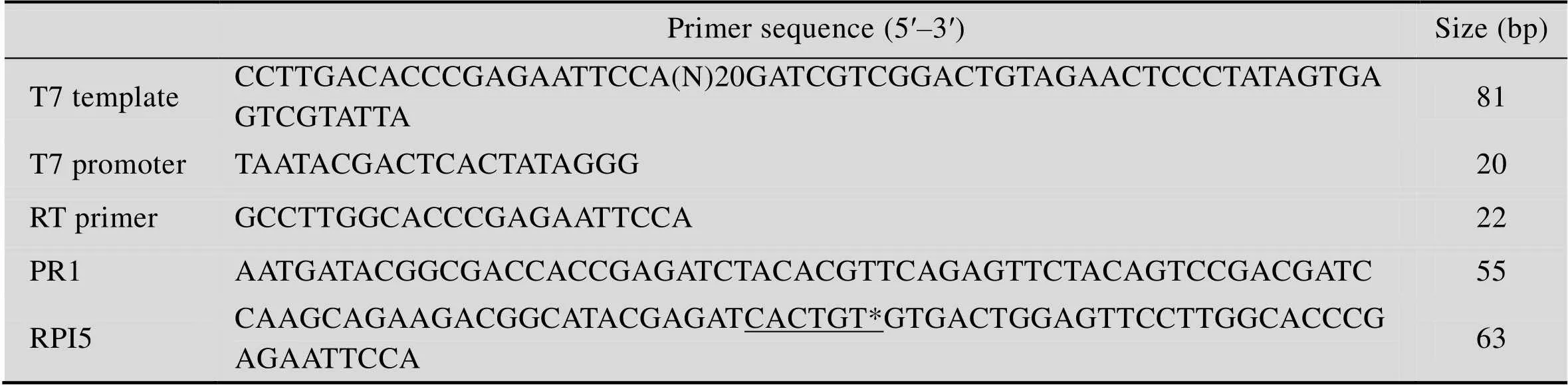
将表达SBP-UP1的重组质粒转化BL21 (DE3),经IPTG诱导表达,高压破菌,收集破菌上清液。蛋白经过HisTrap、Heparin和Superdex75纯化而获得,凝胶阻滞筛峰图显示SBP-UP1的出峰体积在59.9 mL,根据本实验室Superdex75 16/60标准曲线公式,计算得到分子量为27 kDa (图2A),SDS-PAGE (图2B) 显示重组SBP-UP1位于27 kDa与理论分子量 (26 180 Da) 相符,且纯度大于95%。
图2 SBP-UP1的Superdex75 16/60凝胶阻滞峰图 (A) 以及15% SDS-PAGE (B) 分析
2.2 T7 RNA转录文库的鉴定
含有20个随机碱基的RNA文库理论大小在62 bp,经16%尿素PAGE胶鉴定,T7体外转录所获得RNA文库与理论值相符。
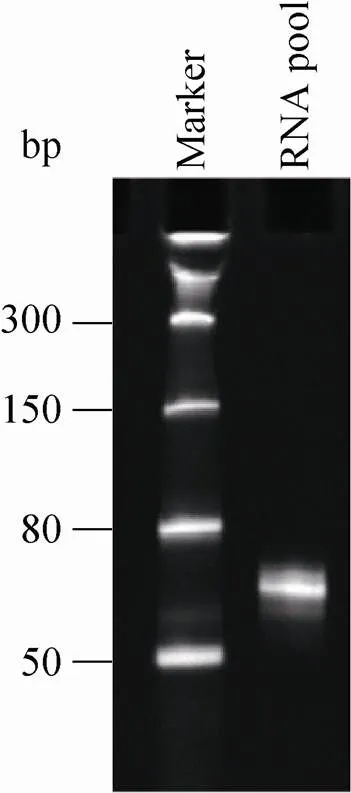
图3 RNA随机文库尿素PAGE胶分析
2.3 二代测序文库构建
经链亲和酶素磁珠筛选获得的特异识别SBP-UP1的RNA文库,通过RT-PCR扩增,所得PCR产物除了含有20个随机碱基序列,随机序列两侧还包含用于二代测序的adapter和barcode,产物大小为138 bp,Native PAGE胶 (图4) 显示PCR产物大小符合理论推测。经PAGE胶纯化所得DNA文库送上海晶能生物公司测序。
图4 SBP-UP1的SELEX文库
2.4 二代测序数据分析
测序数据经去掉两端adapter,得到1 950万条序列的数据量,再利用SKA算法分析含有 20个随机序列的测序数据,设置k=8,将每一个8 mer motif的权重排序,获得UP1结构域最优识别的RNA motif,5′-AGGCAGCA-3′。同时,通过分析SBP-UP1优先识别的8条RNA motif (表3),发现AGG和AG二种RNA序列出现的频率最高,在这8条RNA motif中都存在,但是这两种RNA序列两侧的序列是可变的、不确定的,因此可以推断 UP1特异识别AGG和AG两种保守RNA序列,而其余的序列则有一定的变化。
表3 SBP-UP1特异识别8 mer motif
Table 3 SBP-UP1 specifically binding of 8 mer motif
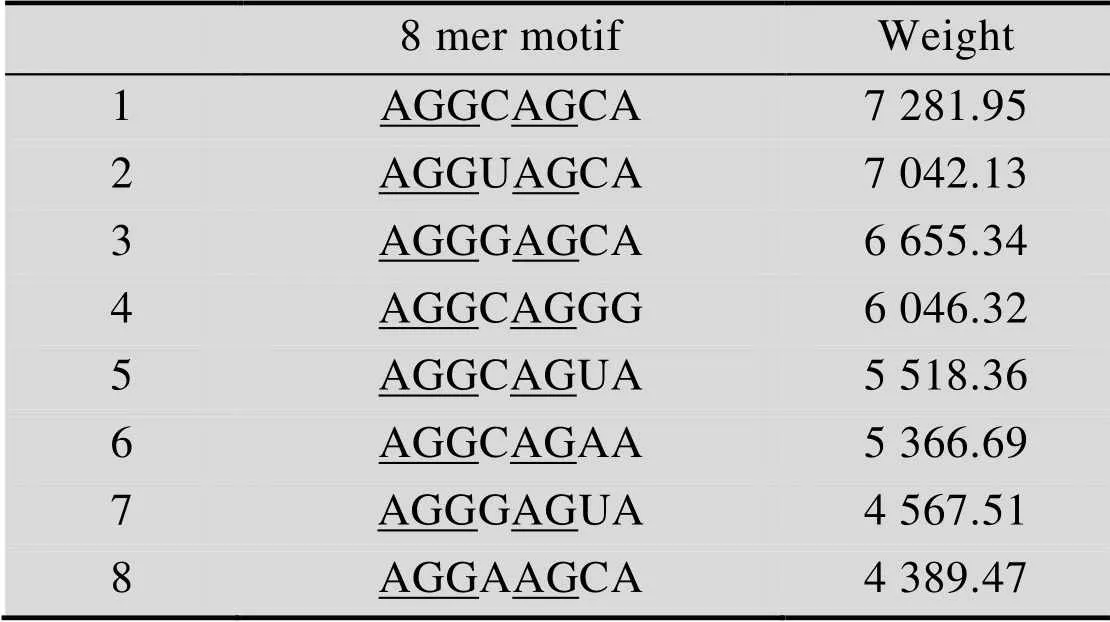
The consensus sequence was underlined.
2.5 EMSA验证SELEX结果
根据SELEX的结果,我们利用亚磷酰胺法化学合成了丰度最高的8 mer的RNA motif:5′–CCA–3′,EMSA的结果显示UP1可以在体外结合5′–CCA–3′,且在UP1∶RNA=2∶1时,RNA都被UP1结合,几乎看不到free-RNA,,因此通过EMSA实验验证了经SELEX结合二代测序技术筛选得到的UP1特异识别RNA motif可以与UP1在体外结合,也说明结合SELEX技术与二代测序技术筛选UP1特异识别RNA motif的方法是可行的。
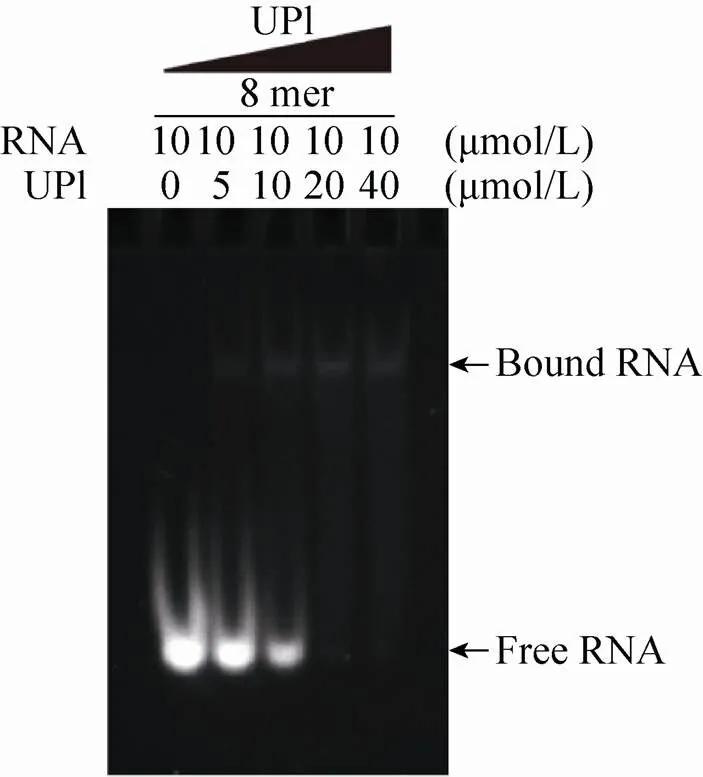
图5 SBP-UP1结合8 mer RNA的EMSA分析结果
3 讨论
本研究将SELEX与二代高通量测序技术相结合,发现hnRNP A1的UP1结构域最优识别的RNA motifCCA,并通过EMSA实验验证了该motif与UP1在体外的特异性结合。经过SKA算法的数据分析,我们不仅获得了UP1的最优识别的RNA motif,还获得一些结合力稍弱的RNA motif,但在这些序列中AGG和AG二种RNA序列出现的频率最高。由于UP1中含有两个连续的RRM结构域,所以我们推断UP1的两个RRM结构域有可能分别倾向于特异性识别AGG和AG,这与通过传统的SELEX方法获得的motif UGA/U中的核心序列相同[16]。hnRNP A1蛋白通过识别不同的RNA底物参与microRNA的形成、mRNA的可变剪切等生物学过程[14-15],其识别底物的序列也会有所差异,之前的研究从20 bp的随机核酸文库中,通过8轮筛选,获得了hnRNP A1最优识别的RNA motif[16],而通过本研究我们可以发现结合力较弱的RNA motif,但是这些RNA motif的生物学意义还有待进一步的验证。
本研究将UP1蛋白片段插入表达载体pET28a中,构建了重组表达质粒pET28a-SUMO- SBP-UP1。SUMO-tag有利于蛋白的表达纯化,在纯化的过程中可以经Ulp1酶切除,通过HisTrap柱、Heparin柱以及Superdex75的纯化获得纯度在98%以上的SBP-UP1。SBP-tag则是用于SELEX体外筛选底物RNA,SBP-tag与链亲和酶素有着较强的亲和力,d=2.5 nmol/L[20],通过带有链亲和酶素标记的磁珠进行筛选,由于磁珠易于洗涤及浓缩富集的优点,可以提高筛选效率[21]。传统的SELEX要经过大概10轮左右的筛选才能获得最适的RNA motif[22],既耗费时间也耗费人力,而我们的方法只需经过1轮筛选,便可得到蛋白特异识别的RNA文库,再经反转录和PCR,在cDNA两端加上用于二代测序的adapter和barcode,即可完成SELEX文库的构建,经二代测序得到的高通量数据,用SKA算法加以分析,我们可得到蛋白特异识别的RNA motif。SKA算法被认为是比MEME更适合于分析SELEX筛选的文库数据,它是通过累计计算每一条kmer RNA在测序数据中的权重,将这些kmer RNA的权重进行排序即可得到RNA motif的特异性的排序,SKA可以准确分辨假阳性的结果,提高数据分析的可靠性[8]。由于高通量测序数据的通量大,信息量丰富,我们不仅可以发现RNA结合蛋白的最适底物,还可以发现结合力次优的RNA motif,因为在生物体内RNA结合蛋白可能会通过结合不同的RNA发挥不同的功能[9-10],这对于我们更全面地了解RNA结合蛋白的调控机制提供了更多的信息。
SELEX技术与高通测序技术的结合,使它具有优于其他研究蛋白-RNA相互作用的研究方法的特点,可以通过测序数据的分析得到蛋白-RNA相互作用的更多信息。传统的SELEX技术,通过多次循环筛选,最后可以得到亲和力最高的靶序列,但是却忽视了较弱的结合靶序列和蛋白RNA结合的特异性,且多轮筛选、PCR扩增均会带入序列偏好性。CLIP,是一种体内筛选技术,它可以真实地反映蛋白与RNA相互作用在体内的情况,但是这种技术难度高,且重复的筛选步骤和交联的过程会引入序列的偏好性,后续的分析也很复杂,无法区别是单一蛋白的结合RNA motif还是蛋白复合物结合的RNA motif[23-24]。研究者通过比较SELEX技术和CLIP技术发现,SELEX技术所发现的motif,在CLIP中也有较高的信号。因此,新型的SELEX技术获得的结果能够一定程度反映体内的情况,也可以一定程度纠正CLIP偏好性,并互补于CLIP技术[8]。
SELEX与高通量测序技术的结合将为RNA结合蛋白识别底物的特异性研究提供快速有效的方法,除了基础生物学研究以外,该方法还可以应用于更多核糖核酸结合分子识别特异性的研究,如生物分析、疾病诊断、治疗、药物筛选等方面。
REFERENCES
[1] Ciesiolka J, Gorski J, Yarus M. Selection of an RNA domain that binds Zn2+. RNA, 1995, 1(5): 538–550.
[2] Nitsche A, Kurth A, Dunkhorst A, et al. One-step selection of vaccinia virus-binding DNA aptamers by MonoLEX. BMC Biotechnol, 2007, 7: 48.
[3] Paige JS, Wu KY, Jaffrey SR. RNA mimics of green fluorescent protein. Science, 2011, 333(6042): 642–646.
[4] Tuerk C, Gold L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science, 1990, 249(4968): 505–510.
[5] Jolma A, Kivioja T, Toivonen J, et al. Multiplexed massively parallel SELEX for characterization of human transcription factor binding specificities. Genome Res, 2010, 20(6): 861–873.
[6] Katz Y, Wang ET, Airoldi EM, et al. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nat Methods, 2010, 7(12): 1009–1015.
[7] Mardis ER. Next-generation DNA sequencing methods. Annu Rev Genomics Hum Genet, 2008, 9: 387–402.
[8] Lambert N, Robertson A, Jangi M, et al. RNA Bind-n-Seq: quantitative assessment of the sequence and structural binding specificity of RNA binding proteins. Mol Cell, 2014, 54(5): 887–900.
[9] Campbell ZT, Bhimsaria D, Valley CT, et al. Cooperativity in RNA-protein interactions: global analysis of RNA binding specificity. Cell Rep, 2012, 1(5): 570–581.
[10] Zykovich A, Korf I, Segal DJ. Bind-n-Seq: high-throughput analysis ofprotein-DNA interactions using massively parallel sequencing. Nucleic Acids Res, 2009, 37(22): e151.
[11] Dreyfuss G, Matunis MJ, Pinol-Roma S, et al. hnRNP proteins and the biogenesis of mRNA. Annu Rev Biochem, 1993, 62: 289–321.
[12] Shamoo Y, Krueger U, Rice LM, et al. Crystal structure of the two RNA binding domains of human hnRNP A1 at 1.75 A resolution. Nat Struct Biol, 1997, 4(3): 215–222.
[13] Lin JY, Shih SR, Pan MJ, et al. hnRNP A1 interacts with the 5' untranslated regions of enterovirus 71 and Sindbis virus RNA and is required for viral replication. J Virol, 2009, 83(12): 6106–6114.
[14] Guil S, Cáceres JF. The multifunctional RNA-binding protein hnRNP A1 is required for processing of miR-18a. Nat Struct Mol Biol, 2007, 14(7): 591–596.
[15] Tange TØ, Damgaard CK, Guth S, et al. The hnRNP A1 protein regulates HIV-1 tat splicinga novel intron silencer element. EMBO J, 2001, 20(20): 5748–5758.
[16] Burd CG, Dreyfuss G. RNA binding specificity of hnRNP A1: significance of hnRNP A1 high-affinity binding sites in pre-mRNA splicing. EMBO J, 1994, 13(5): 1197–1204.
[17] Barrette-Ng IH, Wu SC, Tjia WM, et al. The structure of the SBP-Tag-streptavidin complex reveals a novel helical scaffold bridging binding pockets on separate subunits. Acta Crystallogr D Biol Crystallogr, 2013, 69(Pt 5): 879–887.
[18] Keefe AD, Wilson DS, Seelig B, et al. One-step purification of recombinant proteins using a nanomolar-affinity streptavidin-binding peptide, the SBP-Tag. Protein Expr Purif, 2001, 23(3): 440–446.
[19] Nilsen TW, Rio DC, Ares M Jr. High-yield synthesis of RNA using T7 RNA polymerase and plasmid DNA or oligonucleotide templates. Cold Spring Harb Protoc, 2013, 2013(11), doi: 10.1101/pdb.prot078535.
[20] Wu SC, Wong SL. Structure-guided design of an engineered streptavidin with reusability to purify streptavidin-binding peptide tagged proteins or biotinylated proteins. PLoS ONE, 2013, 8(7): e69530.
[21] Oh SS, Ahmad KM, Cho M, et al. Improving aptamer selection efficiency through volume dilution, magnetic concentration, and continuous washing in microfluidic channels. Anal Chem, 2011, 83(17): 6883–6889.
[22] Nieuwlandt D, Wecker M, Gold L.selection of RNA ligands to substance P. Biochemistry, 1995, 34(16): 5651–5659.
[23] Sugimoto Y, König J, Hussain S, et al. Analysis of CLIP and iCLIP methods for nucleotide-resolution studies of protein-RNA interactions. Genome Biol, 2012, 13(8): R67.
[24] Sugnet CW, Srinivasan K, Clark TA, et al. Unusual intron conservation near tissue-regulated exons found by splicing microarrays. PLoS Comput Biol, 2006, 2(1): e4.
Screening specific recognition motif of RNA-binding proteins by SELEX in combination with next-generation sequencing technique
Lu Zhang, Jinhao Xu, and Jinbiao Ma
State Key Laboratory of Genetics, Department of Biochemistry, School of Life Sciences, Fudan University, Shanghai 200438, China
RNA-binding protein exerts important biological function by specifically recognizing RNA motif. SELEX (Systematic evolution of ligands by exponential enrichment), anselection method, can obtain consensus motif with high-affinity and specificity for many target molecules from DNA or RNA libraries. Here, we combined SELEX with next-generation sequencing to study the protein-RNA interaction. A pool of RNAs with 20 bp random sequences were transcribed by T7 promoter, and target protein was inserted into plasmid containing SBP-tag, which can be captured by streptavidin beads. Through only one cycle, the specific RNA motif can be obtained, which dramatically improved the selection efficiency. Using this method, we found that human hnRNP A1 RRMs domain (UP1 domain) bound RNA motifs containing AGG and AG sequences. The EMSA experiment indicated that hnRNP A1 RRMs could bind the obtained RNA motif. Taken together, this method provides a rapid and effective method to study the RNA binding specificity of proteins.
SELEX, next-generation sequencing, hnRNP A1, RNA motif
October 12, 2015; Accepted: December 11, 2015
生物技术与方法
张璐, 徐晋暤, 麻锦彪. 一种结合SELEX与二代测序技术筛选RNA结合蛋白特异识别序列新方法的建立. 生物工程学报, 2016, 32(7): 966–974.
Zhang L, Xu JH, Ma JB. Screening specific recognition motif of RNA-binding proteins by SELEX in combination with next-generation sequencing technique. Chin J Biotech, 2016, 32(7): 966–974.
Corresponding author: Jinbiao Ma. Tel: +86-21-51630542; Fax: +86-21-51630541; E-mail: majb@fudan.edu.cn
(本文责编 陈宏宇)

