一种浸矿混合菌种的筛选、鉴定及浸矿的研究
聂毅磊 陈宏 罗立津 贾纬 陈星伟
(福建省微生物研究所 福建省新药(微生物)筛选重点实验室,福州 350007)
一种浸矿混合菌种的筛选、鉴定及浸矿的研究
聂毅磊 陈宏 罗立津 贾纬 陈星伟
(福建省微生物研究所 福建省新药(微生物)筛选重点实验室,福州 350007)
旨在从某低品位硫化铜矿酸性矿坑水中分离获得浸矿效果优良的细菌,同时对细菌的浸矿行为进行初步的研究。采集福建某低品位硫化铜矿酸性矿坑水样品,9K培养基富集获得混合菌种FIM-201304。利用9K固体平板,从混合菌FIM-201304中分离获得优势浸矿菌D-1。应用高通量Illumina Miseq测序技术分析混合菌FIM-201304 的群落结构。通过表型特征和16S rRNA基因序列分析鉴定菌株D-1。采用摇瓶培养对比研究混合菌和单菌对低品位硫化铜矿和低品位硫化镍矿的浸出效果。结果显示,高通量测序技术分析结果表明,混合菌FIM-201304 的优势菌是嗜酸氧化亚铁硫杆菌(Acidithiobacillus ferrooxidans,A. f errooxidans)(丰度占比85.02%)、铜绿色假单胞菌(Pseudomonas sp.)(丰度占比10.07%)、嗜酸异养菌(Acidiphilium acidophilum,A.acidophilum)(丰度占比1.30%)和鞘氨醇单胞菌(Sphingomonas sp.)(丰度占比1.21%)。表型特征和16S rRNA 基因序列分析确定菌株D-1属于氧化亚铁硫杆菌。摇瓶实验结果表明,浸出反应20 d后,接种混合菌的浸出体系中铜、镍浸出率分别为65%和56%,相同条件下,接种单菌的浸出体系中铜、镍的浸出率为41%和36%,接种混合菌的浸出体系比接种单菌的浸出体系中铜、镍浸出率分别提高了24%和20%。混合菌对矿石的浸出效果显著优于单菌。异养菌促进了自养菌对矿石中金属元素的浸出。
嗜酸氧化亚铁硫杆菌;生物浸出;混合菌;高通量测序;16S rRNA 基因
生物湿法冶金是利用微生物对矿石进行直接、间接或者两者共同作用浸出矿石中有用金属的一种工艺。与传统冶金技术相比,生物冶金具有成本低、工艺简单、对环境友好等优点,尤其对于采用传统的冶金方法难以处理的低品位矿石、原矿和尾矿等,生物冶金更显其独特之处[1-5]。生物冶金的核心技术是筛选获得高效、适应能力强、选择性好的微生物菌种。
嗜酸氧化亚铁硫杆菌(Acidithiobacillus ferrooxidans,A. ferrooxidans)被认为是酸性环境中浸矿的主导菌种。但该类微生物生长缓慢,这也是导致生物浸出周期长和浸出效率低的主要原因[6]。研究表明,浸矿微生物的一些代谢产物对其自身的生长具有抑制作用[7,8]。若浸矿体系中含有以有机底物作为能源物质的异养菌,例如嗜酸异养菌(Acidiphilium acidophilum,A. acidophilum),可以在一定程度上解除代谢产物对自养菌的抑制作用,有利于主要自养浸矿菌的生长,充分发挥其浸矿作用,提高浸出率[9,10]。像这种两类或多类微生物在浸矿过程中相互作用,更好地进行单类种群不能完成或不能高效完成的物质转化,增强各自的生长代谢活动,从而提高金属浸出速率和浸出率的微生物作用机制称为协同作用[11]。微生物协同作用在生物冶金中发挥着重要的作用[12,13]。
本研究从福建某低品位硫化铜矿酸性矿坑水样品中富集到一种浸矿混合菌种FIM-201301和一株嗜酸氧化亚铁硫杆菌D-1,分析混合菌种的优势菌,旨在为开发利用以氧化亚铁硫杆菌为主的浸矿混合菌种的应用及丰富浸矿微生物协同作用奠定理论基础。
1 材料与方法
1.1材料
1.1.1培养基 9K液体培养基:A液:(NH4)2SO43.0 g,K2HPO40.5 g,KCl 0.1 g,MgSO4·7H2O 0.5 g,Ca(NO3)20.01 g,800 mL去离子水溶解,H2SO4调节pH至2.0,121℃高压灭菌 20 min;B液:FeSO4·7H2O 44.78 g,去离子水200 mL溶解,H2SO4调节pH至2.0,孔径为0.22 μm的滤膜过滤除菌。将灭菌后的A液与B液混匀后分装待用。
9K固体培养基:A液:(NH4)2SO41.5 g,KCl 0.1 g,K2HPO40.5 g,MgSO4·7H2O 0.5 g,Ca(NO3)20.01 g,KSCN 15.4 g,450 mL去离子水溶解,H2SO4调节pH至3.0,121℃高压灭菌20 min;B液:FeSO4·7H2O 22.5 g,去离子水50 mL溶解,H2SO4调节pH至2.0,孔径为0.22 μm的滤膜过滤除菌;C液:琼脂糖 10 g,500 mL去离子水溶解,121℃高压灭菌20 min。
待A液和C液冷却到60-70℃时将A液、B液和C液混合均匀,迅速倒入培养皿中,每个培养皿中倒入10-15 mL,冷却凝固后即成固体平板。
1.1.2矿石样 实验所用硫化铜矿石经过缩分、筛分、破碎与磨碎等流程,矿样的粒度<0.075 mm,主要矿物成分为黄铜矿、斑铜矿、黄铁矿和方铅矿,铜品位为0.47%。试样化学组成成分见表1。

表1 硫化铜矿石化学组成分析结果
所用低品位硫化镍矿为矿样,矿样的粒度<0.075 mm,主要矿物成份为镍磁黄铁矿、镍黄铁矿和黄铜矿。镍品位为0.73%。试样化学组成成分见表2。

表2 硫化镍矿石化学组成分析结果
1.1.3主要试剂和仪器 所用化学试剂均为分析纯。ZHWY-3112B摇床(上海智城分析仪器制造有限公司)。Miseq/E250高通量测序平台(美国Illumina公司)。PCR扩增仪(S1000,Bio-Rad公司)。电泳仪(6003EN,上海申能博彩生物科技有限公司)。WFX-120原子吸收分光光度计(北京瑞利分析仪器有限公司)。HH-4恒温水浴锅(上海江星仪器有限公司)。
1.2方法
1.2.1样品采集 细菌样品采自福建某铜矿矿山酸性矿坑水,取样时矿坑水温度27℃,pH≈2.6。将矿坑水移至灭菌容器,容器上方留有一定的空气,加入少量灭菌的硫酸亚铁。置于-20℃冰箱保存。
1.2.2菌种分离 将10 mL采集的原始细菌样品加入到盛有200 mL 9K液体培养基的三角瓶中,30℃,180 r/min 恒温振荡培养,直至培养液的颜色呈棕红色,获得第一代富集培养物。将第一代富集培养物转代培养,方法同上,振荡培养,但细菌接种量要逐代减少。转代培养10次左右,获得最终的富集培养物,此时,镜检观察有大量运动细菌存在。取0.2 mL富集培养物划线培养于9K固体培养基上,30℃培养,挑取优势单菌落接入9K液体培养基中恒温振荡培养,至培养液的颜色呈棕红色,再划线于平板固体培养,重复上述操作3次,分离到一株具有Fe2+氧化活性的嗜酸性细菌。将纯化菌株保存于9K固体培养基中,待用。
1.2.3菌群总DNA的提取、扩增 取培养72 h的富集培养物,过滤去除黄钾铁矾,8 000 r/min离心15 min,倾去上清液,收集菌体,用TENP缓冲液(50 mmol/L Tris,20 mmol/L EDTA,100 mmol/L NaCl,0.01 g/mL PVP,pH10)洗涤至基本无色,再用PBS缓冲液(8 g NaCl,0.2 g KCl,1.44 g Na2HPO4,0.24 g KH2PO4,溶于1 L水中,pH7.4)洗涤菌体2次,去除腐殖质;采用CTAB/NaCl法[14]提取菌群基因组DNA。步骤如下:腐殖质去除后的富集培养物中加入500 mL TE溶液(pH8.0),悬浮,加入60 μL 10%十二烷基硫酸钠(SDS)和6 μL 10 mg/mL蛋白酶K,混匀,60℃水浴1 h,加2.5 mL 5 mol/L NaCl 和2 mL CTAB/NaCl溶液,65℃水浴10 min,以等体积的苯酚/氯仿/异戊醇、氯仿/异戊醇、各抽提1次,异丙醇沉淀DNA,DNA溶于50 μL TE中,-20℃保存备用。利用1%琼脂糖凝胶电泳检测抽提的基因组DNA完整性。采用16S rRNA基因的通用引物27F(5'-AGAGTTTGATCCTGGCTCAG-3')和1492R (5'-GGTTACCTTGTTACGACTT-3')对提取的DNA进行PCR扩增。将扩增后的DNA产物送至上海锐翌生物科技有限公司采用高通量Illumina MiSeq测序技术分析微生物群落结构。
1.2.4菌群高通量Illumina MiSeq测序技术分析
1.2.4.1测序与数据处理过滤 对16S rRNA基因V3-V4区序列进行测序,对rawreads按照以下标准进行过滤拼接,得到Long reads:(1)将ambiguous bases>6的reads过滤掉;(2)将平均质量Q<25的reads过滤掉;(3)对所有reads切除正反向引物序列;(4)将进行以上三步处理后的序列长度小于200 bp的reads过滤掉。
1.2.4.2OTU聚类 为便于下游物种多样性分析,将Tags聚类为OTU(Operational Taxonomic Units)。首先把拼接的Tags中的singletons(对应reads只有一条的序列)过滤掉,利用usearch在0.97相似度下进行聚类,对聚类后的序列进行嵌合体过滤后,得到用于物种分类的OTU。
1.2.4.3物种注释 从各个OTU中挑选出一条序列,作为该OTU的代表序列。将该代表序列,用RDP方法,与已知物种的16S数据库(GreenGenes,http://greengenes.lbl.gov)进行比对,从而对每个OTU进行物种归类。归类后,根据每个OTU中序列的条数,得到OTU丰度表。
1.2.4.4Profiling 在门、纲、目、科、属水平,将每个注释上的物种或OTU在不同样品中的序列数整理在一张表格,形成profiling柱状图及统计表。
1.2.4.5单个样品复杂度分析 利用QIIME软件计算样品的Alpha多样性值,并利用已测得的16S rRNA序列中已知的各种OTU的相对比例,来计算抽取n个(n小于测得reads序列总数)reads时各Alpha指数的期望值,然后根据一组n值(一般为一组小于总序列数的等差数列)与其相对应的Alpha指数的期望值做出稀释曲线。
1.2.5纯菌株DNA提取、扩增和分析 取培养至对数生长期的菌液,离心收集菌体,同上述方法提取菌株基因组DNA和对提取的DNA进行PCR 扩增。将扩增后的DNA产物送至上海生物工程有限公司进行测序。用BLAST软件与NCBI的GenBank数据库收录的的16S rRNA序列进行比对分析。
1.2.6浸矿实验 将分离获得的浸矿混合菌种和单菌分别接入9K培养基中,30℃,180 r/min培养至对数生长期。菌液在8 000 r/min下离心10 min,弃上清液得到沉淀,用pH2.0预先灭过菌的硫酸溶液洗涤沉淀,离心弃除上清液,如此反复操作3次,以达到去除菌液中Fe3+的目的,得到无铁细胞悬液,在生物显微镜下进行计数,调整细胞浓度为1×109cell/mL。取9K培养基(不含Fe2+)180 mL置于500 mL锥形瓶中,称取20 g矿样放入锥形瓶中,硫酸调节矿浆pH值,控制初始pH值在1.8-2.0。加入浸矿菌(单菌和混合菌)(5%接种量),30℃,180 r/min震荡培养。每个条件均包括3组平行实验,并设无菌空白对照。浸出过程中的酸耗通过10 mol/L的硫酸补充,蒸发损失的水分通过添加蒸馏水补充。浸出20 d后用原子吸收光谱测定浸出液中的目标金属元素浓度。
2 结果
2.1富集、分离培养
利用9K培养基从酸性矿坑水中获得最终的富集培养物,将该富集培养物命名为FIM-201304。从固体平板分离获得一株具有Fe2 +氧化活性的菌株,命名为D-1。光学显微镜观察(图1)表明,分离纯化的菌株D-1具有嗜酸氧化亚铁硫杆菌的特征,个体形状为杆状,两端钝圆,以单个、双个或几个成短链状存在。
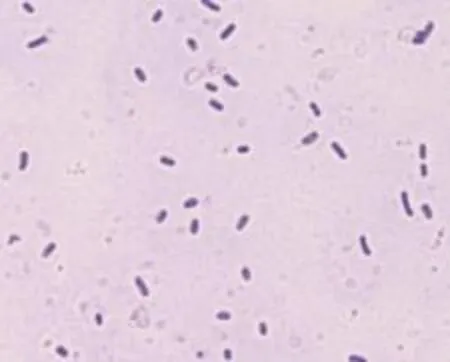
图1 菌株D-1结晶紫染色形态(100×)
2.2菌群的群落结构分析
2.2.1数据统计 通过Illumina平台(Miseq)进行Paired-end测序,下机数据经过数据过滤去除低质量reads,数据产出详细统计结果见表3。

表3 样品测序数据统计
2.2.2OTU 聚类 将序列完全一样的HQ reads归为一种tag,并统计每条tag对应的丰度(即reads数目),然后将tags根据其丰度大小进行排序,考虑到singletons可能由于测序错误造成,故将其中的singletons(对应reads只有一条的序列)这部分序列去除,不进行后期OTU(Operational Taxonomic Units)聚类。利用usearch在0.97相似度下进行聚类,对聚类后的序列进行嵌合体过滤后,得到用于物种分类的OTU,最后将所有HQ reads比对到OTU序列上,将能比对上OTU的reads提取出来,得到最终的Clean reads。统计样品每个OTU中的丰度信息,OTU的丰度初步说明了样品的物种丰富程度。样品OTU统计结果见表4。

表4 样品OTU统计
2.2.3OTU 抽平处理 由于不同样本对应的reads数量差距较大,为了保证后期分析结果合理,对每个样本的数据进行随机抽平处理,每个样品随机抽取71 545条reads。抽平后的样品统计表明OTU数量为215个。
2.2.4物种归类分析 从各个OTU中挑选出丰度最高的一条序列,作为该OTU的代表序列。使用RDP方法,将该代表序列与已知物种的16S数据库进行比对,从而对每个OTU进行物种归类,结果表明共归属51个科,141个属。
2.2.5样品复杂度分析 Alpha多样性反映的是单个样品内部的物种多样性,包括observed species指数、chao指数、shannon指数、simpson指数以及PD_ whole_tree指数等。反映样品中群落的丰富度(species richness)的observed species指数和chao指数对应的稀释曲线(图2)表明,曲线趋于平缓,可以认为测序深度已经基本覆盖到样品中所有的物种。
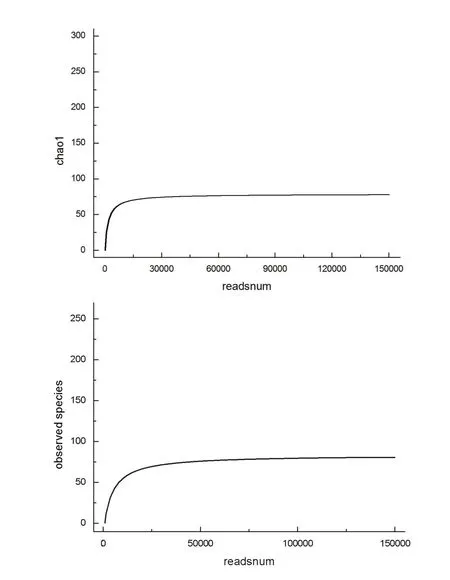
图2 Alpha多样性指数稀释曲线图
高通量Illumina Miseq测序技术分析结果(图3)表明,富集体系FIM-201301中优势种群主要有嗜酸氧化亚铁硫杆菌(A. ferrooxidans)(丰度占比85.02%),铜绿色假单胞菌(Pseudomonas sp.)(丰度占比10.07%),嗜酸异养菌(A. acidophilum)(丰度占比1.30%)和鞘氨醇单胞菌(Sphingomonas sp.)(丰度占比1.21%)。菌株D-1与A. ferrooxidans sp. KCT17,A. ferrooxidans sp. ST2和A. ferrooxidans sp. PS107 的16S rRNA基因序列的相似性均为100%,因而确定D1属于氧化亚铁硫杆菌。
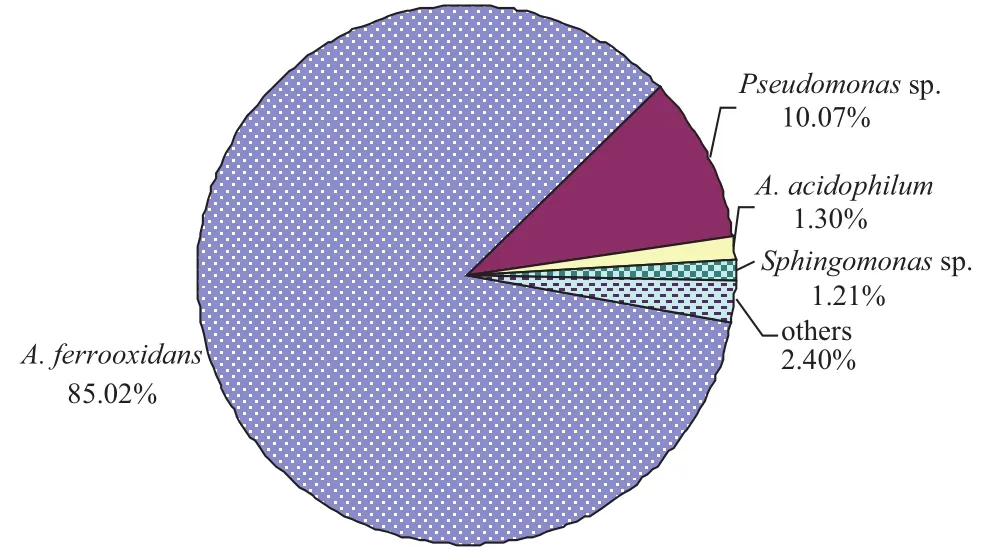
图3 富集体系FIM-201301种群结构丰度占比图
2.2.6矿石浸出效果 混合菌FIM-201301和单菌D1对低品位硫化铜矿和低品位硫化镍矿的摇瓶浸出实验表明,浸出反应20 d后,接种混合菌的浸出体系中铜、镍浸出率分别为65%和56%,在相同条件下,接种单菌的浸出体系中铜、镍的浸出率分别为41%和36%。接种混合菌的浸出体系比接种单菌的浸出体系中铜、镍浸出率分别提高了24%和20%。混合菌对矿石的浸出效果显著优于单菌,表明异养菌促进了自养菌对矿石中金属元素的浸出。自养菌和异养菌组成的混合菌通过协同作用,能更好地完成物质转化,提高各自的生长代谢活动,从而提高金属浸出率。
3 讨论
与传统微生物多样性分析手段相比,高通量测序技术具有通量高、读长长且准确率高等特点,能在测定浸矿微生物群落结构多样性上检测到丰度较低的微生物[15],因而本实验采用高通量Illumina Miseq测序技术对筛选获得的混合菌种FIM-201304的群落结构进行分析。后续我们将探讨浸矿过程中细菌的群落结构动态变化,为改善生物浸矿工艺提供一定理论依据。
浸矿微生物种类繁多,按不同营养类型可划分为自养菌和异养菌。目前,在微生物浸矿中研究比较多,在生产中得到应用的主要是氧化亚铁硫杆菌、氧化硫硫杆菌和氧化亚铁钩端螺旋菌等化能自养微生物[16-20]。该类微生物在生长繁殖过程中不需要任何有机营养,完全靠各种矿物盐生存。但该类微生物生长较为缓慢。浸矿体系中有机物的存在会一定程度上抑制自养菌的生长,尤其是生物浸出后期,微生物代谢产物积累和自身衰亡都会产生较多的有机物,从而抑制自养菌的氧化活性,最终降低金属浸出效率。研究表明自然环境浸矿体系中自养菌和异养菌往往共同存在[21-23]。异养菌,如A. acidophilum,不仅能够利用自养菌代谢产生的有机物作为能源,一定程度上消除有机物对自养菌的抑制作用,而且在生长过程中会产生有机酸等代谢物,此类产物的生成对矿石的溶解有重要的作用。自养菌和异养菌的混合菌对有价金属的浸出率往往高于自养菌单一菌种。Liu等[12]研究了异养菌A. acidophilum 在转录水平上对A. ferrooxidans 生长代谢的影响。结果显示,混合培养时A. ferrooxidans 的指数期延长2 d,细胞数量是纯培养的5倍多,培养液中亚铁离子的浓度增大,与铁离子氧化有关的基因表达量上调。混合培养时,A. acidophilum激活了A. ferrooxidans 中的铁离子氧化相关基因及固定CO2的相关基因,对A. ferrooxidans的生长代谢起到了极大的促进作用。Yang等[24]考察了A. ferrooxidans单菌和A. ferrooxidans 与A. acidophilum 的混合菌对低品位铜矿矿石的柱浸效果,结果表明,A. ferrooxidans单菌浸矿体系中铜的浸出率为14.8%,而混合菌浸矿体系中铜的浸出率为20.11%。A. acidophilum 的加入促进了A. ferrooxidans对铜的浸出率。王秀美[25]进行了A. ferrooxidans单独菌和A. ferrooxidans与A. acidophilum的混合菌对铁闪锌矿的浸出行为研究,结果发现混合菌的浸出效果要好于单独菌,异养菌的加入提高了自养菌的浸矿效率。
目前还未见采用A. ferrooxidans、Pseudomonas sp.、A. acidophilum和 Sphingomonas sp.的混合菌种浸矿的研究报道。该混合菌种是从自然环境中筛选而来,各菌种之间是一个相对稳定和相容的整体,它们通过协同效应在浸矿中发挥作用。下一步我们将对其进行驯化以及浸矿工艺优化的研究,以期更好地提高金属元素的浸出速率和浸出率。
本实验为开发利用以氧化亚铁硫杆菌为主的浸矿混合菌种的应用打下了基础,同时丰富了浸矿微生物协同作用理论。
4 结论
本实验从福建某低品位硫化铜矿酸性矿坑水中分离获得一种浸矿菌群FIM-201304和一株嗜酸氧化亚铁硫杆菌D-1。菌群FIM-201304的优势菌是自养型嗜酸氧化亚铁硫菌和异养型铜绿色假单胞菌、嗜酸异养菌及鞘氨醇单胞菌。采用摇瓶方法对比研究混合菌和嗜酸氧化亚铁硫杆菌单菌对低品位硫化铜矿和低品位硫化镍矿的浸出效果。结果表明,接种混合菌的浸出体系比接种单菌的浸出体系中铜、镍浸出率分别提高了24%和20%。异养菌促进了自养菌对矿石中金属元素的浸出。
[1]喻连香, 周丹, 石丽娟, 等. 国内某低品位难选铜矿的生物浸出研究[J]. 现代生物医学进展, 2014, 14(10):1861-1864.
[2]Wang J, Zhu S, Zhang YS, et al. Bioleaching of low-grade copper sulfide ores by Acidithiobacillus ferrooxidans and Acidithiobacillus thiooxidans[J]. Journal of Central South University, 2014, 21(2):728-734.
[3]Davis-Belmar CS, Cautivo D, Demergasso C, et al. Bioleaching of copper secondary sulfide ore in the presence of chloride by means of inoculation with chloride-tolerant microbial culture[J]. Hydrometallurgy, 2014, 21(150):308-312.
[4]Shabani MA, Irannajad M, Azadmehr AR, et al. Bioleaching of copper oxide ore by Pseudomonas aeruginosa[J]. International Journal of Minerals, Metallurgy and Materials, 2013, 20(12):1130-1133.
[5]Ramanathan T, Ting YP. Selective copper bioleaching by pure and mixed cultures of alkaliphilic bacteria isolated from a fly ash landfill site[J]. Water Air Pollut, 2015, 226(11):373-387.
[6] 朱永光, 杨柳, 张火云, 等. 微生物菌剂的研究与开发现状[J].四川环境, 2004, 23(3):5-12.
[7]Johnson DB. Importance of microbial ecology in the development of new mineral technologies[J]. Hydrometallurgy, 2001, 59(2):147-157.
[8] Johnson DB. Biodiversity and ecology of acidophilic microorganisms [J]. FEMS Microbiol Ecol, 1998, 27(4):307-317.
[9]钱林. Acidithiobacilius ferrooxidans和Acidiphilium spp. 细菌的分离鉴定及其协同浸出黄铜矿能力研究[D]. 长沙:中南大学,2008.
[10]Romo E, Weinacker DF, Zepeda AB, et al. Bacterial consortium for copper extraction from sulphide oreconsisting mainly of chalcopyrite [J]. Brazilian Journal of Microbiology, 2013, 44(2):523-528.
[11]Jain R, Ashish P, Sreekrishnan TR, Dastidr MG. Autoheated thermophilic aerobic sludge digestion and metal bioleaching in a two-stage reactor system[J]. Journal of Environmental Sciences,2010, 22(2):230-236.
[12]Liu HW, Yin HQ, Dai YX, et al. The co-culture of Acidithiobacillus ferrooxidans and Acidiphilium acidophilum enhances the growth,iron oxidation, and CO2 fixation[J]. Archives of Microbiology,2011, 193(12):857-866.
[13]余润兰, 石丽娟, 周丹, 等. 生物浸出过程中微生物协同作用机制的研究进展[J]. 中国有色金属学报, 2013, 23(10):3006-3014.
[14]邵碧英, 陈彬, 汤敏英, 等. 沙门氏菌DNA提取及PCR反应条件的优化[J]. 食品科学, 2007, 28(7):331-334.
[15]郝晓东, 曾伟民, 彭堂见, 等. 高通量测序技术分析不同温度下赞比亚低品位铜矿生物浸出过程中的微生物多样性[J].中国有色金属学报, 2015, 25(9):2558-2564.
[16]Xin BP, Li T, Li X, et al. Reductive dissolution of manganese from manganese dioxide ore by autotrophic mixed culture under aerobic conditions[J]. Journal of Cleaner Production, 2015, 92:54-64.
[17]Nkulu G, Gaydardzhiev S, Mwema E, et al. SEM and EDS observations of carrollite bioleaching with a mixed culture of acidophilic bacteria[J]. Minerals Engineering, 2015, 75:70-76.
[18]Ahmadi A, Mousavi SJ. The influence of physicochemical parameters on the bioleaching of zinc sulfide concentrates using a mixed culture of moderately thermophilic microorganisms[J]. International Journal of Mineral Processing, 2015, 135(10):32-39.
[19]Zhao HB, WangJ, Gan XW, et al. Bioleaching of chalcopyrite and bornite by moderately thermophilic bacteria:an emphasis on their interactions[J]. International Journal of Minerals, Metallurgy and Materials, 2015, 22(8):777-787.
[20]Wang J, Zhu S, Zhang YS, et al. Bioleaching of low-grade copper sulfide ores by Acidithiobacillus ferrooxidans and Acidithiobacillus thiooxidans [J]. Journal of Central South University, 2014, 21(2):728-734.
[21]聂立, 税刘杨, 王建英. 包头泉山金矿浸矿酸性微生物群落优势度变化的比较研究[J]. 微生物学通报, 2014, 41(1):43-49.
[22]Zeng WM, Qiu GZ, Zhou HB, et al. Community structure and dynamics of the free and attached microorganisms during moderately thermophilic bioleaching of chalcopyrite concentrate[J]. Bioresource Technology, 2010, 101(18):7068-7075.
[23]杨国斌. PCR-测序法分析铀生物堆浸过程中微生物群落结构变化特征[J]. 广东化工, 2014(9):21-23.
[24]Yang Y, Diao MX, Liu K, et al. Column bioleaching of low-grade copper ore by Acidithiobacillus ferrooxidans in pure and mixed cultures with a heterotrophic Acidophile acidiphilium sp. [J]. Hydrometallurgy, 2013, 131(1):93-98.
[25]王秀美. 自养与异养浸矿细菌的分离鉴定、浸矿及其生长代谢热的基础研究[D]. 长沙:中南大学, 2008.
(责任编辑 马鑫)
Screening and Identification of Mixed Culture,and Its Bioleaching Capacity
NIE Yi-lei CHEN Hong LUO Li-jin JIA Wei CHEN Xing-wei
(Fujian Institute of Microbiology,Fujian Provincial Key Laboratory of Screening for Novel Microbial Products,Fuzhou 350007)
It is aimed to screen the highly efficient leaching bacteria from acid mining water of low-grade copper sulfide mine,and also preliminarily investigate the bioleaching capacities of the bacteria. Firstly,a mixed culture FIM-201304 was obtained by 9K enrichment from acid mining water of a low-grade copper sulfide mine in Fujian province. Then,one dominated bacterium D-1 was isolated from the FIM-201304 using 9K solid culture medium. Sequencing technology by high-throughput Illumina Miseq was used to analyze the community structure of FIM-201304. D-1 was identified by phenotypic characteristics and 16S rRNA gene sequencing analysis. Flask experiment was utilized to compare the effects of leaching low-grade copper- and nickel-sulfide between the mixed culture FIM-201304 and a single bacterium D-1. Results from high-throughput sequencing analysis showed that the mixed culture FIM-201304 contained the dominated bacteria of Acidithiobacillus ferrooxidans(Abundance accounted for 85.02%),Pseudomonas sp.(Abundance accounted for 10.07%),Acidiphilium acidophilum(Abundance accounted for 1.30%)and Sphingomonas sp.(Abundance accounted for 1.21%). According to phenotypic characteristics and 16S rRNA gene sequence analysis,strain D-1 was identified as Acidithiobacillus ferrooxidans.The results after 20 days of bioleaching with flasks showed that leaching ratio of copper and nickel by FIM-201304 was 65% and 56% respectively,while the leaching ratio by D-1 was 41% and 36% under the same conditions,i.e.,the bioleaching ratio of copper and nickel by mixed culture increased 24% and 20% compared to that by single bacterium,respectively. Conclusively,the mixed culture resulted in the better leaching effect as heterotrophic bacteria promoted the metalleaching capacity of autotrophic bacteria from the ore.
Acidithiobacillus ferrooxidans;bioleaching;mixed culture;high-throughput sequencing;16S rRNA gene
10.13560/j.cnki.biotech.bull.1985.2016.08.026
2015-11-17
福建省科技计划重点项目(2013Y0017)
聂毅磊,男,硕士,副研究员,研究方向:生物工程;E-mail:nie2050@sina.com
陈宏,硕士,高级工程师,研究方向:环境微生物;E-mail:peaceful88@163.com

