大蒜半胱氨酸合成酶的mRNA表达及生物信息学分析
郭天璐张欢欢杜建中郝曜山王亦学孙毅
(1. 山西大学生物工程学院,太原 030006;2. 山西省农业科学院生物技术研究中心,太原 030031)
大蒜半胱氨酸合成酶的mRNA表达及生物信息学分析
郭天璐1张欢欢2杜建中2郝曜山2王亦学2孙毅2
(1. 山西大学生物工程学院,太原 030006;2. 山西省农业科学院生物技术研究中心,太原 030031)
半胱氨酸合成酶的产物半胱氨酸是植物中含硫氨基酸的重要来源,大蒜中含硫氨基酸丰富,研究大蒜半胱氨酸合成酶的性质及生物学功能以明确其在大蒜硫代谢中的作用。利用生物信息学技术分析从NCBI数据库中获得的3个大蒜的半胱氨酸合成酶基因(AsGCS2、AsGCS3和AsGCS4)的开放阅读框,预测其蛋白序列、分子量大小、蛋白质特性、亚细胞定位、系统进化等特征;通过荧光定量PCR分析了3个半胱氨酸合成酶的组织表达特性。结果显示,AsGCS2、AsGCS3和AsGCS4的开放态读码框长度分别为1 152 bp、1 296 bp和1 236 bp;其编码蛋白的理论相对分子量分别为40.6 kD、34.1 kD和36.1 kD。AsGCS2被亚细胞定位于叶绿体中,而AsGCS3和AsGCS4均被定位于细胞质中。氨基酸比对结果表明,AsGCS2与AsGCS3相似度为70%,与AsGCS4相似度为59%;而AsGCS3与AsGCS4相似度为68%。进化分析图表明,AsGCS2属于Bsas2亚家族,AsGCS3属于Bsas1亚家族,AsGCS4属于Bsas6亚家族。荧光定量PCR结果表明,大蒜不同CSase的组织特异性是不同的,AsGCS2在叶中表达量最高,ASGCS3在根中表达量最高,而ASGCS4在叶和根中都有较高的表达量。3个半胱氨酸合成酶基因分别属于不同的Bsas亚家族,它们的组织表达特性也不相同,它们在大蒜不同组织中的半胱氨酸合成途径中起作用。
大蒜;半胱氨酸合成酶;荧光定量PCR
大蒜(Allium sativum)是百合科葱属的多年生草本植物,自古以来一直是食药两用的食品,其中蒜素(Allicin)被认为有较强的抗微生物活性[1]。此外,大蒜中还含有20多种含硫化合物,而这些含硫化合物的生成都离不开半胱氨酸。半胱氨酸是蛋白及谷胱甘肽中的重要成分,也是甲硫氨酸与植物含硫化合物的硫供体,是蒜氨酸合成的必要成分。半胱氨酸生物合成是由丝氨酸乙酰转移酶(Serine acetyltransferase,SATase)和半胱氨酸合成酶(Cysteine synthase,CSase)完成[2]。半胱氨酸合成酶也称作O-乙酰丝氨酸硫解酶(O-acetylserine(thiol)lyase,OAS),催化O-乙酰丝氨酸和硫化氢合成半胱氨酸。在半胱氨酸合成酶的催化下H2S与O-乙酰丝氨酸反应生成Cys,且半胱氨酸合成酶能与丝氨酸乙酰转移酶形成复合物[3,4]。半胱氨酸合成酶属于β-取代丙氨酸合成酶家族(β-substituted alanine synthases family,Bsas)需要磷酸吡哆醛作为辅酶[5]。目前,从许多植物中都克隆到了半胱氨酸合成酶,分别位于细胞质、线粒体、叶绿体这些亚细胞结构中[6]。半胱氨酸合成酶基因的表达受到氮、硫、光照及重金属等环境因素调节[7-9]。过表达半胱氨酸合成酶的烟草,由于提高了体内半胱氨酸及谷胱甘肽的含量,增加了其对重金属、氧化、除草剂等逆境的耐受性[10-13]。
本研究从NCBI 数据库检索到3个大蒜的半胱氨酸合成酶基因,通过生物信息学软件分析这些基因的性质,通过荧光定量PCR分别检测其在不同组织部位的相对表达量,旨在为大蒜半胱氨酸合成酶的生物功能研究及应用提供参考。
1 材料与方法
1.1材料
山东金乡白皮大蒜,购买自当地农贸市场。
1.2方法
1.2.1序列分析及系统发育树的构建 在NCBI数据库中检索到3条大蒜CSase的cDNA序列。利用ExPASy网站的Translate工具将大蒜CSase的cDNA翻译成氨基酸序列;利用ExPASy服务器ProtParam、ProtScale工具预测CSase蛋白的亲疏水性和理化性质;利用TMHMM工具分析了CSase蛋白的跨膜结构域;利用SignalP4.1 预测CSase蛋白的信号肽;利用PSORT 预测ScCBF1的亚细胞定位;并在PSIpred网站上预测CSase的二级结构(表1)。利用ClustalX与其他物种的CSase氨基酸序列进行多序列比对;利用MEGA5.1(Neighbor-Joining)构建系统进化树。

表1 生物信息学研究的数据库及软件网址
1.2.2大蒜CSase和Alliinase的mRNA表达特性 用Primer Primer5在获得的cDNA序列中选择合适的区域,设计大蒜CSase的mRNA表达引物及Alliinase的mRNA的表达引物(表2)。

表2 大蒜CSase及Alliinase研究中使用的引物
将大蒜种植于花盆中,取发芽后7 d大蒜的根、茎、叶的组织。每3株的组织混合为一个重复,每个样品取3次重复。所有的材料用75%酒精擦拭表面后液氮速冻,于-70℃冰箱保存。采用RNAiso (TaKaRa)提取以上材料的总RNA,经 BioPhotometer plus核酸蛋白仪(Eppendorf)定量后,取2 μg总RNA作为模板用Reverse TranscriptaseM-MLV(RNase H-)(TaKaRa)反转录合成cDNA第一链。
以上述的反转录的cDNA(5×稀释)为模板,以EF1a作为内参基因(表2),在ABI公司的7300 Real-time PCR仪完成定量PCR扩增。每个样品设置3次重复。SYBR Green试剂盒采用TransGen公司的TransStart Green qPCR SuperMix UDG,扩增程序为:50℃ 2 min;95℃ 10 min;95℃ 15 s,60℃ 45 s,40个循环。结果采用2-ΔΔCt法处理数据。
2 结果
2.1生物信息学分析
通过NCBI数据库获得3个大蒜CSase基因,分别是AsGCS2(GenBank:AY766093)AsGCS3(Gen-Bank:AY766094)、AsGCS4(GenBank:AY766095),对其进行生物信息学分析,结果见表3。

表3 大蒜CSase生物信息学分析结果
2.1.1AsGCS2生物信息学分析 从NCBI数据库获得大蒜的AsGCS2的cDNA序列长度为1 424 bp,其中ORF长度为1 152 bp(36-1 187 bp),其对应的多肽为383个氨基酸。Protparam分析表明,AsGCS2理论的分子质量为40.6 kD,理论的等电点为8.73,不稳定系数为35.74,表明其为相对稳定蛋白,脂肪系数为92.27,总平均疏水性为0.019,表明该蛋白可能是一种疏水蛋白。
利用TMHMM在线分析,结果表明该蛋白未发现跨膜结构域,因此AsGCS2不是跨膜蛋白。利用SignalP4.1Server在线软件对大蒜的AsGCS2蛋白信号肽进行预测分析结果表明,该蛋白N端不存在任何信号肽序列,是非分泌蛋白。利用PSORT在线软件分析得知AsGCS2定位于叶绿体中。在PSIpred网站上预测AsGCS2的二级结构,发现AsGCS2包含12个α螺旋,10个β折叠。
2.1.2AsGCS3生物信息学分析 从NCBI数据库获得大蒜AsGCS3的cDNA序列长度为1 296 bp,其中ORF长度为972 bp(125-1 096 bp),其对应的多肽为323个氨基酸。Protparam分析表明,AsGCS3理论的分子质量为34.1 kD,理论的等电点为5.80,不稳定系数为29.98,表明其为相对稳定蛋白,脂肪系数为103.9,总平均疏水性为0.12,表明该蛋白可能是一种疏水蛋白。
利用TMHMM在线分析,结果表明该蛋白未发现任何的跨膜结构域,表明AsGCS3也不是跨膜蛋白。利用SignalP4.1Server在线软件对大蒜的AsGCS3蛋白的信号肽进行预测分析,结果表明该蛋白N端不存在信号肽序列,同样是非分泌蛋白。利用PSORT在线软件分析得知AsGCS3定位于细胞质中。在PSIpred网站上预测AsGCS3的二级结构,发现AsGCS3包含11个α螺旋,10个β折叠。
2.1.3AsGCS4生物信息学分析 从NCBI数据库获得大蒜AsGCS4的cDNA序列长度为1 236 bp,其中ORF长度为1 020 bp(60-1 079 bp),其对应的多肽为343个氨基酸。Protparam分析表明,AsGCS4理论的分子质量为36.1 kD,理论的等电点为5.30,不稳定系数为36.4,表明其为相对稳定蛋白,脂肪系数为89.76,总平均疏水性为-0.071,表明该蛋白可能是一种亲水蛋白。
利用TMHMM在线分析,结果表明AsGCS4蛋白未发现任何的跨膜结构域,表明此蛋白不是跨膜蛋白。利用SignalP4.1Server在线软件对大蒜的AsGCS4蛋白的信号肽进行预测分析结果表明,AsGCS4蛋白N端不存在信号肽序列,同样也是非分泌蛋白。利用PSORT在线软件分析得知AsGCS4定位于细胞质中。利用PSIpred网站软件预测AsGCS4的二级结构,发现AsGCS4包含10个α螺旋,11个β折叠。
2.2蛋白多序列比对及系统进化树分析
用GeneDoc将从NCBI数据库中找到的3个半胱氨酸合成酶基因:AsGCS2、AsGCS3、AsGCS4进行比对发现,AsGCS2与AsGCS3相似性为70%,与AsGCS4相似性为59%,而AsGCS3与AsGCS4相似性为68%,说明它们之间具有很高的保守性。Saito等[14]在1993年已经证明赖氨酸是与辅酶磷酸吡哆醛连接的重要位点。如图1所示,第98-116位氨基酸是半胱氨酸合成酶的PLP连接位点,并具有保守序列KXEXXXPXXSVKDR(方框部分),其中活性位点中心的第二个赖氨酸残基(Lys109)连接PLP(图1圆圈标注),酶活性位点的19个氨基酸残基在植物中高度保守。
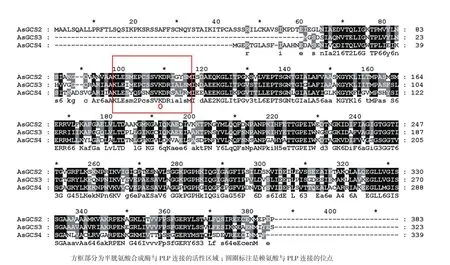
图1 大蒜CSase氨基酸序列比对结果
本实验通过将已知亚细胞定位的其他物种的半胱氨酸合成酶基因进行序列比对后,用ClustalX、MEGA5.1(Neighbor-Joining)构建系统进化树。结果(图2)显示,按照它们亚细胞定位的不同大体可以分为6个亚家族,我们所研究的AsGCS2属于Bsas2(叶绿体CSase),AsGCS3属于Bsas1(细胞质CSase),AsGCS4位于细胞质,但是属于Bsas6,一个功能未知的亚家族。
2.3CSase(AsGCS2、AsGCS3、AsGCS4)和Alliinase(AsALL)基因的组织特异性表达
通过对大蒜萌发时期根、假茎、叶组织的mRNA表达水平进行Real-time PCR分析可以看出(图3),大蒜不同的组织部位CSase的表达程度是不同的,AsGCS2在叶部位表达量达到最高,AsGCS3表达量最高的部位为根,AsGCS4在叶和根中都有较高的表达量。总体而言CSase在根部都有较高的表达量,假茎部表达量较低,与之相反,AsALL在根部表达量较低。
3 讨论
半胱氨酸合成酶属于Bsas家族,基于一级结构及亚细胞定位,Hatzfeld将其分成6个亚家族:Bsas1,细胞质CSase;Bsas2,叶绿体CSase;Bsas3,线粒体CAS;以及未定位的Bsas4,Bsas5 和Bsas6[6]。Bsas1、Bsas2主要的活性都是合成Cys[15]。Bsas3类的β-氰丙氨酸合成酶(β-Cyanoalanine synthase,β-CAS)催化氢氰酸与L-半胱氨酸结合生成β-氰丙氨酸,具有使氰化物脱毒的功能,且都需要磷酸吡哆醛(pyridoxal 5'-phosphate,PLP)作为辅基。其序列及结构与半胱氨酸合成酶相似,定位于线粒体中[16]。有些Bsas3中的β-CAS具有合成半胱氨酸与β-氰丙氨酸的双重功能[16-18]。Bsas4的Case活性很低,没有CAS活性[6]。CSase-like 蛋白At5g28030被证明具有半胱氨酸脱硫酶的活性[19]。Bsas5的酶具有S-硫胱氨酸合成酶(S-sulfocysteine synthase)活性,没有半胱氨酸合成酶活性[20]。Bsas6的酶定位于细胞质中,但其功能尚不清楚。AsGCS4与Bsas6的酶序列相近,但与所有已知的半胱氨酸合成酶的差异较大,也可能属于一个新的Bsas亚家族Bsas7,由于缺乏相关功能研究结果与AsGCS4相关序列的支持,暂时将AsGCS4归入Bsas6中。
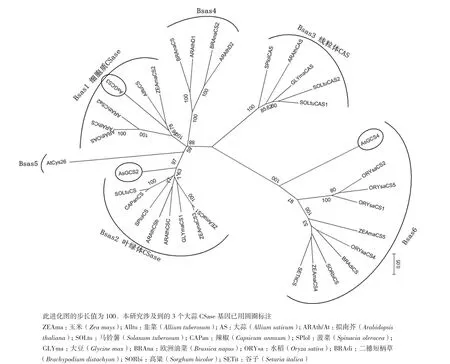
图2 Bsas家族的系统进化分析
半胱氨酸是合成蒜氨酸及蒜素的重要前体,为了研究哪个半胱氨酸合成酶与蒜氨酸及蒜素合成有关,比较了3种CSase酶与蒜氨酸酶在大蒜不同组织部位的表达,未发现与蒜氨酸酶表达完全吻合的基因在叶与根中表达量均较高。蒜氨酸酶在叶和假茎中表达较高,半胱氨酸合成酶的表达模式与其均不相同,可能大蒜中还存在其他未知的半胱氨酸合成酶,拟南芥中有至少8个CSase-like蛋白,玉米与水稻中有5个CSase-like蛋白。
半胱氨酸合成酶催化半胱氨酸合成,其产物半胱氨酸对无机硫的同化起中心作用,为甲硫氨酸、谷胱甘肽及植物中的多种含硫次级代谢物的生物合成提供唯一的硫化物供体[21]。半胱氨酸合成酶的表达受到氮、硫、盐等非生物胁迫的调节,过量表达半胱氨酸合成酶能够增加植物体内谷胱甘肽含量,提高植物对逆境的抵抗力[22]。半胱氨酸在植物免疫调节中具有重要作用,拟南芥OAS-Al的突变体导致对致病菌的免疫力下降[23]。由于半胱氨酸的重要作用,植物中的半胱氨酸合成酶是冗余的,OAS-TL A,OAS-TL B贡献了半胱氨酸合成酶活性的绝大部分,半胱氨酸合成酶对花粉发育具有重要作用,OAS-TL A,OAS-TL B,OAS-TL C全部缺失的花粉发育受到影响[24]。这些研究表明半胱氨酸合成酶在植物抗逆,抗损伤以及免疫调节方面具有重要作用。大蒜中半胱氨酸、谷胱甘肽等含硫化合物的组分远高于其他植物,因此,研究其半胱氨酸合成酶的表达及生物学功能具有重要意义。
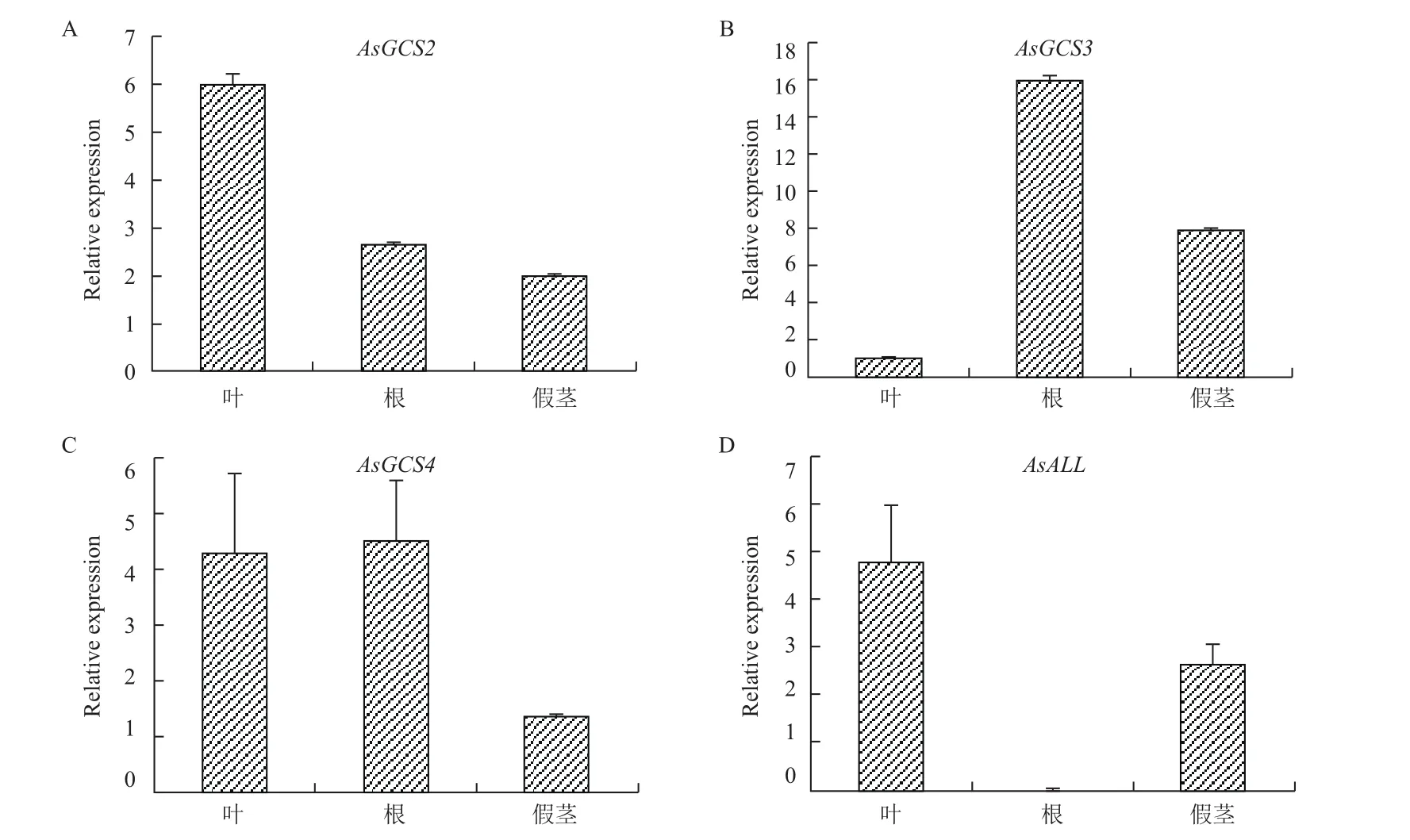
图3 CSase(AsGCS2、AsGCS3、AsGCS4)和Alliinase基因的不同组织特异性表达
4 结论
AsGCS2属于Bsas2叶绿体CSase亚家族,亚细胞定位于叶绿体,在叶中表达量最高;AsGCS3属于Bsas1线粒体CAS亚家族,亚细胞定位于细胞质,在根中表达量最高;AsGCS4属于Bsas6亚家族,亚细胞定位于细胞质,在叶和根中都有较高的表达量。除此之外,它们在大蒜不同组织中的半胱氨酸合成途径中起作用。
[1]Yoshimoto N, Yabe A, Sugino Y, et al. Garlic gamma-glutamyl transpeptidases that catalyze deglutamylation of biosynthetic intermediate of alliin[J]. Front Plant, 2015, 5:758-768.
[2]Urano Y, Manabe T, Noji M, Saito K. Molecular cloning and functional characterization of cDNAs encoding cysteine synthase and serine acetyltransferase that may be responsible for high cellular cysteine content in Allium tuberosum[J]. Gene, 2000, 257(2):269-277.
[3] Ravina CG, Chang CI, Tsakraklides GP, et al. The sac mutantsof chlamydomonas reinhardtii reveal transcriptional and posttranscriptional control of cysteine biosynthesis[J]. Plant Physiol, 2002,130:2076-2084.
[4]Ikegami F, Itagaki S, Murakoshi I. Purification and characterization of two forms of cysteine synthase from Allium tuberosum[J]. Phytochemistry, 1992, 32:31-34.
[5]Ikegami F, Murakoshi I. Enzymic synthesis of non-protein β-substituted alanines and some higher homologues in plants[J]. Phytochemistry, 1994, 35(5):1089-1104.
[6]Hatzfeld Y, Maruyama A, Schmidt A, et al. β-Cyanoalanine synthase is a mitochondrial cysteine synthase-like protein in spinach andArabidopsis[J]. Plant Physiology, 2000, 123(3):1163-1172.
[7]Takahashi H, Saito K. Subcellular localization of spinach cysteine synthase isoforms and regulation of their gene expression by nitrogen and sulfur[J]. Plant Physiology, 1996, 112(1):273-280.
[8]CintiaGoulart K, Masaaki N, Michimi N, et al. Heavy metal tolerance of transgen ic tobacco plants over- expressing cysteine synthase[J]. Biotechnology Letters, 2004, 26:153-157.
[9]Nakamura T, Yamaguchi Y, Sano H. Four rice genes encoding cysteine synthase:isolation and differential responses to sulfur,nitrogen and light[J]. Gene, 1999, 229(1-2):155-161.
[10]Noji M, Saito M, Nakamura M, et al. Cysteine synthase overexpression in tobacco confers tolerance to sulfur-containing environmental pollutants[J]. Plant Physiology, 2001, 126(3):973-980.
[11]Youssefian S, Nakamura M, Orudgev E, et al. Increased cysteine biosynthesis capacity of transgenic tobacco overexpressing an O-acetylserine(thiol)lyase modifies plant responses to oxidative stress[J]. Plant Physiology, 2001, 126(3):1001-1011.
[12]Domínguez-Solís JR, Gutiérrez-Alcala G, Romero LC, et al. Cytosolic O-acetylserine(thiol)lyase gene is regulated by heavy metals and can function in cadmium tolerance[J]. Journal of Biological Chemistry, 2000, 276(12):9297-9302.
[13]Nakamura M, Ochiai T, Noji M, et al. An improved tolerance to cadmium by overexpression of two genes for cysteine synthesis in tobacco[J]. Plant Biotechnology, 2014, 31(2):141-147.
[14]Saito K, Kurosawa M, Murakoshi I. Determination of a functional lysine residue of a plant cysteine synthase by site-directed mutagenesis, and the molecular evolutionary implication[J]. FEBS Lett, 1993, 328:111-114.
[15]Hell R, Bork C, Bogdanova N, et al. Isolation and characterization of two cDNAs encoding for compartment specific isoforms of O-acetylserine(thiol)lyase from Arabidopsis thaliana[J]. FEBS Lett, 1994, 351:257-262.
[16]Marrero-Degro J, Marcano-Velázquez J, Siritunga D. Isolation and characterization of novel β-cyanoalanine synthase and cysteine synthase genes from cassava[J]. Plant Molecular Biology Reporter, 2011, 29(3):514-524.
[17]王小芳, 杨玲娟, 董晓宁, 等. 植物半胱氨酸合成及调控研究进展[J]. 植物生理学报, 2011, 47(1):37-48.
[18]Ikegami F, Takayama K, Murakoshi I. Purification and properties of β-cyano-l-alanine synthase from Lathyrus latifolius[J]. Phytochemistry, 1988, 27:3385-3389.
[19]Álvarez C, Calo L, Romero LC, et al. An Oacetylserine(thiol)lyase homolog with L-cysteine desulfhydrase activity regulates cysteine homeostasis in Arabidopsis thaliana[J]. Plant Physiol,2010, 152:656-669.
[20]Bermudez MA, Galmes J, Moreno I, et al. Photosynthetic adaptation to length of day is dependent on S-sulfocysteine synthase activity in the Thylakoid lumen[J]. Plant Physiol, 2012, 160(1):274-288.
[21]Bonner ER, Cahoon RE, Knapke SM, et al. Molecular basis of cysteine biosynthesis in plants:Structural and functional analysis of O-acetylserine sulfhydrylase from Arabidopsis thaliana[J]. Journal of Biological Chemistry, 2005, 280(46):38803-38813.
[22]许奕, 金志强, 宋顺, 等. 香蕉MaCSase 基因的克隆和表达分析[J]. 中国农学通报, 2012, 28(34):202-210.
[23]Álvarez C, Ángeles Bermúdez M, Romero LC, et al. Cysteine homeostasis plays an essential role in plant immunity[J]. New Phytologist, 2012, 193(1):165-177.
[24]Birke H, Heeg C, Wirtz M, et al. Successful fertilization requires the presence of at least one major O-acetylserine(thiol)lyase for cysteine synthesis in pollen of Arabidopsis[J]. Plant Physiology,2013, 163(2):959-972.
(责任编辑 马鑫)
mRNA Expression and Bioinformatics Analysis of Cysteine Synthase in Allium sativum
GUO Tian-lu1ZHANG Huan-huan2DU Jian-zhong2HAO Yao-shan2WANG Yi-xue2SUN Yi2
(1. College of Bio-engineering,Shanxi University,Taiyuan 030006;2. Research Center of Biotechnology,Shanxi Academy of Agricultural Sciences,Taiyuan 030031)
The cysteine synthesized by cysteine synthase is an important source of amino acids containing sulfur in plants. The amino acid with sulfur in garlic is abundant,thus we studied the properties and biological functions of garlic cysteine synthase in order to clarify its role in the metabolism of garlic. Three cysteine synthase genes of garlic,AsGCS2,AsGCS3 and AsGCS4 were found from NCBI database,and then bioinformatics analysis and RT-PCR were conducted. The lengths of their open reading frames were 1 152 bp,1 296 bp,and 1 236 bp,respectively. The theoretical relative molecular weights of their encoded enzymes were 40.6 kD,34.1 kD,and 36.1 kD,respectively. By subcellular localization,AsGCS2 was localized in the chloroplasts,while AsGCS3 and AsGCS4 were localized in the cytoplasm. The results of the amino acid sequence alignment revealed that the similarity of AsGCS2 with AsGCS3 was 70%,and that with AsGCS4 was 59%,while it was 68% between AsGCS3 and AsGCS4. Phylogenetic analysis showed that AsGCS2 belonged to Bsas2,AsGCS3 belonged to Bsas1,but AsGCS4 was different from all known cysteine synthases,possibly belonging to Bsas6. RT-PCR expression analysis indicated that the expression levels of CSase varied in various tissues. The expression level of AsGCS2 was the highest in the leaves,the highest expression of AsGCS3 was in the root,and AsGCS4 expressed highly in both leaves and roots. In conclusion,3 cysteine synthase gene AsGCS2,AsGCS3,and AsGCS4 belong to the different subfamilies of Bsas,their tissue expression characteristics were not the same,and they play a role in different tissues of garlic in cysteine biosynthesis.
Allium sativum;cysteine synthase;qRT-PCR
10.13560/j.cnki.biotech.bull.1985.2016.08.015
2015-10-21
转基因生物新品种培育重大专项(2014ZX08003001-002-003)
郭天璐,女,硕士研究生,研究方向:植物转基因技术;E-mail:421489192@qq.com
孙毅,男,博士,研究方向:植物转基因技术;E-mail:sunyi692003@163.com张欢欢,男,硕士,研究方向:转基因抗虫技术;E-mail:frank.red@163.com

