利用水稻抗旱种质研究驯化和育种对遗传多样性的影响
卫海滨,夏 辉,楼巧君,冯芳君,马孝松,梅捍卫,罗利军(上海市农业生物基因中心,上海 201106)
利用水稻抗旱种质研究驯化和育种对遗传多样性的影响
卫海滨,夏 辉,楼巧君,冯芳君,马孝松,梅捍卫*,罗利军*
(上海市农业生物基因中心,上海201106)
利用高通量测序获取SNP标记技术对上海市农业生物基因中心99个水稻抗旱种质进行全基因组重测序,分析其遗传多样性和遗传基础。结果表明:在所有SNPs位点中,多态性比率为68.98%(1 927 238/2 793 702),SNPs标记密度在12条染色体上分布范围为4.5—6.2个SNP/kb;供试品种间的遗传距离分布具有两个密度高峰;邻接法构建系统发育树将99个品种划分为籼稻和粳稻两个类群。综合SNPs和进化分析结果表明:水稻在自然选择和人工驯化过程形成复杂进化史,99个抗旱种质在DNA水平上的差异呈现两个极端,遗传距离分布范围较大(0.003—0.998),具有较宽遗传基础和丰富的遗传多样性。本研究以期为水稻节水抗旱新品种选育的亲本选配提供理论依据。
水稻;遗传多样性;抗旱性;育种;单核苷酸多态性
水稻(Oryzasativa L.)是世界上最古老的主要粮食作物之一,在全球农业生产中占有十分重要的地位。当前世界人口不断增长,耕地面积逐年减少,水资源的竞争日益激烈,全球面临的粮食危机凸显[1]。然而,上世纪末至今水稻产量却没有大的突破。主要原因在于栽培稻驯化过程中,为了获得一种优良性状而使得很多优秀农艺性状的杂合位点在人工选择过程中丢失[2]。栽培稻的驯化和育种中,基因组的遗传多态性发生了明显的变化,导致水稻种质的遗传多样性降低,供体亲本的遗传背景变窄[3]。目前已收集的水稻抗旱种质品种包括具有抗旱或高产性能的地方种和节水抗旱稻育成种,其遗传多样性的丰富程度将是重要遗传资源加以充分利用的基础。
近年来,随着高通量测序技术的普及,SNP分子标记被广泛用于研究物种的遗传多样性与遗传结构以及不同品种间的亲缘关系[4]。例如,通过对水稻全基因组SNP数据的分析,揭示了水稻的驯化与起源,发现粳稻首先从普通野生稻中驯化出来,然后在栽培稻的传播过程中粳稻与普通野生稻反复发生基因流最终形成了籼稻[5]。对我国水稻主产区的1495份杂交水稻品种进行基因组测序,分析揭示了基因组区段在籼稻和粳稻间的遗传多样性特征[6]。因此,利用SNP分子标记研究水稻群体的遗传多样性及其在驯化过程中发生的变化,是完全可行的。
为了有效利用淡水资源,实现从高产到节水抗旱的转型,本世纪初我国启动了“绿色超级稻新品种培育”的863项目,本单位承担了水稻节水抗旱种质创新子课题。上海市农业生物基因中心初步建立了我国节水抗旱核心种质资源,通过对各种材料的评价和鉴定,并进行遗传多样性分析。本研究采用Illumina高通量测序SNP标记对本中心建立的抗旱种质(99个栽培稻品种)进行研究,旨在揭示这些抗旱种质在全基因组水平上的遗传多样性和亲缘关系,快速获取大量有益的种质评价信息,以期为水稻节水抗旱新品种选育的亲本选配提供理论依据。
1 材料与方法
1.1水稻品种及其DNA提取
供试品种来自上海市农业生物基因中心种质资源,包括通过节水抗旱性能的评价和鉴定的地方品种和节水抗旱稻育成种,共计99个(表1)。各品种的系谱资料源自品种参试时育种单位提供的信息,可以参考国家水稻数据中心的资料(www.ricedata.com)。上海市农业生物基因中心水稻遗传育种温室种植,取水稻苗期幼嫩叶片,采用天根生化科技(北京)有限公司植物基因组抽提试剂盒抽提基因组DNA。

表1 用于SNP分析的水稻品种Table 1 Rice varieties used forsNP analysis

(续表1)
1.2高通量测序及SNP分析
利用Illumina HiSeq-2000对水稻抗旱种质的99个品种进行全基因组重测序,由苏州众信生物技术有限公司完成,测序深度为5×,序列比对的参考基因组为日本晴msu7.0(http://rice.plantbiology.msu.edu/)。获取基因组SNP信息后,将SNP分型数据用Clustalw 2软件进行完全比对,应用Phylip 3.6.5软件的Dnadist程序计算品种间的遗传距离,最后采用邻接法构建遗传关系树状图。
2 结果与分析
2.1多态性SNP位点的分布
99个水稻抗旱种质的重测序基因组序列与日本晴Nipponbare参考基因组进行比对,检测出群体SNPs。在分布于12条染色体的2793702个SNP位点中,1 927 238个被检测到多态性,多态性比率为68.98%。不同染色体上分布的多态性位点数量范围为124070—214941个,分布差异较大,且在1号染色体上分布最多(图1)。但每条染色体的SNPs标记密度差异较小,分布区间为4.5—6.2个SNP/kb,平均标记密度为5.16个SNP/kb。
2.2水稻品种间的遗传变异
基于SNP标记的品种两两之间遗传距离分析,其变异范围为0.003—0.998,平均值为0.527,其中‘MILT_1444’(R11)和‘IRAT_13’(R16)间的遗传距离最小(0.003),而‘LIANGGUOZAO’(R85)与‘TODOROKIWASE’(R102)间的遗传距离最大(0.998)。99个供试品种两两计算,共获得4 851个遗传距离数字,以0.05为组距进行次数分布分析,可见99个品种间的遗传距离具有两个分布密度高峰(图2)。在0.2与0.9处分布密度最大,两侧不对称。大于0.7的遗传距离数字有1 922个(39.62%),小于0.3的有1 323个(27.27%),分布呈现两个极端的趋势。
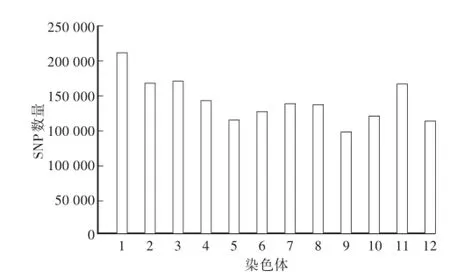
图1 多态性位点在水稻12条染色体上的分布Fig.1 Distribution of polymorphicsites in 12 chromosomes of rice

图2 品种间遗传距离的次数分布Fig.2 Frequencydistribution ofgeneticdistances between varieties
2.3聚类分析
根据水稻SNP遗传距离矩阵进行聚类分析,99个品种被划分为籼稻(黑色)和粳稻(灰色)两大类群。其中育种常用到的两个代表性品种:籼稻‘珍汕97B’(R158)和粳稻‘IRAT 109’(R109),分别位于籼粳群体中部,图3中分别以红色和绿色表示;两个AUS类型的品种‘LAMBAYEQUE 1’(R25)和‘AUS 454’(R35)被分类至为粳稻,图中以蓝色表示,与‘IRAT 109’的遗传距离分别为:0.182和0.387,表明在遗传基础上具有很高的亲缘性,且‘IRAT 109’(R109)与‘LAMBAYEQUE 1’(R25)具有更近的亲缘关系。

图3 水稻抗旱种质99个品种的聚类图Fig.3 Cluster analysis of 99drought-resistant rice varieties
3 讨论
水稻基因组精细图的完成和高通量测序技术的迅猛发展,为DNA水平上利用SNP标记研究水稻的遗传多样性创造了良好基础[5-8]。本研究选用的水稻抗旱种质99个品种,是来源于国内外的优良品种,具备一定的代表性。虽然群体材料数量较少,却具备较高的遗传多样性水平,例如每200个碱基就有一个SNP(图1),并与已有研究报道相吻合[9]。同时,经过群体多态性筛选(MAF>0.05),发现了较高的SNP多态性比率,SNP密度较大。以上结果表明该抗旱种质群体具有遗传多样性变异丰富的特点,因此在抗旱性以外,还可能具有较丰富的优异农艺性状。
水稻的驯化会使遗传多样性降低,尤其是一些地方品种及育成种遗传多态性下降水平更加严重[10]。本研究通过遗传距离推断遗传变异程度,发现水稻抗旱种质群体遗传距离分布具有双峰特征(图2)。其中遗传距离位于0.2—0.3出现的峰,表明驯化获得优良农艺性状的同时,当代栽培稻在驯化过程中经历了驯化瓶颈,导致栽培群体相对于祖先野生稻整体遗传多态性有所降低。另一个遗传距离峰位于0.9附近,表明部分种质资源具有相对较为丰富的遗传多样性。亲缘关系结果表明(图3),来源于籼稻和粳稻两大类群的品种具有各自的种质抗性优势,育种分别从籼稻和粳稻中选择一个亲本,更有利于节水抗旱稻新品种的培育[11]。利用这批核心种质,上海市农业生物基因中心已培育出一大批节水抗旱稻,包括常规节水抗旱稻品种:‘中旱2号’‘沪旱3号’‘沪旱15号’‘沪旱7号’,及三系杂交节水抗旱稻不育系与恢复系:‘沪旱1A’‘沪旱2A’‘旱恢2号’‘旱恢3号’等[12-13]。因此这批抗旱种质确实能够成为培育节水抗旱稻的资源宝库。
目前,在节水抗旱稻育种中被广泛深入应用的材料依旧不多,而这些抗旱种质资源却具备较高程度的遗传差异与多样性,提示我们应该进一步挖掘该种质资源群体的育种潜力。在这个过程中,本研究中应用的SNP可以成为很好的分子标记进行分子标记辅助选育与育种,为选育节水抗旱稻新品种提供重点关注的亲本资源。另一方面,发掘与SNP相关的潜在抗旱等优良农艺性状基因,将会成为下一步的研究方向,并将为阐明节水抗旱的分子遗传基础提供有利的参考。
[1]LUO L J,ZHANG Q F.Thestatus andstrategy ondrought resistance of rice(Oryzasativa L.)[J].Chinese J Ricesci.,2001,15:209-214.
[2]CHEN H,XIE W,HE H,et al.A high-densitysNPgenotyping array for rice biology and molecular breeding[J].Mol Plant,2014,7:541-553.
[3]LI Z K,FU B Y,GAO Y M,et al.Genome-wide introgression lines and their use ingenetic and moleculardissection of complex phenotypes in rice(Oryzasativa L.)[J].Plant Mol Biol.,2005,59(1):33-52.
[4]VARSHNEY R K,MAHENDAR T,AGGARWAL R K,et al.Genic molecular markers in plants:Development and applications[M]// VARSHNEY R K,TUBEROSA R.Genomics-Assisted Crop Improvement.Dordrecht,The Netherlands:Springer Netherlands,2007:13-29.
[5]HUANG X H,KURATA N,WEI X H,et al.A map of ricegenome variation reveals the origin of cultivated rice[J].Nature,2012,490:497-501.
[6]HUANG X H,YANGs H,GONG J Y,et al.Genomic analysis of hybrid rice varieties reveals numeroussuperior alleles that contribute to heterosis[J].Nature Communications,2015,6:6258.
[7]SI L,CHEN J,HUANG X,et al.OsSPL13 controlsgrainsize in cultivated rice[J].Natgenet.,2016,48(4):3518.
[8]邓宏中,王彩红,徐群,等.中国水稻地方品种与选育品种的遗传多样性比较分析[J].植物遗传资源学报,2015,16(3):433-442.
[9]SHEN Y J,JIANG H,JIN J P,et al.Development ofgenome-widedNA polymorphismdatabase for map-based cloning of ricegenes[J].Plant Physiol.,2004,135(3):1198-1205.
[10]GARRIS A J,TAI T H,COBUMN J,et al.Geneticstructure anddiversity in Oryzasativa L[J].Genetics,2005,169(3):1631-1638.
[11]LUO L J.Breeding for water-saving anddrought-resistance rice(WDR)in China[J].J Exp Bot.,2010,61(13):3509-3517.
[12]余新桥,梅捍卫,刘康,等.优质节水抗旱雄性不育系‘沪旱1A’的选育与利用[J].上海农业学报,2006,22(2):32-35.
[13]杨华,龙萍,夏辉,等.上海水稻种质资源的研究与利用进展[J].植物遗传资源学报,2015,16(2):245-249.
(责任编辑:程智强)
Impacts ofdomestication and breeding ongeneticdiversity in utilizingdrought-resistant ricegermplasms
WEI Hai-bin,XIA Hui,LOU Qiao-jun,FENG Fang-jun,MA Xiao-song,MEI Han-wei*,LUO Li-jun*
(Shanghai Agrobiologicalgene Center,Shanghai 201106,China)
Thegenome-widesequencing of the 99drought-resistant rice varieties collected by this center was conducted by using high throughoutgenomesequence togetsNP markers and thegeneticdiversity was analyzed.The resultsshowed that the percentage of polymorphicsites was 68.98%(1 927 238/2 793 702)among all the identifiedsNPs,and thedensity ofsNP markers in 12 chromosomes was 4.5—6.2sNP/kb;Thedistribution ofgeneticdistances between the tested varieties had twodensity peaks;The 99 rice varieties could be classified into indica and japonica types in the phylogenetic tree established by the neighbor-joining method. ThesNP and phylogenetic analyses indicated that a complicated history of rice evolution formedduring naturalselection and artificialdomestication,thedifferences of 99drought-resistant rice varieties atdNA level presented two extremes,thedistribution ofgeneticdistances was relatively wide in range(0.003—0.998),and thus rice had abundantgeneticdiversity.The findings could provide a theoretical basis for parentalselection in breeding newdrought-resistant rice cultivars.
Rice;Geneticdiversity;Drought resistance;Breeding;SNP
S511
A
1000-3924(2016)04-001-05
2016-03-30
上海市自然科学基金项目(14ZR1437000);上海市种业发展项目[沪农科种字(2014)第1号,沪农科种字(2016)第1-6号]
卫海滨(1977—),男,博士,助理研究员,主要从事节水抗旱稻遗传研究。E-mail:whb@sagc.org.cn
,E-mail:hmei@sagc.org.cn,lijun@sagc.org.cn

