浓香型白酒窖泥变质前后古菌群落差异分析
于春涛,项 明,王 蕾,许郑林,刘振江
(1.沧州医学高等专科学校,河北沧州061001; 2.河北省宁晋县泥坑酒业有限责任公司,河北石家庄054000)
浓香型白酒窖泥变质前后古菌群落差异分析
于春涛1,项明1,王蕾1,许郑林1,刘振江2
(1.沧州医学高等专科学校,河北沧州061001; 2.河北省宁晋县泥坑酒业有限责任公司,河北石家庄054000)
窖泥是浓香型白酒的生命线,窖泥变质则为酒厂带来巨大损失。采用Illumina Miseq高通量测序DNA技术对窖泥变质前后古菌群落结构进行分析。通过构建古菌16S rDNA基因文库,结果表明,变质后窖泥共扩增出52876条序列,变质前窖泥扩增出11703条序列,古菌丰度有明显提高。对序列进行聚类、测序、分析,窖泥变质前优势古菌为:甲烷短杆菌属(35.66%)、甲烷杆菌属(10.25%)、类芽孢杆菌属(4.27%)、芽孢杆菌属(3.39%)、甲烷八叠球菌属(3.27%)等;窖泥变质后优势菌为:产甲烷袋菌属(38.18%)、甲烷杆菌属(34.21%)、甲烷袋状菌属(6.90%)等。结果揭示了甲烷菌是窖泥变质前后古菌的优势菌,为进一步研究窖泥微生物全貌及窖泥培养、养护提供依据。
窖泥; 变质; 古菌群落; 差异分析; 高通量测序; 白酒
窖泥是浓香型白酒生产的基础,窖泥微生物通过复杂的物质能量代谢,产生具有独特风味和品质的白酒,因此窖泥的好坏直接影响产酒的质量。窖泥微生态中的微生物多样性十分复杂,包括细菌、真菌及古菌,这些微生物群落会随着生长环境的变化(如温度、湿度、pH值、C/N等)而变化[1]。较早开展浓香型白酒微生物研究的是法国人A.Calmette的探索性实验——“阿米露法”[2]。目前我国学者在窖泥功能菌研究方面取得了不少可喜成绩,主要以细菌为主,真菌和古菌研究较少,如邓依[3]等通过细菌rDNA ITS-AFLP结合聚类分析技术比较了多粮发酵新老窖池间的细菌群落差异,罗惠波[4]通过PCR-SSCP技术研究窖池微生物群落等。
由于北方四季温差大,气候干燥,窖泥中微生物菌群演替快,再加上管理不善,致使窖泥中微生态平衡被破坏,代谢出现异常,导致窖泥出现板结变硬、腐败气味等变质现象[5],用于生产致使酒质下降,给酒厂带来了巨大损失。已有研究表明,一些古菌在浓香型白酒“产香”过程中起着重要作用[6]。因此弄清窖泥古菌群落的动态变化,检测出窖泥变质前后的主要优势古菌,对人工窖泥培养和提高浓香型白酒质量尤为重要。但由于古菌独特的生长方式,窖泥中大部分古菌是未培养的,即使得到了纯培养,其形态和生理也可能发生了很多变化[7],因此,采用传统的分离培养方法研究窖泥古菌存在很大的局限性[8-9]。
近几年发展起来一项全新的高通量DNA测序技术——Illumina Solexa合成测序,能检测出土壤中80%以上的优势菌群,具有高准确性、高通量、高灵敏度和低运行成本等突出优势,可以同时分析样本内菌群丰度和样本间菌群丰度差异,已广泛应用于探索土壤、海洋和湿地,以及食品发酵和废水处理反应器等微生物区系研究,并取得了丰硕的研究成果[10-11]。目前,利用Illumina Solexa高通量DNA测序技术对窖泥中古菌群落结构的研究还未见文献报道。鉴于此,本实验通过Illumina Solexa高通量DNA测序技术,分析了浓香型白酒窖泥变质前后古菌群落差异,以期全面、系统认识窖泥微生物群落组成与窖泥质量之间的关系,为揭示窖泥微生态全貌提供帮助。
1 材料与方法
1.1样品采集
窖泥样品取自河北某知名酒厂酿酒车间优质窖泥(HQ1)和变质窖泥(HU1)各100 g。分装后密封冷冻保存。
1.2实验方法
1.2.1窖泥预处理及总DNA提取[12]
称取200 mg的样品,放入灭菌的2 mL离心管中,加入1 mL70%vol乙醇,振荡混匀,10000 r/min室温离心3 min,弃置上层液体。加入1 xPBS溶液,振荡混匀,10000 r/min室温离心3 min,弃置上层液体。倒置2 mL管于吸水纸上1 min,直至没有液体流出。将样品管放入55℃烘箱中维持10 min,使残留酒精完全挥发,保证后续实验操作。
DNA提取步骤按OMEGA试剂盒E.Z.N.ATM Mag-Bind Soil DNAKit的试剂盒使用说明书。
1.2.2PCR扩增及对产物进行琼脂糖凝胶电泳
利用Qubit2.0 DNA检测试剂盒对基因组DNA精确定量,以确定PCR反应应加入的DNA量。古菌引用槽式PCR扩增3轮,第1轮使用M-340F,GU1ST-1000R引物扩增:古1st-340F CCCTAYGGGGYGCASCAG,古1st-1000R GGCCATGCACYWCYTCTC,PCR体系按照如下进行:10×PCR buffer 5 μL,dNTP(10 mM each)0.5 μL,Genomic DNA 10 ng,Bar PCR primer F(50 μM)0.5 μL,Primer R(50 μM)0.5 μL,Plantium Taq(5 U/μL)0.5 μL,H20 add to 50 μL。配制好的PCR体系按照如下反应条件进行扩增:94℃变性3 min,接下来5个循环,分别是94℃变性30 s,45℃退火20 s,65℃延伸30 s;再接着20个循环,分别是94℃变性20 s,55℃退火20 s,72℃延伸30 s,72℃延伸5 min,然后进行第2轮扩增,使用第1轮PCR产物进行扩增,PCR所用的引物已经融合了Miseq测序平台的V3—V4通用引物,349F引物:CCCTACACGACGCTCTTCCGATCTN(barcode)GYGCASCAGKCGMGAAW,806R引物GACTGGAGTTCCTTGGCACCCGAGAATTCCAGGACTACVSGGGTATCTAAT,PCR体系和扩增条件均同第1轮。第3轮扩增引入Illumina桥式PCR兼容引物。PCR结束后,产物进行琼脂糖凝胶电泳,对DNA进行回收。
1.2.316S rDNA扩增片段的测序、质控和聚类
采用Misep测序平台对扩增片段进行双端测序,测得的基因序列经质量控制软件Prinsep处理,去除barcode、两端primer以及部分低质量基因序列,经质控后的基因序列长度大部分分布在400~600 bp之间,基本满足分析需要。
对测得的基因序列采用uclust软件进行OUT聚类,通常域值的序列相似性定位0.97,操作分类单元被认为可能属于属。
水压试验应注意事项:试压时管道内不应有气泡,否则当管道发生漏水时,会导致压力下降现象,不易从压力表上反映出来;管道升压过程中,压力表摇摆不定,摆幅较大,读数不稳,且升压又较慢时,示意管道内气体没有排尽,应重新排气再升压;管道内注水后,必须让其充分浸泡,才能保证管道试压准确;管道顶部土方回填时,宜留出接口位置,以方便检查和修理;当管道有压力时,检查管道不得用手锤敲打管壁和接口,严禁修理管道缺陷,遇有缺陷,应做出标志,卸压后再修补;水压试验过程中,管道危险区域,例如后背、支撑、管端等严禁站人,确保人员安全。
1.2.4对处理后序列进行物种分类,比较窖泥变质前后菌群结构差异
物种分类采用的软件为RDP classifier,基于OUT聚类的结果,获取每一个OUT聚类的代表序列,分别是长度最长序列(length)、丰度最大序列(abundance)和所有序列(ALL)形成3份结果,并对各类RDP分析[13-14]。本文所有的展示,均使用OUT_ALL中的数据,genus水平进行展示,采用柱形图及表格的形式对比窖泥变质前后群落结构差异。
1.2.5MEGAN分析
使用MEGAN软件,通过交互式搜索NCBI中的分类数据库信息,以树状图形式表现物质丰度情况与菌落结构,反应窖泥变质前后真菌的组成情况。
2 结果与分析
2.1窖泥变质前后古菌16S rDNA的PCR产物电泳图谱(见图1)
结合图1中的3条泳道对比可知,泳道1和泳道2有较清晰的条带,条带大小位于400~600 bp之间,基本能够满足分析需要。DNA提取时一般都通过试剂盒过柱提取,这种柱子有固定孔径,从而决定了基因组片段的大小。
2.2窖泥变质前后16S rDNA多样性分析
扩增后的PCR产物去除嵌合体和靶区域序列,得到的基因序列数见表1。
将多条序列按其序列间的距离对它们进行聚类,后根据序列之间的相似性作为域值分成操作分类单元(OTU),通常域值的序列相似性定为0.97,操作分类单元被认为属于属[15-16]。采用OUT VENN分析图统计样本中共有的和独有的OUT数目,直观展示出窖泥变质前后OUT数目的差异(图2)。HU1有1218个OTUs,HQ1有783个OTUs,两者共有192个OTUs。窖泥变质后,古菌的种类和丰度有所提高。说明窖泥变质后的生境更适合一些古菌的生长。
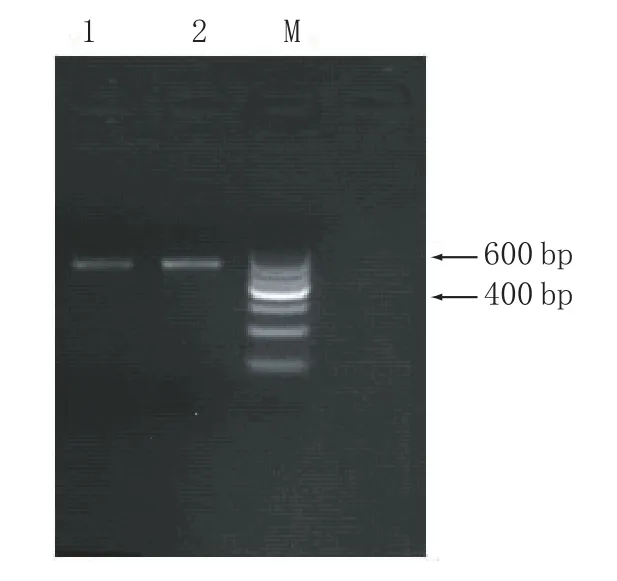
图1 古菌16S rDNA电泳图谱

表1 处理后基因序列统计表
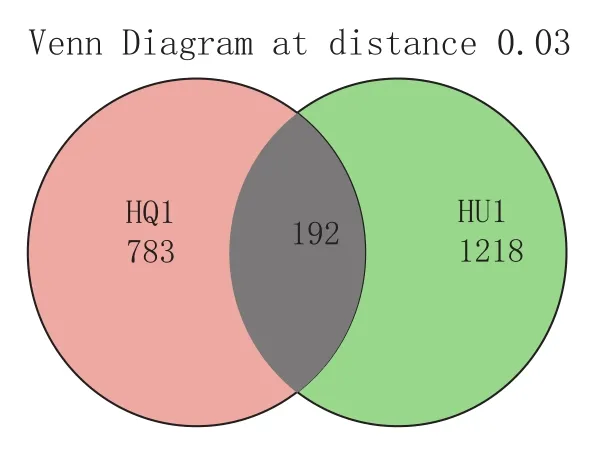
图2 窖泥变质前后OUT数
2.3古菌群落结构分析
对处理后序列,采用RDP classifier软件对每条序列在genus水平上计算其分配到此rank中的概率值,一般概率值大于0.8,即RDP分类域值。根据分类学分析结果,可以得知样品在属分类水平的数据,样本中菌群的reads数目,也就是菌群的丰度值(表2、表3)。
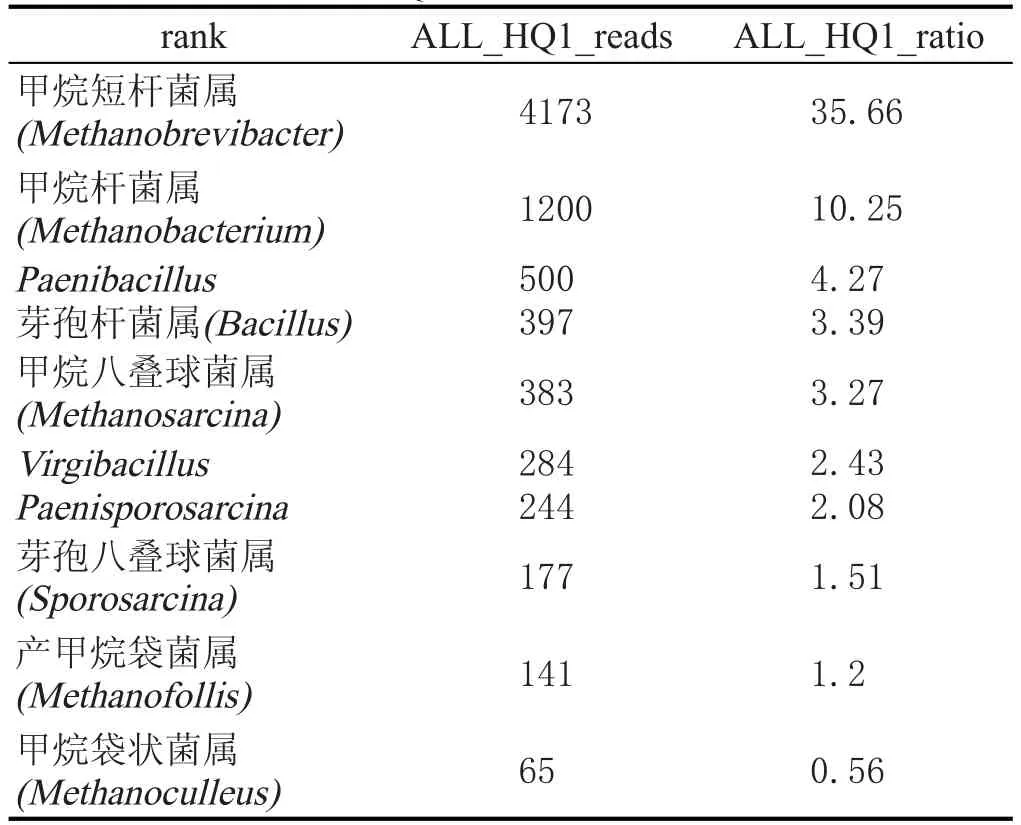
表2 HQ1古菌种类及丰度值
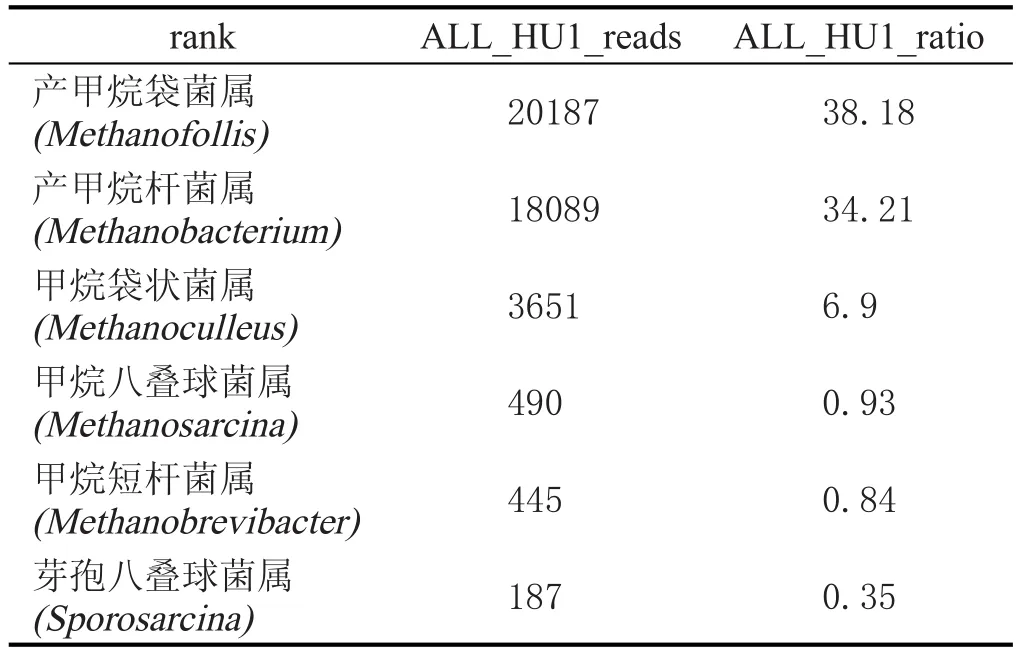
表3 HU1古菌种类及丰度值
由表2、表3可知,窖泥变质前后的古菌群落结构、丰度差异显著,选择优势菌群进行比较。窖泥变质前的绝对优势菌属为甲烷短杆菌属(35.66%)和甲烷杆菌属(10.25%),窖泥变质后产甲烷袋菌属(38.18%)和产甲烷杆菌属(34.21%)成为绝对优势菌属。Thomton[17]认为:在原始土壤中,部分优势菌种的存在都是有特定的有利于其生长环境的结果,这个结论普遍被人们所接受。窖泥中古菌群落结构发生变化,归根结底是由于窖泥生境的变化导致。Widden[18]认为,土壤生境的变化很复杂,其中包括生物因素和非生物因素,非生物因子如Ca2+、温度、湿度、K+、窖泥的形成过程等,可能是少数或几个主要真菌种出现的首要调节者。但有更多的证据证实生物因子有可能是主要的调节者,Gochenaur[19]研究后得出结论:窖泥中微生物种间竞争,将会导致微生态群落发生演替,适者生存。窖泥变质后产甲烷袋菌属及产甲烷杆菌属更显示出对环境的适应性。
已有研究表明[20],古菌发酵有促进己酸发酵进程多产己酸的作用,从而增加己酸乙酯含量,提高酒质。随着酿造周期的延长,窖泥中的古菌经历了一个长期驯化、变化的过程,古菌的群落结构会随着窖泥生境的改变而发生改变。甲烷菌作为窖泥古菌域的优势菌,其以乙酸、H2、CO2作为营养,这几种物质窖泥中很丰富,因此,甲烷菌群落结构变化与营养关系不大。许宝孝等[21]研究认为,甲烷短杆菌生长和产甲烷的最适温度为40℃,最适pH值为7.6。窖泥在变质后pH值小于5.5,抑制甲烷短杆菌的生长,而产甲烷袋状菌和产甲烷杆菌则表现出适应这种酸性环境。
2.4窖泥变质前后古菌分类树
使用MEGAN软件,通过交互式搜集NCBI中的分类数据库信息,以树状图形式表现窖泥变质前后古菌群落结构(图3、图4)。
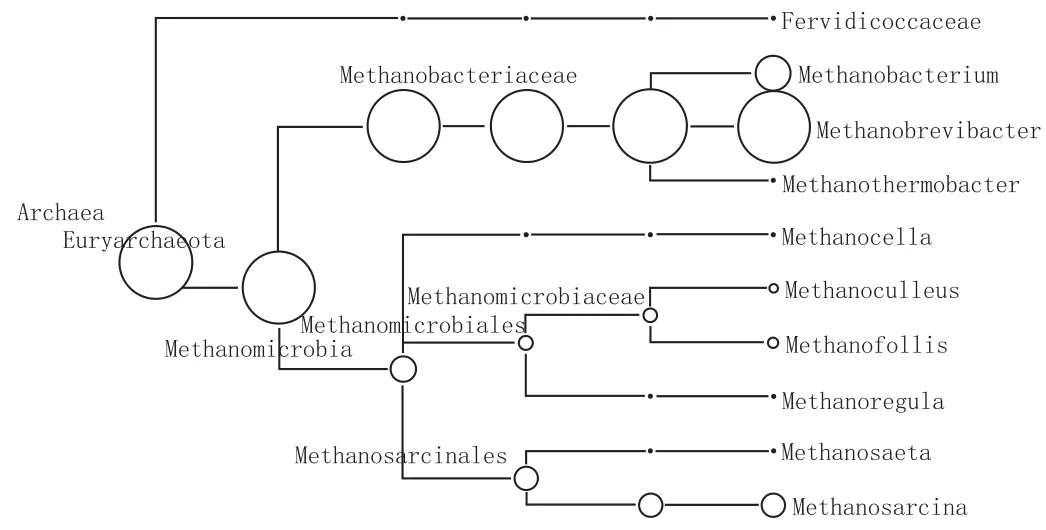
图3 HQ1属水平MEGANF丰度图
如图3所示,窖泥变质前古菌群落有广古菌门(Euryarchaeota)和泉古菌门(Crenarchaeota)的各级菌属,主要是甲烷菌。甲烷菌在自然界中分布很广,如人类消化系统、水稻田、湖泊、湿地污泥等[22]。目前大多数产甲烷古菌依然没有被分离,关于这些菌在窖泥中的生理、微生态之间的关系不得而知。甲烷菌在厌氧条件下合成甲烷,形成维持细胞生存所需的能量,合成甲烷的底物有3种:乙酸占60%以上、H2和CO2占30%,甲基化合物占10%,这些底物在窖泥中很丰富,因此窖泥中还有数量众多、种类多样的甲烷菌。
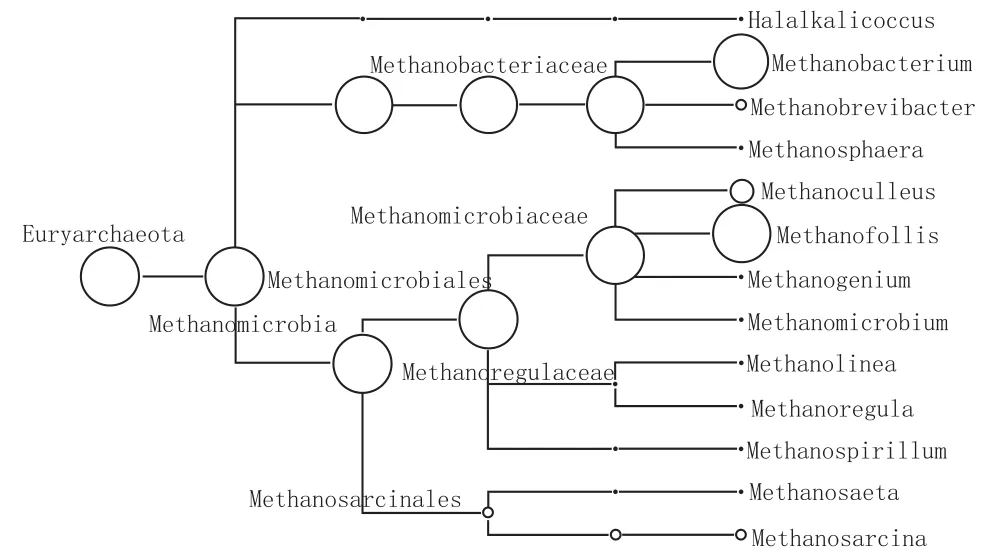
图4 HU1属水平MEGANF丰度图
如图4所示,窖泥变质后古菌群落主要由广古菌门(Euryarchaeota)构成,甲烷菌依然是优势菌属,只是种属间丰度发生了较大的变化。由于古菌独特的代谢方式和极强的环境适应能力,使得同一地域的古菌群落种属多样性差异较小,但由于温度、pH值、C/N等因素的影响,种属之间丰度差异比较显著。窖泥变质前后甲烷微球菌目(Methanomicrobiales)的含量分别为1.78%和45.15%,说明甲烷微球菌目更适应窖泥变质后的环境。Brauer等研究表明,甲烷微球菌目的一个种属最佳生长pH值在4.5~5之间[23],而窖泥变质后的pH值也在这个区间,由此可以得知,pH值可能是引起甲烷菌群落结构变化的主因。
3 讨论
窖泥被许多酒厂视为具有独特风格和酒质的基础,对窖泥功能菌的研究有助于认识窖泥微生物的代谢过程。窖泥变质后,微生态群落发生变化,酒质下降,给酒厂带来巨大损失。古菌域的产甲烷菌与己酸菌互利共生,对发酵浓香型白酒具有重要的作用[24]。因此,了解窖泥变质前后古菌群落差异,对于研究窖泥变质有重要意义。由于传统培养技术和显微镜观察方法的局限性,对窖泥微生物区系古菌群落组成研究还远远不够。采用Illumina Miseq测序平台的高通量DNA测序技术,对窖泥变质前后古菌群落组成、结构、动态方面有了更深入的了解,为研究窖泥功能古菌及窖泥变质的古菌影响提供有利支持。
通过16S rDNA序列分析表明,窖泥变质前后16S rDNA扩增序列为11703条和52876条,聚类结果为783个OTUs和1218个OTUs,说明古菌的种类和丰度在窖泥变质后均有较大的提高。窖泥的物理环境条件对古菌群落产生一定的影响(如土壤质地、pH值、C/N、光照、湿度、温度等),它们或是单个因子或是多个因子共同起作用影响着窖泥古菌的群落组成[25]。从扩增序列和聚类OUT数量分析,窖泥变质后生境更适合一些古菌的生长。
采用RDP classifier软件对16S rDNA序列分析得知,窖泥变质前优势菌属甲烷短杆菌属(35.66%)和甲烷杆菌属(10.25%),窖泥变质后优势菌属为产甲烷袋菌属(38.18%)和产甲烷杆菌属(34.21%)。产甲烷袋菌属和产甲烷杆菌属是否在窖泥变质过程中起到直接或间接作用,有待于认识窖泥微生物全貌后再作综合分析。
浓香型白酒窖泥微生物的研究有很高的理论价值和应用价值。通过高通量DNA测序技术可以快速直观地得到窖泥古菌的多样性并且获得大量未知种类,为我们了解窖泥古菌群落组成提供技术保障。然而,要真正了解窖泥古菌群落及其生理生化特性、形态结构必须将传统培养方法和现代分子生物学方法结合起来。
[1]Widden P.Fungal communities in soils along an elevationgradient in northern England[J].Mycologia,1987,79:298-309.
[2]陈宪仪.生物与中国酿造文化[J].中国酿造,2001(3):33-35.
[3]邓依,唐云容,张文学.16S-23S rRNAITS-AFLP指纹图谱分析在白酒窖泥原核微生物多样性分析中的应用[J].酿酒科技,2010(3):46-48.
[4]罗惠波,甄攀,黄治国.浓香型白酒窖池细菌群落[J].微生物学通报,2010,37(11):1621-1627.
[5]吴三多,赖凳燡,温宽和,等.北方地区窖泥退化原因及管理养护的研究[J].酿酒科技,2014(9):71-74.
[6]吴衍庸,卢世珩.利用窖泥甲烷菌与己酸菌二元发酵技术提高泸型曲酒质量的研究[J].酿酒科技,2011(5):38-41.
[7]Amann R I,Ludwig W,Schleifer K H.Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J].Microbiol Rev,1995,59(1):143-169.
[8]易彬,任道群,唐玉明,等.不同窖龄窖泥微生态变化研究[J].酿酒科技,2011(6):32-34.
[9]陶勇,芮俊鹏,李家宝,等.浓香型白酒窖泥中细菌和古菌的组成与多样性[J].化工学报,2014,65(5):1800-1804.
[10]Lauber C L,Hamady M,Knight R,et al.Pyrosequencingbased assessment of soil pH as a predictor of soil bacterial community structure at the continental scale[J].Appl Environ Microbio,2009,75(15):5111-5120.
[11]曾祥勇,董雅舒,胡贝,等.不同年份窖泥细菌16S rDNA系统发育分析[J].四川大学学报,2014(9):71-74.
[12]黄永光,黄平,涂华彬.窖泥微生物总DNA的提取纯化研究[J].酿酒科技,2004(3):41-42.
[13]Lee S H,Kang H J,Lee Y H,et al.Monitoring bacterial community structure and variability in time scale in full-scale anaerobic digesters[J].Environ Monit,2012,14(7):1893-1905.
[14]YannarellAC,Triplett E W.Geographic and environmental sources of variation in lake bacterial community composition [J].Applied and Environmental Microbiology,2005,71(1):227-239.
[15]Sun Y Z,Mao S Y,Zhu WY.Rumen chemical and bacterial changes during stepwise adaptation to a high-concentrate diet in goats[J].Animal,2010,4(2):210-217.
[16]Good I J.The population frequencies of species and the estimation of population parameters[J].Biometrika,1953,40:237-264.
[17]Thomton R H.Fungi occurring in mixed oakwood and heath soil profiles[J].Trans Brit Mycol Soc,1956,39:484--494.
[18]Gochenaur S E.Fungi of a Long Island oak-birch forest II. Population dynamics and hydrolase patterns for the soil penicillia[J].Mycologia,1984,76:218-231.
[19]吴衍庸.论甲烷发酵在传统酿制泸型曲酒中的特殊意义[J].酿酒科技,2000(5):33-34.
[20]许宝孝,奚明权,金秀其.甲烷短杆菌属HX菌株的分离和特性[J].微生物学报,1985,25(4):283-288.
[21]朱晨光,许政暟,宋任涛.产甲烷古菌[J].生命的化学,2009,29 (1):129-133.
[22]Brauer S L,Cadillo-Quiroz H,Yashiro E,et al.Isolation of a novel acidiphilic methanogen from an acidic peat bog[J]. Nature,2006,442:192-194.
[23]罗青春,刘超兰.不同年份窖泥中主要产甲烷菌的荧光定量PCR研究[J].酿酒科技,2013(12):17-19.
[24]Kobayashi T,Yan F,Takahashi S,et al.Effect of starch addition on the biological conversion and microbial community in a methanol-fed UASB reactor during long-term continuous operation[J].Bioresource technol,2011,102:7713-7719.
Analysis of the Difference in Archaea Communities in Pit Mud of Nongxiang Baijiu before and after Mud Deterioration
YU Chuntao1,XIANG Ming1,WANG Lei1,XU Zhenglin1and LIU Zhenjiang2
(1.Cangzhou Medical College,Cangzhou,Hebei 061001;2.Nikeng Distillery Co.Ltd.,Shijiazhuang,Hebei 054000,China)
Pit mud is the lifeline of Nongxiang Baijiu and its deterioration might bring huge loss for distilleries.The structure of archaea communities in pit mud before and after mud deterioration was analyzed by Illumina Miseq high throughput sequencing DNA technology.By constructing 16S rDNA gene bank of archaea,the results showed that there were 52876 sequences amplified in pit mud after the deterioration,11703 sequences had amplified in pit mud before the deterioration,and the abundance of archaea increased evidently.The clustering,sequencing and analysis of the sequence suggested that the dominant archaea before mud deterioration included Methanobrevibacter(35.66%),Methanobacterium(10.25%),Paenibacillus(4.27%),Bacillus(3.39%),Methanosarcina(3.27%),etc;and the dominant archaea after mud deterioration included Methanofollis(38.18%),Methanobacterium(34.21%),Methanoculleus(6.90%),etc.The results revealed that methane-producing bacteria was the dominant bacteria of archaea before and after mud deterioration.This study provided scientific evidence for pit mud culture/ maintenance and further research on pit mud microbes.
pit mud;deterioration;archaea community;analysis of the difference;high throughput sequencing;Baijiu
TS262.3;TS261.4;TS261.1;Q93-3
A
1001-9286(2016)08-0060-05
10.13746/j.njkj.2016114
2016-04-06
于春涛(1979-),男,研究生,研究方向为微生物发酵,E-mail:aimiao818@163.com。
优先数字出版时间:2016-05-26;地址:http://www.cnki.net/kcms/detail/52.1051.TS.20160526.1007.003.html。

