中国野生葡萄叶绿体分离及叶绿体DNA提取的研究
谢海坤,焦 健,樊秀彩,张 颖,姜建福,孙海生,刘崇怀
(中国农业科学院郑州果树研究所,郑州450009)
中国野生葡萄叶绿体分离及叶绿体DNA提取的研究
谢海坤,焦健,樊秀彩,张颖,姜建福,孙海生,刘崇怀*
(中国农业科学院郑州果树研究所,郑州450009)
以中国野生刺葡萄、山葡萄、桑叶葡萄和东南葡萄的成熟叶片为材料,比较柱式植物叶绿体DNAout试剂盒和改良的高盐-低pH法分离叶绿体及提取cpDNA效果。结果显示:(1)2种方法均分离得到了中国野生葡萄的叶绿体,但与柱式植物叶绿体DNAout试剂盒相比,改良的高盐-低pH法得到的叶绿体浓度高、杂质少,更适合中国野生葡萄叶绿体分离。(2)柱式植物叶绿体DNAout试剂盒提取的cpDNA,其OD260/OD280在1.28~1.36之间,质量浓度为4.2~7.8 ng·μL-1,质量低,不能满足后续测序要求;而改良的高盐-低pH法提取的cpDNA,其OD260/OD280值在1.84~1.90之间,质量浓度为2 514.4~4 133.7 ng·μL-1,质量高,纯度好,能满足后续测序要求。研究表明,改良的高盐-低pH法能简便、快速获得中国野生葡萄完整叶绿体及高质量cpDNA,并且提取的cpDNA可满足后续测序要求,为葡萄属植物叶绿体基因组的深入研究奠定了基础。
中国野生葡萄;叶绿体分离;cpDNA提取;柱式植物叶绿体DNAout;改良的高盐-低pH法
叶绿体是植物进行光合作用的重要细胞器,具有相对独立的遗传物质(cpDNA)。cpDNA一般长120 160 kb[1],多数是共价闭合双链分子,少数为线状分子。叶绿体基因组由4部分组成,大单拷贝区(LSC)、小单拷贝区(SSC)和2个反向重复序列(IRs)。叶绿体基因组序列高度保守,且属于母系遗传,后代遗传稳定,已在遗传结构分析[2]、分子标记[3]、核质互作和叶绿体基因工程[4]、起源演化[5]等领域开展了广泛研究。
常用的植物叶绿体的分离及cpDNA提取的方法主要有DNaseI法[6]、无水法[7]、蔗糖密度梯度离心法[8]、Percoll密度梯度离心法[9]、氯化铯密度梯度离心法[10]和改良的高盐-低pH法[11]。DNaseI法难以使叶绿体膜保持完整,导致cpDNA的得率很低[11]。无水法的报道相对较少,目前只应用在C4植物黍子[12]等植物叶绿体的分离上。传统的密度梯度离心法主要用于提取豆科和禾本科植物的cpDNA,其成本高、耗时长、产率低,对设备要求也较高,不宜广泛应用[13]。而改良的高盐-低pH法,可以简单、快速获得完整叶绿体和高质量cpDNA,目前已成功在杨树[14]、雷蒙德氏棉[15]、小麦[16]、茶树[17]、枣[18]、割手密[19]等植物中得到验证。
野生葡萄叶片内富含色素和单宁类物质,在采用常规的植物叶绿体分离及cpDNA提取方法时,会产生大量泡沫或沉淀,影响分离及提取效果。1994年,吴俊辉等[20]采用蔗糖密度梯度离心法,对几种中国野生葡萄的叶绿体进行了研究,克服了色素和单宁等对叶绿体分离的影响,成功提取和纯化了叶绿体及其DNA。但是,此方法耗时长、操作繁琐、对设备要求也高,不易广泛应用。而改良的高盐-低pH法[11]结合了蔗糖密度梯度离心法、DNaseI法和盐沉淀法的优点,结果表明,它可以简便、快速地获得完整叶绿体及高质量cpDNA,已广泛应用在其他被子植物中。本研究将此方法应用于中国野生葡萄叶绿体分离及cpDNA提取,同时与柱式植物叶绿体DNAout试剂盒提取方法比较,探寻一个简便、快速获得中国野生葡萄完整叶绿体及高质量cpDNA的方法,为葡萄属植物叶绿体的分离及其DNA的提取提供方法指导,这对葡萄属植物叶绿体基因组的深入研究有重大意义。
1 材料和方法
1.1实验材料
中国野生刺葡萄(Vitisdavidii)、山葡萄(V.amurensis)、桑叶葡萄(V.heyneana)和东南葡萄(V.chunganensis)的成熟叶片采自中国农业科学院郑州果树研究所国家果树种质,郑州葡萄圃。2015年5月摘取成熟健康的葡萄叶片,带回实验室,经蒸馏水洗净,用滤纸吸干水分,并用湿润的纱布包裹。处理后的叶片于4 ℃冰箱黑暗处饥饿处理24 h,然后液氮速冻处理,存于-80 ℃超低温冰箱中保存备用。
1.2方法
1.2.1叶绿体的分离和cpDNA提取柱式植物叶绿体DNAout试剂盒(CAT#: 120406-15)购自北京天恩泽基因科技有限公司,所有步骤均按照此试剂盒的使用说明书操作。
改良高盐-低pH法参考Shi等[11]的方法,略调整。所有操作均在冰上进行,所用玻璃器皿要事先预冷。
叶绿体的分离步骤如下:
(1)匀浆:取饥饿处理的中国野生葡萄叶片20 g,去粗叶脉,剪成1 cm长短,装入预冷的九阳料理机(JYL-C012)中,迅速加入预冷的Buffer A[(1.25 mol/L NaCl,0.25 mol/L抗坏血酸,10 mmol/L焦亚硫酸钠,0.012 5 mol/L 硼砂,50mmol/L Tris-HCl (pH 8.0),7 mmol/L Na2EDTA(pH 8.0),1% PVP-K40即聚乙烯吡咯烷酮(w/v),0.1%BSA即牛血清蛋白(w/v),1 mmol/L DTT即二硫苏糖醇(BSA和DTT现用现加,pH 3.8)]400 mL,匀浆过程尽量避免发热,先低速匀浆2次,每次5 s,后高速匀浆57次,每次10 s,最终使材料匀浆完全。
(2)过滤:用200目单层尼龙网过滤匀浆液至预冷的500 mL量筒中,再等分到6个预冷的50 mL塑料离心管中,静置2 min。
(3)弃细胞残骸:4 ℃、180 g(原文献中200 g),离心20 min,取上清;重复离心1次。
(4)粗提:4 ℃、3 070 g(原文献中3 500 g),离心20 min,弃上清。
(5)纯化:每管沉淀中加入42 mL预冷的Buffer B[1.25 mol/L NaCl,0.012 5 mol/L硼砂,1% PVP-K40(w/v),50 mmol/L Tris-HCl (pH 8.0),25 mmol/L Na2EDTA (pH 8.0),0.1%BSA(w/v),1mmol/L DTT(BSA和DTT现用现加),pH 8.0],用大小适中的无菌软毛刷轻轻洗刷,使沉淀充分悬浮;4 ℃、3 070 g(原文献中3 500 g),离心20 min,弃上清。
(6)再纯化:再次向每管沉淀中加入42 mL Buffer B,用无菌软毛刷使沉淀轻轻悬浮,4 ℃、3 300 g,离心20 min,弃上清。管中即为所得叶绿体沉淀,此步骤是新增的。
(7)重复上步操作1次。随后,用显微镜观察叶绿体的完整性。
叶绿体的裂解和cpDNA提取步骤如下:在6管纯化的叶绿体沉淀中均加入2 mL Buffer B,并合为一管,在4 ℃、3 300 g(原文献中3 750 g),离心20 min,弃上清。再向叶绿体沉淀中加入8 mL Buffer C[100 mmol/L NaCl,100 mmol/L Tris-HCl (pH 8.0),50 mmol/L Na2EDTA,1 mmol/L DTT(DTT现用现加)]、1.5 mL 20% SDS、20 μL β-巯基乙醇、30 μL蛋白酶K(10 mg/mL),充分混匀,55 ℃水浴锅中培养过夜,使叶绿体充分裂解,释放出cpDNA。
裂解结束后,将裂解液冰浴5 min,后加入1.5 mL KAc(pH 5.7),颠倒混匀,再冰浴30 min;4℃、5 260 g(原文献中10 000 g),离心15 min,用移液枪吸上清。加入与上清等体积的酚/氯仿/异戊醇(25∶24∶1)抽提2次,每次需充分颠倒混匀,4 ℃、5 260 g(原文献中10 000 g),离心20 min。离心后的液体包括上层水相、中层变性蛋白和下层的有机溶剂。小心移取上清液至新的50 mL离心管中,加入等体积预冷的异丙醇,轻轻颠倒混匀,-20 ℃过夜沉淀cpDNA。
4 ℃、5 260 g(原文献中10 000 g),离心20 min,弃上清,得到cpDNA。用弯钩针头将沉淀挑出,置于2 mL离心管中。cpDNA用预冷的70%乙醇、无水乙醇洗涤。自然风干沉淀,加入50 μL 1×TE Buffer(pH 8.0),4 ℃溶解过夜,轻轻混匀后,即得到cpDNA原液。
加0.5 μL RNase A(去Dnase,10 mg/mL)处理cpDNA原液,37 ℃温育30 min,自然冷却后,得到cpDNA溶液,置于-20 ℃保存备用。
1.2.2叶绿体显微观察取2 mL Buffer B溶液加入提取的叶绿体沉淀中,制作临时装片,在尼康显微镜(ECLIPSE Ti-S/L100型)100×油镜下观察叶绿体的状况。
1.2.3中国野生葡萄cpDNA质量检测从提取的cpDNA溶液中取2 μL,在核酸蛋白检测仪(美国Thermo公司,ND-1000型)上测定核酸含量,用1×TE Buffer(pH 8.0)作空白对照,记录OD260/OD280、OD260/OD230及质量浓度。
1.2.4中国野生葡萄cpDNA电泳检测取2 μL cpDNA溶液,加2 μL稀释10×的荧光染料和1 μL 6×loading buffer(上样缓冲液)混匀,在0.7%(w/v)琼脂糖凝胶上电泳,电泳缓冲液为1×TAE,恒压126 V,恒流120 mA,电泳大约60 min后,在紫外凝胶成像分析仪(Carestream公司,GL212PRO型)下检测其条带情况,并拍照。
2 结果与分析
2.1中国野生葡萄叶绿体显微观察
采用柱式植物叶绿体DNAout(方法Ⅰ)和改良高盐-低pH法(方法Ⅱ),分别分离得到了刺葡萄、山葡萄、桑叶葡萄和东南葡萄的叶绿体(图1)。对比发现,方法Ⅱ得到的叶绿体浓度高、数量多、杂质少、背景清晰,更适宜中国野生葡萄叶绿体的分离。
2.2中国野生葡萄cpDNA质量检测
从中国野生葡萄cpDNA的OD260/OD280、OD260/OD230和质量浓度参数(表1)可以看出,2种方法得到的cpDNA各种指标差异显著。方法I提取的cpDNA质量浓度在4.2~7.8 ng·μL-1之间,OD260/OD280值在1.28~1.36之间,低于纯净DNA的比值1.8~2.0,而OD260/OD230值在0.44~0.67之间,低于污染物的评估值2.0,这表明样品中含有较多的蛋白质、酚类及多糖,cpDNA断裂严重,纯度很低,不能满足后续的建库测序要求。而方法Ⅱ提取的cpDNA,质量浓度高达2 514.4~4 133.7 ng·μL-1,OD260/OD280值在1.84~1.90之间,居纯净DNA的比值1.8~2.0之间,OD260/OD230值为在2.07~2.14之间,大于污染物的评估值2.0,说明多糖、蛋白质、碳水化合物的污染轻,可以满足建库测序要求。因此,方法Ⅱ提取的中国野生葡萄cpDNA的各种指标显著高于方法Ⅰ。由上述可知,改良的高盐-低pH方法所提取的cpDNA更能满足测序要求,此方法更适合葡萄属植物cpDNA提取。

图1 柱式植物叶绿体DNAout(Ⅰ)和改良高盐-低pH法(Ⅱ)分离几种野生葡萄叶绿体显微图片Fig. 1 Chloroplast micrographs of 4 grapes species seperated by column plant chloroplast DNAout(Ⅰ)and modified high-salt low-pH method(Ⅱ)
2.3中国野生葡萄cpDNA电泳检测
从琼脂糖凝胶电泳结果(图2)可以看出:方法I提取的cpDNA无条带,而方法Ⅱ提取的cpDNA主带清晰、明亮、整齐一致,无降解现象,且无RNA、多糖及其他次生物等杂质的干扰。对比可知,方法Ⅱ所得cpDNA的质量显著好于方法Ⅰ,与核酸蛋白检测仪分析结果相一致。
3 讨 论
高质量cpDNA是叶绿体基因组测序的基础。相对于全基因组DNA的提取,cpDNA的提取难度更大,因为叶绿体的分离较为困难,并且存在核DNA和线粒体DNA的污染。要提取高质量的cpDNA,首先要分离得到完整的叶绿体,并进行纯化。此外,叶绿体的裂解和cpDNA 的提取也需要精细操作。

表1 2种方法提取的cpDNA紫外分析结果
注:Ⅰ、Ⅱ分别对应柱式植物叶绿体DNAout和改良的高盐-低pH方法
Note: Ⅰ and Ⅱ stand for the column plant chloroplast DNAout and the modified high-salt low-pH method
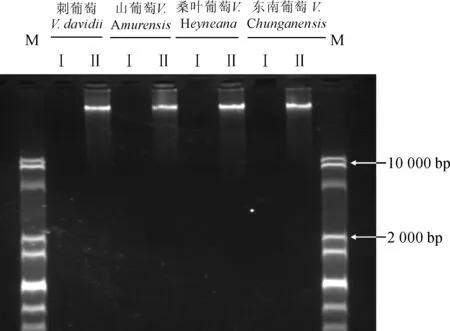
图2 柱式植物叶绿体DNAout(Ⅰ)和高盐-低pH法(Ⅱ)提取几种葡萄cpDNA电泳图谱Fig. 2 Electrophoresis pattern of cpDNA of 4 grapesextracted by column plant chloroplast DNAout(Ⅰ)and modified high-salt low-pH method(Ⅱ)
目前市场上还没有针对分离葡萄叶绿体及提取其DNA的试剂盒,本实验所用的柱式植物叶绿体DNAout试剂盒主要是针对大豆、菠菜、烟草等一年生草本植物。从提取结果上看柱式植物叶绿体DNAout试剂盒不适合中国野生葡萄叶绿体的分离和cpDNA的提取,而改良的高盐-低pH法却能简便、
快速获得中国野生葡萄完整叶绿体及高质量叶绿体cpDNA。这可能是因为改良的高盐-低pH法的缓冲液中,不仅含有大量的NaCl,使细胞和叶绿体膜有充足的韧性经受离心、洗涤等操作,而且缓冲液中的维生素C、焦亚硫酸钠、BSA和DTT等还原剂能抑制溶液中的游离氧化单宁和其它次生物质呈显色反应,从而维持匀浆液最初的颜色[20]。此缓冲液还能抑制核DNA与叶绿体之间的吸附,从而减少核DNA的污染[21]。此外,采摘叶片后的黑暗饥饿处理,充分消耗了叶片中的淀粉,减少了其对叶绿体分离和cpDNA提取的干扰[18]。
本研究在改良的高盐-低pH法的基础上,对离心转速和相关步骤略微调整,成功分离出中国野生刺葡萄、山葡萄、桑叶葡萄和东南葡萄的完整叶绿体,并提取得到高质量cpDNA。改良的高盐-低pH法分离出中国野生葡萄的叶绿体及提取的cpDNA显著好于柱式植物叶绿体DNAout试剂盒所分离的叶绿体及提取的cpDNA,其OD260/OD280值在1.84~1.90之间,质量浓度为2 514.4~4 133.7 ng·μL-1,质量高,纯度好,RNA消化完全,无降解现象,能满足后续测序要求,为葡萄属植物叶绿体基因组的深入研究奠定了基础。
[1]PALMER J D. Comparative organization of chloroplast genomes [J].AnnualReviewofGenetics, 1985,19(1):325-354.
[2]陈涛,王小蓉,罗华,等.9个野生中国樱桃群体叶绿体DNAtrnQ-rps16序列变异及其遗传结构分析[J].遗传,2012,34(11):1 475-1 483.
CHEN T, WANG X R, LUO H,etal. Chloroplast DNAtrnQ-rps16 variation and genetic structure of nine wild Chinese cherry (CerasuspseudocerasusLindl.) populations [J].Hereditas, 2012,34(11): 1 475-1 483.
[3]TOMOTARO N, VAUGHAN DA, KOH-LCHI K. Phylogenetic analysis ofOryzaspecies, based on simple sequence repeats and their flanking nucleotide sequences from the mitochondrial and chloroplast genomes [J].TheoreticalandAppliedGenetics, 2005,110(4): 696-705.
[4]LELIVELT C L C, MCCABE M S, NEWELL C A,etal. Stable plastid transformation in lettuce (LactucasativaL.) [J].PlantMolecularBiology, 2005,58(6): 763-774.
[5]XU D, ABE J, GAI J,etal. Diversity of chloroplast DNA SSRs in wild and cultivated soybeans: evidence for multiple origins of cultivated soybean [J].TheoreticalandAppliedGenetics, 2002,105(5): 645-653.
[6]HALLIWELL B. Methods in Chloroplast Molecular Biology [M]. Elsevier Biomedical Press, 1982,158(1): 190-191.
[7]WU N H, COTE J C, WU R. Structure of the chloroplastpsbAgene encoding the QB protein fromOryzasativaL. [J].DevelopmentalGenetics, 1987,8(8): 339-350.
[8]HIRAI A, ISHIBASHI T, MORIKAMI A,etal. Rice chloroplast DNA: a physical map and the location of the genes for the large subunit of ribulose 1, 5-bisphosphate carboxylase and the 32 KD photosystem II reaction center protein [J].TheoreticalandAppliedGenetics, 1985,70(2): 117-122.
[9]HEINHORST S, GANNON G C, GALUN E,etal. Clone bank and physical and genetic map of potato chloroplast DNA [J].TheoreticalandAppliedGenetics, 1988,75(75): 244-251.
[10]胡华萃,王进,沈桂芳,等.氯化铯密度梯度离心法纯化叶绿体DNA[J].浙江大学学报(理学版),1985,12(3):370-372.
HU H C, WANG J, SHEN G F,etal. Purification of chloroplast DNA by the method of CsCl-Density gradient centrifugation [J].JournalofZhejiangUniversity, 1985,12(3): 370-372.
[11]SHI C, HU N, HUANG H. An improved chloroplast DNA extraction procedure for whole plastid genome sequencing [J].PLOSOne, 2012,7(2): e31468.
[12]马建忠,娄世庆,匡廷云,等.黍子叶绿体中存在质粒状DNA[J].自然杂志,1992,(7):556-557.
MA J Z, LOU S Q, KUANG T Y,etal. Plastid DNA in broomcorn millet chloroplast [J].ChineseJournalofNature, 1992,(7):556-557.
[13]盖树鹏,孟祥栋.简易、快速提取大葱叶绿体、线粒体DNA的方法[J].农业生物技术学报,2000,8(3):262-262.
GAI S P, MENG X D. A simple and rapid method for the isolation of chloroplast and mitochondrial DNA from welsh onion (AlliumfistulosumL.) [J].JournalofAgriculturalBiotechnology, 2000,8(3): 262-262.
[14]崔彬彬,李云,冯大领,等.杨树叶绿体分离及叶绿体DNA提取方法研究[J].保定师范专科学校学报,2006,19(2):25-27.
CUI B B, LI Y, FENG D L,etal. A study for isolation ofPopuluschloroplast and cpDNA [J].JournalofBaodingTeachersCollege, 2006,19(2): 25-27.
[15]李鹏波,薛龙飞,王彦霞,等.雷蒙德氏棉叶绿体基因组Fosmid文库构建[J].棉花学报,2011,23(1):10-14.
LI P B, XUE L F, WANG Y X,etal. Construction of a fosmid library of chloroplast genome inGossypiumraimondii[J].CottonScience, 2011,23(1): 10-14.
[16]侯典云,马占强,徐虹,等.小麦叶绿体DNA提取方法研究[J].河南农业科学,2011,40(10):38-40.
HOU D Y, MA Z Q, XU H,etal. Study on extraction method of wheat chloroplast DNA [J].JournalofHenanAgriculturalSciences, 2011,20(10): 38-40.
[17]陈春梅,陈亮.茶树叶绿体DNA 提取方法研究[J].分子植物育种,2014,12(3):562-566.
CHEN C M, CHEN L. Extraction method of chloroplast DNA of tea plant (Camelliasinensis) [J].MolecularPlantBreeding, 2014,12(3): 562-566.
[18]杨晓婷,黄建,张春梅,等.枣叶绿体基因组DNA提取方法研究[J].西北林学院学报,2015,30(2):105-110.
YANG X T, HUANG J, ZHANG C M,etal. An optimized chloroplast DNA extraction protocol for Chinese Jujube [J].JournalofNorthwestForestryUniversity, 2015,30(2): 105-110.
[19]田春艳,李富生,何丽莲,等.割手密叶绿体DNA的高效提取方法[J].分子植物育种,2016,14(1):86-91.
TIAN C Y, LI F S, HE L L,etal. An efficient method for isolation of chloroplast DNA inSaccharumspontaneum[J].MolecularPlantBreeding, 2016,14(1): 86-91.
[20]吴俊辉,舒煦,李朝銮.中国葡萄属植物叶绿体DNA的提取、纯化及分子量测定[J].植物分类与资源学报,1994,(2):178-186.
WU J H, SHU X, LI C L. Isolation, purification and measure of molecular weight of cpDNA fromVitisspecies in Chinese[J].ActaBotanicaYunnanica, 1994,(2): 178-186.
[21]刘少林,靖深蓉,郭立平,等.用改进的高盐低pH法分离和纯化棉花叶绿体及叶绿体DNA[J].棉花学报,1997,9(5):239-241.
LIU S L, JING S R, GUO L P,etal. A preliminary study on genetic diversity of upland cotton cultivars in China [J].CottonScience, 1997,9(5): 239-241.
(编辑:宋亚珍)
An Optimized Chloroplast Isolation and Chloroplast DNA Extraction Protocol for Chinese Wild Grapes
XIE Haikun, JIAO Jian, FAN Xiucai, ZHANG Ying, JIANG Jianfu,SUN Haisheng, LIU Chonghuai*
(Zhengzhou Fruit Research Institute, Chinese Academy of Agricultural Sciences, Zhengzhou 450009, China)
Mature leaves collected fromVitisdavidii,V.amurensis,V.heyneanaandV.chunganensiswere used for chloroplast isolation and cpDNA extraction in this study. The two methods were the column plant chloroplast DNAout and modified high-salt low-pH method, and the results were compared with each other. (1) Both methods had separated the chloroplast of Chinese wild grapes, but the modified high-salt low-pH method obtained higher concentration and less impurity of chloroplast than that of column plant chloroplast DNAout. So the modified high-salt low-pH method was more suitable for chloroplast isolation. (2) The value of OD260/OD280of cpDNA extracted by the column plant chloroplast DNAout was between 1.28 and 1.36, and the concentration was between 4.2 ng·μL-1and 7.8 ng·μL-1, which did not meet the demand of subsequent chloroplast genome sequencing. In contrast, the value of OD260/OD280of cpDNA extracted by the modified high-salt low-pH method was between 1.84 and 1.90 and the concentration was between 2 514.4 ng·μL-1and 4 133.7 ng·μL-1, so the cpDNA extracted in this way was extremely high-quality and pure. As a result, the cpDNA extracted by the modified high-salt low-pH method meet the demand of subsequent chloroplast genome sequencing. As a conclusion, the modified high-salt low-pH method isolated intact chloroplast and extract high-quality cpDNA of Chinese wild grapes simply and quickly. And the cpDNA meet the demand of subsequent chloroplast genome sequencing. It was also a critical step to make further research of chloroplast genomes ofVitisL.
Chinese wild grapes; chloroplast isolation; cpDNA extraction; column plant chloroplast DNAout; modified high-salt low-pH method
1000-4025(2016)07-1464-06
10.7606/j.issn.1000-4025.2016.07.1464
2016-04-19;修改稿收到日期:2016-06-04
现代农业产业技术体系建设专项资金(CARS-30-yz-1);中国农业科学院科技创新工程专项(CAAS-ASTIP-2015-ZFRI);农业部物种保护项目(2130135-34)
谢海坤(1989-),女, 在读硕士生,主要从事葡萄种质资源研究。E-mail:1379226793@qq.com
刘崇怀,研究员,博士生导师,主要从事葡萄种质资源研究。E-mail: liuchonghuai@caas.cn
Q781; S663.1
A

