拟南芥抗盐突变体的筛选
李超群,王文静,葛晓春
(1. 复旦大学 生命科学学院 上海 200438;2. 复旦大学 遗传工程国家重点实验室,上海200438)
拟南芥抗盐突变体的筛选
李超群1,2,王文静1,2,葛晓春1,2
(1. 复旦大学 生命科学学院 上海 200438;2. 复旦大学 遗传工程国家重点实验室,上海200438)
盐胁迫是影响植物生长发育的非生物胁迫因素之一,目前发现的盐胁迫信号途径的基因还非常有限.发掘新的耐盐相关基因对于了解植物的盐胁迫响应机制和改善作物的耐盐性具有非常重要的意义.从EMS突变体库中筛选突变体是一种经典的正向遗传学方法,有可能筛选到从T-DNA插入突变体库中无法发现的新基因.本实验用160mmol/L NaCl作为盐胁迫的筛选条件,以在含盐平板上长出绿色子叶作为筛选指标筛选抗盐(Salt Resistant, ST)的EMS突变体,初步筛选到了140株ST突变体,其中84株收到了种子,第二代进行抗盐表型确定后,得到了15株表型稳定的ST突变体,均表现出明显的抗盐表型,并对其中的一些突变体做了初步的表型及基因定位分析.
拟南芥; EMS诱变; 抗盐筛选; ST突变体
盐胁迫是抑制植物生长、降低农作物粮食产量的非生物胁迫因素之一[1].海水倒灌、降雨量减少、土壤荒漠化等环境因素使得全球土地盐渍化程度日益严重,全球范围内大约有20%的灌溉土地受到不同程度盐碱化的影响[2].盐胁迫几乎影响了植物所有的重要生命过程,如生长发育、光合作用、蛋白质合成、能量和脂类代谢等[3],因此研究植物如何在盐胁迫下生存具有非常重要的现实意义.
植物在受到盐胁迫后,会伴随出现离子胁迫、渗透胁迫以及氧化胁迫等.盐胁迫破坏了植物体内的离子平衡,导致细胞质中有毒性的Na+积累和必需离子(如K+)的减少[4].盐胁迫下,植物细胞通过多种Na+转运载体限制Na+流入和促进Na+排出细胞,或者将Na+贮存在液泡中.
SOS(Salt Overly Sensitivity)途径是植物应对盐胁迫信号途径中非常重要的一条通路,这条途径中的主要基因都是由筛选盐敏感突变体发现的,包括SOS1, SOS2, SOS3, AKT1, HKT1等.SOS1是一种细胞质膜上的Na+/H+反向转运蛋白(Na+/H+antiporter)[5-6],可以将细胞质中的 Na+外排出去;SOS2是一种Ser/Thr蛋白激酶,其C末端是调节结构域,N末端是催化结构域[7],它可结合SOS3,通过磷酸化调控SOS1的转运活性;SOS3是一种钙离子结合蛋白,能够特异地感知盐胁迫诱导的钙离子信号[8-9],与SOS2形成复合体,传递盐胁迫信号.当植物受到盐胁迫时,细胞内的钙离子浓度升高,SOS3结合钙离子使SOS2-SOS3蛋白激酶复合体磷酸化,激活SOS1的活性,从而将Na+运出细胞.SOS2-SOS3蛋白激酶复合体还能下调高亲和性钾离子转运蛋白(HKT1)的活性,限制Na+流入细胞[10-13].因此,植物主要通过SOS途径调控离子平衡.
盐胁迫引起的渗透胁迫途径主要包括了ABA依赖和ABA非依赖途径.这些途径中的基因也主要通过对突变体表型分析获得.NAC(HD-ZIP)和DREB2转录因子主要参与ABA非依赖信号途径;而ABRE/ABF(bZIP),NAC(RD26),MYB2和MYC2转录因子主要参与ABA依赖信号途径.另外,磷脂信号途径和蛋白激酶信号途径也会参与到调节渗透胁迫的过程中,磷脂酶C(PLC)和D(PLD)水解膜磷脂产生三磷酸肌醇(IP3)、二酰基甘油(DAG)和磷脂酸(PA)等第二信使,参与渗透平衡的调节;钙离子依赖的蛋白激酶(CDPK)和MAP激酶通过蛋白激酶信号途径调节渗透平衡[14-16].
拟南芥的生活周期短、子代数量众多、基因组简单且基因组序列已进行了测定,这些优点使得它成为植物学研究的模式生物.化学诱变剂甲基磺酸乙脂(EMS)是一种烷化剂,可以诱发植物基因组发生点突变.与T-DNA插入突变不一样的是,它可能会产生一些基因功能局部受影响的弱突变体;而T-DNA插入突变则是由于T-DNA大段插入引起基因功能全部或局部丧失,或者表达水平严重下降引起的突变.EMS突变体中的局部氨基酸变化产生的表型,可以弥补T-DNA插入突变体表型研究的不足.用EMS处理拟南芥种子,构建突变体库是分离和研究基因功能非常有效的方法.
为了筛选抗盐途径相关的新基因,从而为植物抗盐机理提供更多信息,本研究利用EMS处理野生型拟南芥(Col-0)种子,构建了突变体库,然后以160mmol/L NaCl作为筛选浓度筛选抗盐突变体,以能长出绿色子叶作为筛选指标.我们筛选得到了15株表型稳定的抗盐突变体,并且将这些突变体与Ler的野生型进行了杂交,构建了图位克隆的作图群体.另外,对其中几个突变体做了遗传学分析,检测了突变基因的显隐性和是否属于单基因突变体.
1 材料和方法
1.1材料
拟南芥(Arabidopsisthaliana)野生型,Columbia-0(Col-0)生态型,Landsberg(Ler)生态型.
1.2方法
1.2.1实验植物材料的种植和生长
用1mL 70% 乙醇旋转灭菌拟南芥种子10min,然后换1mL 无水乙醇+0.01% Triton X-100,振荡几次后置于灭菌的干净滤纸上,待乙醇挥发后,点播或均匀撒在培养基上.将平板置于4℃冰箱中春化2d,转移至长日照(16h光照/8h黑暗)温室,(22±2)℃条件下进行培养.幼苗生长8~9d后移植到土壤中继续生长.
1.2.2EMS诱变
称重1万粒的野生型(Col-0)种子大约0.2g,置于50mL的离心管中用蒸馏水浸泡6h.离心管离心10min,吸出水(尽量不要吸出种子).向其中加入100mL 的100mmol/L的磷酸缓冲液(pH 7.5),并加入300μL的EMS,终浓度为0.3%(体积比),盖上管盖.将离心管置于摇床室温轻轻震荡12h.处理完后,离心吸除EMS,用蒸馏水清洗10~20次,每次5min.清洗完后,将种子倒入干净的离心管中,加入0.1% 琼脂糖悬浮种子,置于4℃低温处理2d.处理结束后,直接点种于土壤中,种30~40粒于每个小穴盘(勿太密,否则育性差).生长约2~3周后,观察土壤中有一些白化苗,说明诱变比较成功.
1.2.3候选突变体的筛选
为了筛选抗盐突变体,预实验中选择的NaCl浓度为160mmol/L,将M1代种子(种子诱变后长成的植株结的种子)以小组为单位均匀地撒种于含160mmol/L NaCl的1/2 MS培养基上,移入温室生长.观察每个培养基上长出来的有绿色子叶的幼苗,做上标记,10d后移植到土中,观察生长表型,单株收种.再将这些单株收的种子(M2)再次点在含有160mmol/L NaCl的1/2 MS培养基上,观察是否能重复M1代表型.
1.2.4拟南芥的杂交
取生长状况良好的拟南芥,从母本植物上挑选未开花的花苞(以白色花瓣刚刚显露为宜),于前一天晚上用尖头镊子去除雄蕊(4+2),注意尽量不要伤害到柱头.第二天10:00~14:00花开最旺盛时取父本的花粉,反复涂抹于母本的柱头上,确保花粉接触到母本的柱头,若柱头第二天变为紫色则说明授粉成功,等种子成熟即可采收,得到杂交F1代种子.F1可用于观察突变基因的显隐性,F2代可统计分离比,判断是否为单基因突变引起的表型.
得到单基因隐性突变体的纯合子后,将其与野生型(Col-0)回交,同时将准备进行图位克隆的候选个体与另一种生态型(Ler)拟南芥杂交,得到F1代种子后让其自交,从F2代群体中选取有表型的个体进行突变体基因定位.
1.2.5植物基因组DNA的提取
取2片叶片,加液氮研磨后加入400μL DNA抽提缓冲液(200mmol/L Tris pH 7.5,250mmol/L NaCl,25mmol/L EDTA,0.5% SDS),离心5min,取上清,加入等体积异丙醇颠倒振荡,静置后离心沉淀DNA,用75%酒精清洗沉淀2次,干燥后加100μL灭菌水溶解.
1.2.6粗定位
从突变体和Ler杂交得到的分离群体F2中,筛选有抗盐表型的株系,然后取其叶片,提取DNA,利用SSLP分子标记进行PCR扩增,根据表型与标记的连锁关系来分析突变基因的定位.本文中用的SSLP分子标记可以参见文献[17].
2 结果与分析
2.1筛选拟南芥抗盐突变体的盐浓度确定
据已有报道,拟南芥Col-0在75mmol/L NaCl的盐胁迫下表现出盐敏感表型,在100mmol/L NaCl或者更高的盐浓度胁迫下,幼苗生长受到严重抑制[18].通过比较不同盐浓度下拟南芥的生长表型,我们确定了抗盐突变体的筛选条件为 160mmol/L NaCl.将野生型(Col-0)和EMS突变体种子撒播在同一个1/2 MS培养基上,培养基中含160mmol/L NaCl,7d后幼苗的生长状况如 图1.由图1可见,野生型(Col-0)在含 160mmol/L NaCl的1/2 MS培养基上,大部分种子都可以萌发,但是不能长出绿色子叶,而抗盐的EMS突变体可以长出绿色子叶(圆圈标记).
2.2抗盐突变体的筛选
2.2.1初筛结果
将EMS突变体M1代种子分成7组,然后,把这些M1代的种子分批撒在含160mmol/L NaCl的 1/2 MS 培养基上,此时野生型子叶全部呈黄色,而抗盐突变体可以在盐平板上长出绿色子叶.通过这种方法,初步筛选得到了140株耐盐ST突变体,将这些突变体幼苗移到土里继续生长,最后有84株长大并且收获了突变体种子M2,将它们命名为ST-1~ST-84.
2.2.2表型重复的结果
将84个(ST-1~ST-84)抗盐突变体M2单株的种子即M3重新点播在含160mmol/L NaCl的1/2 MS培养基上,发现只有15个ST(ST-1,ST-2,ST-7,ST-10,ST-14,ST-25,ST-27,ST-47,ST-53,ST-58,ST-59,ST-60,ST-61,ST-68,ST-70)突变体具有稳定的抗盐的表型.
2.3抗盐突变体的抗盐表型
将经过重复筛选后得到的有稳定抗盐表型的突变体进行抗盐表型分析.首先,将这些突变体种子点播在含160mmol/L NaCl的1/2 MS培养基上,温室生长14d,然后拍照.本文选取了其中3个ST突变体(ST-1,ST-25和ST-70)为例说明它们的抗盐表型(图2,见第100页).
另外,我们还统计了这些突变体在盐平板上的鲜重,发现这些ST突变体的鲜重明显大于野生型(图3,见第100页).
在平板上直立生长ST-1和ST-2突变体,培养基中加入150mmol/L 或160mmol/L NaCl,以不加 NaCl的平板作为对照.结果发现,ST-1和ST-2突变体在正常条件下的根比野生型短,不同浓度盐处理后,其根长变化到与野生型差不多(图4,见第100页).
对它们的根长和鲜重做了定量统计,发现处理组中突变体的鲜重明显大于野生型,且突变体能长出绿色的子叶(图5,见第100页).
2.4ST-1的遗传分析
为了确定ST-1突变体基因的显隐性关系及突变体是否为单基因突变体,我们将ST-1与Col-0进行回交,得到的F1和F2代种子分别点播在含160mmol/L NaCl的1/2 MS培养基上,发现回交得到的F1代种子在盐平板上的表型和Col-0一样,都长不出绿色子叶,回交得到的F2代种子中筛到了抗盐的突变体如图6所示,对F2代中有抗盐表型的个体和与野生型表型的个体进行了统计,结果如表1所示.分离比接近1∶3,说明ST-1是一个隐性单基因突变体.
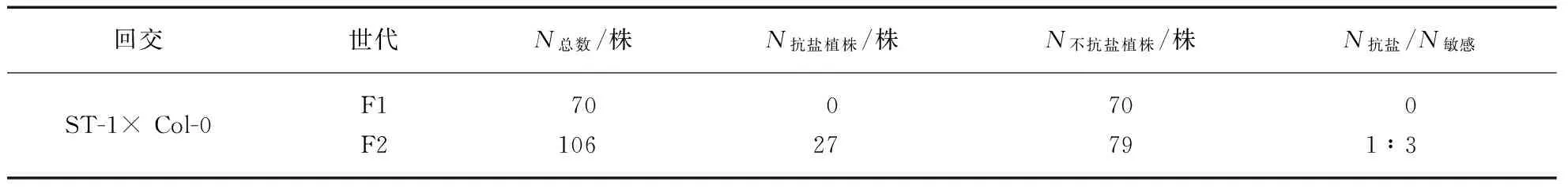
表1 ST-1的遗传表型分析
2.5ST-1的图位克隆
2.5.1作图群体的构建
利用拟南芥另一种生态型(Ler)与ST-1进行了杂交,得到F1代,然后让F1代的种子进行自交得到F2代,收种.将收到的F2代种子同样用160mmol/L NaCl的筛选条件进行筛选.将抗盐表型明显的突变体移到土里生长,生长一个月后单株取材,作为进行图位克隆的作图群体.
2.5.2ST-1的粗定位
根据5条染色体上不同的粗定位SSLP分子标记,通过PCR扩增的手段对杂交二代的纯合子群体进行鉴定.统计用不同SSLP引物进行PCR扩增的结果,发现杂交二代的重组率在F21M12标记附近最低(表2),因此基本确定了ST-1中的突变基因位于1号染色体的F21M12标记附近,具体结果如图7(见第102页)所示.由图可知,其他的染色体上的SSLP分子标记作为引物的PCR扩增产物电泳条带几乎都有分离,而只有以F21M12作为引物的PCR扩增产物电泳条带几乎和Col-0一样,没有分离,这说明F21M12与突变位点连锁.
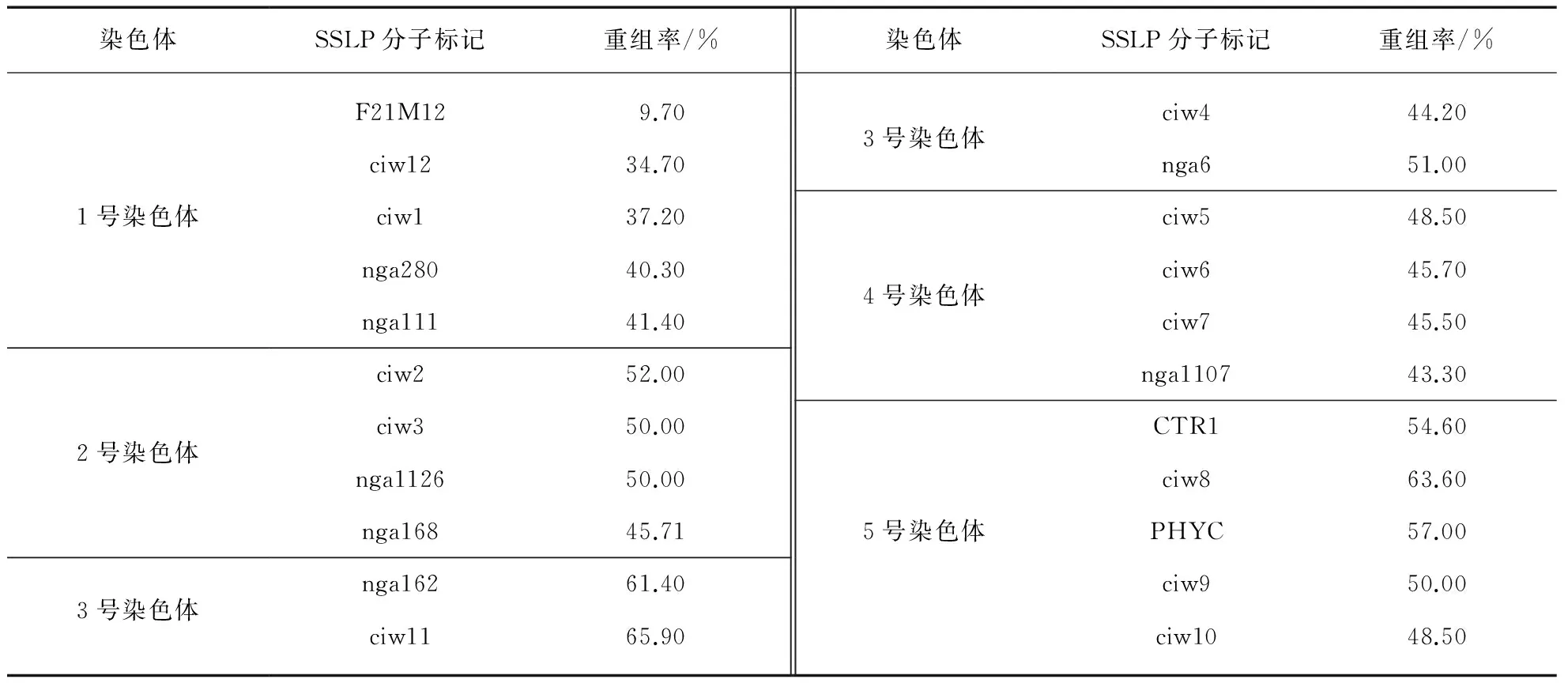
表2 ST-1突变体粗定位时各种SSLP分子标记的重组率
3 讨 论
构建突变体库是分离和研究基因功能的非常有效的方法.合适的筛选方法和筛选浓度是突变体筛选成功的关键,因此筛选方法和筛选浓度的确定是一个非常重要的问题: NaCl浓度过低起不到区分野生型和突变体的作用;浓度过高则严重影响突变体的萌发率.朱健康课题组用50mmol/L NaCl作为筛选浓度,采用弯根实验筛选拟南芥EMS突变体库(Col-0为背景)得到了对盐胁迫敏感的SOS突变体,进而成功地解析了SOS信号途径[5-6, 19].还有一些研究是用EMS诱变甘薯愈伤组织,然后用200mmol/L NaCl 筛选抗盐的突变体[20],也成功地得到了一些突变体.
将EMS突变体种子直接撒播到盐平板上,观察能不能长出绿色子叶,这种方法较为直观和快捷.拟南芥Col-0野生型在160mmol/L NaCl的盐胁迫下,萌发率受到的影响不是很大,但不能长出绿色子叶,因此是一个较为合适的筛选浓度.本实验中,我们采用160mmol/L NaCl来筛选抗盐突变体.
本实验中筛选的突变体,多为盐胁迫信号途径的负调控因子,因为基因突变后,抗盐表型会增强,而朱健康及其他多数课题组等筛选盐敏感突变体,多为盐胁迫信号途径的正调控因子.目前对于盐胁迫途径的负调控因子了解还较少,我们希望通过这种方法,能够克隆到该途径一些新的基因.EMS突变一般为随机点突变,因此每个实验室都有可能会筛到一些不同的突变体.
目前从筛选突变体到定位突变基因变得越来越简捷,特别是随着深度测序技术的发展.我们已经将一些突变体的突变基因进行了初步定位,接下来将会采用深度测序的方法对200株以上的杂交F2代抗盐突变体的混合样品进行全基因组测序,通过生物信息学分析Col-0和Ler的SNP在杂交F2代的分离情况,将可以精确获知突变基因的信息.
盐胁迫和其他非生物胁迫,例如干旱和氧化胁迫等有着密切的联系,它们之间的信号通路存在着交叉,存在一些共同的应对胁迫的机制[21].因此,研究盐胁迫相关基因,也有可能对其他非生物胁迫途径的研究提供参考.
[1]HUANG X. A previously unknown zinc finger protein, DST, regulates drought and salt tolerance in rice via stomatal aperture control[J].Genes&Development, 2009,23(15): 1805-1817.
[2]JOHNSON J M. AnArabidopsismutant impaired in intracellular calcium elevation is sensitive to biotic and abiotic stress[J].BMCPlantBiology, 2014,14(1): 162.
[3]KURBAN H. Effect of salinity on growth, photosynthesis and mineral composition in leguminous plant Alhagipseudoalhagi(Bieb.)[J].SoilScienceandPlantNutrition, 1999,45(4): 851-862.
[4]BINZEL M, RATAJCZAK R. Function of membrane transport systems under salinity: Tonoplast[J].Salinity:EnvironmentPlantsMolecules, 2002: 423-449.
[5]SHI H. TheArabidopsisthalianasalt tolerance gene SOS1 encodes a putative Na+/H+antiporter[J].ProcNatlAcadSciUSA, 2000,97(12): 6896-6901.
[6]QIU Q. Regulation of SOS1, a plasma membrane Na+/H+exchanger inArabidopsisthaliana, by SOS2 and SOS3[J].ProcNatilAcadSciUSA, 2002,99(12): 8436-8441.
[7]LIU J. TheArabidopsisthalianaSOS2 gene encodes a protein kinase that is required for salt tolerance[J].ProcNatlAcadSciUSA, 2000,97(7): 3730-3734.
[8]ISHITANI M. SOS3 function in plant salt tolerance requiresN-myristoylation and calcium binding[J].ThePlantCellOnline, 2000,12(9): 1667-1677.
[10]HALFTER U, ISHITANI M, ZHU J,etal. TheArabidopsisSOS2 protein kinase physically interacts with and is activated by the calcium-binding protein SOS3[J].ProcNatlAcadSciUSA, 2000,97(7): 3735-3740.
[11]LIU J, ZHU J. A calcium sensor homolog required for plant salt tolerance[J].Science, 1998,280(5371): 1943-1945.
[12]ZHU J. Salt and drought stress signal transduction in plants[J].AnnualReviewofPlantBiology, 2002,53: 247.
[13]RUS A. AtHKT1 is a salt tolerance determinant that controls Na+entry into plant roots[J].ProcNatlAcadSciUSA, 2001,98(24): 14150-14155.
[14]TAKAHASHI S. Hyperosmotic stress induces a rapid and transient increase in inositol 1, 4, 5-trisphosphate independent of abscisic acid inArabidopsiscell culture[J].PlantandCellPhysiology, 2001,42(2): 214-222.
[15]MIZOGUCHI T ICHIMURA K, YOSHIDA R, et al. MAP kinase cascades inArabidopsis: Their roles in stress and hormone responses, in MAP kinases in plant signal transduction[J].ResultsProblCellDiffer, 2000,27: 29-38.
[16]SHEEN J. Ca2+-dependent protein kinases and stress signal transduction in plants[J].Science, 1996,274(5294): 1900-1902.
[17]LUKOWITZ W, GILLMOR C S, SCHEIBLE W,etal. Positional cloning inArabidopsis. Why it feels good to have a genome initiative working for you[J].PlantPhysiology, 2000,123(3): 795-806.
[18]MUNNS R, TESTER M. Mechanisms of salinity tolerance[J].AnnuRevPlantBiol, 2008,59: 651-681.
[19]WU S, DING L, ZHU J,etal. SOS1, a genetic locus essential for salt tolerance and potassium acquisition[J].ThePlantCellOnline, 1996,8(4): 617-627.
[20]LUAN Y. Mutation induced by ethylmethanesulphonate(EMS),invitroscreening for salt tolerance and plant regeneration of sweet potato(IpomoeabatatasL.)[J].PlantCell,TissueandOrganCulture, 2007,88(1): 77-81.
[21]MAHAJAN S, TUTEJA N. Cold, salinity and drought stresses: An overview[J].ArchivesofBiochemistryandBiophysics, 2005,444(2): 139-158.
Identification of Arabidopsis Salt-Resistant Mutants
LI Chaoqun1,2, WANG Wenjing1,2, GE Xiaochun1,2
(1. School of Life Sciences, Fudan University, Shanghai 200438, China;2.StateKeyLaboratoryofGeneticEngineering,FudanUniversity,Shanghai200438,China)
Salinity is one of the major abiotic stresses that affects the growth and development of plants. So far very limited salt-resistant genes have been cloned and studied. Identifying new genes involved in salt tolerance are very important for understanding the salt signaling pathway and improving salt tolerance of plants. EMS mutagenesis is a classic method of reverse genetics. It can be used to discover novel salt-resistant genes which may not be identified by screening T-DNA insertion lines. In this study, 160mmol/L NaCl was used to screen for salt-tolerant mutants. The leaves of salt-tolerant mutants were green but that of the wild type plants turned yellow. 140 strains of salt-tolerant mutants were identified initially, however, only 84 of them set seeds. After confirmation of the salt resistant phenotype using T2 generation, 15 strains of stable salt-tolerant mutants(ST mutants) were obtained. Genetic and phenotypic analysis and preliminary mutated gene mapping were done for some of the identified mutants.
Arabidopsis; EMS mutagenesis; salt-resistant screening; ST mutant
0427-7104(2016)01-0097-07
2015-04-07
上海市基础科研重点项目(12JC1400500),国家自然科学基金(31170169)
李超群(1987—),男,硕士研究生;葛晓春,女,教授,通讯联系人,E-mail: xcge@fudan.edu.cn.
Q 943.2
A

