婴儿配方乳粉中克罗诺杆菌的分离鉴定及分型
张丽丽(上海邦德职业技术学院酒店管理分院,上海200444)
婴儿配方乳粉中克罗诺杆菌的分离鉴定及分型
张丽丽
(上海邦德职业技术学院酒店管理分院,上海200444)
采集了市场中的220份婴儿配方乳粉,利用生理生化和分子鉴定的方法对采集样品中的克罗诺杆菌进行了分离鉴定,检出10株克罗诺杆菌,污染率为4.55%。利用16S rRNA和ompA基因对克罗诺杆菌进行分型分析,都将10株克罗诺杆菌分为了2个簇,但是不同分型方法各个簇中的菌株不同。结果表明16S rRNA和ompA基因可以用于克罗诺杆菌的分型,但是辨识度不高。
婴儿配方乳粉;克罗诺杆菌;鉴定;分型
中国是婴儿配方乳粉生产和销售的大国,近年来消费者对婴儿配方乳粉的安全问题也越来越关注。克罗诺杆菌是囊括了之前阪崎肠杆菌的一个新的属,属于肠杆科的食源性致病菌,也是婴儿配方乳粉中的A类致病菌[1-2]。该致病菌能够通过污染婴儿配方乳粉感染新生儿,从而导致坏死性小肠结肠炎、脓血症、脑膜炎等严重的疾病,甚至导致患儿的死亡[3-4]。目前在国内的市售的婴儿配方乳粉中克罗诺杆菌具有较高的污染率,这与克罗诺杆菌丰富的遗传多样性有着密切的联系[5-6]。
本研究对国内市场中市售的婴儿配方乳粉进行了克罗诺杆菌的分离鉴定,并利用生理生化和16S rRNA的鉴定方法对克罗诺杆菌进行了鉴定,最后采用16S rRNA和ompA基因对分离到的克罗诺杆菌进行了分子分型研究,从而有助于我们从分子层面了解污染我国婴儿配方乳粉中克罗诺杆菌的分子特性,为揭示克罗诺杆菌的遗传多样性提供了理论依据。
1 材料与仪器
1.1主要材料
1.1.1培养基
溶菌肉汤(Lysogeny Broth,LB)、缓冲蛋白胨水(Buffered Peptone Water,BPW)、胰酪胨大豆琼脂培养基(Tryptose Soya Agar,TSA)、改良月桂基硫酸盐胰蛋白胨肉汤-万古霉素(Modified lauryl sulfate tryptose vancomycin medium,mLST-Vm)、阪崎肠杆菌显色培养基(DFI):上述培养基均购买自青岛海博生物有限公司。
1.1.2主要试剂
试验所需引物:由诺禾致源诺禾致源生物信息科技有限公司合成;细菌全基因组DNA试剂盒:北京天根生化科技有限公司;API20E试剂盒:上海百舜生物科技有限公司;2×PCR Master Mix:北京天根生化科技有限公司;D2000:百川开泰生物技术(北京)有限公司;双蒸水:上海榕柏生物技术有限公司;琼脂糖:南京生兴生物技术有限公司;1×TAE缓冲液:上海双螺旋生物科技有限公司。
1.2试验仪器
生物安全柜:山西天朗科技发展有限公司;高压蒸汽灭菌锅:济南思卓医疗器械有限公司;恒温培养箱:广州市中誉仪器有限公司;高速离心机:上海卢湘仪离心机仪器有限公司;普通PCR仪:赛飞(中国)有限公司;电泳仪:北京六一仪器厂。
2 方法
2.1采样
在本市各个超市采购婴儿配方乳粉,共采购220份婴儿配方乳粉。
2.2克罗诺杆菌的分离鉴定
根据参考GB 4789.40-2010《食品安全国家标准食品微生物学检验阪崎肠杆菌检验》对克罗诺杆菌进行分离鉴定。
2.3克罗诺杆菌DNA的提取及16S rRNA分子分型
利用细菌全基因组DNA试剂盒提取克罗诺杆菌的DNA,利用16S rRNA的通用引物对DNA进行扩增,并送样测序,在Genbank中对比测序后的基因序列得到菌株的鉴定结果,为了进一步利用16S rRNA分子方法对分离菌株进行分子分型,试验方法参照娄彬彬等的报道[7]。
2.4克罗诺杆菌ompA基因的分子分型
利用克罗诺杆菌ompA的特异性引物ompA F:5’-TAGACTTTACATCGCCAGGG-3’ 和 ompA R:5’-GAGCTTTCACGTTGTCACAG-3’,对克罗诺杆菌进行特性性的扩增,扩增条件如下:94℃预变性2 min;变性94℃15 s,复性60℃15 s,延伸72℃30 s,30个循环;最后72℃延伸5 min[8]。得到的扩增产物经过琼脂糖凝胶电泳验证后送样测序,最后得到克罗诺杆菌ompA的基因序列。
2.5生物信息学分析
利用MEGA6.0软件对克罗诺杆菌16S rRNA和ompA基因序列进行最大似然算法系统发育分析。%%%%%%
3 结果与分析
3.1婴儿配方乳粉中克罗诺杆菌的分离鉴定
婴儿配方乳粉中克罗诺杆菌分离鉴定的结果如表1所示。
利用通过GB 4789.40-2010《食品安全国家标准食品微生物学检验阪崎肠杆菌检验》的方法对婴儿配方乳粉中的克罗诺杆菌进行分离鉴定,结果显示利用该方法,220份婴儿配方乳粉中检测12株疑似菌株,即在阪崎肠杆菌显色培养基DFI中显示出蓝绿色菌落,且在TSA培养基中显示为黄色可疑菌落。上述疑似克罗诺杆菌利用API20E试剂盒进行生化鉴定,结果显示12株疑似克罗诺杆菌中11株为克罗诺杆菌即为阳性,1株为阴性。最后利用分子生物学的方法对生化鉴定结果进行验证,将16S rRNA扩增产物送样测序,并将测序所得到的序列在Genbank中比对,结果显示11株被生化鉴定为克罗诺杆菌的菌株10株为克罗诺杆菌,其中CE8虽然在生化检验中为克罗诺杆菌,但是经过分子水平的比对后发现其为阴沟肠杆菌。可见220份婴儿配方乳粉中分离到了10株克罗诺杆菌,该致病菌的污染率为4.55%。
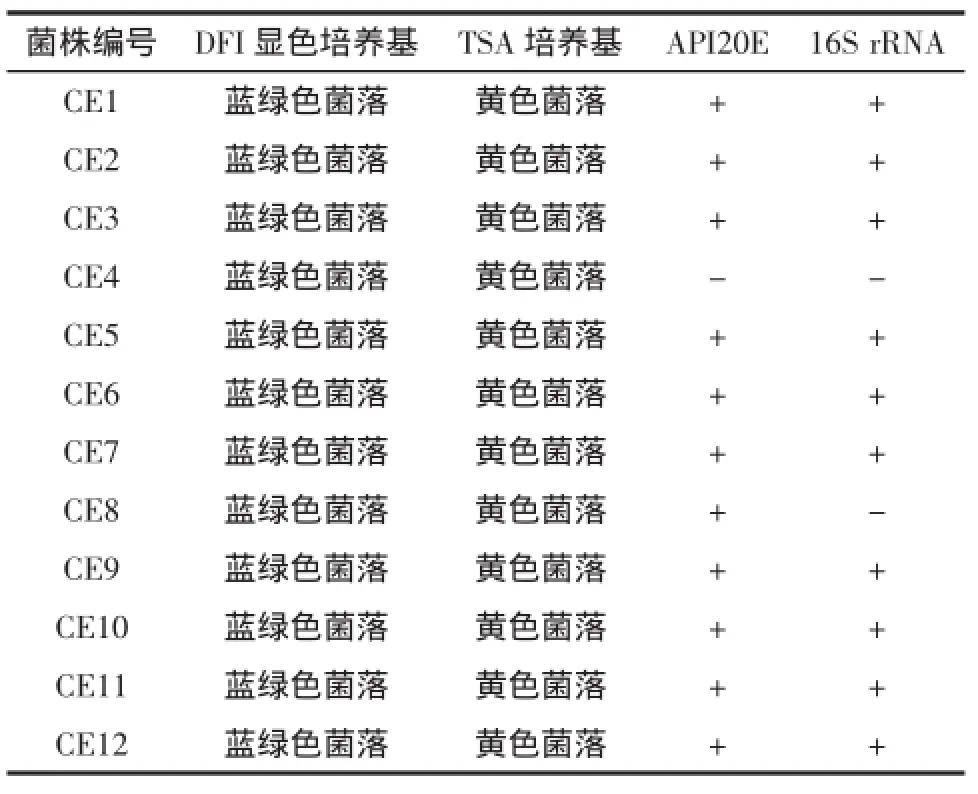
表1 婴儿配方乳粉中克罗诺杆菌鉴定结果Table 1 The result of identification ofthe Cronobacter spp.in powdered infantformula
3.2克罗诺杆菌16S rRNA分型
利用MEGA6.0软件对分离得到的10株克罗诺杆菌的16S rRNA序列进行系统发育分析,结果如图1所示。
由图1可知,10株克罗诺杆菌被分为了2个簇,其中 CE10、CE11、CE9、CE1和 CE2为一簇,CE7、CE12、CE3、CE5和CE6为一簇。同时标尺为0.0005说明就整体而言,10株克罗诺杆菌的系统发育关系是很近的,因此虽然16S rRNA可以用于简单的分型,但是辨识度并不高。
3.3克罗诺杆菌ompA基因的分型
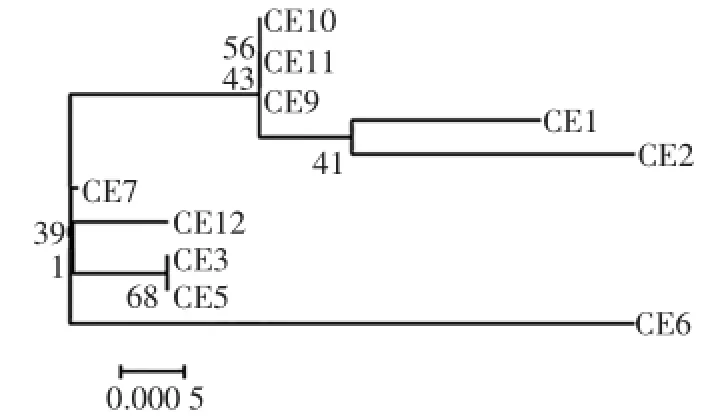
图1 克罗诺杆菌16S rRNA序列的最大似然系统发育树Fig.1 Maximum-likelihood tree of 16S rRNA gene for Cronobacter strains
利用MEGA6.0软件对10株克罗诺杆菌ompA基因进行了系统发育分析,并得到了清晰的系统发育关系,如图2所示。
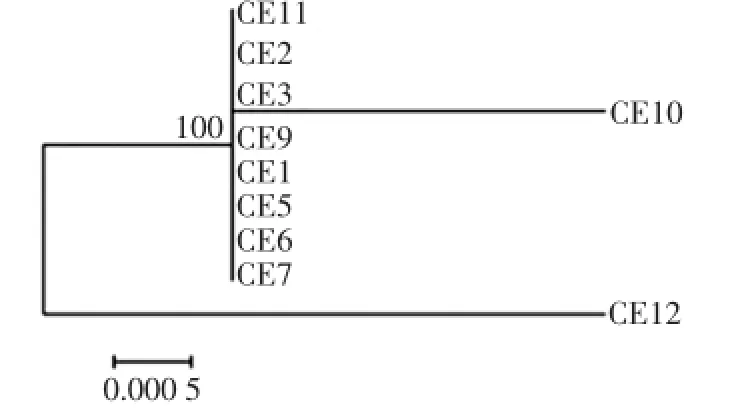
图2 克罗诺杆菌ompA基因序列的最大似然系统发育树Fig.2 Maximum-likelihood tree of ompA gene for Cronobacter strains
由图2可知,10株克罗诺杆菌的ompA基因被分为了两个簇,一簇为CE11、CE2、CE3、CE10、CE9、CE1、CE6、CE5和CE7,一簇为CE12,且CE12明显远离了其他的9株克罗诺杆菌,这说明CE12的ompA基因明显区别于其他9株菌。ompA基因与克罗诺杆菌的侵染能力和致病能力有关,因此CE12可能具有特殊的侵染能力。此外我们发现利用ompA基因对克罗诺杆菌分型的辨识度较低。
4 结论与讨论
在本研究中市售的婴儿配方乳粉克罗诺杆菌的污染率为4.55%,这表明克罗诺杆菌仍是婴儿配方乳粉中重要的致病菌。在2014年的调查中克罗诺杆菌的污染率为4.3%[11],这与我们的研究结果相似,此外一些研究也表明在婴儿配方乳粉的生产过程中包装环节是最易被克罗诺杆菌污染的环节之一[1,11]。在本研究中,我们利用了生理生化和分子鉴定的方法对婴儿配方乳粉中的克罗诺杆菌进行分离鉴定,利用GB 4789.40-2010《食品安全国家标准食品微生物学检验阪崎肠杆菌检验》的方法,12株克罗诺杆菌的疑似菌株被分离鉴定,经过API20E的20个生理生化指标的鉴定发现12个疑似菌株中11个为克罗诺杆菌,而16S rRNA分子鉴定的结果则表明12株克罗诺杆菌疑似菌株中10个为克罗诺杆菌,这表明API20E的鉴定方法优于显色性培养基的鉴定方法,但是16S rRNA的分子鉴定方法可以从分子层面对克罗诺杆菌进行鉴定,比API20E的生理生化鉴定更具优越性。
16S rRNA是最常见的用于克罗诺杆菌分型的基因之一[1]。娄彬彬等利用16S rRNA对分离自婴儿配方乳粉中的阪崎克罗诺杆菌进行了分子分型,并发现16S rRNA分子分型的辨识度有限,但是仍能对阪崎克罗诺杆菌进行分子分型[7]。ompA基因是编码克罗诺杆菌外膜蛋白A的重要基因,与克罗诺杆菌的致病能力有关[8]。我们的研究表明此基因也可以用于克罗诺杆菌的分子分型,且具有一定的辨识度。此外,在目前克罗诺杆菌分子分型的研究中,ITS序列分型、脉冲场电泳分型、多位点序列分型等方法也已经用于克罗诺杆菌的分型中[6,9-10]。
[1]Peng F,Chaoxin M,Binbin L,etal.Genotyping and source tracking of Cronobacter sakazakii and C.malonaticus isolates from powdered infant formula and an infantformula production factory in China[J]. Applied&EnvironmentalMicrobiology,2015,81(16):5430-5439
[2]董晓晖,李程思,吴清平,等.食品污染克罗诺杆菌(阪崎肠杆菌)的分离及鉴定[J].微生物学报,2013,53(5):429-436
[3]王翔,祝长青,徐幸莲,等.阪崎肠杆菌(克罗诺杆菌属)分子检测方法研究进展[J].食品科学,2011(13):350-354
[4]董晓晖,吴清平,莫树平,等.克罗诺杆菌检测方法研究进展[J].华中农业大学学报,2013,32(1):130-136
[5]Sumyya H,Susan J,Forsythe S J.Cronobacter sakazakii ST4 Strains and Neonatal Meningitis,United States[J].Emerging Infectious Diseases,2013,19(1):175-177
[6]Joseph S,SonbolH,HaririS etal.Diversity ofthe Cronobacter genus as revealed by multilocus sequence typing[J].Journal of Clinical Microbiology,2012,50(9):3031-3039
[7]娄彬彬,满朝新,费鹏,等.阪崎克罗诺杆菌的16S rDNA鉴定分型[J].中国食物与营养,2015,21(7):39-42
[8]Ks M N M V.Cloning and sequencing of the ompA gene of Enterobacter sakazakii and development of an ompA-targeted PCR for rapid detection of Enterobacter sakazakii in infant formula[J].Applied&Environmental Microbiology,2006,72(4):2539-2546
[9]牛婕婷,满朝新,费鹏,等.阪崎克罗诺杆菌的ITS序列分析[J].中国食物与营养,2015,21(3):27-30
[10]柴云雷,满朝新,卢雁,等.PFGE与16S rDNA对阪崎克罗诺杆菌分型的研究[J].中国乳品工业,2014,42(10):11-14
Isolation,Identification and Type of the Cronobacter spp.in Powdered Infant Formula
ZHANG Li-li
(Hospitality Elite School,Shanghai Bangde Vocational College,Shanghai200444,China)
220 powdered infant formula samples were collected from markets.10 Cronobacter strains were isolated and identified using the physiological,biochemical and molecular identification methods,with a contamination rate of 4.55%.10 Cronobacter strains were divided into two clusters respectively by the 16S rRNA and ompA genes analysis,however,the composition of strains were different in each cluster by different methods. The above results showed that the 16S rRNA and ompA genes could be used for the Cronobacter type with the nothigh distinguishability.
powdered infantformula;Cronobacter spp.;identification;type
10.3969/j.issn.1005-6521.2016.12.039
张丽丽(1982—),女(汉),讲师,硕士,研究方向:食品科学。
2016-03-02

