基于RNA-seq技术开发中间偃麦草基因组特异分子标记
崔 雨,鲍印广,王洪刚,2,李兴锋,2
(1.山东农业大学作物生物学国家重点实验室,山东泰安 271018; 2.国家小麦改良中心泰安分中心,山东泰安 271018)
基于RNA-seq技术开发中间偃麦草基因组特异分子标记
崔 雨1,鲍印广1,王洪刚1,2,李兴锋1,2
(1.山东农业大学作物生物学国家重点实验室,山东泰安 271018; 2.国家小麦改良中心泰安分中心,山东泰安 271018)
摘要:中间偃麦草(Thinopyrum intermedium,2n=6x=42,JJJSJSStSt或EeEeEbEbStSt)是小麦遗传改良的重要近缘植物,高抗锈病、白粉病等病害,耐逆性强。开发中间偃麦草基因组特异分子标记对于加快其优异基因向普通小麦中的转移和利用具有重要意义。为建立快速准确开发中间偃麦草基因组特异分子标记的新体系,首先利用RNA-seq技术对中间偃麦草及其基因组供体二倍体长穗偃麦草、百萨偃麦草、假鹅观草的苗期叶片进行转录组测序,然后根据测序结果设计EST-SSR引物,对普通小麦、中间偃麦草及其基因组供体材料进行DNA扩增分析,从中筛选鉴定中间偃麦草基因组特异标记。结果表明,在所合成的200对EST-SSR引物中,有75对引物(37.5%)在中间偃麦草、二倍体长穗偃麦草、百萨偃麦草和假鹅观草中具有特异扩增,引物多态率较高。说明利用RNA-seq技术开发EST-SSR引物可以高效地用于筛选中间偃麦草基因组特异分子标记,这一技术体系也可应用于其他作物及其近缘物种的特异分子标记开发。
关键词:中间偃麦草;小麦;RNA-seq;特异分子标记
中间偃麦草为多年生野生草本植物,是小麦重要的近缘植物之一,其先后被划入小麦属(Triticum)、冰草属(Agropyron)、偃麦草属(Elytrigia)和类麦属(Thinopyrum)。尽管后来颜 济和杨俊良[1]将其归入毛麦属(Trichopyrum),但是因为习惯的原因,中间偃麦草的名称一直被延用下来。吉万全等[2]认为,中间偃麦草染色体组有两个亲缘关系较近的染色体组,可能分别来源于二倍体长穗偃麦草(EeEe)和百萨偃麦草(EbEb),另外一个染色体组与其他两个染色体组亲缘关系较远,其原始亲本供体可能是假鹅观草(StSt)。Chen等[3]认为中间偃麦草的染色体组为JJJSJSStSt,原始供体同样可能为二倍体长穗偃麦草、百萨偃麦草和假鹅观草。目前,一般将中间偃麦草染色体组构成表示为EeEeEbEbStSt或者JJJSJSStSt[4]。
中间偃麦草对小麦的“三锈”、白粉病等病害免疫,对小麦黑穗病、叶枯病、根腐病和条纹花叶病等病害高抗,同时又是大麦黄矮病的良好抗源,且易与小麦杂交,因此它已成为小麦遗传改良中具有重要利用价值的种质资源。国内外不同研究者利用普通小麦与中间偃麦草杂交,目前已创制出一批八倍体、异附加系、异代换系及易位系等小偃麦异染色体系,为小麦遗传改良提供了丰富的种质材料,并成功将中间偃麦草的多种优异基因转移到普通小麦遗传背景当中[5-7]。
小麦远缘杂交后代材料遗传组成的精确鉴定是对其优异基因进行开发利用的前提,分子标记的发展为小麦异源种质系的鉴定提供了新的辅助选择技术,大大提高了小麦异染色体系特别是小片段易位系的选择效率,为更好地转移和利用小麦近缘物种有利基因提供了便利[8]。目前一些新技术的应用加快了小麦近缘植物分子标记开发的进程,如陈世强等[9]基于SLAF-seq技术开发出一批长穗偃麦草染色体特异分子标记,代 程等[10]由冰草转录组EST序列开发了130个冰草6P特异标记。基于高通量测序的RNA-Seq可以快速获得大量表达基因序列,高效应用于分子标记开发、功能基因发掘、分子机理研究以及转录组比较等方面[11-15]。鉴于目前开发或筛选的中间偃麦草特异分子标记多态率较低、数量有限且定位复杂,无法满足中间偃麦草研究的需求,因此大批量开发中间偃麦草基因组(Ee、Eb、St)特异标记对于丰富中间偃麦草标记数目、明确标记的基因组来源、鉴定小偃麦遗传组成和分析偃麦草属内系统关系等有重要意义。本研究利用RNA-seq技术获得中间偃麦草及其基因组供体二倍体长穗偃麦草、百萨偃麦草和假鹅观草的转录组数据,拟开发一批特异性强、稳定性高的中间偃麦草基因组EST-SSR标记,旨在为高效地开发中间偃麦草基因组特异分子标记建立新的技术体系,为小偃麦新种质的鉴定和中间偃麦草优异基因的进一步利用奠定基础。
1材料与方法
1.1试验材料
供试材料包括中间偃麦草(Thinopyrumintermedium,2n=6x=42,EeEeEbEbStSt或JJJSJSStSt)、二倍体长穗偃麦草(Th.elongatum,2n=2x=14,EeEe)、假鹅观草(Pseudoroegneriastrigosa,2n=2x=14,StSt)、百萨偃麦草(Th.bessarabicum,2n=2x=14,EbEb)及普通小麦(TriticumaestivumL.,6n=2x=42,AABBDD)中国春和烟农15。其中,中间偃麦草引自原中国科学院西北植物研究所,由李振声院士提供;假鹅观草引自中国农业科学院作物科学研究所,由李立会研究员提供;百萨偃麦草引自南京农业大学,由亓增军教授提供;其余材料均由国家小麦改良中心泰安分中心保存。
1.2RNA-seq及引物设计
所用的转录组数据均由中间偃麦草、二倍体长穗偃麦草、假鹅观草和百萨偃麦草苗期叶片总RNA经IlluminaHiSeqTM2000平台测序获得。用Trinity将测序序列进行拼接,以此作为后续分析的参考序列,取每条基因中最长的转录本作为unigene。采用MISA(1.0版,默认参数)对unigene进行序列分析,搜索SSR位点,所检测SSR位点包括单核苷酸重复、二核苷酸重复、三核苷酸重复、四核苷酸重复、五核苷酸重复和六核苷酸重复6类,判断标准为单核苷酸重复不少于10次,二核苷酸重复不少于6次,三核苷酸、四核苷酸、五核苷酸和六核苷酸重复不少于5次。找出SSR位点之后,采用Primer 3(2.3.5版)进行SSR引物批量设计,主要参数设置如下:产物大小100~280 bp,引物长度18~27 bp,GC含量40%~65%,退火温度57~63 ℃,上下游引物Tm值相差小于5 ℃。
1.3引物筛选及PCR扩增
根据中间偃麦草、二倍体长穗偃麦草、假鹅观草和百萨偃麦草的EST-SSR数据,每个材料选取50对引物,共计200对,由上海生物工程公司合成。扩增体系(10 μL):模板DNA(33 ng·μL-1)3 μL、2×PowerTaqPCR MasterMix 5 μL、引物(5 μmol·L-1)2 μL。扩增反应在T100TM(BIO-RAD,USA)PCR仪上进行。扩增程序:94 ℃预变性5 min;94 ℃变性1 min,55 ℃退火40 s,72 ℃延伸1 min,30个循环;72 ℃延伸5 min。扩增产物经8%聚丙烯酰胺非变性凝胶电泳分离,银染显影,拍照分析。
2结果与分析
2.1转录组测序及SSR检测结果
高通量测序(IlluminaHiSeqTM2000)得到的原始数据及利用MISA对unigene进行SSR检测的结果见表1和图1。

表1 RNA-seq的基本情况

A:中间偃麦草;B:二倍体长穗偃麦草;C:百萨偃麦草;D:假鹅观草;Mono:单核苷酸重复;Di:二核苷酸重复;Tri:三核苷酸重复;Tetra:四核苷酸重复;Penta:五核苷酸重复;Hexa:六核苷酸重复
A:Th.intermedium; B:Th.elongatum; C:Th.bessarabicum; D:Ps.strigosa; Mono:Mononucleotide repeat; Di:Dinucleotide repeat; Tri:Trinucleotide repeat; Tetra:Tetranucleotide repeat; Penta:Pentanucleotide repeat; Hexa:Hexanucleotide repeat
图1不同材料SSR基元的分布
Fig.1Distribution of SSR motifs in different materials
经检测,在中间偃麦草中搜索到6 033个SSR位点,分布在5 390条unigene中,561条unigene含有1个以上SSR,252条unigene含有复合SSRs,平均每6.55 kb有1个SSR;在二倍体长穗偃麦草中搜索到5 367个SSR位点,分布在4 831条unigene中,456条unigene含有1个以上SSR,194条unigene含有复合SSRs,平均每6.82 kb有1个SSR;在百萨偃麦草中搜索到7 232 个SSR位点,分布在6 299条unigene中,794条unigene含有1个以上SSR,317条unigene含有复合SSRs,平均每5.83 kb有1个SSR;在假鹅观草中搜索到6 648个SSR位点,分布在5 830条unigene中,708条unigene含有1个以上SSR,301条unigene含有复合SSRs,平均每6.02 kb有1个SSR。
中间偃麦草、二倍体长穗偃麦草、百萨偃麦草和假鹅观草等4个材料的SSR发生频率(检出SSR的unigene与总unigene数目之比)分别为8.6%、8.3%、11.6%和9.9%,SSR出现频率(SSR数目与总unigene数目之比)分别为9.6%、9.3%、13.3%和11.3%,在本试验中百萨偃麦草及假鹅观草的SSR检测效率高于中间偃麦草和长穗偃麦草。从SSR基元类型上来看,三核苷酸重复比例最大,占50.0%左右;从SSR基元的重复次数上来看,重复5~8次的序列占比超过六成,其中二倍体长穗偃麦草达到74.0%。
2.2EST-SSR标记分析结果
在中间偃麦草、二倍体长穗偃麦草、假鹅观草、百萨偃麦草的EST-SSR数据中分别选取50对引物,共计合成200对引物,然后利用中国春、烟农15、中间偃麦草、二倍体长穗偃麦草、百萨偃麦草及假鹅观草对新开发的引物进行了多态性验证。具体结果如下。
2.2.1中间偃麦草EST-SSR的扩增结果
利用50对基于中间偃麦草RNA-seq开发的EST-SSR引物对不同材料进行扩增,共有45对引物可以在不同材料中扩增出清晰的条带,其中,thinta47等21对引物在中国春、烟农15和中间偃麦草中扩增出相同条带;thinta19在二倍体长穗偃麦草中扩增出特异条带;thinta48等23对引物在中国春、烟农15和中间偃麦草间有多态性扩增,可以作为中间偃麦草的特异引物加以利用,在这些特异引物中,thinta8等7对引物在中间偃麦草和百萨偃麦草间扩增出相同条带,thinta5等6对引物在中间偃麦草、二倍体长穗偃麦草、百萨偃麦草、假鹅观草间扩增出不同条带。部分引物扩增结果见图2。
2.2.2二倍体长穗偃麦草EST-SSR的扩增结果
利用50对基于二倍体长穗偃麦草RNA-seq开发的EST-SSR引物对不同材料进行扩增,共有49对引物可以在不同材料中扩增出清晰的条带,其中,thelo12等29对引物在中国春、烟农15和二倍体长穗偃麦草中扩增出相同条带;thelo5和thelo37在百萨偃麦草中扩增出特异条带;thelo11等18对引物在中国春、烟农15和二倍体长穗偃麦草间有多态性扩增。部分引物扩增结果见图3。

M:DL2000;1:烟农15;2:中国春;3:中间偃麦草;4:二倍体长穗偃麦草;5:百萨偃麦草;6;假鹅观草。图3~5同
M:DL2000; 1:Yannong 15; 2:Chinese Spring; 3:Th.intermedium; 4:Th.elongatum; 5:Th.bessarabicum; 6:Ps.strigosa. The same in fig.3-5
图2部分中间偃麦草分子标记在普通小麦和中间偃麦草及其基因组供体中的扩增
Fig.2Amplication inT.aestivumL.,Th.intermediumand its genome donor with part molecular markers fromTh.intermedium
2.2.3百萨偃麦草EST-SSR的扩增结果
利用50对基于百萨偃麦草RNA-seq开发的EST-SSR引物对不同材料进行扩增,共有31对引物可以在不同材料中扩增出清晰的条带,其中,thbes4等18对引物在中国春、烟农15和百萨偃麦草中扩增出相同条带;thbes23和thbes42引物在二倍体长穗偃麦草和百萨偃麦草中扩增出相近条带;thbes33在假鹅观草中扩增出特异条带;thbes37等10对引物在中国春、烟农15和百萨偃麦草间有多态性扩增,在这些特异引物中,thbes39等3对引物在中间偃麦草和百萨偃麦草间扩增出相同条带。部分引物扩增结果见图4。
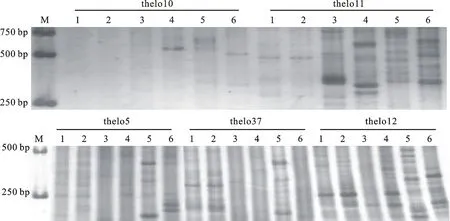
图3 部分二倍体长穗偃麦草分子标记在普通小麦和中间偃麦草及其基因组供体中的扩增
2.2.4假鹅观草EST-SSR的扩增结果
利用50对基于假鹅观草RNA-seq开发的EST-SSR引物对不同材料进行扩增,共有40对引物可以在不同材料中扩增出清晰的条带,其中,psstr20等20对引物在中国春、烟农15和假鹅观草中扩增出相同条带;其中,psstr39引物在百萨偃麦草中扩增出特异条带;psstr32等19对引物在中国春、烟农15和假鹅观草间有多态性扩增,在这些特异引物中,psstr17和psstr23引物在中间偃麦草和假鹅观草间扩增出相同条带,psstr38和psstr44引物在百萨偃麦草中扩增出特异条带。部分引物扩增情况见图5。
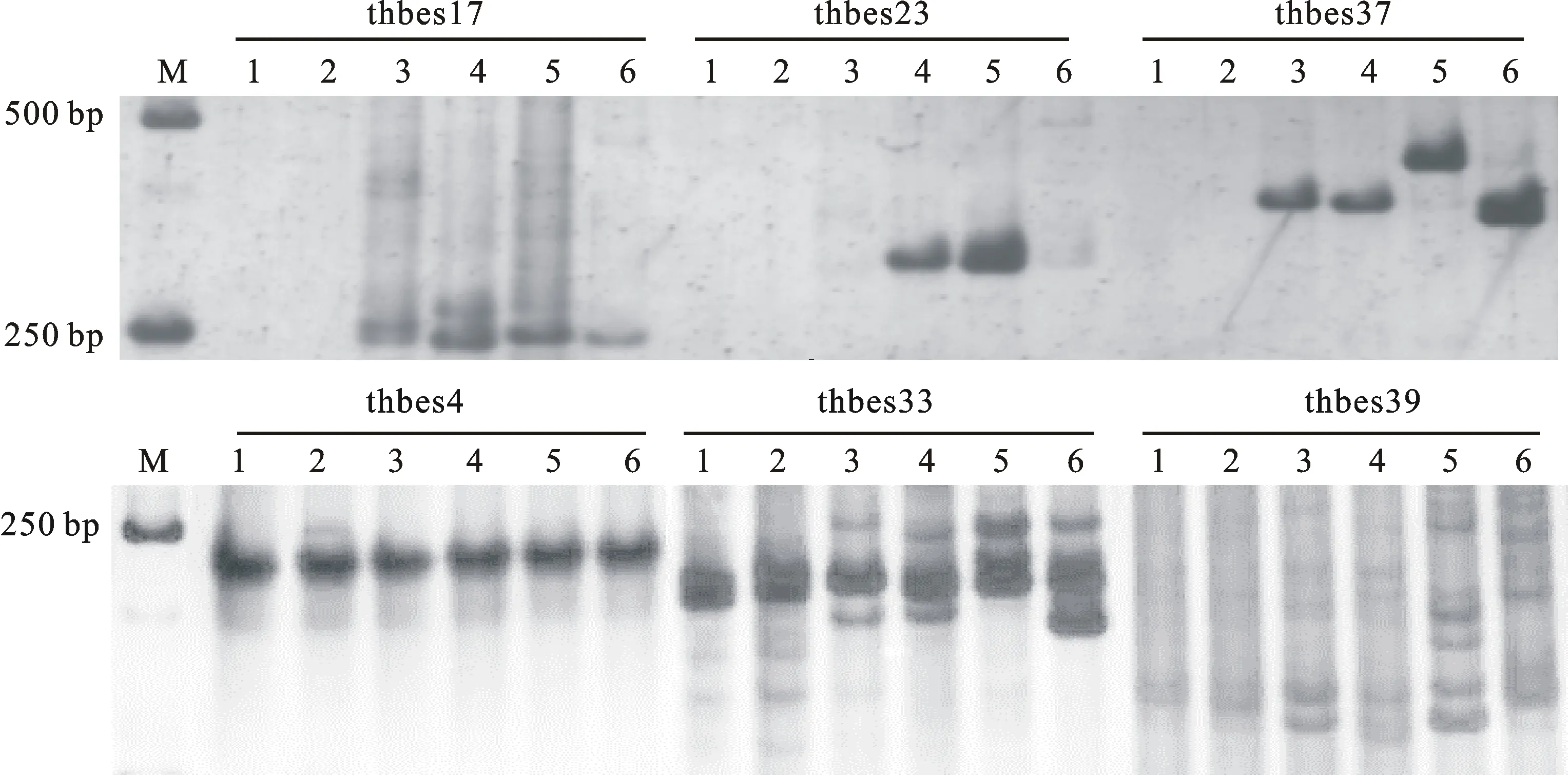
图4 部分百萨偃麦草分子标记在普通小麦和中间偃麦草及其基因组供体中的扩增

图5 部分假鹅观草分子标记在普通小麦和中间偃麦草及其基因组供体中的扩增
通过引物多态性验证,并利用中间偃麦草、二倍体长穗偃麦草、百萨偃麦草及假鹅观草对多态性引物进行基因组归类统计,最终开发出的中间偃麦草、二倍体长穗偃麦草、百萨偃麦草、假鹅观草基因组特异分子标记数量依次为28个、19个、22个和20个。标记序列见表2。

表2 各材料特异分子标记名称及序列
(续表2Continued table 2)

材料Materials分子标记Marker引物序列(5'→3')Primersequence(5'→3')分子标记Marker引物序列(5'→3')Primersequence(5'→3')二倍体长穗thelo1F:CGCAACTCACAAGTGATGCCthelo22F:AGATCGACACCGGCAAAAGT偃麦草R:AACACGATAAAGGCAGGCCAR:CCGATGCTCTCACCACGTAATh.elongatumthelo3F:AGCCTCCAGAATGACCTTGCthelo23F:ACAATCGGGTCCAAACAGCTR:CATCATCTTCTTGCTGCCGCR:GAGAAGAACTCCGTGTCCCGthelo6F:CACGTGAGATTCCGCTGAGGthelo34F:GCCCAACTAGTGCACTCAGGR:AGTGGGACTTGTTGGTGAGCR:GCACGTTCCCTAGAAAGGCTthelo7F:GGAGATGATGCCGGTTCTGAthelo39F:TGCTGCTCCTTGCTCTGAAAR:ACATACAGAAGCACGGCACAR:ACTCTCGAGCCCTTCAAGGAthelo8F:ATGAACTGCATCGACTCGCTthelo41F:CCTCTCCCGACCTCTCTCTCR:GCAAAGGATAGAGGGGGCATR:GTGATCACCTCGTTGTTGCGthelo10F:CCTCCCATTCTTGAACGCCTthelo44F:CTTCACTTCGTCGTCCAGCTR:CTTCTTCCTCATGCGCATGCR:CGAGGAGCGTGATGACATGAthelo11F:CCATGATTGTCCGCCTACGAthelo45F:CACTCCACCACTCTGCTTCCR:AACCCCACAACGGAGATCACR:CCTTTCCCTCCATGTGACCCthelo14F:AATGGCCAAACGGGAGAGAGthelo50F:TTTGAGTCTTGGGTCAGGGCR:ACACTAAGCCGGCAATCACAR:TCTTGGTTCTGTGTCGTACCAthelo17F:CACGTCATCAGCTCCACTCAthinta19F:TGAGATGCTCCATGTGGTCTR:AGTTGTCTGGCAGGATGGTCR:GCGTGGTCAACTTGCAGAAGthelo18F:ATCCTCGCCTCTGTCTGCTAR:CGGGAACTTGGAGGCATTCT百萨偃麦草thbes17F:AGGCCCCACTCTCACACATAthinta9F:TGGCGAGGGAACAATCAGAGTh.bessarabicumR:TGCCTTAGCTGTACACTGGCR:CTGCCCCTCCTTTCTAGAAAAthbes19F:GGGCAATCACCACAAACAGGthinta16F:TGGCCTCAGAGCAAAACACAR:CGCAAAGGTTTTTCACTCGGTR:GGCTATCTTACAAGGAGGAGGTGthbes27F:GCACACGCAACGAACTTACAthinta45F:AAGCTGGTGCGGAACTCTTTR:TGGAGAAAGTTGGCATCTCCGR:GCAGAGATGAAAGTAGTACAAGCGthbes36F:CGACACCTGCGCAACATTACthinta46F:CTCCTCCTCCTCTCCTTGCTR:ATGGGTTGGCTGCAAGAAGAR:AGGAGGAGGGGCATGATGTthbes37F:ACGAAACACACGGGGGTATCthinta48F:CGCGGAAGAAATGCCGTATCR:TGCTAGGCACCTGCTAATCGR:CTCTCTCATCTTGGCGTGCAthbes38F:CGTATCCCACGCGCTTTGTAthinta49F:CTCACCGTTGCTCACTGGATR:GCTCATGCGACACAAATCCAR:CATCATCTTCTTGCTGCCGCthbes39F:GAGGAGAAGGTGGTGGAAGCpsstr38F:GACGGCAAGATTATGGCAGCR:AACCGGAGCCATAACCACTGR:CTACACCTGCGTCATCTCCCthbes40F:TGCACCAAGATGAGGAGCAGpsstr39F:GGGAGATGACGCAGGTGTAGR:TGGCATTGCTCTGGTGTCTTR:GGCTCTTGTCAGATCGTGCTthbes41F:AGCTGAACCAACGAGACCTGthelo5F:CCAGCTCAGCGGCAAACTAAR:TGCTGGTGTGGCCACATTATR:GCTTCTCCTTCACCTTCCCGthbes45F:TTATGGTTGGGGGTGATGCCthelo37F:GCTGGTCCATTGGTTTGCTGR:GCCCTTTTGTGAGAGCTGTTR:CTTCGGCCATCCCTCCGGthinta8F:TCATCCTCGAAGACGTGCTGpsstr44F:CGGCTTCGGTTCCTCTTTCTR:CTTCCCACCTCCGACGAAAAR:TCATGCCAGTTGTGAGGGAC假鹅观草psstr2F:GGAAGGAAGCAAATCGCAGCpsstr23F:TCGCAGCGGTAGTTGTTCTTPs.strigosaR:AAAATCCCTTCCTCGTCCCGR:CGCCACTCGACTTAAACCCTpsstr4F:GTCCTTCCTCTCCGTTGGTGpsstr25F:AGGCGGACATACAAATGGCAR:GCCGTCACACCACACACAR:CTTTCCCTTCTTCTTCTTCCTCTCpsstr6F:CACCTATCCCCGTTCCGTTCpsstr26F:TGTGCACCTGTACTGTGTCGR:AGGCCTTCTTGTCGAAGTCGR:TAAACAATGCCACCGCGAAGpsstr9F:GGAGATCGTGGTACTCGCTGpsstr29F:AACGCAGATGCCCACATACAR:ATGGAGCAGTTGGTGGGATGR:TGTCTATCCCACCTACCCCApsstr11F:CTCCGGTCACCACCTTTTCTpsstr32F:AGGATCAGTGGCCACCTACTR:TGAACCTCCATTGCCTTGGTR:CCTCACAAGCCATGGGATGTpsstr13F:TTACTCTCTGCCACCACCCApsstr38F:GACGGCAAGATTATGGCAGCR:CAAAACGCCAGCCCTACAACR:CTACACCTGCGTCATCTCCCpsstr16F:CTCCTCCTAACAACAGGGCGpsstr43F:GTCCATACGTAGACACACAGCAR:CGCCATTTTATCTGCCTGCCR:AGCTACCACACGTATACGCGpsstr17F:GCAATTTCCGTTGGCCAGAGpsstr44F:CGGCTTCGGTTCCTCTTTCTR:TGGAAAACCAATTACTGCCAACAR:TCATGCCAGTTGTGAGGGACpsstr19F:ACTGACTCATGCTCTCTTCACApsstr47F:CGGCTTCGGTTCCTCTTTCTR:AGGTCCACCTGTTGTTGTCGR:GAGACCAAGGCCAGTGTTCApsstr22F:GCCACTCTGAATCCCGTCTCthbes33F:TACCACCGCCCAGCAAATAGR:TAGCACTGAATCGCCGGATGR:GAAGACGACGAGCGGTACTC
3讨 论
3.1基于RNA-seq开发中间偃麦草基因组特异标记的特点
对同一物种EST序列进行SSR搜索时,所得结果可能不同,如小麦的SSR出现频率为3.2%[16]与4.1%[17],这是由于不同软件在搜索SSR时所采取的算法及标准不同,搜索到的结果存在差异;不同物种的SSR出现频率也有很大差异,如党参(16.1%)[18]较小麦高出几倍。本研究中,中间偃麦草、二倍体长穗偃麦草、百萨偃麦草和假鹅观草的SSR出现频率(9.6%、9.3%、13.3%和11.3%)相比其他禾本科植物,非常适合大批量开发标记。
利用通过高通量测序获得的中间偃麦草及其基因组供体材料转录组数据设计出的200对引物中,共75对引物在中国春、烟农15和中间偃麦草、二倍体长穗偃麦草、百萨偃麦草、假鹅观草间具有多态性扩增。仅计算由某材料转录组数据开发的引物在该材料中的多态率,所开发引物在中间偃麦草、二倍体长穗偃麦草、百萨偃麦草及假鹅观草中的多态率分别达到46.0%、36.0%、20.0%和38.0%,多态率远高于根据谷类作物的共线性,利用小麦G-SSR引物1 255对、EST-SSR引物3 012对、STS引物 293 对、RAPD引物1 200个产生的多态性引物在中间偃麦草(7.6%)、二倍体长穗偃麦草(7.5%)、百萨偃麦草(7.5%)及假鹅观草(5.9%)中的多态率(课题组前期试验)。因此,基于RNA-seq开发EST-SSR具有多态率高、特异性强的特点。有35对引物在中国春和中间偃麦草等材料中没有稳定的扩增产物,这可能由于采用部分单核苷酸重复序列设计引物、转录组EST拼接出现误差等原因导致。
设计引物采用的序列直接关系引物扩增多态率,中间偃麦草等材料和小麦存在较近的亲缘关系,序列有一定的相似性,这会降低特异标记的开发效率,本研究中,44.0%的引物在中国春、烟农15和中间偃麦草等材料间具有相同的扩增片段也印证了这一点。下一步可通过中间偃麦草与小麦EST序列比对,得到中间偃麦草的特异序列,由此设计出的引物应该特异性更高、针对性更强,同时这些特异序列对于偃麦草的分子遗传和进化研究很有裨益。
3.2中间偃麦草基因组特异标记的应用
利用7个中国春-百萨偃麦草染色体异附加系可以将本研究开发的22个百萨偃麦草特异标记,分别定位于 1Eb到7Eb染色体上,同样,可以将本研究中开发的其他材料的特异分子标记定位到相关的染色体上,这项工作尚在进行之中。下一步希望通过开发大量的引物并验证更多新特异分子标记,使其覆盖到Ee基组、Eb基组及St 基组每一条染色体上,建立Ee、Eb及St基组特异分子标记体系。开发的中间偃麦草特异标记可以应用于鉴定小麦-中间偃麦草小片段易位系或渐渗系以及染色体物理作图中。
本研究中有122对引物(61.0%)在中国春、烟农15上有稳定的扩增位点,因而也可以作为小麦的分子标记,这进一步丰富了小麦的标记基础。另外,在特异引物中,有10对引物在中间偃麦草和百萨偃麦草间扩增出相同特异条带,有2对引物在中间偃麦草和假鹅观草间扩增出相同特异条带;还有一些非特异引物在中间偃麦草与其基因组供体间或在二倍体长穗偃麦草、百萨偃麦草、假鹅观草间扩增出相同条带。说明中间偃麦草、二倍体长穗偃麦草、百萨偃麦草、假鹅观草间有较高的同源性,这对于研究偃麦草属内的物种亲缘关系也有着一定帮助。
通过转录组测序和分子标记的验证,建立了高效开发中间偃麦草特异标记的新方法,开发的标记可应用于小偃麦后代材料遗传组成的鉴定,加速中间偃麦草优异基因的转移与利用。
参考文献:
[1]颜 济,杨俊良.小麦生物系统学 [M].北京:中国农业出版社,2013:473-516.
Yan J,Yang J L.Biosystematics of Wheat [M].Beijing:China Agriculture Press,2013:473-516.
[2]吉万全,薛秀庄,王秋英,等.中间偃麦草的GISH分析 [J].西北植物学报,2001,21(3):401-405.
Ji W Q,Xue X Z,Wang Q Y,etal.GISH analysis ofThinopyrumintermedium[J].ActaBo-tanicaBorealiOccidentaliaSinica,2001,21(3):401-405.
[3]Chen Q,Conner R L,Laroche A,etal.Genome analysis ofThinopyrumintermediumandTh.ponticumusing genomicinsituhybridization [J].Genome,1998,41(4):580-586.
[4]亓晓蕾.八倍体小偃麦混合基因组形成的遗传基础研究和小偃麦种质系的鉴定 [D].泰安:山东农业大学,2014.
Qi X L.Genetic basis of the formation of mixed genomes in octoploidTrititrigiaand identification ofTrititrigiagermplasm [D].Tai’an:Shandong Agricultural University,2014.
[5]王洪刚,刘树兵,亓增军,等.中间偃麦草在小麦遗传改良中的应用研究 [J].山东农业大学学报,2000,31(3):333-336.
Wang H G,Liu S B,Qi Z J,etal.Application studies ofElytrigiaintermediumin hereditary improvement of wheat [J].JournalofShandongAgriculturalUniversity,2000,31(3):333-336.
[6]李振声,容 珊,陈漱阳.小麦远缘杂交 [M].北京:科学出版社,1985.
Li Z S,Rong S,Chen S Y.Wheat Distant Hybridizadion [M].Beijing:Science Press,1985.
[7]李晴祺.冬小麦种质创新与评价利用 [M].济南:山东科学技术出版社,1998.
Li Q Q.Creation Evaluation and Utilization of Winter Wheat Germplasm [M].Jinan:Shandong Science and Technology Press,1998.
[8]Colmer T D,Flowers T J,Munns R.Use of wild relatives to improve salt tolerance in wheat [J].JournalofExperimentalBotany,2006,57(5):1059-1078.
[9]陈士强,秦树文,黄泽峰,等.基于SLAF-seq技术开发长穗偃麦草染色体特异分子标记 [J].作物学报,2013,39(4):727-734.
Chen S Q,Qin S W,Huang Z F,etal.Development of specific molecular makers forThinopyrmelongatumchromosome using SLAF-seq technique [J].ActaAgronomicaSinica,2013,39(4):727-734.
[10]代 程,张锦鹏,武晓阳,等.小麦背景下冰草6P染色体特异EST标记的开发 [J].作物学报,2012,38(10):1791-1801.
Dai C,Zhang J P,Wu X Y,etal.Development of EST markers specific toAgropyroncristatumchromosome 6P in common wheat background [J].ActaAgronomicaSinica,2012,38(10):1791-1801.
[11]Wu H L,Chen D,Li J X,etal.Denovo characterization of leaf transcriptome using 454 sequencing and development of EST-SSR markers in tea(Camelliasinensis) [J].PlantMolecularBiologyReporter,2013,31:524-538.
[12]Chung J W,Kim T S,Sundan S,etal.New cDNA-SSR markers in the narrow-leaved vetch(Viciasativasubsp.nigra) using 454 pyrosequencing [J].MolecularBreeding,2014,33(3):749-754.
[13]Russell J R,Bayer M,Booth C,etal.Identification,utilisation and mapping of novel transcriptome-based markers from blackcurrant(Ribesnigrum) [J].PlantBiology,2011,11:147-158.
[14]Iehisa J C M,Shimizu A,Sato K,etal.Genome-wide marker development for the wheat D genome based on single nucleotide polymorphisms identified from transcripts in the wild wheat progenitorAegilopstauschii[J].TheoreticalandAppliedGenetics,2014,127(2):261-271.
[15]李晨旭,刘志涛,庄丽芳,等.基于RNA-seq的百萨偃麦草染色体特异分子标记开发与应用 [J].中国农业科学,2015,48(6):1052-1062.
Li C X,Liu Z T,Zhuang L F,etal.Development and application of specific molecular markers ofThinopyrumbessarabicumbased on RNA-seq [J].ScientiaAgriculturaSinica,2015,48(6):1052-1062.
[16]Kantety R V,Rota M L,Matthews D E,etal.Data mining for simple sequence repeats in expressed sequence tags from barley,maize,rice,sorghum and wheat [J].PlantMolecularBiology,2002,48:501-510.
[17]陈军方,任正隆,高丽锋,等.从小麦EST序列中开发新的SSR引物 [J].作物学报,2005,31(2):154-158.
Chen J F,Ren Z L,Gao L F,etal.Developing new SSR makers from EST of wheat [J].ActaAgronomicaSinica,2005,31(2):154-158.
[18]王 东,曹玲亚,高建平.党参转录组中SSR位点信息分析 [J].中草药,2014,45(16):2390-2394.
Wang D,Cao L Y,Gao J P.Data mining of simple sequence repeats inCodonopsispilosulatranscriptome [J].ChineseTraditionalandHerbalDrugs,2014,45(16):2390-2394.
收稿日期:2015-12-15修回日期:2016-01-22
基金项目:山东省良种工程农业生物资源利用项目
通讯作者:李兴锋(E-mail:lixf@sdau.edu.cn)
中图分类号:S512.1;S330
文献标识码:A
文章编号:1009-1041(2016)06-0699-09
Development of Specific Molecular Markers forThinopyrumintermediumUsing RNA-seq Data
CUI Yu1,BAO Yinguang1,WANG Honggang1,2,LI Xingfeng1,2
(1.College of Agronomy,Shandong Agriculture University,Tai’an,Shandong 271018,China;2.Tai’an Sub-center of National Wheat Improvement Center,Tai’an,Shandong 271018,China)
Abstract:Thinopyrum intermedium,one of the important relatives of common wheat,has good tolerance to environmental stress and resistance to diseases such as rust and powdery mildew.Development of specific molecular markers is valuable for transformation and application of the favorable genes from Th.intermedium.In order to develop an efficient method of screening the specific molecular markers in Th.intermedium,the RNA-seq data of Th.intermedium and its genome donors,including Th.elongatum,Th.bessarabicum and Pseudoroegneria strigosa were used for developing EST-SSR markers.Two hundred pairs of EST-SSR primers were synthesized and verified through amplifying different DNA materials,and 75(37.5%) primers have specific amplification in Th.intermedium,Th.elongatum,Th.bessarabicum and Ps.strigosa with good polymorphism.The result indicates that developing EST-SSR primers from RNA-seq would be efficient in screening specific molecular markers of Th.intermedium,which can be used in developing specific molecular markers for other crops and their related species.
Key words:Thinopyrum intermedium; Wheat; RNA-seq; Specific molecular markers
网络出版时间:2016-05-30
网络出版地址:http://www.cnki.net/kcms/detail/61.1359.S.20160530.1535.006.html
第一作者E-mail:cuiyu112311@126.com

