应用不对称PCR提高PRRSV-CSFV-JEV共检芯片的杂交效率
张 仙,邓 静,常晓霞,杨国淋,马 锐,滑 翔,赵玉佳,欧阳达,文心田,2,黄小波,2,曹三杰,2,文翼平,2,伍 锐,2
应用不对称PCR提高PRRSV-CSFV-JEV共检芯片的杂交效率
张仙1,邓静3,常晓霞1,杨国淋1,马锐1,滑翔1,赵玉佳1,欧阳达1,文心田1,2,黄小波1,2,曹三杰1,2,文翼平1,2,伍锐1,2
1.四川农业大学动物医学院,动物传染病与基因芯片实验室,雅安625014;2.四川农业大学人兽共患病研究室与猪病防治研究中心,成都611130;3.四川华神兽用生物制品有限公司,四川畜科生物制品有限公司,成都610299
摘要:目的比较采用普通RT-PCR和不对称PCR分别进行样品的直接荧光标记,应用于cDNA芯片杂交,分析其对芯片杂交效率的影响。方法以本实验室建立保存的重组质粒和病毒为模板,进行不对称RT-PCR引物浓度优化,应用普通RT-PCR与不对称RT-PCR进行直接荧光标记,将标记的产物与制备的cDNA芯片杂交,在相同条件下完成杂交后芯片的洗涤与扫描,记录扫描图片与数据。结果与普通RT-PCR标记方法相比,应用不对称RT-PCR技术标记单链靶基因,能特异有效提高芯片的杂交效率。结论应用不对称RT-PCR技术对样品进行荧光标记后,与cDNA芯片杂交能提高芯片的杂交效率,有利于cDNA芯片的检测应用。
关键词:不对称PCR;cDNA芯片;标记
Supported by the Special Fund for Agro-scientific Research in the Public Interest of China(No. 201203056)
基因芯片技术是近年来新兴起的一种生物高新技术,该技术可以同时定量或定性的检测成千上万个基因信息,具有无选择性、客观、高通量的特点。已有的资料表明随着基因组数据的增多、分子生物学的快速发展,基因芯片技术在疾病诊断[1]、新药开发[2]、基因组研究[3]等生物科学领域中广泛应用。
基因芯片的杂交原理是点制在固相载体上的核苷酸探针与其标记互补的样本核酸杂交。单链DNA由于不存在互补双链的竞争性结合,其杂交效率及检测灵敏性高。单链DNA制备的方法主要包括:磁珠捕获法[4]、人工合成法、M13噬菌体扩增法[5]和不对称PCR法[6]。不对称PCR(asymmetric PCR)是用不等量的一对引物,PCR扩增后产生大量的单链DNA。不对称PCR法操作简便,成本低而应用比较广泛[7-9]。本研究选用直接引物标记进行常规PCR和不对称PCR靶基因扩增,杂交后根据扫描图片及数据分析不对称PCR对cDNA芯片杂交效率的影响。
1材料与方法
1.1重组质粒菌T/ PS9,PRRSV 5’UTR,US-type;T/Yll,PRRSV ORF6,US-type;T/CSF1,CSFV 5’UTR;T/PS9,CSFV/PS8;T/PRM,JEV;T/JEV,JEV 均由本实验室构建并保存。
1.2引物探针制备和靶基因扩增引物参照本实验室张焕容[10],肖驰[11]报道的方法设计的特异性引物(表1),定位基因的扩增参照曹三杰[12]报道的方法设计引物(表2),引物由宝生物工程有限公司合成,其中反向引物合成两条,一条无标记,一条5’端用Cy3标记修饰。

表1 探针制备和靶基因扩增引物

表2 定位基因引物
1.3PRRSV-CSFV-JEV共检芯片的制备将保存的探针基因重组质粒菌(T/Yll、T/ PS9、T/PS8、T/CSF1、T/PRM、T/JEV)分别进行复苏,然后按照Omega小量质粒提取试剂盒说明方法提取质粒。将提取的质粒作1∶50稀释后作为模板进行PCR扩增,同时按照曹三杰[10]的报道,以λDNA为模板进行定位探针的PCR扩增,扩增产物采用乙醇醋酸钠法进行纯化。将纯化好的探针浓度稀释至500 ng/μL,然后用点样缓冲液将探针浓度调整至250 ng/μL。按照设计好的检测阵列的排布(每张基片设置4个相同的9×9点阵,如图1)和性能参数,用晶芯®SmartArrayerTM16微阵列芯片点样仪在氨基基片上进行接触式点样。点制完成的基片在预设为80 ℃的PCR仪中烘焙8 h,紫外交联30 min,然后60~80 ℃重新水化10 s后,1 000 r/min;0.2%的SDS剧烈清洗3 min后,再用蒸馏水清洗2 min后离心干燥,密封、4 ℃保存。
1 and 9: Positive quality; 2: Negative control; 3: PRM; 4: JEV;5: PS8; 6: CSF1; 7: PS9; 8: Y11.
1.4基因芯片的有效性检测单独以λDNA为模板进行常规的PCR扩增标记,再以保存的T/Yll、T/PS9、T/PS8、T/CSF1、T/PRM、T/JEV重组质粒为模板进行PCR扩增标记,将标记产物与制备的共检芯片进行初步的杂交检测,验证基因芯片的有效性。
1.5病毒核酸的提取采用天根总RNA提取试剂盒(柱)对处理的组织样本进行总RNA提取,并将提取的RNA反转录为cDNA,反转录产物于-20 ℃保存备用。
1.6单链和对照样品的制备用dd H2O将未进行荧光标记的引物(即上游引物)进行稀释,使得PRM、JEV、CSFV、PS8、Y11、PS9基因的反向引物和非荧光标记引物比例分别为50∶1、40∶1、80∶1、80∶1、20∶1、10∶1,用浓度不同的引物分别进行PCR扩增,反应体系为:10×Ex buffer(Free Mg2+)25.0 μL,25 mmol/L MgCl22.0 μL,10.0 mmol/L dNTP Mixture 2.0 μL,5 U/μL Ex Taq 0.3 μL,非荧光标记的上游引物1 μL,5′端Cy3标记的下游引物1 μL,cDNA模板1 μL,ddH2O加至25 μL。扩增条件为: 94 ℃ 变性3 min; 94 ℃ 变性30 s,56.4 ℃退火30 s,72 ℃延伸30 s,共35个循环;72 ℃延伸10 min,12 ℃保持。
1.7芯片杂交及洗涤取制备保存的基因芯片,95~100 ℃双蒸水中变性1~2 min。其间上下抽提以免在玻片表面形成气泡,立即置95%乙醇中快速冷却后离心干燥,将芯片置于杂交舱中,于芯片点样区加入预杂交液25 μL,将盖玻片轻放其上,以免形成气泡,使预杂交液均匀覆盖芯片区,将芯片置于密封的湿盒内于44 ℃下预杂交1 h。吸除预杂交液,将利用单重PCR的标记好的核酸样品和多重PCR标记的核酸样品分别于95 ℃变性3 min 后,立即置冰中冷却5 min,再与一定量预冷的杂交缓冲液混匀,取该混合液20~40 μL 加到芯片区,以盖玻片轻放其上,将芯片放入杂交舱内于一湿盒内避光杂交,于48 ℃杂交温度下杂交2 h 时间。杂交完全后,在50 ℃预热的洗液1、洗液2、洗液3、双蒸水依次洗涤3 min,1 000 g离心干燥后备用。
1.8扫描及结果分析本实验选用的标记物为Cy3,故选用绿色532 nm单通道荧光,扫描图像以JPG格式保存,扫描数据以数据分析软件LuxScan3.0获取信息,分析结果以*.LSR文件格式保存。本研究引入SNRm(样点信号值的以2.0为底的对数与背景信号值对数的比值),来判定每个样点的杂交结果,信号值大于1 000并且SNRm值高于2.0则判定为阳性,否则为阴性。
2结果
2.1探针质粒鉴定结果各探针基因重组质粒菌(T/Yll、T/PS9、T/PS8、T/CSF1、T/PRM、T/JEV)分别进行复苏,提取的质粒分别扩增出了预期大小的片段(Y11:155 bp、JEV:238 bp、PRM:451 bp、PS8:248 bp、PS9:179 bp、CSFV:143 bp),2%琼脂糖凝胶电泳如图2所示:
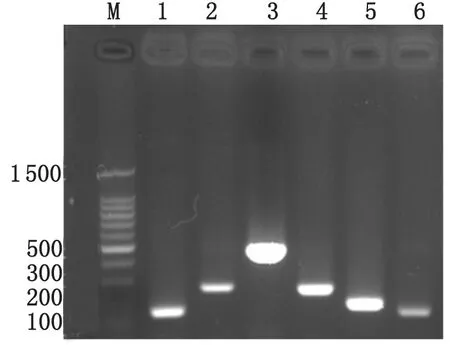
M: 100 bp Ladder DNA Marker; Lane 1: Y11; Lane 2: JEV; Lane 3: PRM; Lane 4: PS8; Lane 5: PS9: Lane 6: CSF1.
2.2基因芯片的有效性检测结果以λDNA定位基因为模板进行靶基因扩增标记,将扩增产物与杂交缓冲液以1∶1混合后与点制芯片杂交。扫描后,图像(图3A)和数据显示,阳性质控λDNA定位基因杂交斑点明显可见,信号值大于1 000,SNRm值高于2.0,阴性对照和其它探针斑点不明显或没有,表明制备的基因芯片、阳性定位基因质量可靠。6个探针基因的全部探针与基因芯片杂交后,相应的探针位点均可以看到明显的杂交斑点,阳性对照斑点明显,信号值均大于1 000,SNRm值高于2.0,表明制备的共检芯片、选用探针基因质量可靠(图3B)。

A:阳性定位基因的有效性检测;B:探针基因的有效性检测
2.3探针质粒杂交结果由图4(A)、(B)、(C)、(D)、(E)、(F)所示cDNA芯片与双链荧光标记样品杂交后,其相应的探针均出现了阳性信号。图4(a)、(b)、(c)、(d)、(e)、(f)所示cDNA芯片与单链荧光标记样品杂交后,其相应的探针也均出现了阳性信号,但信号明显强于常规PCR标记杂交(表3)。
3讨论
目前,临床上常用的检测方法主要有3种:病原分离、血清学方法和分子生物学方法。血清学检测如酶联免疫吸附试验(ELISA)、免疫组化(IHC)和荧光抗体试验(FAT)等;分子生物学检测聚合酶链式反应(PCR)、原位杂交(ISH)、基因芯片检测和环介导等温扩增技术(LAMP)等。基因芯片被首次研究报道是在1995年,从此之后得到了快速的发展和应用[13]。基因芯片根据探针的不同可分为寡聚核苷酸芯片和cDNA芯片,寡聚核苷酸芯片由于探针较短,灵敏性更高,但是容易产生假阳性;cDNA芯片由于探针和靶基因是互补的片段,因此准确性更高[14]。

A: Conventional PCR result of PRM gene; a: asymmetric PCR result of PRM gene; B: Conventional PCR result of JEV gene; b: asymmetric PCR result of JEV gene; C: Conventional PCR result of PS8 gene; c: asymmetric PCR result of PS8 gene; D: Conventional PCR result of CSF1 gene; d: asymmetric PCR result of CSF1 gene; E: Conventional PCR result of PS9 gene; e: asymmetric PCR result of PS9 gene; F: Conventional PCR result of Y11 gene; f: asymmetric PCR result of Y11 gene.
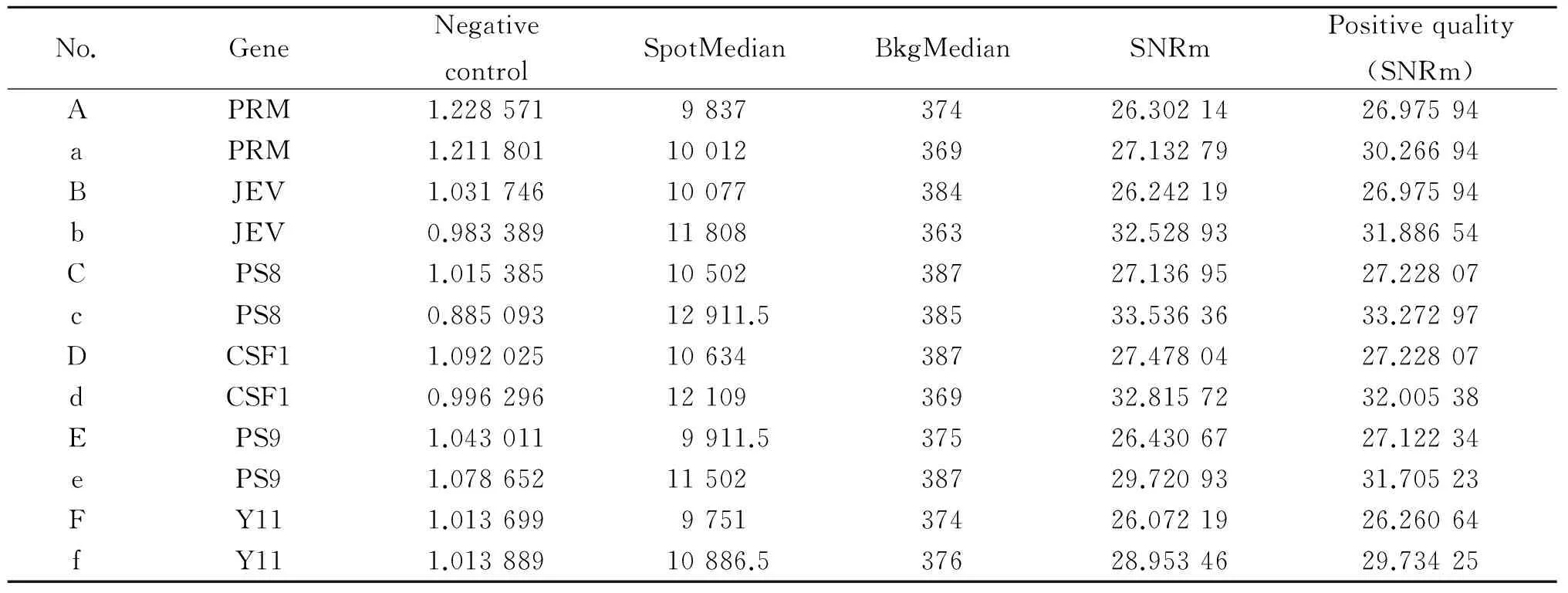
表3 常规PCR与不对称PCR的杂交结果
基因芯片在动物疫病的诊断方面有其独特的优势。1)同时对多个样品进行平行检测;2)分析过程中可以根据研究目的选用多色荧光(最多为4种)还是单色荧光对样品进行标记;3)检测所需样本量小,减少试剂损耗;4)灵敏度高,特异性强;5)研究的整个过程可完全自动化,使检测结果更为客观、准确。
由于生物芯片技术发展时间较短,很多技术还处于初级研发阶段,要成为实验室研究或临床可以普遍采用的技术仍有诸多关键问题亟待解决。如:1)基因芯片的特异性的提高;2)增加信号检测的灵敏度;3)简化样品制备和标记操作;4)设备与操作人员的要求较高;5)应用标准的统一等。本研究致力于提高信号检测的灵敏度而进行不对称PCR标记与常规PCR标记进行比较。
基因芯片的杂交原理是点制在固相载体上的核苷酸探针与其标记互补的样本核酸杂交。稳定的DNA双链与探针基因杂交时存在竞争性抑制,影响探针和靶基因的结合能力和稳定性,进一步影响cDNA芯片的分辨能力和结果的可信度。另外,高温解链后的单链PCR产物,一部分单链直接又复性成双链,一部分引物与单链靶序列复性,在TaqDNA聚合酶作用下又合成双链,两种反应相互竞争进一步导致单链靶基因的量减少。因此直接合成只用于杂交的一条链用于杂交可以排除双链的的干扰,提高杂交效率。
Wang等[15]运用不对称PCR方法分析了关于提高芯片杂交效率的研究,结果显示,在相同条件下不对称PCR标记方法相对于常规PCR更能扩增出大量的荧光标记的产物。孟祥贤等[16]应用金纳米颗粒介导不对称PCR,产生了大量的单链。不对称PCR技术可以通过调节上下游引物的量、引物的退火温度(Tm值)以及引物的长度的不同来扩增大量的单链DNA。而其中应用最广也最为关键的是限制性引物与非限制性引物的比例,因为应用不对称PCR虽可以获得大量的单链DNA,但非限制性引物的量多或少均可以影响PCR的扩增效果,所以摸索出引物最适比例,对提高杂交效率十分重要。
本研究在本实验室前期研究的基础上选用直接引物标记进行常规PCR和不对称PCR靶基因扩增。不对称PCR电泳结果、芯片扫描图像和数据分析显示,应用不对称PCR技术对样品进行标记后,芯片的杂交效率有所提高。PRM、JEV、CSFV、PS8、Y11、PS9基因的反向引物和非荧光标记选用的引物比例分别为50∶1、40∶1、80∶1、80∶1、20∶1、10∶1,这是在本研究进行之前经过长期摸索而探索的条件。研究结果显示在上述条件下杂交检测信号值和信噪比均为最强。相对于常规PCR,不对称PCR扩增可以明显提高杂交效率。
参考文献:
[1]Luo Y, Du J, Zhan Z, et al. A diagnostic gene chip for hereditary spastic paraplegias[J]. Brain Res Bull, 2013, 97: 112-118. DOI: 10.1016/j.brainresbull.2013.07.002
[2]Fu Y, Pan Y, Pan M, et al. Development of a high-throughput DNA microarray for drug-resistant gene detection and its preliminary application[J]. J Microbiol Methods, 2012, 89(2): 110-118. DOI: 10.1016/j.mimet.2012.02.010
[3]Dally S, Lemuth K, Kaase M, et al. DNA microarray for genotyping antibiotic resistance determinants inAcinetobacterbaumannii clinical isolates[J]. Antimicrob Agents Chemother, 2013, 57(10): 4761-4768. DOI: 10.1128/AAC.00863-13
[4]Wang CH, Chang CJ, Wu JJ, et al. An integrated microfluidic device utilizing vancomycin conjugated magnetic beads and nanogold-labeled specific nucleotide probes for rapid pathogen diagnosis[J]. Nanomed: Nanotechnol, Biol Medi, 2014, 10(4): 809-818. DOI : 10.1016/j.nano.2013.10.013
[5]Jin HE, Farr R, Lee SW. Collagen mimetic peptide engineered M13 bacteriophage for collagen targeting and imaging in cancer[J]. Biomaterials, 2014, 35(33): 9236-9245. DOI: 10.1016/j.biomaterials.2014.07.044
[6]Venkatesan V, Hoti SL, Kamaraj N, et al. Optimisation of an asymmetric polymerase chain reaction assay for the amplification of single-stranded DNA fromWuchereriabancroftifor electrochemical detection[J]. Memorias do Instituto Oswaldo Cruz, 2013, 108(6): 804-807. DOI: 10.1590/0074-0276108062013020
[7]Hopkins M, Kailasan S, Cohen A, et al. Diversity of environmental single-stranded DNA phages revealed by PCR amplification of the partial major capsid protein[J].ISME J, 2014. DOI: 10.1038/ismej.2014.43
[8]Hong T, Wang T, He Z, et al. Qualitative and quantitative detection of methylation at CpG sites using the fluorescein-dGTP incorporated asymmetric PCR assay strategy[J]. Chem Commun, 2014, 50(50): 6653-6655. DOI: 10.1039/C4CC01933D
[9]Dally S, Rupp S, Lemuth K, et al. Single-stranded DNA catalyzes hybridization of PCR-products to microarray capture probes[J]. PLoS One, 2014, 9(7): e102338. DOI: 10.1371/journal.pone.0102338
[10]Zhang HR, Cao SJ, Wen XT, et al. Preparation PRV, PPV, and JEV detection microarray[J].Chin J Vet Sept, 2007, 27(5): 667-670.(in Chinese)
张焕容,曹三杰,文心田,等. 伪狂犬病病毒, 猪细小病毒和流行性乙型脑炎病毒检测基因芯片的制备[J].中国兽医学报, 2007, 27(5): 667-670.
[11]Xiao C, Cao SJ, Wen XT, et al. Development of PRRS-CSF-PCV2 Diagnostic DNA Microarray[J].Acta Veterinaria et Zootechnica Sinica, 2006, 37(8): 799-803.(in Chinese)
肖 驰,曹三杰,文心田,等. PRRS-CSF-PCV2诊断DNA芯片构建研究[J]. 畜牧兽医学报, 2006, 37(8): 799-803.
[12]Cao SJ, Wen XT, Xiao C, et al. Preparation and preliminary application of diagnostic gene chip of several avian infectious diseases[J].Acta Veterinaria et Zootechnica Sinica, 2006, 37(4): 356-360.(in Chinese)
曹三杰,文心田,肖驰,等. 几种主要禽疫病诊断基因芯片的制备及初步应用[J]. 畜牧兽医学报, 2006, 37(4): 356-360.
[13]Schena M, Shalon D, Davis RW, et al. Quantitative monitoring of gene expression patterns with a complementary DNA microarray[J].Science, 1995, 270(5235): 467-470. DOI: 10.1126/science.270.5235.467
[14]Margalit O, Somech R, Amariglio N, et al. Microarray-based gene expression profiling of hematologic malignancies: basic concepts and clinical applications[J].Blood Rev, 2005, 19(4): 223-234. DOI: 10.1016/j.blre.2004.11.003
[15]Wang XW, Zhang L, Jin LQ, et al. Development and application of an oligonucleotide microarray for the detection of food-borne bacterial pathogens[J]. Appl Microbiol, 2007, 76(1): 225-233. DOI: 10.1007/s00253-007-009-x
[16]Meng XX, Yang XH, Wang KM, et al. Gold nanoparticle-based asymmetric PCR for single-strand DNA[J]. Acta Chimica Sinica, 2010, 68(9): 917-920.(in Chinese)
孟祥贤,羊小海,王柯敏,等. 金纳米颗粒介导不对称 PCR: 制备单链核酸的新方法[J].化学学报, 2010, 68(9): 917-920.
DOI:10.3969/j.issn.1002-2694.2016.01.011
通讯作者:文心田,Email:xintian@sicau.edu.cn
中图分类号:Q789
文献标识码:A
文章编号:1002-2694(2016)01-0051-05
Corresponding author:Wen Xin-tian, Email:xintian@sicau.edu.cn
收稿日期:2015-04-27;修回日期:2015-08-17
Enhancing the hybridization efficiency of the PRRSV-CSFV-JEV microarray by asymmetric PCR
ZHANG Xian1,DENG Jing3,CHANG Xiao-xia1,YANG Guo-lin1,MA Rui1,HUA Xiang1,ZHAO Yu-jia1,OUYANG Da1,WEN Xin-tain1,2,HUANG Xiao-bo1,2,CAO San-jie1,2,WEN Yi-ping1,2,WU Rui1,2
(1.LaboratoryofAnimalInfectiousDiseaseandMicroarray/KeyLaboratoryofAnimalDiseaseandHumanHealthofSichuanProvince,CollegeofVeterinaryMedicine,SichuanAgriculturalUniversity,Ya’an625014,China;2.LaboratoryofZoonosisandSwineDiseasePreventionResearchCenter,SichuanAgriculturalUniversity,Chengdu611130,China;3.SichuanHuashenVeterinaryBiologicalProductsCo.,Ltd,SichuanXukeBiologicalProductsCo. ,Ltd.,Chengdu610299,China)
Abstract:Microarray is wildly used in every field of biology as a tool of molecular biology research, with advantages of accuracy, fast and high-throughput. The hybridization efficiency is the critical factor of the microarray technology. We studied the hybridization efficiency of two fluorescence labeling methods by microarray hybridization. On the basis of our laboratory research, we selected T/PRRS-PS9, T/Yll, T/CSF1, T/PS8, T/PRM, and T/JEV as template, to optimize the concentration of asymmetric RT-PCR primer. The templates were come from recombinant plasmids which were established and saved in our laboratory. The templates would be amplified by traditional PCR and the amplification would be the probes of the cDNA microarrays. The samples used to be labeled by fluorescence, we amplified and labeled the samples from clinic by means of asymmetric PCR and traditional PCR labeling technology, they hybridized with the cDNA microarrays we prepared respectively, followed by washing, scanning and analysis. Results indicated that compared to the traditional RT-PCR labeling method, the asymmetric RT-PCR labeling method showed superior results with enhance hybridization efficiency. The hybridization efficiency could be enhanced by asymmetric PCR labeling method, which improving the detection applicability of microarray technology.
Keywords:asymmetric PCR; cDNA microarray; fluorescence labeling
国家公益性行业(农业)科研专项(No.201203056)

