椰纤果产生菌株分离鉴定和小型菌种库的建立
刘龙祥,刘四新,2,陈华美,余敏华,陈白羽,毕继才,李从发,2
椰纤果产生菌株分离鉴定和小型菌种库的建立
刘龙祥1,刘四新1,2,陈华美1,余敏华1,陈白羽1,毕继才1,李从发1,2
(1.海南大学食品学院,海南 海口570228;2.海南大学热带生物资源教育部重点实验室,海南 海口570228)
摘 要:从海南省内外的椰纤果生产企业代表性的布点采样、分离以及交换等方式共获得145株细菌纤维素产生菌,对其中93株采用持家基因、16SrDNA 基因等序列分析结合形态特征进行鉴定,引入近缘的7株不同种的模式菌株作为参照。结果显示,这些菌株被鉴定为驹形氏杆菌属(Komagataeibacter),其中79株分别鉴定为3个已知种:K. nataicola、K. europaeus、K. hansenii,另14株为未知种。采用持家基因dnaK、rpoB和groEL对14株未知菌进行鉴定,发现它们两两序列相似度均大于种间差异的临界值99.0 %、98.8%和99.2%,推测该14株菌可能为驹形氏杆菌属的同一个新种。实验室目前的细菌纤维素产生菌包含模式种在内至少有9个已知种、还有至少1个疑似新种。小型菌种库还在持续建设中,这为保护我国椰纤果生产的菌种资源、优良菌株的遗传选育、椰纤果发酵生产的技术升级以及细菌纤维素的相关理论研究等奠定了基础。
关键词:椰纤果;细菌纤维素;菌种分离鉴定;菌种库建立;持家基因;16S rDNA序列分析
椰纤果为食品工业中广泛应用的细菌纤维素(Bacterial cellulose,BC),在生物医药和纳米材料等方面具有重要应用价值[1-3]。国内外对BC的研究一直很活跃[4-7],其中提高产量是目前研究的热点(选育优良菌株)。海南省是我国椰纤果商品化生产基地,随着市场需求的不断扩大,省外也建有相关生产企业,在发展高峰期企业数达近百家。但业界对菌种的来源、种属关系、生物学特性、安全性等重视程度不够。目前,普遍认为产BC的微生物都是木醋杆菌(Acetobacter xylinum)或木葡糖酸醋杆菌(Gluconacetobacter xylinus),并往往随意更换、混用菌种或长期传代,导致生产性能退化、发酵不稳定、产率降低等问题。越南生产的椰纤果因椰子水充足、劳动力低廉而具有明显价格优势。近年随着边贸不断活跃,椰纤果违规进口的现象也不在少数,巨大的价格差一度对海南椰纤果生产行业产生巨大冲击,不少企业被迫关停椰纤果发酵生产车间,改为进口压缩果片,进行简单复水切割后再销售。椰纤果生产企业这种逐渐收缩的现状对盛产椰子的海南的经济发展是很不利的。另一方面,国内生产企业缺少对菌种资源的妥善保护,随意管理甚至遗失菌种,缺少对BC产生菌分类地位、独特生物学性质的重视[5,9-12],尽管近十多年来国内已有大量BC产生的菌株筛选和鉴定的报道[5,8-12],但缺少类似国外的系统研究[13-15],因此对BC产生菌进行收集、分离鉴定和菌种资源保护具有重要研究价值和实际应用意义。
早在20世纪末,日本Yamada等[16-17]将产生泛醌Q10的菌株从醋杆菌属(Acetobacter)中划分出来,建立了一个新属Gluconacetobacter(葡糖酸醋杆菌属),根据形态和化学特征,结合16S rRNA基因系统发育分析,将葡糖酸醋杆菌属中一些种分出,并归入新属驹形氏杆菌属(Komagataeibacter)中。因此,企业界熟知的模式种木醋杆菌(A. xylinum)或木葡糖酸醋杆菌(G. xylinus)应是木驹形氏杆菌(K. xylinus)。
本实验室长期从事BC研究,通过分离、交换和购买保存了一些BC产生菌,鉴于企业界椰纤果菌种的不良管理现状,本文拟建立BC产生菌的菌种资源库,为深入研究其系统发育关系、进行高产高效菌株选育和BC其他相关理论研究提供菌种资源保证,为最终提高BC发酵水平奠定基础。
1 材料与方法
1.1 试验材料
菌株:除本实验室保存的菌株外,先后从国内外BC生产企业或研究机构收集、交换的样品为BC发酵液、种子液、或受污染的发酵液以及斜面种子等形式。于冰箱暂存以备分离、纯化。
从日本理化研究所生物资源中心的菌种保藏中心JCM(Japan Collection of Microorganisms)和日本国家技术和评估研究所的生物资源中心NBRC(Biological Resource Center,NITE)分别购买了5株和2株模式菌株作为鉴定用参照菌株,分别为Komagataeibacter nataicola JCM 25120T、Komagataeibacter xylinus JCM 7644T、Komagataeibacter sucrofermentans JCM 9730T、Komagataeibacter swingsii JCM 17123T、Komagataeibacter rhaeticus JCM 17122T和Komagataeibacter maltaceti NBRC 14815T、Gluconacetobacter diazotrophicus NBRC 110704T。
培养基:菌种分离培养基:(NH4)SO43 g/L,MgSO40.3 g/L,KH2PO40.3 g/L,新鲜椰子水(V/V)50%,加糖至折光糖度5%,琼脂粉 15 g/L,pH 5.0。
BC液体发酵试验培养基:(NH4)SO43 g/L,MgSO40.3 g/L,KH2PO40.3 g/L,发酵椰子水(V/V)50%,折光糖度5%,pH 4.0。其中,新鲜椰子水为市售近成熟的椰子,破壳取椰子水而得。发酵椰子水为新鲜椰子水经装瓶自然发酵7 d而得。二者均冻存于冰柜备用。
1.2 试验方法
1.2.1 菌种分离和形态观察 将采集的样品(斜面菌种先经活化)进行平板分离、纯化,挑取菌落形态各异的菌株接入斜面、适温培养。对污染物样品的分离物进行液体发酵培养,确认是否产生纤维素,然后进行革兰氏染色、镜检并拍照、测量菌体大小。对纯化培养的菌落进行特征观察、测量和拍摄。
1.2.2 基因序列扩增和分析 采用康为世纪生物科技有限公司的“细菌基因组DNA提取试剂盒”提取各菌株的基因组DNA。
扩增16S rDNA基因的引物为P1:AGAGTT TGATCCTGGCTCAG,Pr:GGCTACCTTGTT ACGACTT。
PCR扩增条件:94℃ 3 min;94℃ 1 min;52℃1 min;72℃ 2 min;72℃ 10 min;30个循环。
扩增dnaK基因的引物为dnaK-F:CTGCG CATCATCAACGAGCC,dnaK-R:CTCACGC TCGCCCTGATAGA。PCR扩增条件为:94℃ 3 min、94℃ 1 min、55℃ 1 min、72℃ 1.5 min、72℃ 10 min,30个循环[20]。
PCR产物送Invitrogen(上海)公司测序。登录EzTaxon Server核酸数据库进行16S rDNA基因序列比对,登录NCBI进行dnaK基因序列Blast比对。系统发育分析采用MAGA 5.0软件进行多序列匹配排列,采用Kimura2-parameter方法计算进化距离,采用Neighbor-joining方法,1000次随机抽样,以自引导值表示可信度,构建系统发育树。
2 结果与分析
2.1 菌株分离情况
先后分离或采集、交换获得各种形式样品18批次,经菌种分离和纯化,并经液体发酵合成BC,获得131个能产生纤维素的纯培养分离物,加上实验室原保存和交换的共计145个菌株(表1),对2015年前获得的93株进行分子鉴定和形态特征记录。
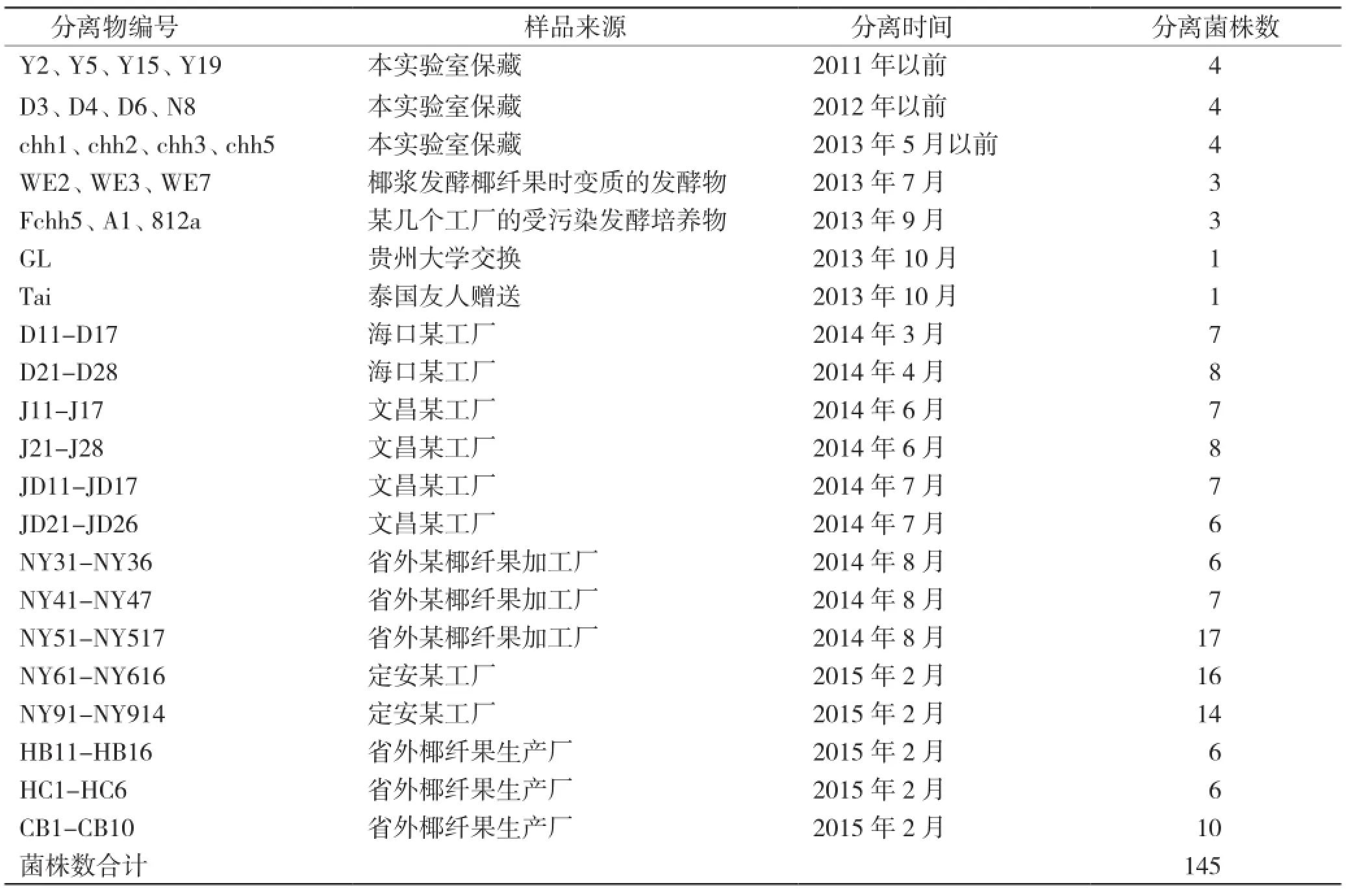
表1 样品来源和分离时间
2.2 菌株的形态特征
经对供试菌株进行培养(3 d)和形态观察,根据菌落是否透明、隆起、粘稠及将菌落挑起置于无菌水中的乳化情况等,将所有产生纤维素的供试菌株分为5种类型(表2)。

表2 供试菌株的菌落特征类型
针对每类型菌落特征的菌株,挑选BC产量最高的菌株进行染色和显微观察,发现所有菌株均呈革兰氏阴性反应,但菌体大小不一、杆状至短杆状,单个或成对排列(表3)。

表3 根据显微形态特征划分的菌株类型
2.3 16S rDNA基因序列分析
本研究对93个菌株的16S rDNA基因进行克隆、测序,其序列长度在1 339~1 408 bp之间,登录EzTaxon Server进行比对分析,发现其序列相似度在99.56%~100%之间。驹形氏杆菌属(Komagataeibacter)内不同种的16S rDNA基因序列相似度极高,达98.2%~100%[20],说明该属内菌种鉴定时若单一使用该指标则存在较大局限,其不具有区分种的鉴定意义。因此本研究利用多位点序列分析技术对分离菌株的某些持家基因进行了分析[20]。它们在确定某些近缘种亲缘关系时更有效,如dnaK基因、rpoB基因和groEL基因的序列分析被用于驹形氏杆菌属的菌种鉴定[20]。
虽然报道称Komagataeibacter属内种的16S rDNA 同源性极高,但其是一个基本的特征数据。本研究对分离获得的菌株都进行了16S rDNA基因的克隆、测序(序列结果略),并结果全部菌株的16S rDNA如报道所述、都极高,说明16S rDNA基因序列特征对区分属内种的鉴定意义不大。
2.4 dnaK基因序列及系统发育分析
本研究首先对购买的7株模式菌株和本实验室前期保藏的12株BC产生菌分别进行了3个持家基因dnaK(编码一种热休克蛋白)、rpoB(编码RNA聚合酶β亚基)和groEL(编码一种伴侣蛋白)[19-21]的扩增、比对及系统发育分析。结果发现所有模式菌株的这3个持家基因的序列相似性与NCBI中的数据比对相似度均为100%或者略低于100%,都高于这3个基因的临界相似值(其临界值依次为99.0 %、98.8 %、99.2 %);同时,12株保藏菌的3个持家基因的相似性结果表明,这3个持家基因分别都可以单独达到种水平的区分。因此本文选用dnaK基因作为其他81个菌株首期的鉴定指标,对其相似度低于99%的分离物则增加rpoB 和groEL 两个基因的序列鉴定。
登录NCBI数据库对所有菌株的dnaK基因序列(798~821bp)进行比对,以Acetobacter aceti LMG 18491T 为外围参考菌株构建系统发育树[22]。结果发现,虽然93株菌之间的16S rDNA基因相似度极高,但其dnaK基因序列与NCBI数据库中菌株的相似度水平却不尽相同,其中有14株菌低于99.0 %的临界值,而其它79株与NCBI数据库中菌株相似度在99%~100%之间,显示为已知种。通过构建系统发育树(图1,只显示了部分代表性菌株)分析可知,该79株可初步鉴定为K. nataicola、K. europaeus和K. hansenii,而有14株与所有已知种都不能聚到一簇,显示其为Komagataeibacter属的疑似新种。再对14株菌的dnaK基因序列进行两两比对分析,结果显示其相似度均大于99.0 %,说明这14株菌可能为Komagataeibacter属内同一个新种。对该14株菌再进行rpoB和groEL 两个基因序列分析鉴定发现,其序列相似性都低于相应区分种的阈值,显示该14株菌为疑似新种。
3 结论与讨论
本研究通过各种方式采集了18批次椰纤果样品或者菌株,通过分离纯化及实验室保藏菌株共获得145株细菌纤维素产生菌,在国内建立了椰纤果生产和BC研究的首个小型菌种库。通过形态观察和分子序列分析鉴定了2015年前获取的93株菌。研究结果显示,在Komagataeibacter内进行菌种鉴定时不宜使用16S rDNA基因序列,而宜采用dnaK、rpoB和groEL等持家基因序列。其次,所有93株菌都被鉴定为驹形氏杆菌属(Komagataeibacter),包含3个已知种:K. nataicola,K. europaeus、K. hansenii(79株、占85%)和1个疑似新种(14株、约占15%)。这14株分离物的菌落形态特征包含B和E两类,菌体形态特征包含a、b和e3类。以持家基因序列分析法发现该14株菌可能是Komagataeibacter属的同一个疑似新种。
本研究中样品多从企业环境收集、分离而来,其中已知种中有77株被鉴定为K. nataicola,未发现被誉为BC生产和研究的“明星菌种”K. xylinus。本文收集样品时被告知是木醋杆菌(A. xylinum)或者木葡糖酸醋杆菌(G. xylinus),可能的原因有:(1)原种K. xylinus中的某些菌株不纯,由于长期传代、用于nata de coco生产,加之含椰子水的培养基对K. nataicola具有富集与筛选作用,使K. xylinus逐渐被“稀释”,而只留下K. nataicola一个种;(2)原来生产实践中流传的名称本不准确。这可能由于K. xylinus最早被研究、被接受和认识,也曾是Acetobacter、Gluconacetobacter研究和生产的模式种,产BC能力很强,因此大家就推测产椰纤果的就应该是K. xylinus;(3)海南也有企业曾送其生产菌种到国内权威机构鉴定,曾被鉴定为Gluconacetobacter xylinus,而这可能与其主要使用16S rDNA基因作为鉴定指标有关。因此,对椰纤果/BC产生菌的鉴定引入基于持家基因测序的MLSA法是必要的,也是可行的。本研究为椰纤果的生产提供了菌种资源,为BC相关深入研究奠定了良好基础,也为企业发酵生产的技术进步、升级改造提供了有益借鉴。

图1 基于dnaK基因的系统发育树
参考文献:
[1] A Svenssona,T Harraha,B Panilaitisa,et al. Bacterial cellulose as a potential scaffold for tissue engineering of cartilage[J]. Biomaterials,2005,26(4):419-431.
[2] Fábia K,Andrade N A,Irina A,et al. Studies on the
biocompatibility of bacterial cellulose[J]. Journal ofBioactive and Compatible Polymers,2013,28(1):97-112.
[3] Sybele S L,Paulo T O,Ana M M,et al. Bacterial cellulose-collagen nanocomposite for bone tissue engineering[J]. Journal of Materials Chemistry,2012,22(41):22102-22112.
[4] 冯劲,施庆珊,冯静. 不同干燥方式对细菌纤维素物理性能的影响[J]. 现代食品科技,2013,29(9):2225-2229.
[5] 许燕娜,张剑恩,黎嘉惠,等. AcetobacterxylinumCG MCC5173发酵生产细菌纤维素的条件优化[J]. 现代食品科技,2012,28(11):1535-1540.
[6] Aleshin A N,Berestennikov A S.,Krylov P S,et al. Electrical and optical properties of bacterial cellulose films modified with conductive polymer PEDOT/PSS [J]. Synthetic Metals,2015,199:147-151.
[7] Rajwade J M,Paknikar K M,Kumbhar J V. Applications of bacterial cellulose and its composites in biomedicine[J]. Applied microbiology and biotechnology,2015,99(6):2491-2511.
[8] 刘四新,李枚秋. 椰子纳塔发酵条件研究[J]. 食品与发酵工业,1999,25(1):36-39.
[9] 邓毛程,王瑶,李静,等. 细菌纤维素产生菌的筛选、鉴定及其产物分析[J]. 河南工业大学学报(自然科学版),2014,5:88-92.
[10] 卢美欢,马英辉,王银存,等. 一株细菌纤维素生产菌的分离鉴定[J]. 中国酿造,2013,32(7):46-49.
[11] 周胜虎,薛齐佳,刘传凤,等. 细菌纤维素高产菌株的筛选和初步鉴定[J]. 湖北农业科学,2013,52 (15):3514-3517.
[12] 马霞,王瑞明,关凤梅,等. 细菌纤维素生产菌株的分离和菌种初步鉴定[J]. 工业微生物,2005,35 (3):23-26.
[13] Yamada Y. Transfer of Gluconacetobacter kakiaceti,Gluconacetobacter medellinensis and Gluconacetobacter maltaceti to the genus Komagataeibacter as Komagataeibacter kakiaceti comb. nov.,Komagataeibacter medellinensis comb. nov. and Komagataeibacter maltaceti comb. nov[J]. International journal of systematic and evolutionary microbiology,2014,64(5):1670-1672.
[14] Yamada Y,Yukphan P,Lan Vu H T,et al. Description of Komagataeibacter gen. nov.,with proposals of new combinations(Acetobacteraceae)[J]. The Journal of general and applied microbiology,2012,58(5):397-404.
[15] Yamada Y,Yukphan P. Genera and species in acetic acid bacteria[J]. International journal of food microbiology,2008,125(1):15-24.
[16] Yamada Y,Hoshino K I,Ishikawa T.The phylogeny of acetic acid bacteria based on the partial sequences of 16S ribosomal RNA the elevation of the subgenus Gluconoacetobacter to the generic level[J]. Bioscience,biotechnology,and biochemistry,1997,61 (8):1244-1251.
[17] Ya m a d a Y,H o s h i n o K I,I s h i k a w a T. Gluconacetobacter nom corrig.(Gluconoacetobacter [sic]). In validation of publication of new names and new combinations previously effectively published outside the IJSB,List No.64[J]. International journal of systematic bacteriology,1998,48(1):327-328.
[18] Yamada Y,Yukphan P,Lan Vu H T,et al. Description of Komagataeibacter gen. nov.,with proposals of new combinations(Acetobacteraceae)[J]. The Journal of general and applied microbiology,2012,58(5):397-404.
[19] Brady C.,Cleenwerck I.,Venter S.,et al. Phylogeny and identification of Pantoea species associated with plants,humans and the natural environment based on multilocus sequence analysis(MLSA)[J]. Syst Appl Microbiol,2008,31(6-8):447-460.
[20] Cleenwerck I,De Vos P,De Vuyst L. Phylogeny and differentiation of species of the genus Gluconacetobacter and related taxa based on multilocus sequence analyses of housekeeping genes and reclassification of Acetobacter xylinus subsp. sucrofermentans as Gluconacetobacter sucrofermentans (Toyosaki et al. 1996) sp. nov.,comb. nov[J]. Int J Syst Evol Microbiol,2010,60(10):2277-2283.
[21] De B K,Schillinger U,Caroline L,et al. Leuconostoc holzapfelii sp. nov.,isolated from Ethiopian coffee fermentation and assessment of sequence analysis of housekeeping genes for delineation of Leuconostoc species[J]. Int J Syst Evol Microbiol,2007,57(12):2952-2959.
[22] Yamada Y ,Yukphan P,Vu H L,et al. Subdivision of the genus Gluconacetobacter Yamada,Hoshino and Ishikawa 1998:the proposal of Komagatabacter gen. nov.,for strains accommodated to the Gluconacetobacter xylinus group in the α-Proteobacteria[J]. Annals of Microbiology,2011,62(2):849-859.
[23] Lisdiyanti P,Navarro R R.,Uchimura T,et al. Reclassification of Gluconacetobacter hansenii strains and proposals of Gluconacetobacter saccharivorans sp. nov. and Gluconacetobacter nataicola sp. nov[J]. Int J Syst Evol Microbiol,2006,56(9):2101-2111.
(责任编辑 白雪娜)
中图分类号:S963.21+1
文献标识码:A
文章编号:1004-874X(2016)03-0142-06
收稿日期:2015-05-28
基金项目:海南省自然科学基金(314048);海南省普通高校研究生创新科研项目(Hy2013-B02);海口市热带农产品深加工重点实验室项目(2013-45);海南大学研究生优秀学位论文培育计划项目
作者简介:刘龙祥(1988-),男,在读硕士生,E-mail:longxiang8802@163.com
通讯作者:李从发(1967-),男,教授,E-mail:Congfa@vip.163.com
Isolation,identification and library establishment of bacterial cellulose-producing strains in Nata de coco
LIU Long-xiang1,LIU Si-xin1,2,CHEN Hua-mei1,YU Min-hua1,CHEN Bai-yu1,BI Ji-cai1,LI Cong-fa1,2
(1. College of Food Science Hainan University,Haikou 570228,China;2. Tropical biological resources,key Laboratory of the ministry of education,Hainan University,Haikou 570228,China)
Abstract:145 bacterial cellulose-producing strains were obtained by isolation and collection from factories both at home and aboard of Hainan. 93 strains gained before 2015 were identified by phenotypic characteristics and molecular sequence features(house-keeping gene and 16S rDNA gene),with another 7 type strains being used as reference strains. The results showed that these strains belonged to 1 genus,Komagataeibacter,and 3 known species,with another 14 strains being unknown species. This essay proposed that we should take the house-keeping genes,such as dnaK,rpoB,groEL gene,as the identification standard of these 93 strains,because the similarity values of 16S rDNA gene were to be above 99.56%. By pair-wise comparisons of dnaK,rpoB and groEL sequence for the 14 strains,the similarity values were to be above 99.0%,98.8%,99.2%,which suggested that the 14 isolates may belong to the same species. Currently,the strains labrary was classified to cover 9 known species and 1 proposed new species. These strains form a small but distinctive library,which laied a solid foundation on the genetic improvement of excellent strains,the conservation of BC-producing culture resource,and intensive study of bacterial cellulose.
Key words:Nata de coco;bacterial cellulose;strain isolation and identification;library establishment;housekeeping gene;16S rDNA sequence analysis

