土壤微生物总活性研究方法进展
车荣晓,王 芳,王艳芬,邓永翠,张 静,马 双,崔骁勇,*
1 中国科学院大学,北京 100049 2 格里菲斯大学,布里斯班 4111 3 南京师范大学,南京 210023
土壤微生物总活性研究方法进展
车荣晓1,2,王芳1,王艳芬1,邓永翠3,张静1,马双1,崔骁勇1,*
1 中国科学院大学,北京100049 2 格里菲斯大学,布里斯班4111 3 南京师范大学,南京210023
摘要:微生物总活性是指在某一时段内微生物所有生命活动的总和,它直接决定着微生物行使生理、生态功能的能力,是微生物学研究的热点,也是难点。迄今为止,还没有建立直接测定微生物总活性的方法,只能用一些相关指标来间接反映它。目前常用的指标主要包括微生物的呼吸速率、生长速率以及胞内RNA含量等。与其它一些基质和环境相比,测定土壤中的微生物总活性更为困难。通过总结研究土壤微生物总活性常用的3种方法,在简略概括传统的土壤微生物呼吸测定法的基础上,详细介绍了放射性同位素标记法和RNA直接表征法的原理和操作流程,整理归纳了一些重要应用案例,比较分析了不同方法的优缺点,以期为选择研究土壤微生物总活性的适宜方法提供依据。
关键词:土壤微生物;微生物总活性;土壤微生物呼吸;微生物生长活性;土壤RNA
当前微生物生态学的研究正从两个方面展开:即微生物多样性和微生物功能,前者关注特定环境中的微生物群落结构,广泛采用分子生物学研究方法,并取得了显著进展;后一方面的研究深化了对微生物介导的特定生态过程的理解。微生物生态学研究的最终目标是建立微观尺度的微生物群落结构与宏观尺度的生态系统功能之间的联系,而微生物活性正是整合两者不可或缺的纽带。鉴于其极端重要性,微生物活性已经成为相关研究必须测定的基本指标,粗略检索发现,近些年每年都有上百篇生态、环境、土壤等方面的研究论文测定了微生物活性,但是基于研究目的和实验条件的不同,微生物活性的内涵和测定方法也有很大的差异,因此迫切需要界定微生物活性的含义,完善和开发微生物活性的测定方法。
微生物活性又称为微生物代谢活力[1],严格意义上是指微生物在某一时段内所有生命活动的总和,或在环境介质中微生物介导的所有过程的总和。从上述定义可以看出,直接测定微生物活性近乎是不可能的。基于此,绝大部分研究用特定代谢过程的速率来反映微生物的特定活性,如跟氮素转化相关的固氮活性[2]、硝化及反硝化活性[3],与碳转化相关的纤维素分解活性[4]等。这些研究为理解微生物在生态系统的特定功能过程中的作用提供了重要基础,但是往往不能反映环境中微生物整体的代谢状态和综合生态系统功能。另一些研究则试图寻找相关指标(proxy),间接反映微生物的总活性[5- 7]。
任何生命活动都需要能量供给,因此与能量供给相关的指标可以用来表征微生物总活性。这些指标主要包括微生物的ATP含量、呼吸速率等。由于ATP指征法存在较大的缺陷,现在更多采用呼吸速率反映微生物总活性[8- 10]。通常认为微生物活性与生长速率有正相关关系,即生长速率快的微生物具有更高的活性,所以微生物的生长速率也已成为表征微生物总活性的重要指标之一。早先的研究多采用平板计数或测量微生物生物量等方法测定微生物的生长速率,这些方法效率较低,已逐渐被放射性同位素标记法取代[6]。RNA与生物体内各种酶的合成密不可分,而酶是生命活动的主要承担者,随着RNA提取和定量方法的发展,用微生物胞内RNA浓度来表征微生物总活性的方法逐步完善,并被推广应用[5]。
相比于其他环境介质,土壤基质具有更为复杂的组成和结构,对微生物总活性的测定有较大的干扰,测定更加困难。目前有关土壤微生物总活性的研究主要采用土壤微生物呼吸测定法、放射性同位素标记法及RNA直接表征法进行,其中土壤微生物呼吸测定法作为研究微生物总活性的经典方法已为国内外研究者所熟知,而放射性同位素标记法和RNA直接表征法出现的时间相对较短,虽然在国际上已有大量的研究和应用[5- 6],但在国内尚未见该方法的研究报道。在此,简要概述土壤微生物呼吸测定法,详细介绍放射性同位素标记法和RNA直接表征法在研究微生物总活性中的应用,比较各方法的优缺点,供微生物生态学、环境科学和微生物工程等领域的相关研究人员参考。
1土壤微生物呼吸测定法
微生物呼吸是指微生物通过分解有机物质产生能量的过程,它是微生物几乎所有生命活动的能量来源。因此,呼吸作用的强弱在很大程度上可以反映微生物的总活性,而基于呼吸速率的微生物活性测定也已成为土壤微生物研究最常用的方法之一。
土壤微生物呼吸速率常用的测定方式有3种:CO2释放速率、O2消耗速率和呼吸引起的温度变化,其中尤以前两种方法最为常用。为排除动、植物的影响,土壤微生物呼吸速率多在非原位状态下测定:即采集土壤样品,去除其中的植物根系后,置于一定的温度、水分等条件下培养。在此过程中CO2或O2浓度的变化一般采用气相色谱仪、红外气体分析仪(IRGA)、氧电极或专门的呼吸仪等仪器测定[7],温度变化的测定则需要高精度测温装置[7]。
微生物呼吸测定能较为准确地反映微生物总活性,且技术要求低,因此最早得到广泛的应用。呼吸作用是碳循环的重要环节,CO2是呼吸作用的主要产物,又是最主要的温室气体之一,随着全球变暖问题日益凸显,微生物在碳素转化中的作用愈发广受关注。测定微生物呼吸既能反映土壤微生物总活性,又能表征其在碳素转化中的生态功能,因此该方法至今仍是测定土壤微生物活性最常用的方法之一。在测定土壤微生物呼吸速率的同时,结合多种土壤酶活性的分析[11- 13],可以更准确地反映和理解土壤微生物总活性。
微生物呼吸测定法也存在明显的缺陷。首先,CO2生成速率或O2消耗速率实际上反映的是有机碳矿化速率和有氧呼吸速率,与真实的微生物呼吸速率之间可能会有较大偏差[14];而测定土壤温度的变化又难以排除环境温度变化的影响,应用范围较窄。其次,要准确测定土壤微生物呼吸必须消除植物根系呼吸和土壤动物呼吸的影响,导致该方法难以实现原位测定。再次,该方法不能获得活性微生物种类的信息,不能建立微生物总活性与活性微生物种类之间的联系。
综上所述,该方法适用于土壤动、植物干扰较少,又不需要了解土壤活性微生物种类的研究。基于CO2生成速率的土壤微生物总活性测定适用于有机碳作为微生物的主要能源的土壤;基于O2消耗速率的方法则更适用于通气性良好、微生物以有氧呼吸为主的土壤;而基于温度变化的测定方法,因为需要设置无呼吸对照,则更适用于室内培养实验。
2放射性同位素标记法
通常生长速率快的微生物也具有更高的代谢活性,因此生长速率也是表征微生物总活性的常用指标之一。微生物大分子的合成速率与微生物的生长速率有很好的相关性,由于放射性同位素检测的灵敏度很高,在环境中只需添加痕量的放射性同位素标记的大分子前体物质,经短时间孵育后,通过测定微生物体内相应大分子物质的放射性活度,就可以反映微生物的生长速率。该类方法由于只添加痕量的前体物质,在孵育阶段对微生物的生长、代谢影响较小,因此成为测定微生物生长速率的常用方法。经过大量尝试和筛选,目前应用最多的前体物质为胸腺嘧啶核苷(TdR)、亮氨酸(Leu)以及醋酸盐。在土壤微生物研究中,放射性胸腺嘧啶核苷标记法、放射性亮氨酸标记法常用于测定土壤细菌的生长速率;放射性醋酸盐标记法常用于测定真菌的生长速率(表1)。
胸腺嘧啶核苷是DNA合成的前体之一,与微生物的分裂、增殖密切相关。早在1974年Thomas等就已使用放射性胸腺嘧啶核苷标记法测定土壤微生物的生长速率[15],该方法曾一度成为研究水生环境中微生物生长速率的标准方法。但近期有研究发现,很大一部分微生物并不能吸收胞外胸腺嘧啶核苷,因此该方法测定的结果不能准确反映微生物总活性[16]。
亮氨酸是蛋白质合成的前体之一,与微生物的生长、代谢关系密切。环境中绝大多数微生物都能直接利用胞外亮氨酸[16],而且微生物胞内蛋白质含量要显著高于DNA含量,所以亮氨酸标记法已有取代胸腺嘧啶核苷标记法的潜势[17]。尽管在有些研究中两种方法得到的结果较为一致[18- 19],但在另外一些研究中这两种方法测得的微生物生长速率差异较大[20- 21]。建议最好将两种方法结合起来进行土壤微生物总活性研究,在条件无法满足时应优先考虑使用放射性亮氨酸标记法。
醋酸盐是真菌麦角固醇合成的前体物质。尽管并不是只有真菌才能利用醋酸盐,但是麦角固醇却是真菌所特有的,是其细胞膜的重要组分。因此,可以通过分离并测定麦角固醇的放射性活度,排除其它微生物的影响。基于此,放射性醋酸盐标记法可用于测定真菌的生长速率。Pennanen等在1998年首次使用这一方法研究土壤中真菌的生长速率[22],此后,该方法被广泛应用于此类研究中[23- 25]。
应用放射性同位素标记法测定土壤微生物总活性的传统方法不需要分离提取土壤微生物,而是直接向用蒸馏水制备的土壤悬浊液中施入放射性标记的前体物质。此后Bååth改进了这一方法,先将土壤中的微生物使用“混匀-离心”的方法提取出来,之后再进行放射性同位素标记[26]。流程改进后可以有效地排除同位素标记物质被土壤颗粒吸附引起的干扰[6]。现在,胸腺嘧啶核苷和亮氨酸标记法一般采用Bååth改进的流程[27],但由于从土壤中提取真菌的效率较低,采用醋酸盐标记法研究土壤真菌总活性时一般采用不提取真菌的传统流程。不管采用传统方法还是Bååth改进的方法,在放射性同位素标记的前体加入之前都要将待测体系置于测试温度下稳定10min左右;在施入放射性同位素标记的前体后,根据实验需要选择合适的孵育时间(一般Leu和TdR标记法为1—2 h;醋酸盐标记法为4 h左右);孵育结束后向体系中加入适量5%的福尔马林,杀死微生物并终止反应;之后选用合适的方法提取相应的大分子物质(Leu标记法需提取蛋白质;TdR标记法需提取DNA;醋酸盐标记法则需要提取麦角固醇),并溶于NaOH溶液[28];最后用液体闪烁仪测定相应大分子物质的放射性活度,并结合孵育时间计算微生物的生长速率[26]。
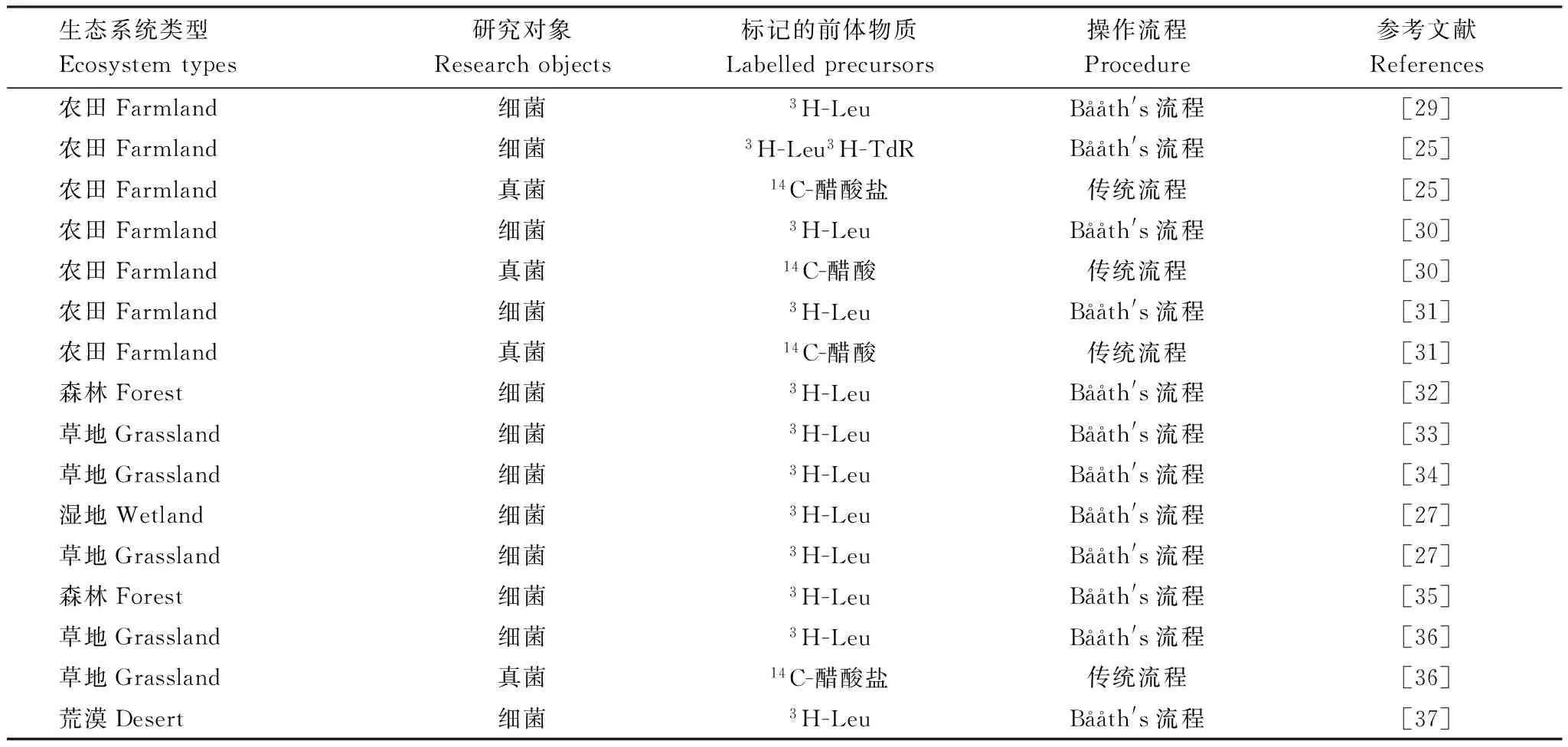
表1 放射性同位素标记法测定土壤微生物活性的主要应用案例
Rousk等已经对放射性同位素标记法的应用案例做了较为详尽的总结[6],但对近年的研究做了一些简单补充(表1)。可以看出,该方法在农田、草地以及森林等主要陆地生态系统类型上都有着广泛的应用。如前所述,现在土壤细菌生长速率和活性研究多采用放射性亮氨酸标记法,仅有一个研究结合了放射性亮氨酸标记法和放射性胸腺嘧啶核苷标记法[25]。为提高同位素标记法的检测效率和灵敏度,现在细菌生长速率的测定一般采用Bååth改进的操作流程;而因为从土壤中提取真菌难度较大,真菌的相关研究多采用传统操作流程的放射性醋酸盐标记法。
应用放射性同位素标记法测定土壤微生物的生长速率和总活性有两个问题备受关注:一是该方法的测量结果能在多大程度上反映原位的状况?针对这一问题开展的研究表明,改变一些重要的环境条件,微生物仍然能够在数小时内维持其原有的生长速率不变,并且该方法的操作环节较易完成,一般对测定结果的影响很小[26,38- 39],因此,其测定结果可以较为准确地反映土壤原位真实的微生物生长速率和总活性。第二个问题是,该方法能否特异性地测定土壤细菌或真菌的总体生长速率和总活性?研究发现,不管采用传统操作流程,还是采用Bååth改进的流程,TdR或Leu标记法都能特异性地测定土壤细菌的生长速率和总活性[39- 40]。由于麦角固醇是真菌所特有的,所以醋酸盐标记法测定真菌生长速率和总活性的特异性也较好。
当然,应用放射性同位素标记法测定土壤微生物总活性也存在3个无法避免的缺陷。首先,放射性同位素可能会给待测微生物造成较大损害,从而影响测定结果。其次,该方法无法给出微生物的种类组成信息,只能得到较单一的土壤微生物总体生长速率结果。第三,微生物生长速率并不完全等价于微生物活性[5],微生物生长速率能在多大程度上表征微生物总活性也是最值得关注的问题和研究课题。事实上,微生物活性由生长活性和非生长活性两部分构成,而微生物生长速率仅能反映微生物的生长活性。已有研究发现微生物在某些胁迫条件下会提高自身的非生长活性[41],而且土壤中的微生物绝大部分都处于非生长状态[42- 43],没有生长活性,只有非生长活性。因此,直接用微生物生长速率表征微生物活性有时会产生较大偏差。
综上所述,放射性同位素标记法在测定微生物生长速率方面有较大优势,但作为微生物总活性的间接指标,由于其只能指示生长活性,存在较大的局限性。可以用于近似表征营养供应及其它环境条件良好、微生物生长活性占主导的土壤中微生物的总活性。
3RNA直接表征法
蛋白质是绝大多数生命活动的直接承担者,在翻译过程中,信使RNA(mRNA)是模板,转运RNA(tRNA)是搬运氨基酸的工具,核糖体RNA(rRNA)是构成翻译工厂的核心组件,它们与蛋白质合成关系极为密切,因此通过RNA来反映微生物活性已成为一种重要的研究手段[5]。在微生物活性的研究中,mRNA常用于表征某一特定代谢过程的活跃程度,而rRNA可用于表征微生物总活性,也是本文讨论的重点。用RNA测定土壤微生物总活性有其独有的优势:能同时获得微生物的代谢活性信息和活性微生物群落结构信息;不需要孵育过程,原位测定简单易行。因此该方法已经被广泛采用,2013年的评述论文报道当时已经检索到用该方法研究微生物总活性的报道100余篇[5]。
rRNA直接表征法测定微生物总活性有着坚实的理论基础和一定的实验依据。首先,rRNA是核糖体的核心组分,是蛋白质合成的催化中心,因此胞内rRNA的量与胞内蛋白质合成速率有着紧密的关系。其次,已有研究显示多种微生物胞内rRNA的含量与其生长速率、代谢活性有高度相关性[44- 46]。再次,微生物的死亡和休眠往往伴随着rRNA的降解[47- 48],只有活性微生物中存在大量rRNA。
早期的研究多测定rRNA的总量来表征微生物总活性,而现在多趋向于直接分析SSU rRNA的拷贝数来表征微生物总代谢活性。这是因为微生物体内SSU rRNA(核糖体小亚基rRNA)与rRNA总量有良好的线性相关关系,而且SSU rRNA基因是微生物研究中应用最广泛的分子标记。通常16S rRNA用于表征土壤细菌和古菌总活性;18S rRNA则用于表征土壤真菌总活性。
SSU rRNA表征法测定微生物总活性的操作流程如下:1)土壤样品采集;2)土壤总RNA和土壤总DNA提取;3)土壤总RNA反转录;4)实时荧光定量PCR测定SSU rRNA基因和SSU rRNA的拷贝数;5)通过克隆文库、末端限制性片段长度多态性(T-RFLP)、高通量测序等技术确定土壤活性微生物的群落组成。其中样品保存、RNA提取及活性表征方式需要特别注意。
(1)样品的保存问题
相比基于DNA的微生物生态学研究,用于RNA研究的土壤样品的保存要求更为严格。通常要求样品在采集后即刻进行液氮速冻;在运输过程中无法实现液氮保存时,可用干冰冻存作为短期的替代选择;实验室内则要求-80 ℃冻存。此外,现在有很多试剂公司已推出RNA保护液类产品,可抑制RNA的合成和降解,实现常温下样品的短期保存。
不当的保存方法可能引起RNA降解,甚至导致试验失败。更为严重的是保存方法不当还会造成实验假象。过高的保存温度——例如简单的冷冻保存或常温保存——并不能完全阻止微生物体内RNA的合成与降解。而通常样品保存的条件与土壤原位环境条件差异较大,这会导致测定时样品中的RNA实际反映的是样品保存条件下的微生物活性状态,而非采样时土壤原位条件下的微生物活性状态。常见的实验假象有16S rRNA表征的活性细菌群落比16S rRNA基因表征的总细菌群落的种间亲缘关系更近,微生物总活性在各处理间无显著差异等。
总结近期用rRNA直接表征法研究土壤微生物活性的报道(表2),结果显示用液氮或-80 ℃保存土壤样品时,活性微生物群落结构能反映试验处理间的差异,我们待发表的数据也证实增温会显著改变活性细菌的群落结构(土样为液氮保存)。在较高温度下保存土壤样品,则可能降低实验结果的可信度。例如,Männistö等研究积雪在不同月份对活性微生物群落的影响[49],5月份采集的土壤样品保存在4 ℃,6月份的样品在环境温度下保存,结果发现5、6月份的土壤活性细菌群落结构差异较大。虽然作者在文中提及他们所采用的土壤保存方法不会影响用rRNA研究土壤细菌群落结构,但是我们很难辨别这种群落差异是不是因为样品保存方式不当所造成的假象。再如Angel等调查沿降水梯度的活性微生物群落[50],仅用冰盒保存土壤样品,结果发现活性微生物群落结构仅在水分含量不同的土壤间表现出差异,但有理由怀疑温度的效应可能被不当的保存方法造成的假象所掩盖。

表2 RNA直接表征法测定土壤微生物总活性的主要应用案例
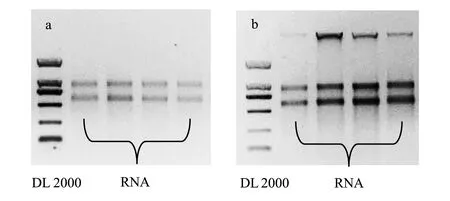
图1 土壤RNA提取电泳图Fig.1 Electrophoretogram of soil RNA a:正常的土壤RNA电泳图;b:有基因组DNA残留的土壤RNA电泳图
(2) 土壤RNA提取问题
土壤RNA有多种提取方法,刘华等总结了包括Trizol法、SDS-Phenol法和试剂盒法在内的多种方法[55]。对于有机质含量较高的土壤,建议优先选择试剂盒法;若采用常规方法则需用核酸纯化柱去除杂质。所提取RNA的质量多用1%的琼脂糖凝胶电泳检测,理论上会出现3条较亮的电泳条带,自上到下分别对应LSU rRNA(核糖体大亚基 rRNA)、SSU rRNA和5.8/5S rRNA,mRNA等则呈弥散状分布于LSU rRNA 条带下方。但实际上5/5.8S rRNA条带一般不可见,故电泳图谱出现两条清晰条带即可证明RNA提取是成功的(图1a)。由于在细胞内LSU rRNA和SSU rRNA的拷贝数之比为1,所以条带的亮度主要由片段的长度决定。因为28S rRNA长度为18S rRNA的2.4倍左右,所以真核生物中判别RNA提取质量的另一方法是根据两个条带的亮度,第一条亮带的亮度达到第二条亮带的两倍即为成功。但23S rRNA与16S rRNA长度比一般在1.8左右,所以原核生物RNA两个条带的亮度差异不是特别明显。此外,若在SSU rRNA 条带上方仍有条带出现则证明所提取的RNA中混有残留的基因组DNA(图1b),需用DNA酶消化去除。
(3) 活性表征的方式
推荐使用单位质量(一般为每克)干土中SSU rRNA的拷贝数表征土壤微生物总活性,用SSU rRNA与SSU rRNA基因拷贝数的比值表征微生物的平均活性。有些研究用每纳克核酸中SSU rRNA的拷贝数表征微生物总活性(表2),这种表示方法实际上反映的是土壤核酸的组成比例,与微生物活性关系不大。采用该指标并不合理,甚至可能会掩盖试验的处理效应,得出错误的结论。
当然,用rRNA直接表征土壤微生物的活性也存在一定的风险[5]。首先,微生物胞内SSU rRNA含量与微生物活性之间的量化关系还未完全确立,仅有的少数研究也发现这一量化关系在不同微生物之间有较大的差异[5,56- 58],因此在群落和生态系统尺度上是否有确定的关系还存在疑问。其次,尚未有研究确定微生物胞内SSU rRNA含量是否与微生物的非生长活性相关,因此SSU rRNA能否表征微生物总活性还需要进一步的试验验证。再次,因为土壤RNA和DNA一般是分别提取的,采用不同的方法体系,用SSU rRNA与DNA的比值表征微生物平均活性会因此产生不确定性[59]。
SSU RNA表征法在指示微生物总活性方面有很强的应用潜力,在表征活性微生物群落结构方面已有较为广泛的应用[60- 61]。但一般认为该方法在微生物处于生长稳态时的使用效果最好,如果要应用于其他条件,则需要辅以其它验证性实验,以确立SSU rRNA拷贝数与微生物总活性的关系。
4结论
综上所述,土壤微生物呼吸测定法、放射性同位素标记法和RNA直接表征法是基于不同的原理来指示土壤微生物总活性的,各有长处,同时也都有明显的缺陷(表3)。土壤微生物呼吸测定法非常适用于表征微生物总活性,但该方法的主要问题在于现存测定方法并不能真实反映微生物总的呼吸强度,但在通气性良好的土壤中,微生物呼吸方式大多为有氧呼吸,可以通过O2消耗速率近似反映微生物总活性;在微生物以有机碳作为最主要的能量来源时,CO2生成速率可以较好地反映微生物总呼吸。放射性同位素标记法测定的指标为生长速率,因此它适合于表征微生物的总体生长活性,也可在有利于微生物良好生长的环境中近似表征微生物总活性。在我国,由于放射性标记物质的购买、运输、环境释放和测定等受到极大的限制,该方法的生态学应用非常困难。RNA直接表征法相比于前两种方法有明显的优势,它既能原位测定微生物总活性,又能提供微生物种类组成信息。但是,目前在实验证据上对它能否准确地反映土壤微生物总活性还存在疑问。在方法的选择上需要根据实际情况综合考量,确定合适的方法开展土壤微生物总活性研究;在条件允许的情况下,建议将几种方法组合使用。

表3 3种土壤微生物活性研究方法的比较
致谢:感谢周小奇博士、Jackie Wilkinson对本文写作的帮助。
参考文献(References):
[1]池振明. 现代微生物生态学. 北京: 科学出版社, 2006: 1- 6.
[2]Cusack D F, Silver W, McDowell W H. Biological nitrogen fixation in two tropical forests: ecosystem- level patterns and effects of nitrogen fertilization. Ecosystems, 2009, 12(8): 1299- 1315.
[3]Zhong L, Du R, Ding K, Kang X M, Li F Y, Bowatte S, Hoogendoorn C J, Wang Y F, Rui Y C, Jiang L L, Wang S P. Effects of grazing on N2O production potential and abundance of nitrifying and denitrifying microbial communities in meadow-steppe grassland in northern China. Soil Biology and Biochemistry, 2014, 69: 1- 10.
[4]Stark S, Männistö M K, Eskelinen A. Nutrient availability and pH jointly constrain microbial extracellular enzyme activities in nutrient-poor tundra soils. Plant and Soil, 2014, 383(1/2): 373- 385.
[5]Blazewicz S J, Barnard R L, Daly R A, Firestone M K. Evaluating rRNA as an indicator of microbial activity in environmental communities: limitations and uses. The ISME Journal, 2013, 7(11): 2061- 2068.
[6]Rousk J, Bååth E. Growth of saprotrophic fungi and bacteria in soil. FEMS Microbiology Ecology, 2011, 78(1): 17- 30.
[7]Kluge B, Peters A, Krüger J, Wessolek G. Detection of soil microbial activity by infrared thermography (IRT). Soil Biology and Biochemistry, 2013, 57: 383- 389.
[8]Zhou X Q, Chen C R, Wang Y F, Xu Z H, Duan J C, Hao Y B, Smaill S. Soil extractable carbon and nitrogen, microbial biomass and microbial metabolic activity in response to warming and increased precipitation in a semiarid Inner Mongolian grassland. Geoderma, 2013, 206: 24- 31.
[9]Chen Q L, Wang H, Yang B S, He F. The combined effects of atrazine and lead (Pb): Relative microbial activities and herbicide dissipation. Ecotoxicology and Environmental Safety, 2014, 102: 93- 99.
[10]Stromberger M E, Shah Z, Westfall D G. High specific activity in low microbial biomass soils across a no- till evapotranspiration gradient in Colorado. Soil Biology and Biochemistry, 2011, 43(1): 97- 105.
[11]Pourreza M, Hosseini S M, Sinegani A S, Matinizadeh M, Dick W A. Soil microbial activity in response to fire severity in Zagros oak (QuercusbrantiiLindl.) forests, Iran, after one year. Geoderma, 2014, 213: 95- 102.
[12]Frossard A, Gerull L, Mutz M, Gessner M O. Litter supply as a driver of microbial activity and community structure on decomposing leaves: a test in experimental streams. Applied and Environmental Microbiology, 2013, 79(16): 4965- 4973.
[13]Esch E H, Hernandez D L, Pasari J R, Kantor R S G, Selmants P C. Response of soil microbial activity to grazing, nitrogen deposition, and exotic cover in a serpentine grassland. Plant and Soil, 2013, 366(1/2): 671- 682.
[14]Bloem J, Hopkins D W, Benedetti A. MicrobiologicalMethods for Assessing Soil Quality. Oxfordshire: CABI Publishing, 2006: 117- 118.
[15]Thomas D R, Richardson J A, Dicker R J. The incorporation of tritiated thymidine into DNA as a measure of the activity of soil micro-organisms. Soil Biology and Biochemistry, 1974, 6(5): 293- 296.
[16]Perez M T, Hortnagl P, Sommaruga R. Contrasting ability to take up leucine and thymidine among freshwater bacterial groups: implications for bacterial production measurements. Environmental Microbiology, 2010, 12(1): 74- 82.
[17]Kirchman D, K′nees E, Hodson R. Leucine incorporation and its potential as a measure of protein synthesis by bacteria in natural aquatic systems. Applied and Environmental Microbiology, 1985, 49(3): 599- 607.
[18]Mahmood S, Finlay R D, Fransson A M, Wallander H. Effects of hardened wood ash on microbial activity, plant growth and nutrient uptake by ectomycorrhizal spruce seedlings. FEMS Microbiology Ecology, 2003, 43(1): 121- 131.
[19]Rousk J, Brookes P C, Bååth E. Contrastingsoil pH effects on fungal and bacterial growth suggest functional redundancy in carbon mineralization. Applied and Environmental Microbiology, 2009, 75(6): 1589- 1596.
[20]Ranneklev S B, Bååth E. Temperature-driven adaptation of the bacterial community in peat measured by using thymidine and leucine incorporation. Applied and Environmental Microbiology, 2001, 67(3): 1116- 1122.
[21]Rousk J, Bååth E. Fungal and bacterial growth in soil with plant materials of different C/N ratios. FEMS Microbiology Ecology, 2007, 62(3): 258- 267.
[22]Pennanen T, Fritze H, Vanhala P, Kiikkila O, Neuvonen S, Bååth E. Structure of a microbial community in soil after prolonged addition of low levels of simulated acid rain. Applied and Environmental Microbiology, 1998, 64(6): 2173- 2180.
[23]Rousk J, Brookes P C, Bååth E. Investigating the mechanisms for the opposing pH relationships of fungal and bacterial growth in soil. Soil Biology and Biochemistry, 2010, 42(6): 926- 934.
[24]Bapiri A, Bååth E, Rousk J. Drying-rewetting cycles affect fungal and bacterial growth differently in an arable soil. Microbial Ecology, 2010, 60(2): 419- 428.
[25]Bengtson P, Sterngren A E, Rousk J. Archaeal abundance across a pH gradient in an arable soil and its relationship to bacterial and fungal growth rates. Applied and Environmental Microbiology, 2012, 78(16): 5906- 5911.
[26]Bååth E. Thymidine incorporation into macromolecules of bacteria extracted from soil by homogenization- centrifugation. Soil Biology and Biochemistry, 1992, 24(11): 1157- 1165.
[27]Ford H, Rousk J, Garbutt A, Jones L, Jones D L. Grazing effects on microbial community composition, growth and nutrient cycling in salt marsh and sand dune grasslands. Biology and Fertility of Soils, 2013, 49(1): 89- 98.
[28]Bååth E, Pettersson M, Soderberg K H. Adaptation of a rapid and economical microcentrifugation method to measure thymidine and leucine incorporation by soil bacteria. Soil Biology and Biochemistry, 2001, 33(11): 1571- 1574.
[29]Rousk J, Elyaagubi F K, Jones D L, Godbold D L. Bacterial salt tolerance is unrelated to soil salinity across an arid agroecosystem salinity gradient. Soil Biology and Biochemistry, 2011, 43(9): 1881- 1887.
[30]Jones D L, Rousk J, Edwards-Jones G, DeLuca T H, Murphy D V. Biochar-mediated changes in soil quality and plant growth in a three year field trial. Soil Biology and Biochemistry, 2012, 45: 113- 124.
[31]Quilliam R S, Marsden K A, Gertler C, Rousk J, DeLuca T H, Jones D L. Nutrient dynamics, microbial growth and weed emergence in biochar amended soil are influenced by time since application and reapplication rate. Agriculture Ecosystems and Environment, 2012, 158: 192- 199.
[32]Rousk J, Frey S D, Bååth E. Temperature adaptation of bacterial communities in experimentally warmed forest soils. Global Change Biology, 2012, 18(10): 3252- 3258.
[33]Walsh J J, Rousk J, Edwards-Jones G, Jones D L, Williams A P. Fungal and bacterial growth following the application of slurry and anaerobic digestate of livestock manure to temperate pasture soils. Biology and Fertility of Soils, 2012, 48(8): 889- 897.
[34]Birgander J, Reischke S, Jones D L, Rousk J. Temperature adaptation of bacterial growth and14C- glucose mineralisation in a laboratory study. Soil Biology and Biochemistry, 2013, 65: 294- 303.
[35]Goeransson H, Godbold D L, Jones D L, Rousk J. Bacterial growth and respiration responses upon rewetting dry forest soils: Impact of drought-legacy. Soil Biology and Biochemistry, 2013, 57: 477- 486.
[36]Meisner A, Bååth E, Rousk J. Microbial growth responses upon rewetting soil dried for four days or one year. Soil Biology and Biochemistry, 2013, 66: 188- 192.
[37]van Gestel N C, Reischke S, Bååth E. Temperature sensitivity of bacterial growth in a hot desert soil with large temperature fluctuations. Soil Biology and Biochemistry, 2013, 65: 180- 185.
[38]Rousk J, Demoling L A, Bååth E. Contrasting short-term antibiotic effects on respiration and bacterial growth compromises the validity of the selective respiratory inhibition technique to distinguish fungi and bacteria. Microbial Ecology, 2009, 58(1): 75- 85.
[39]Bååth E. Thymidine incorporation into soil bacteria. Soil Biology and Biochemistry, 1990, 22(6): 803- 810.
[40]Rousk J, Demoling L A, Bahr A, Bååth E. Examining the fungal and bacterial niche overlap using selective inhibitors in soil. FEMS Microbiology Ecology, 2008, 63(3): 350- 358.
[41]Schimel J, Balser T C, Wallenstein M. Microbial stress-response physiology and its implications for ecosystem function. Ecology, 2007, 88(6): 1386- 1394.
[42]Lennon J T, Jones S E. Microbial seed banks: the ecological and evolutionary implications of dormancy. Nature Reviews Microbiology, 2011, 9(2): 119- 130.
[43]Stenström J, Svensson K, Johansson M. Reversible transition between active and dormant microbial states in soil. FEMS Microbiology Ecology, 2001, 36(2/3): 93- 104.
[44]Schaechter M, Maaloe O, Kjeldgaard N O. Dependency on medium and temperature of cell size and chemical composition during balanced growth of Salmonella typhimurium. Journal of General Microbiology, 1958, 19(3): 592- 606.
[45]Ramos C, Molbak L, Molin S. Bacterial activity in the rhizosphere analyzed at the single-cell level by monitoring ribosome contents and synthesis rates. Applied and Environmental Microbiology, 2000, 66(2): 801- 809.
[46]Perez-Osorio A C, Williamson K S, Franklin M J. Heterogeneous rpoS and rhlR mRNA levels and 16S rRNA/rDNA (rRNA gene) ratios withinPseudomonasaeruginosabiofilms, sampled by Laser Capture Microdissection. Journal of Bacteriology, 2010, 192(12): 2991- 3000.
[47]Lahtinen S J, Ahokoski H, Reinikainen J P, Gueimonde M, Nurmi J, Ouwehand A C, Salminen S J. Degradation of 16S rRNA and attributes of viability of viable but nonculturable probiotic bacteria. Letters in Applied Microbiology, 2008, 46(6): 693- 698.
[48]Segev E, Smith Y, Ben-Yehuda S. RNAdynamics in aging bacterial spores. Cell, 2012, 148(1/2): 139- 149.
[49]Männistö M K, Kurhela E, Tiirola M, Häggblom M M.Acidobacteriadominate the active bacterial communities of Arctic tundra with widely divergent winter-time snow accumulation and soil temperatures. FEMS Microbiology Ecology, 2013, 84(1): 47- 59.
[50]Angel R, Pasternak Z, Soares M I M, Conrad R, Gillor O. Active and total prokaryotic communities in dryland soils. FEMS Microbiology Ecology, 2013, 86(1): 130- 138.
[51]Yarwood S, Brewer E, Yarwood R, Lajtha K, Myrold D. Soil microbe active community composition and capability of responding to litter addition after 12 years of no inputs. Applied and Environmental Microbiology, 2013, 79(4): 1385- 1392.
[52]Barnard R L, Osborne C A, Firestone M K. Responses of soil bacterial and fungal communities to extreme desiccation and rewetting. The ISME Journal, 2013, 7(11): 2229- 2241.
[53]Schindlbacher A, Rodler A, Kuffner M, Kitzler B, Sessitsch A, Zechmeister-Boltenstern S. Experimental warming effects on the microbial community of a temperate mountain forest soil. Soil Biology and Biochemistry, 2011, 43(7): 1417- 1425.
[54]Baldrian P, Kolarik M, Stursova M, Kopecky J, Valaskova V, Vetrovsky T, Zifcakova L, Snajdr J, Ridl J, Vlcek C, Voriskova J. Active and total microbial communities in forest soil are largely different and highly stratified during decomposition. The ISME Journal, 2012, 6(2): 248- 258.
[55]刘华, 申天琳, 李学坤, 付合才, 戴九兰. 土壤RNA提取方法研究进展. 应用与环境生物学报, 2011, 17(6): 918- 922.
[56]Worden A Z, Binder B J. Growth regulation of rRNA content inProchlorococcusandSynechococcus(marine cyanobacteria) measured by whole-cell hybridization of rRNA-targeted peptide nucleic acids. Journal of Phycology, 2003, 39(3): 527- 534.
[57]Sukenik A, Kaplan-Levy R N, Welch J M, Post A F. Massive multiplication of genome and ribosomes in dormant cells (akinetes) ofAphanizomenonovalisporum(Cyanobacteria). The ISME Journal, 2012, 6(3): 670- 679.
[58]McCoy G R, Kegel J U, Touzet N, Fleming G T A, Medlin L K, Raine R. An assessment of RNA content inPrymnesiumparvum,Prymnesiumpolylepis,cf.Chattonellasp. andKarlodiniumveneficumunder varying environmental conditions for calibrating an RNA microarray for species detection. FEMS Microbiology Ecology, 2014, 88(1): 140- 159.
[59]Sessitsch A, Gyamfi S, Stralis-Pavese N, Weilharter A, Pfeifer U. RNA isolation from soil for bacterial community and functional analysis: evaluation of different extraction and soil conservation protocols. Journal of Microbiological Methods, 2002, 51(2): 171- 179.
[60]Wilhelm L, Besemer K, Fasching C, Urich T, Singer G A, Quince C, Battin T J. Rare but active taxa contribute to community dynamics of benthic biofilms in glacier-fed streams. Environmental Microbiology, 2014, 16(8): 2514- 2524.
[61]Charvet S, Vincent W F, Lovejoy C. Effects of light and prey availability on Arctic freshwater protist communities examined by high-throughput DNA and RNA sequencing. FEMS Microbiology Ecology, 2014, 88(3): 550- 564.
A reviewon the methods for measuring total microbial activity in soils
CHE Rongxiao1,2, WANG Fang1, WANG Yanfen1, DENG Yongcui3, ZHANG Jing1, MA Shuang1, CUI Xiaoyong1,*
1UniversityofChineseAcademyofSciences,Beijing100049,China2GriffithUniversity,Brisbane4111,Australia3NanjingNormalUniversity,Nanjing210023,China
Abstract:Total microbial activity (TMA) in soils is vital for understanding the roles of microorganisms in ecosystem processes. It can be defined as the sum of physiological activities of all the microbes at a given moment. As TMA is difficult to measure directly, a series of proxies, such as respiration rates, growth rates, and cellular RNA concentration, have been proposed. Here, methods used to measure soil TMA are synthesized and compared. (1) Respiration may be the process most closely related to life activities. Thus, respiration rates are the most commonly used proxies of soil TMA. The main limitation is that current methods to determine respiration rates usually cannot accurately reflect actual respiration rates. When respiration rates are measured using CO2 production or O2 consumption rates, they indicate carbon mineralization or aerobic respiration rates, respectively. (2) Microbes with higher growth rates are usually more active. Thus, growth rates are also widely used to indicate soil TMA. As biomacromolecule synthesis is approximately proportional to microbial growth rates, incorporation of radioactive isotope labeled precursors (i.e., thymidine, leucine, and acetate) can be employed to estimate microbe growth rates. Generally, trace radioactively labeled precursors are added to slurries (traditional methods) or extracted microbial suspensions (Bååth′s methods). After a brief incubation, microbes are killed and the corresponding biomacromolecules are extracted to measure their radioactivity. Thymidine and leucine incorporation are commonly used to measure heterotrophic bacterial growth rates, while acetate incorporation is used to estimate growth rates of saprotrophic fungi. Radioactive isotope labeling methods are robust tools to estimate the growth rates of soil microbes. However, one critical problem is that TMA includes both growth activity and non-growth activity, whereas these methods only reflect the former. (3) Evidently, none of the methods based on respiration rates or incorporation of radioactive isotope labeled precursors can accurately link microbial activity with their identities. However, this issue can be resolved through using methods based on RNA. RNA correlates closely with protein synthesis, which is involved in most metabolic processes. Therefore, RNA concentration is assumed an ideal indicator of microbial activity. Generally, mRNA can be used to indicate the activity of specific metabolic processes, including nitrogen fixation and denitrification, whereas rRNA is a proxy of soil TMA. As cellular concentrations of small subunit rRNA (SSU rRNA) are proportional to total cellular rRNA concentrations, SSU rRNA copies can serve as an indicator of soil TMA and the ratio of SSU rRNA copies to SSU rRNA gene copies can be used to determine average microbial activity in soil. Additionally, active microbial community structure can be illustrated using profiling methods, such as clone library, T-RFLP, and high-throughput sequencing, based on SSU rRNA. These approaches can simultaneously identify soil microbes and their activity via in situ measurements. However, there is still no adequate evidence to support the assertion that the methods based on SSU rRNA can accurately reflect microbial activity, especially for non-growth activity. In conclusion, none of these methods are perfect to determine soil TMA; and a combination of suitable methods should be selected for individual ecosystems.
Key Words:soil microbes; total microbial activity; soil microbial respiration; microbial growth activity; soil RNA
基金项目:国家自然科学基金项目(41230750); 中国科学院战略先导专项B课题(XDB05010200)
收稿日期:2014- 10- 26; 网络出版日期:2015- 08- 24
*通讯作者
Corresponding author.E-mail: cuixy@ucas.ac.cn
DOI:10.5846/stxb201410262093
车荣晓,王芳,王艳芬,邓永翠,张静,马双,崔骁勇.土壤微生物总活性研究方法进展.生态学报,2016,36(8):2103- 2112.
Che R X, Wang F, Wang Y F, Deng Y C, Zhang J, Ma S, Cui X Y.A reviewon the methods for measuring total microbial activity in soils.Acta Ecologica Sinica,2016,36(8):2103- 2112.

