棉花不同GbU6启动子截短克隆及功能鉴定
雷建峰李 月徐新霞阿尔祖古丽·塔什蒲 艳张巨松刘晓东,*
1新疆农业大学农学院/新疆农业大学农业生物技术重点实验室, 新疆乌鲁木齐830052;2新疆巴州农业技术推广中心, 新疆库尔勒841000
棉花不同GbU6启动子截短克隆及功能鉴定
雷建峰1李 月1徐新霞2阿尔祖古丽·塔什1蒲 艳1张巨松1刘晓东1,*
1新疆农业大学农学院/新疆农业大学农业生物技术重点实验室, 新疆乌鲁木齐830052;2新疆巴州农业技术推广中心, 新疆库尔勒841000
摘 要:U6启动子是CRISPR/Cas9基因组编辑载体系统中驱动sgRNA转录的重要元件, 而使用较短序列的启动子也是构建CRISPR/Cas9基因组编辑载体的基本要求之一。将已经克隆的海岛棉GbU6-5P启动子(长度为1166 bp), 采用Transfer PCR方法成功地截出6个长度不同的U6启动子, 其长度分别为672、468、358、280、202和105 bp, 并分别构建了6个启动子驱动的GUS融合植物表达载体。将构建好的6个GbU6-5Ps∶∶GUS-pCAMBIA1300与初始克隆的GbU6-5P∶∶GUS-pCAMBIA1300植物表达载体一起利用农杆菌真空渗透转化法分别转染棉花花粉。GUS组织化学染色显示, 克隆到的7个不同截短大小的GbU6-5P启动子均能驱动GUS基因在棉花花粉中转录, 棉花花粉被染成蓝色但颜色深浅存在显著差异。结果显示启动子长度越短, 其转录活性越高。而且另外两种棉花U6启动子GbU6-1P和 GbU6-7P也表现出类似的结果。本研究克隆了3个短小的、在棉花花粉细胞中具有转录功能的GbU6启动子。结果显示更短的U6启动子具有更高的转录活性, 而且这一特点在不同U6启动子上具有共性。这预示着使用更短U6启动子不仅符合构建CRISPR/Cas9基因组编辑载体的要求, 而且会提高sgRNA的转录水平, 进而可能提高基因组编辑效率。
关键词:棉花; GbU6启动子; 截短克隆; 真空渗透; 棉花花粉
本研究由国家自然科学基金项目(31560534)和新疆维吾尔自治区研究生科研创新项目(XJGRI2015084)资助。
This study was supported by the National Natural Science Foundation of China (31560534) and the Science and Technology Innovation Projects for Graduate Students in Xinjiang Uygur Autonomous Region (XJGRI2015084).
第一作者联系方式∶ E-mail∶ 15299175640@163.com
URL∶ http∶//www.cnki.net/kcms/detail/11.1809.S.20160218.1503.008.html
基因组编辑技术是一种研究基因功能的重要工具, 它能精确改变受体细胞染色体特定位点的基因序列, 可以高效产生特定基因的功能失活突变体,能为生物功能基因组研究提供优质的遗传材料。所以该技术自诞生以来, 就受到广大生物学家的青睐。ΙΙ型CRISPR/Cas9基因组编辑系统是继锌指核酸酶(ZFN)和TALE核酸酶(TALEN)之后, 另一种可以对基因组高效定点修饰的新技术[1-3]。sgRNA和 Cas9是该技术系统的2个必要成员, 其中sgRNA是由crRNA和tracerRNA(trans-activating crRNA)融合而成, 具有靶向结合目的基因位点的功能。在CRISPR/Cas9技术系统中, sgRNA的转录通常由U6启动子或者 U3启动子完成, 二者转录活性相对较高, 且都有明确的转录起始位点∶ 前者以 G碱基开头, 后者以 A碱基开头。精确起始转录具有靶向功能的sgRNA能消除无关DNA序列的转录, 从而大大减少脱靶效应的产生[4-6]。
多项研究结果表明 U6启动子是驱动带有靶向位点sgRNA转录的理想启动子。Hyun等[7]构建了由AtU6启动子驱动sgRNA转录, 靶向修饰拟南芥FT基因和SPBPlike-4基因的CRISPR/Cas9基因组编辑系统, 并在 T1代转化子中检测到靶位点突变。Thomas等[8]克隆了大豆U6启动子, 并成功修饰了9个大豆内源基因。Jiang等[9]克隆了水稻U6启动子驱动 sgRNA的转录, 并在水稻原生质体中敲除了OsSWEET14和OsSWEET11基因。U6启动子虽然具有高效转录的特点, 但是它在同源关系较远的不同物种之间不一定适用[10], 而且同一物种基因组中存在多个U6启动子, 这些启动子的转录效率也不相同[6,11]。
棉花是我国重要的经济作物和纺织工业原料。目前用于棉花 CRISPR/Cas9基因组编辑系统的 U6启动子还未见报道。预测棉花基因组中至少有10种 U6启动子。本课题组在前期工作中已克隆了 5种 U6启动子, 并利用基因枪瞬时转化棉花花粉的方法从中筛选出一个转录效率相对较高的GbU6-5P启动子(长度为 1166 bp)[12], 但已有研究表明, 用于CRISPR/Cas9基因组编辑技术的 U6启动子通常只有 200~400 bp长[13-14], 甚至短于 100 bp[15]。由于Cas9基因巨大(大于4000 bp), 而且驱动Cas9基因表达的35S启动子长度也在1000 bp左右[14-15], 这2个片段上分布着绝大多数常用的限制性内切酶位点。因此在构建sgRNA和Cas9共表达载体时, 可用于sgRNA重组片段连接的酶切位点非常少。所以这就要求U6启动子上不能再出现这些酶切位点, 而只有更短的片段才有可能满足这个要求。因此克隆的 U6启动子片段越短越便于CRISPR/Cas9载体的构建。
本研究以前期获得的5种棉花U6启动子为模板,克隆不同截短大小的 GbU6启动子并构建相应的GUS融合表达载体。采用农杆菌真空渗透法[16-17]转化棉花花粉, 并验证它们的转录活性, 以克隆出较短的而且具有高效转录活性的棉花U6启动子。
1 材料与方法
1.1 试验材料
试验所用AtU6-26P∶∶sgRNA[18]、pBI101载体、农杆菌GV3101、棉花品种新海16均为新疆农业大学农业生物技术重点实验室保存。各种限制性内切酶购于Fermentas公司; T4 DNA连接酶、Blunt Zero平端载体、Trans1-T1感受态细胞、琼脂糖凝胶回收试剂盒、Taq DNA聚合酶、RNase A、KD Plus DNA聚合酶、1 kb Plus DNA Ladder均购自于北京全式金生物技术有限公司; Phusion超保真 DNA聚合酶购自于纽英伦生物技术(北京)有限公司; 其他常规试剂均为国产分析纯; 引物合成及测序均由上海杰李生物技术有限公司完成。
1.2 GbU6-5P不同截短大小启动子的克隆
根据前期已克隆的 GbU6-5P启动子序列和AtU6-26P∶sgRNA载体序列设计了6对截短GbU6-5P启动子的Transfer PCR引物(表1)。本课题组于2014年已完成对GbU6-5P (1787 bp)的第一轮克隆。第二轮是精确克隆不同截短的GbU6-5P启动子, 以第一轮PCR克隆的GbU6-5P启动子质粒为供体质粒模板, 以拟南芥AtU6-26P∶sgRNA的质粒为受体质粒模板进行由两轮PCR组成的Transfer PCR[19], 对 GbU6-5P启动子进行不同长度的截短。对电泳检测正确的Transfer PCR产物进行Dpn I (4 U)酶切处理,37℃酶切3 h以上, 取10 µL消化产物转化大肠杆菌。对获得的不同截短的GbU6-5P质粒采用BamH I 和 Hind III酶切鉴定及测序验证, 将正确的克隆命名为T-GbU6-5Ps。

表1 本研究中使用的引物序列Table 1 Primer sequences used in this study
1.3 不同截短 GbU6-5P::GUS融合的植物表达载体的构建
将已克隆的GUS基因和T-GbU6-5Ps质粒用Bbs I酶切, 回收目标片段, 连接, 转化, 然后进行菌落PCR和酶切鉴定, 酶切正确的质粒命名为 GbU6-5Ps∶GUS。分别用Hind III和BamH I酶切植物表达载体 pCAMBIA1300和 6个构建好的 GbU6-5Ps∶ GUS的融合表达载体, 回收目标片段, 连接, 转化,对重组质粒进行酶切鉴定, 酶切正确的质粒被命名为 GbU6-5Ps∶GUS-P1300。不同截短的 GbU6-5Ps启动子对应的GbU6-5Ps∶∶GUS融合表达载体示意图见图1。将鉴定正确的表达载体转入农杆菌GV3101,用于下一步侵染。
1.4 农杆菌真空渗透转化棉花花粉
将构建好的不同GbU6-5Ps∶∶GUS-P1300以及阳性对照CaMV35S∶GUS和阴性对照pCAMBIA1300空载体的农杆菌按1∶100比例接种于LB培养基(含50 µg mL-1Kan和25 µg mL-1Rif)中活化。28℃, 180 转 min-1摇菌, 摇至菌液的OD600=0.6~1.2时, 分别吸取不同体积的活化菌液接种到6 mL LB培养基(含50 µg mL-1Kan和25 µg mL-1Rif)中, 使得每种农杆菌起始菌液OD值相同, 再次28℃, 180转 min-1摇菌, 统一摇至菌液的OD600=1.6时, 12 000×g离心5 min收集菌体, 弃上清液, 重悬于花粉萌发培养基[17]中[0.1% H3BO3、0.3% Ca(NO3)2、0.2% MgSO4·7H2O、0.1% KNO3、45%蔗糖], 收集新鲜棉花花粉, 等量分成多份。每份各加入上述不同的农杆菌重悬液, 并在-0.05 MPa压力下抽真空30 min。每种农杆菌转化进行技术重复3次, 生物学重复2次。

图1 不同截短GbU6-5P启动子驱动报告基因GUS的融合表达载体构建Fig.1 Construction of GUS reporter gene fusion expression vector driven by different truncated GbU6-5P promoters
1.5 不同截短 GbU6-5Ps::GUS在棉花花粉中的瞬时表达分析
将抽完真空后的棉花花粉悬浮液 100×g离心 1 min, 弃上清液, 收集转化后的花粉, 并用蒸馏水漂洗4~5次, 每次100×g离心1 min, 弃上清液, 以去除农杆菌。然后加入适量的 GUS染液(0.5 mol L-1磷酸缓冲液, pH 7.0; 0.5 mol L-1EDTA, pH 8.0; 10% Triton X-100; 20 mmol L-1X-Gluc), 37℃, 180转 min-1振荡染色3~4 h, 100×g离心1 min, 吸取上清GUS染液保留备用。用蒸馏水漂洗染色后的花粉4~5次, 每次100×g离心1 min, 以确保去除残留的GUS染液。最后收集花粉在体视显微镜下观察拍照。同时取每种花粉GUS染液的蓝色上清液在620 nm波长(蓝色上清液的最大吸收波长)处测定光吸收值。
1.6 农杆菌真空渗透与非真空渗透转化效率的比较
根据上一试验结果选取2个转录效率相对较高的截短启动子GbU6-5P6和GbU6-5P7进行真空渗透与非真空渗透转化处理, 分别统计在真空渗透和非真空渗透条件下棉花花粉的转化效率, 转化效率(%)=(被染成蓝色花粉的个数/花粉的总个数)×100,每个处理重复3次, 以探究农杆菌介导转化棉花花粉的试验中, 在转化效率上, 真空渗透与非真空渗透是否存在显著差异。
1.7 其他4种海岛棉U6启动子以及相应截短启动子的克隆与转录活性分析
采用两轮PCR方法, 在海岛棉中分别克隆其他4种U6启动子, 分别命名为GbU6-1P、GbU6-3P、 GbU6-7P、GbU6-9P。然后采用Transfer PCR方法对这4种U6启动子进行2种长度的截短克隆(表1)。以截短的GbU6-5P2∶∶GUS为受体质粒模板, 分别截短 GbU6-1P、GbU6-3P、GbU6-7P、GbU6-9P启动子。同时完成U6启动子与GUS基因融合载体的构建, 克隆测序正确后再连入植物表达载体pCAMBIA1300中并酶切鉴定。分别命名正确的质粒为 GbU6-1P4∶GUS、GbU6-1P5∶GUS、GbU6-3P4∶ GUS、GbU6-3P5∶∶GUS、GbU6-7P4∶∶GUS、GbU6-7P5∶∶GUS、GbU6-9P4∶GUS、GbU6-9P5∶∶GUS。最后转化农杆菌GV3101。同样采用上述农杆菌真空渗透法转化棉花花粉, 分析启动子转录活性。
2 结果与分析
2.1 不同截短GbU6-5P启动子扩增产物鉴定
以测序正确的GbU6-5P启动子克隆质粒为模板,进行 Transfer PCR, 成功克隆了6个不同截短的GbU6-5P启动子片段(图2), 截取的启动子大小分别为GbU6-5P2∶ 672 bp、GbU6-5P3∶ 468 bp、GbU6-5P4∶ 358 bp、GbU6-5P5∶ 280 bp、GbU6-5P6∶ 202 bp、 GbU6-5P7∶ 105 bp。以上截短的启动子质粒经BamH I和Hind III酶切鉴定与预期目标片段大小相符(图略),经测序比对为预期的截短GbU6-5P启动子。

图2 6个不同截短GbU6-5P启动子的Transfer PCR扩增产物电泳检测Fig.2 Transfer PCR amplification of six truncated GbU6-5P promoters
2.2 不同截短 GbU6-5P::GUS融合表达载体的构建
将6个截短的GbU6-5Ps启动子与GUS基因片段连接, 对重组的质粒进行酶切鉴定∶ 用EcoR I单酶GbU6-5P2∶GUS重组质粒切, 产生了3272 bp和 2608 bp两条带; 用EcoR V分别单酶切GbU6-5P3∶∶GUS、GbU6-5P4∶GUS、GbU6-5P5∶∶GUS重组质粒,结果为GbU6-5P3∶GUS产生4096、1495和231 bp条带, GbU6-5P4∶GUS产生3986、1495和231 bp条带, GbU6-5P5∶GUS产生3908、1495和231 bp条带;GbU6-5P6∶GUS、GbU6-5P7∶GUS重组质粒分别用Pst I和Mfe I双酶切, 结果是GbU6-5P6∶GUS产生3404 bp和2149 bp条带, GbU6-5P7∶GUS产生3307 bp和2149 bp条带(图3), 所有酶切结果与预期相符。将6个构建好的GbU6-5P∶GUS的重组基因片段连入植物表达载体pCAMBIA1300, 经酶切鉴定, 酶切结果与预期酶切片段大小相符, 表明不同截短的GbU6-5P驱动GUS基因的植物表达载体GbU6-5P∶ GUS-P1300构建成功(图略)。

图3 GbU6-5Ps::GUS表达载体的酶切鉴定
2.3 不同截短GbU6-5P启动子功能分析
通过GUS组织化学染色发现, GbU6-5P启动子以及6个截短的GbU6-5P启动子均能驱动GUS表达, 棉花花粉被染成蓝色, 但其染色水平存在明显差异。GbU6-5P6∶∶GUS和GbU6-5P7∶GUS染色相对较深, 接近于阳性对照, 而其他长度启动子驱动GUS基因的转化花粉染色相对较弱。其中一个明显的趋势是启动子长度越短, 花粉染色越深(图4)。
2.4 GUS活性测定
为了进一步量化不同截短GbU6-5P启动子的转录活性, 取每种处理转化后的GUS染液上清液, 在其最大光吸收波长 620 nm处测定其光吸收值, 以 pCAMBIA1300的GUS染液为对照。3次重复结果表明, CaMV35S启动子的GUS染色液光吸收值最大,其次是GbU6-5P7和GbU6-5P6, 其他截短启动子随着长度的增加, 其光吸收值也依次降低(图 5), 这与棉花花粉本身染色的结果相吻合。
2.5 真空渗透与非真空渗透转化效率比较
为了探究农杆菌抽真空和不抽真空转化棉花花粉的转化效率是否存在差异, 选用转录活性相对较高的GbU6-5P6和GbU6-5P7试验。结果表明, 抽真空和不抽真空处理存在显著差异(图 6)。GbU6-5P6真空渗透转化效率为 23.7%, 非真空转化效率为12.1%, 真空渗透转化效率比非真空转化效率提高了95.9%; GbU6-5P7真空渗透转化效率为25.1%, 非真空转化效率为 14.5%, 真空渗透转化效率比非真空转化效率提高了73.1%。
2.6 其他4种海岛棉U6启动子的相应截短启动子的转录活性分析
其他4种海岛棉U6启动子的相应截短片段被分别命名为GbU6-1P4 (440 bp), GbU6-1P5 (192 bp);GbU6-3P4 (460 bp), GbU6-3P5 (211 bp); GbU6-7P4(415 bp), GbU6-7P5 (190 bp); GbU6-9P4 (470 bp),GbU6-9P5 (200 bp), GUS融合表达载体酶切鉴定结果图略。通过农杆菌真空渗透转化棉花花粉, 经 GUS组织化学染色发现, 对于 GbU6-1P4∶∶GUS、 GbU6-1P5∶GUS、GbU6-7P5∶GUS可以检测到蓝色花粉, 而其他克隆的不同截短的 GbU6启动子并没有检测到蓝色花粉(图略)。结果还显示较短的GbU6-1P5∶GUS、GbU6-7P5∶GUS比对应长片段的GbU6-1P4∶GUS、GbU6-7P4∶GUS染色显著增强(图7)。在620 nm处光吸收值的结果也显示, GbU6-1P5显著高于GbU6-1P4; 在GbU6-7P4几乎没有光吸收值的情况下, GbU6-7P5的光吸收值却很高(图8)。

图4 不同截短GbU6-5P启动子驱动GUS在棉花花粉中的瞬时表达Fig.4 GUS expression driven by different truncated GbU6-5P promoters in cotton pollen

图5 不同截短GbU6-5P启动子对应的花粉GUS染色后染液光吸收值Fig.5 Light absorbance value at 620 nm of staining solution after histochemical stain corresponding to different truncated GbU6-5P promoters
3 讨论
ΙΙ型CRISPR/Cas9基因组编辑系统自2013年诞生以来, 已在水稻[9,20-22]、小麦[20,23]、玉米[24]、番茄[25]、大豆[8]等作物中成功应用。U6启动子作为利用CRISPR/Cas9基因编辑技术进行棉花分子育种的重要组成部分, 需要它能在生殖细胞中具有高效转录活性, 产生可遗传的新种质。研究表明, 在真核生物中, USE和TATA框是U6启动子发挥转录功能的2个重要元件[6,26]。本课题组在前期的工作中已克隆了5种棉花GbU6启动子, 这5种GbU6启动子同样含有比较保守的-60 bp位置的USE元件和-30 bp位置的TATA框, 利用基因枪瞬时转化的方法从克隆的5种GbU6启动子中筛选出一个转录效率相对较高的 GbU6-5P启动子(1166 bp)。然而由于构建CRISPR/Cas9基因组编辑载体的需要, 所用的 U6启动子在保证具有高转录活性的基础上, 通常不宜过长。因此有必要克隆有效截短的棉花U6启动子。首先将 GbU6-5P启动子截成 6种长短不等的片段, 但都包含完整的USE和 TATA框元件。转录活性分析结果显示, 随着启动子长度的不断截短, 启动子的转录活性却逐渐增强(图4和图5)。在另外2种棉花U6启动子中的分析结果表明, 短片段的 U6启动子的转录活性也显著高于长片段的U6启动子(图7)。不仅如此, 本实验室在番茄 U6启动子克隆与功能分析中也发现了同样的现象(未发表)。以上结果暗示较长的U6启动子上可能含有抑制转录活性的调控元件, 而且这种负调控机制可能在不同物种中非常保守。然而到目前为止, 除USE元件和TATA框外, 在U6启动子上还没有鉴定出其他调控转录的序列元件, 因此有必要对这一现象进一步深入研究。Nekrasov等[15]在基因组编辑技术研究中使用了79 bp长度的U6启动子, 并且取得了理想的实验结果。本研究截短克隆的GbU6-5P7功能启动子长度仅为105 bp, 这也暗示植物中U6启动子可以克隆的更短。是否可以仅保留U6启动子-60 bp位置的USE元件和-30 bp位置的TATA框就能达到理想的转录活性还需要进一步研究。另外结果显示GbU6-3P和GbU6-9P即使启动子被截短后也依然没有表现出转录活性, 原因一方面可能是GbU6-3P和GbU6-9P完全没有功能, 另一种可能是其 U6启动子的功能具有组织特异性,它们在棉花花粉中没有转录活性, 而在其他组织可能具有功能, 这些问题也需要在后续工作中进一步研究。

图6 真空与非真空处理转化效率比较Fig.6 Comparison of transformation efficiency between vacuum and non-vacuum treatments
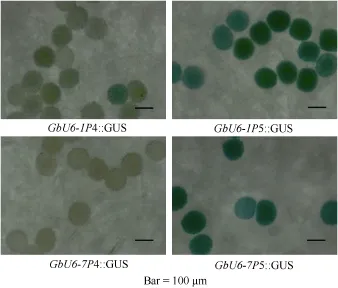
图7 其他截短GbU6启动子花粉GUS染色Fig.7 GUS staining analysis of other truncated GbU6 promoter in cotton pollen

图8 其他截短GbU6启动子GUS染液光吸收值Fig.8 Light absorbance value of GUS staining solution for other truncated GbU6 promoter
棉花是双子叶植物, 易受农杆菌感染, 转化效率较高, 以GUS作为标记基因辅助显微分析等手段在棉花中对瞬时表达跟踪分析能更加快速地鉴定出启动子的转录活性。因此本研究采用农杆菌介导法转化花粉, 对棉花U6启动子的功能进行分析。前人研究表明, 在农杆菌介导的转化过程中, 改变受体材料所处环境的压强以负压真空渗透的形式可以显著提高农杆菌对植物的转化效率[17,27]。棉花花粉在适当的负压下, 组织表面会形成许多微小的创伤,同时分泌一些酚类物质, 有利于对农杆菌的吸附,使得农杆菌通过细胞间隙转移到细胞组织中, 提高转化效率。将构建好的 GbU6-5P∶∶GUS的表达载体利用基因枪瞬时转化法转化棉花花粉, 虽然能够转化成功, 但是存在明显不足, 转化效率较低, 实验成本较高。本研究充分借鉴前人鉴定花粉特异性启动子的方法[16-17], 采用农杆菌真空渗透法转化棉花花粉, 结果显示抽真空处理与不抽真空处理存在显著差异, GbU6-5P6和GbU6-5P7真空处理转化效率分别较非真空处理提高了95.9%和73.1%。由此可见,在农杆菌介导转化法中, 人为增加负压的方式可以明显提高棉花花粉转化的效率。
以上结果显示有效截短的 GbU6启动子转录活性更高, 采用较短的U6启动子可提高sgRNA的转录水平, 最终可能会大幅度提高 CRISPR/Cas9系统基因编辑的效率。
4 结论
克隆了 3个短小的、在棉花花粉细胞中具有转录功能的GbU6启动子; 同一GbU6启动子不同片段大小的转录活性并不相同, 较短的启动子转录活性更强。研究结果也暗示在较长片段的棉花U6启动子上, 可能含有抑制转录活性的调控元件。短的具有转录活性的GbU6-5P7、GbU6-1P5、GbU6-7P5片段可为构建棉花CRISPR/Cas9基因组编辑载体提供更多有效的启动子。
References
[1]Liu L, Fan X D.CRISPR-Cas system∶ a powerful tool for genome engineering.Plant Mol Biol, 2014, 85∶ 209-218
[2]Fichtner F, Urrea Castellanos R, Ülker B.Precision genetic modifications∶ a new era in molecular biology and crop improvement.Planta, 2014, 239∶ 921-939
[3]Cong L, Ran F A, Cox D, Lin S, Barretto R, Habib N, Hsu P D,Wu X, Jiang W, Marraffini L A, Zhang F.Multipl exgenome engineering using CRISPR/Cas systems.Science, 2013, 339∶819-823
[4]Belhaj K, Chaparro-Garcia A, Kamoun S, Nekrasov V.Plant genome editing made easy∶ targeted mutagenesis in model and crop plants using the CRISPR/Cas system.Plant Methods, 2013, 9∶ 39
[5]Belhaj K, Chaparro-Garcia A, Kamoun S, Patron N J, Nekrasov V.Editing plant genomes with CRISPR/Cas9.Curr Opin Biotechnol,2015, 33∶ 76-84
[6]Li X, Jiang D H, Yong K L, Zhang D B.Varied transcriptional efficiencies of multiple Arabidopsis U6 small nuclear RNA genes.J Integr Plant Biol, 2007, 49∶ 222-229
[7]Hyun Y, Kim J, Cho S W, Choi Y, Kim J S, Coupland G.Site-directed mutagenesis in Arabidopsis thaliana using dividing tissue-targeted RGEN of the CRISPR/Cas system to generate heritable null alleles.Planta, 2015, 241∶ 271-284
均为H2。由图3、图4可以看出,D2和H2的取值对于等效应力的影响较大,D2的影响曲线类似于一个二次函数,取值360mm取左右时,等效应力最小;而等效应力总是随着H2的减小而增大。在总变形方面,D1、D2、H2对其起着比较显著的影响。D1和D2的影响曲线可以近似可以认为是二次函数,分别在420mm和345mm左右取得最小值;总形变量随着H2的增大而减小。
[8]Jacobs T B, LaFayette P R, Schmitz R J, Parrott W A.Targeted genome modifications in soybean with CRISPR/Cas9.BMC Biotechnol, 2015, 15∶ 16
[9]Jiang W, Zhou H, Bi H, Fromm M, Yang B, Weeks D P.Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis, tobacco, sorghum and rice.Nucl Acids Res, 2013, 41∶ e188
[10]Wang M B, Helliwell C A, Wu L M, Waterhouse P M, Peacock W J, Dennis E S.Hairpin RNAs derived from RNA polymerase II and polymerase III promoter-directed transgenes are processed differently in plants.RNA Biol, 2008, 14∶ 903-913
[11]Domitrovich A M, Kunkel G R.Multiple, dispersed human U6 small nuclear RNA genes with varied transcriptional efficiencies.Nucl Acids Res, 2003, 31∶ 2344-2352
[12]雷建峰, 伍娟, 陈晓俊, 於添平, 倪志勇, 李月, 张巨松, 刘晓东.棉花花粉中高效转录U6启动子的克隆及功能分析.中国农业科学, 2015, 48∶ 3794-3802 Lei J F, Wu J, Chen X J, Yu T P, Ni Z Y, Li Y, Zhang J S, Liu X D.Cloning and functional analysis of cotton U6 promoter with high transcription activity in cotton pollen.Sci Agric Sin, 2015, 48∶3794-3802 (in Chinese with English abstract)
[13]Fauser F, Schiml S, Puchta H.Both CRISPR/Cas-based nucleases and nickases can be used efficiently for genome engineering in Arabidopsis thaliana.Plant J, 2014, 79∶ 348-359
[14]Feng Z Y, Mao Y F, Xu N F, Zhang B T, Wei P L, Yang D L,Wang Z, Zhang Z J, Zheng R, Yang L, Zeng L, Liu X D, Zhu J K.Multigeneration analysis reveals the inheritance, specificity, and patterns of CRISPR/Cas-induced gene modifications in Arabidopsis.Proc Natl Acad Sci USA, 2014, 111∶ 4632-4637
[15]Nekrasov V, Staskawicz B, Weigel D, Jones J D, Kamoun S.Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guide dendonuclease.Nat Biotechnol, 2013, 31∶691-693
[16]李晓, 王学德.根癌农杆菌转化棉花花粉的研究.棉花学报,2004, 16∶ 323-327 Li X, Wang X D.Study on transformation of cotton pollen using Agrobacterium tumefaciens.Cotton Sci, 2004, 16∶ 323-327 (in Chinese with English abstract)
[17]张燕红, 黄乐平, 周小云, 王冬梅.农杆菌真空渗透法转化棉花花粉的初步研究.棉花学报, 2008, 20∶ 354-358 Zhang Y H, Huang L P, Zhou X Y, Wang D M.The preliminary study on transformation of cotton pollen using Agrobacteriummediated vacuum infiltration.Cotton Sci, 2008, 20∶ 354-358 (in Chinese with English abstract)
[18]Feng Z Y, Zhang B, Ding W, Liu X D, Yang D L, Wei P, Cao F, Zhu S, Zhang F, Mao Y, Zhu J K.Efficient genome editing in plants using a CRISPR/Cas system.Cell Res, 2013, 23∶ 1229-1232
[19]Erijman A, Shifman J M, Peleg Y.A single-tube assembly of DNA using the transfer-PCR (TPCR) platform.Methods Mol Biol, 2014, 1116∶ 89-101
[20]Shan Q, Wang Y, Li J, Gao C.Genome editing in rice and wheatusing the CRISPR/Cas system.Nat Prot, 2014, 9∶ 2395-2410
[21]Endo M, Mikami M, Toki S.Multi-gene knockout utilizing off-target mutations of the CRISPR/Cas9 system in rice.Plant Cell Physiol, 2014, 56∶ 41-47
[22]Miao J, Guo D, Zhang J, Huang Q, Qin G, Zhang X, Wan J, Gu H,Qu L J.Targeted mutagenesis in rice using CRISPR-Cas system.Cell Res, 2013, 23∶ 1233-1236
[23]Santosh K U, Jitesh K, Anshu A, Rakesh T.RNA-guided genome editing for target gene mutations in wheat.Genes Genomes Genetics (Bethesda), 2013, 3∶ 2233-2238
[24]Liang Z, Zhang K, Chen K, Gao C.Targeted mutagenesis in Zea mays using TALENs and the CRISPR/Cas System.J Genet Genomics, 2014, 41∶ 63-68
[25]Christopher B, Vladimir N, Zachary B.Lippman, Joyce V E.Efficient gene editing in tomato in the first generation using the clustered regularly interspaced short palindromic repeats/CRISPR-associated9 system.Plant Physiol, 2014, 166∶1292-1297
[26]Marshallsay C, Kiss T, Filipowicz W.Amplification of plant U3 and U6 snRNA gene sequences using primers specific for an upstream promoter element and conserved intragenic regions.Nucl Acids Res, 1990, 18∶ 3459-3466
[27]Tomson M, Manjula S.Optimization of Agrobacteriummediated transient gene expression and endogenous gene silencing in Piper colubrinum Link.by vacuum infiltration.Plant Cell Tissue Organ Cult, 2011, 105∶ 113-119
DOI:10.3724/SP.J.1006.2016.00675
*通讯作者(
Corresponding authors)∶ 刘晓东, E-mail∶ xiaodongliu75@aliyun.com; 张巨松, E-mail∶ xjndzjs@163.com
收稿日期Received()∶ 2015-10-25; Accepted(接受日期)∶ 2016-01-11; Published online(网络出版日期)∶ 2016-02-18.
Cloning and Functional Analysis of Different Truncated GbU6 Promoters in Cotton
LEI Jian-Feng1, LI Yue1, XU Xin-Xia2, AERZUGULI·Tashi1, PU Yan1, ZHANG Ju-Song1, and LIU Xiao-Dong1,*
College of Agronomy, Xinjiang Agricultural University, Laboratory of Agricultural Biotechnology of Xinjiang Agricultural University, Urumqi 830052, China; Center of Bazhou Agricultural Technology Promotion, Korla 841000, China
Abstract:U6 promoter is an important element for the transcription of sgRNA in the CRISPR/Cas9 genome editing system.Using a short promoter is also one of the basic requirements for the construction of CRISPR/Cas9 genome editing vector.According to the sequence of GbU6-5P promoter cloned, six different truncated U6 promoters were successfully cloned using Transfer PCR method and their length were 672, 468, 358, 280, 202, and 105 bp, respectively.Together with GbU6-5P∶GUS-pCAMBIA1300, GUS fusion expression vectors driven by corresponding truncated promoter were constructed and transformed into cotton pollen by the vacuum in filtration transformation method.Results of GUS histochemical staining showed that the cloned seven truncated GbU6-5P promoters could drive GUS expression in cotton pollen and all corresponding cotton pollen could be stained blue, but there exist different shades among them.Results showed that the shorter promoter, the stronger transcription activity, and so did the two other GbU6 promoter.In this study three short GbU6 promoters with transcription activity in cotton pollen were cloned.GUS staining results showed that the shorter U6 promoter had higher transcriptional activity, which was the common characteristics in different U6 promoter.The above results indicated that using shorter U6 promoter not onlyconform to the requirement of constructing CRISPR/Cas9 genome editing vector, but also improve the transcription of sgRNA,which may enhance the efficiency of genome editing finally.
Keywords:Cotton; GbU6 promoter; Truncated cloning; Vacuum infiltration; Cotton pollen

