基于基因组编辑技术的大片段克隆新策略
曾哲,任晓丹,杨晟
中国科学院上海生命科学研究院 植物生理生态研究所 中国科学院合成生物学重点实验室,上海 200032
基于基因组编辑技术的大片段克隆新策略
曾哲,任晓丹,杨晟
中国科学院上海生命科学研究院 植物生理生态研究所 中国科学院合成生物学重点实验室,上海200032
曾哲, 任晓丹, 杨晟. 基于基因组编辑技术的大片段克隆新策略. 生物工程学报, 2016, 32(4): 401–408.
Zeng Z, Ren XD, Yang S. New methods for cloning large gene clusters based on CRISPR/cas9. Chin J Biotech, 2016, 32(4): 401–408.
摘要:基因组大片段克隆技术是合成生物学研究领域关键的使能技术。传统的大片段克隆技术获取目的大片段的手段存在各种缺陷,比如随机建库克隆需要依靠高通量筛选;PCR难以扩增10 kb以上片段,从小片段拼装费时费力且突变率高;基于限制性酶切连接难以找到片段两端适宜的限制性内切酶酶切位点。最近全基因组合成等前沿研究创造了全新的高性能大片段克隆方法,比如CRISPR/cas9系统中cas9可识别并切割20 bp核酸序列解决了识别位点设计难题,可用来获取任意目的基因片段;组合Gibson或者酵母偶联重组技术组装技术,可高效克隆大片段基因。本文将分类介绍基因组大片段克隆技术,并提出适用不同尺度大小基因克隆的技术选择参考标准。
关键词:大片段克隆,合成生物学,基因组编辑,Gibson组装
Received: November 19, 2015; Accepted: January 22, 2016
Supported by: National Natural Science Foundation of China (No. 31170102).
国家自然科学基金 (No. 31170102) 资助。
1 应用价值及意义
基因组大片段克隆是一项重要的合成生物学技术,在构建高价值产物的工程菌株中更是不可或缺[1-3]。基因组大片段克隆技术已被广泛运用于抗菌药物、生物燃料和高价值次级代谢产物等方面[4-7];如在大肠杆菌中异源克隆表达生产青蒿酸[2]、在天蓝链霉菌中克隆表达异源沉默基因获得新型抗生素[8]、在产黄青霉菌Penicillium chrysogenum中克隆表达青霉素合成基因簇完成工业产量提升[9-10]等。表1列举大片段克隆的成功实例。

表1 大片段基因克隆技术的应用实例Table 1 Examples of large gene clusters cloning methods
基因克隆技术虽然经过四十多年的发展,但这项技术仍存在一些局限,特别是对于大片段基因克隆[1,5]。大片段基因克隆主要包括目的基因片段的切割获得或PCR扩增获得和目的基因片段的组装及体内克隆。其中目的大片段的组装及体内克隆已有Gibson组装[20]和TAR酵母转化偶联重组[21]等多项有效技术手段;而目的大片段的获得却受限于传统的限制性内切酶切割获得或者PCR扩增等,这也是实现大片段基因克隆的“拦路虎”。
值得庆幸的是近年热门的CRISPR-cas9系统中,“分子剪刀”cas9可特异性识别20个碱基并实现精准切割[22-23]。因此,cas9已被成功开发用于切割获取目的基因片段,并且组合了Gibson组装或者TAR酵母转化偶联重组等技术[18-19,24],这必将促进基因组大片段克隆技术的进一步发展。本文将区别于国内现有CRISPR综述,主要论述基于基因组编辑技术而发展的大片段克隆新策略。
2 分类介绍及评价
基因克隆技术经过40多年的发展,早已形成多套技术手段。根据目的基因片段的获得方式,可分为随机建库克隆、PCR扩增和定点酶切3类 (图1);根据目的基因片段的组装方式,可分为连接和重组两类 (图1)。以下将对此展开介绍克隆技术。

图1 大片段克隆技术的分类介绍Fig. 1 The classification of cloning technology for large gene clusters. Cloning large clustersshould include the two steps, obtaining gene fragments and assembling them. Using Cas9 to cut genome and get an objective gene fragment is a new method for cloning large gene clusters.
随机建库克隆通过部分限制性内切酶切割或者机械剪切力使基因组断裂成合适大小的基因片段,再利用装载质粒组装全部基因片段,最后导入至克隆宿主中大量复制[25]。因此,随机建库耗费大量劳力,更是必须依赖高通量筛选方法将含有目的片段转化子挑选出来,同时基因片段的完整性不确定[2]。
PCR扩增需要依靠部分序列信息设计扩增引物,这是体外克隆基因的最简便方法[25]。但是,PCR很难扩增获得10 kb以上片段,因为DNA聚合酶体外扩增活性受限于扩增长度和扩增片段的GC含量等。同时,PCR扩增容易引入核苷酸点突变造成基因片段失真。利用PCR分级扩增小片段再组装获得完整大片段的方法费时费力,并不是一种高效的克隆技术[1,26]。表2笔者罗列比较现有常见基因克隆技术的优劣。
限制性内切酶切割获取目的片段很难在片段两端选择到合适的酶切位点。因为限制性内切酶只识别6−8个碱基位点,在庞大的基因组上必然存在多个酶切位点 (统计意义上,一个具有8 bp识别位点的限制性内切酶在含有65 536个碱基的随机序列内将至少含有两个识别位点),即我们很难在所需克隆的大片段两端选取到合适的限制性内切酶酶切位点[24,27]。
因此,我们希望有一项技术可以从基因组特异切割获得目的片段。而近年来热门的CRISPR/cas9系统中,来自化脓性链球菌的cas9核酸酶可与引导RNA (gRNA) 形成复合体识别20个核酸序列进行切割,最终形成双链DNA断裂 (DSB)。可见cas9具有定点识别序列进行切割的功能。cas9通过与gRNA形成复合体,早已被证实可具有体外切割活性;cas9复合物识别20个核酸序列,具有极强的位点识别特异性,远胜于限制性内切酶的位点识别特异性。因此,cas9已被开发用于切割获取目的基因片段,并且组合Gibson组装或者TAR酵母转化偶联重组等技术,形成完整的大片段基因克隆技术。下文将对CRISPR/cas9组合TAR和CRISPR/cas9组合Gibson技术展开介绍。
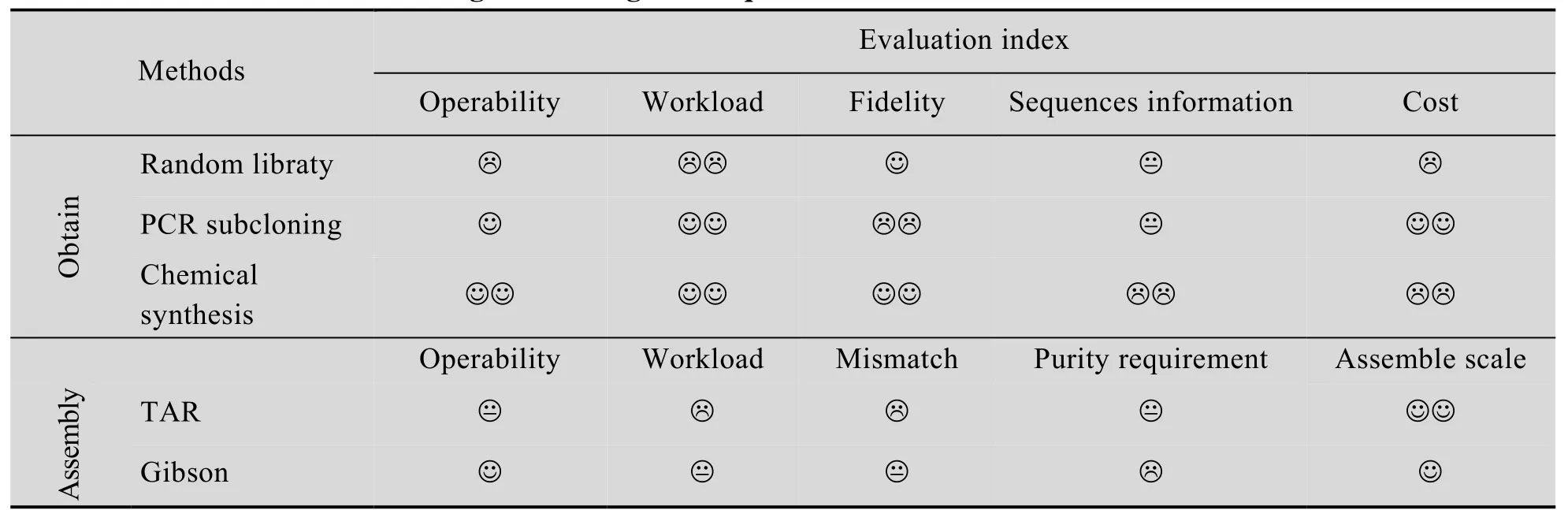
表2 常见基因克隆技术的优劣对比Table 2 Evaluation of common gene cloning techniques
3 优化思路及策略
基于组合优化的思路,科研人员通过在目的大片段的获得和目的大片段的组装两个方面进行技术组合优化,已形成以下3种组合策略(图2):1) 限制性酶切组合同源质粒骨架PCR;2) CRISPR/cas9组合TAR;3) CRISPR/cas9组合Gibson。
3.1限制性酶切组合同源质粒骨架PCR
该项组合策略由清华大学陈国强课题组于2012年开发,即单链重复配对退火扩增(Single-strand overlapping annealing, SSOA)[28],该策略中的同源重组连接得益于张友明等2000年所开发的Red/ET同源重组系统[29]。同源重组连接需要依赖目的片段和用于克隆线性化质粒骨架两端的同源臂匹配;一般采用固定的克隆线性化质粒骨架和通过大片段两端PCR扩增引物带入少于50 bp的同源臂。但由于大片段很难通过PCR扩增获得,SSOA采用直接限制性内切酶切割获得片段,同时以大片段两端为引物扩增获得与大片段同源匹配的克隆线性化质粒骨架。简单地说,原来同源重组连接难以通过PCR在大片段两端加上同源臂,SSOA逆向通过PCR扩增在质粒骨架上加上大片段的同源臂。该方法操作流程详见图4。
该方法把原来大片段两端难以扩增加入质粒骨架同源臂的难题转化为可实行的扩增质粒骨架加入大片段两端同源臂。SSOA可实现在大肠杆菌中完成小于28 kb的基因克隆。当然该方法仍无法克服限制性内切酶识别位点特异性差的难题。

图2 大片段基因克隆技术优化策略操作示意[18-19,28]Fig. 2 Workflow of the optimized cloning strategies for large gene clusters[18-19,28].
3.2CRISPR/cas9+TAR
2015年初报道的CRISPR/cas9组合TAR的大片段克隆方法通过cas9复合体过夜体外切割基因组获得特异性高的目的片段,然后直接将cas9切割产物和两端带有300 bp同源臂的细菌人工染色体 (BAC) 导入酵母,利用酵母较高的同源重组能力,最终完成将目的大片段与BAC连接[18]。带有目的大片段的BAC通过筛选转化子获得。该方法利用cas9获得特异性高的目的片段,减少假阳性转化子,将原来TAR转化子阳性率由2%提高至32%;实验耗时也压缩至4 d。
3.3CRISPR/cas9+Gibson
2015年9月清华大学朱听和中国科学院微生物研究所娄春波等联合开发CRISPR/cas9组合Gibson的大片段克隆策略[19]。该方法通过将菌体固定在低熔点琼脂糖胶内,经过裂解菌体和缓冲液清洗使菌体基因组存于低熔点胶内。在低熔点胶环境下,cas9复合体体外过夜切割基因组获得特异性高的目的片段,然后利用Gibson同源重组与PCR克隆获得的质粒骨架连接 (30 bp overlap),导入大肠杆菌后可获得转化子。该方法成功克隆最大150 kb的目的片段,实验耗时也只需2 d。当然cas9复合物切割获得大片段基因也可在水溶液中操作,2015年4月发表于BioTechniques上的文章采用cas9在水溶液中切割并基于Gibson组装,最终只能实现最大22 kb的基因克隆[24]。从大片段克隆尺度上看,cas9在水溶液中切割基因组明显劣于低熔点胶内切割基因组,低熔点胶制备基因组有助于保持基因组的完整性。
4 技术选择参考标准
上文介绍多种大片段基因克隆技术,那么对于不同基因尺度,我们应该如何选用以上技术呢?基于对以上技术的效率对比和笔者实验室的大片段克隆经验[30],我们提出以下选择参考标准 (图3)。

图3 基因组大片段克隆技术选择参考标准Fig. 3 The choice criteria of cloning strategies for large gene clusters.
当所需克隆基因簇小于15 kb时,首选PCR扩增分段获得小于5 kb的小片段,再利用同源重组连接将小片段拼装成完整的目的片段。在放线菌属等高GC含量的物种中,利用基因组模板进行PCR扩增时,可加入5%的二甲基亚砜以降低PCR退火温度。
当所需克隆基因簇大于15 kb且小于25 kb时,可利用PCR分级扩增获得小片段,再利用Gibson逐级组装[30]。在设计Gibson组装两端连接区时,应该尽量选择GC含量低的区域,以减少克隆载体自连的情况。
当所需克隆基因簇大于25 kb时,应当选用CRISPR/cas9切割基因组获得目的片段。而目的片段与克隆载体的连接及组装可选择Gibson或者TAR;其中Gibson组装需要大于100 ng的目的片段且纯度要求高,TAR组装对目的片段的含量和纯度没有较高要求但转化子阳性率较低。
因此,在进行大片段基因克隆时,我们应当根据所需克隆基因片段的大小、GC含量和所获得片段情况等选择恰当的基因克隆技术。
致谢:感谢中国科学院微生物研究所娄春波研究员在论文写作中的宝贵意见及建议。
REFERENCES
[1] Cobb RE, Zhao HM. Direct cloning of large genomic sequences. Nat Biotechnol, 2012, 30(5): 405–406.
[2] Keasling JD. Synthetic biology and the development of tools for metabolic engineering. Metabol Eng, 2012, 14(3): 189–195.
[3] Weber T, Charusanti P, Musuol-Kroll EM, et al. Metabolic engineering of antibiotic factories: new tools for antibiotic production in actinomycetes. Trends Biotechnol, 2015, 33(1): 15–26.
[4] Rutledge J, Challis GL. Discovery of microbial natural products by activation of silent biosynthetic gene clusters. Nat Rev Microbiol, 2015, 13(8): 509–523.
[5] Cobb RE, Ning JC, Zhao HM. DNA assembly techniques for next-generation combinatorial biosynthesis of natural products. J Ind Microbiol Biotechnol, 2014, 41(2): 469–477.
[6] Izawa M, Kawasaki T, Hayakawa Y. Cloning and heterologous expression of the thioviridamide biosynthesis gene cluster from Streptomyces olivoviridis. Appl Environ Microbiol, 2013, 79(22): 7110–7113.
[7] Gronenberg LS, Marcheschi RJ, Liao JC. Next generation biofuel engineering in prokaryotes. Curr Opin Chem Biol, 2013, 17(3): 462–471.
[8] Yamanaka K, Reyniolds KA, Kersten RD, et al. Direct cloning and refactoring of a silent lipopeptide biosynthetic gene cluster yields the antibiotic taromycin A. Proc Natl Acad Sci USA, 2014, 111(5): 1957–1962.
[9] Thykaer J, Nielsen J. Metabolic engineering of β-lactam production. Metabol Eng, 2003,5(1): 56–69.
[10] Newbert RW, Barton B, Gresves P, et al. Analysis of a commercially improved Penicillium chrysogenum strain series: involvement of recombinogenic regions in amplification and deletion of the penicillin biosynthesis gene cluster. J Ind Microbiol Biotechnol, 1997, 19(1): 18–27.
[11] Miao V, Coёffet-LeGalMF,Brian P, et al. Daptomycin biosynthesis in Streptomyces roseosporus: cloning and analysis of the gene cluster and revision of peptide stereochemistry. Microbiology, 2005, 151(5): 1507–1523.
[12] Chen WH, Qin ZJ, Wang J, et al. The MASTER (methylation-assisted tailorable ends rational) ligation method for seamless DNA assembly. Nucl Acids Res, 2013, 41(8): e93.
[13] Gibson DG, Bender GA, Andrews-Pfannkoch C, et al. Complete chemical synthesis, assembly, and cloning of a Mycoplasma genitalium genome. Science, 2008, 319(5867): 1215–1220.
[14] Zhang HR, Wang Y, Wu JQ, et al. Complete biosynthesis of erythromycin A and designed analogs using E. coli as a heterologous host. ChemBiol, 2010, 17(11): 1232–1240.
[15] Shao ZY, Rao GD, Li C, et al. Refactoring the silent spectinabilin gene cluster using a plug-and-play scaffold. ACS Synth Biol, 2013, 2(11): 662–669.
[16] Zhang L, Zhao GP, Ding XM. Tandem assembly of the epothilone biosynthetic gene cluster by in vitro site-specific recombination. Sci Rep, 2011, doi:10.1038/srep00141.
[17] Westfall PJ, Pitera DJ, Lenihan JR, et al. Production of amorphadiene in yeast, and its conversion to dihydroartemisinic acid, precursor to the antimalarial agent artemisinin. Proc Natl Acad Sci U S A, 2012, 109(3): E111–E118.
[18] LeeNCO, LarionovV, Kouprina N. Highly efficient CRISPR/Cas9-mediated TAR cloning of genes and chromosomal loci from complex genomes in yeast. Nucl Acids Res, 2015, 43(8): e55. doi: 10.1093/nar/gkv112.
[19] Jiang WJ, Zhao XJ, Gabrieli T, et al. Cas9-Assisted targeting of CHromosome segments CATCH enables one-step targeted cloning of large gene clusters. Nat Commun, 2015, 6: 8101. doi:10.1038/ncomms9101.
[20] Gibson DG, Young L, Chuang RY, et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Meth, 2009, 6(5): 343–345.
[21] Larionov V, Kouprina N, Graves J, et al. Specific cloning of human DNA as yeast artificial chromosomes by transformation-associated recombination. Proc Natl Acad Sci USA, 1996, 93(1): 491–496.
[22] Garneau JE, Dupuis MÈ, Villion M, et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature, 2010, 468(7320): 67–71.
[23] Hsu DP, Lander E, ZhangF. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262–1278.
[24] Wang JW, Wang A, Li K, et al. CRISPR/Cas9 nuclease cleavage combined with Gibson assembly for seamless cloning. Biotechniques, 2015, 58(4): 161–170.
[25] Karas BJ, Suzuki Y, Weyman PD. Strategies for cloning and manipulating natural and synthetic chromosomes. Chromosome Res, 2015, 23(1): 57–68.
[26] Hommelsheim CM, Frantzeskakis L, Huang MM, et al. PCR amplification of repetitive DNA: a limitation to genome editing technologies and many other applications. Sci Rep, 2014, 4: 5052. doi: 10.1038/srep05052.
[27] ThiemeF, Engler C, Kandzia R, et al. Quick and clean cloning: a ligation-independent cloning strategy for selective cloning of specific PCR products from non-specific mixes. PLoS ONE, 2011, 6(6): e20556.
[28] WangRY, Shi ZY, Chen JC, et al. Cloning large gene clusters from E. coli using in vitro single-strand overlapping annealing. ACS Synth Biol, 2012, 1(7): 291–295.
[29] Zhang YM, Muyrers JPP, Testa G, et al. DNA cloning by homologous recombination in Escherichia coli. Nat Biotechnol, 2000, 18(12): 1314–1317.
[30] LiL, Zhao YW, Ruan LJ, et al. A stepwise increase in pristinamycin II biosynthesis by Streptomyces pristinaespiralis through combinatorial metabolic engineering. Metabol Eng, 2015, 29: 12–25.
(本文责编 陈宏宇)
New methods for cloning large gene clusters based on CRISPR/cas9
Zhe Zeng, Xiaodan Ren, and Sheng Yang
CAS Key Laboratory of Synthetic Biology, Institute of Plant Physiology and Ecology, Shanghai Institutes for Biological Sciences, Chinese Academy of Sciences (CAS), Shanghai 200032, China
Abstract:Cloning of large genomic sequences is an enabling technology in synthetic biology. To obtain large gene fragments, traditional cloning methods are faced with various defects, for instance, random library cloning relies always on high-throughput screening. It is difficult to get gene fragments more than 10 kb by PCR amplification. Assembly of small fragments is labor intensive with high mutation rates. It is difficult to find suitable cleavage sites on the fragment ends byrestriction endonuclease. Recently genome-wide editing creates a new high-performance large fragments cloning methods. For example, CRISPR/cas9 system can identify and cut 20 bp nucleic acid sequences recognition sites used to obtain any desired gene fragments; if combined with Gibson or transformation associated recombination (TAR) assembly technology, these methods can efficiently clone large fragments. This article introduces large fragments cloning technology by classification, then proposes the choice criteria of methods for cloning gene fragments of different sizes.
Keywords:cloning large gene cluster, synthetic biology, CRISPR/cas9, Gibson assembly
DOI:10.13345/j.cjb.150491
Corresponding author:Sheng Yang. Tel/Fax: +86-21-54924175; E-mail: syang@sibs.ac.cn

