SUFFIX TREE 文件生成器
Aleksejs+Udris 刘岩


摘要:后缀树是一个功能强大的数据结构,可以用于计算机科学执行字符串后处理操作。使用树结构的一个挑战是,随着树的生长、树的结构变得难以想象。该文的项目就是针对后缀树的这一问题,通过使用三维空间来改善树的呈现效果。项目的目的将允许用户在没有重叠显示的情况下,大幅增加从屏幕上获得的数据量。这个项目将着眼于渲染定向图,如在双曲空间的后缀树。
中图分类号:TP311 文献标识码:A 文章编号:1009-3044(2016)13-0077-03
1目标
这个项目是为了在屏幕上通过提供一个有效的数据管理方法,从而改善当前DNA字符串后缀树结构的可视化水平。为了实现这个项目,从输入DNA字符串样本和翻译获得结构到LibSea图格式都使用BioJava生物信息学库来构造后缀树结构。这种格式是为了使处理资源消耗最小化,并且可以在双曲空间里用作海象源工具来显示和导航指示图。
2 介绍
在过去的几年中,可用的生物数据结构体积,如DNA和蛋白质序列大大增加。计算机硬件的不断发展使得它可以处理和分析越来越多从生物中检索到的数据信息。这种增长使生物信息学领域得到提升和发展。随着领域的发展,要求新数据结构能有效的存储和分析,从而获得所需信息。
后缀树是一个有向图的数据结构,在生物信息学领域被用于支持高效和强大的运作。[1] 例如,模式匹配, 近似模式匹配, 寻找共同的子字符串, 文本压缩等。所有这些都可以应用于研究和分析显示为字符串的DNA序列。[2-3]然而,当后缀树被用于构造信息结构图的时候,是非常大的,例如DNA序列,加工信息的大小为显示结果创造了一个重大的困难。就是数据显示可能因过大而不可读。
3 后缀树
字符串S的符号m的后缀树T是一个带根节点的有向树。这样一个后缀树具有精确的m叶子被标记为从1到m的值(图1)。后缀树的每一个内部节点都至少有两个子节点,每一个树的边缘都包含了一个非空的S子串。同一个节点不同边缘的符号不能拥有相同的标签。一个后缀树结构的关键特性是每一个叶节点i, 根点到i的标签串联通常会返回从节点i位置开始的S的准确后缀。这意味着,这个路径写为S[i...m]。[2,10]
通常终止符号$被加到S的末尾,并被用于防止S最后一个后缀与另外一个给定的字符串的后缀的前缀相配。在这类事例当中,树可能无法满足上述结构的定义。为了防止S最后一个后缀与终止符的给定输入串的前缀匹配。终止符$被添加到了开始符列。
3.1 Ukkonen后缀树算法
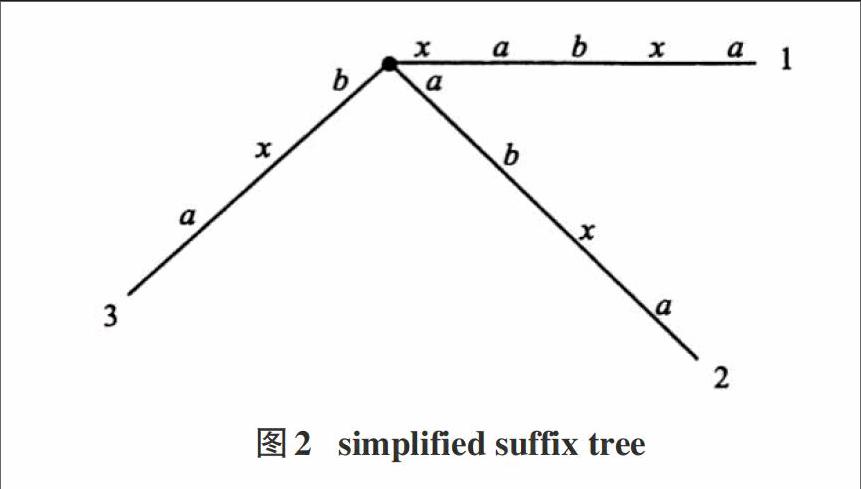
Ukkonen算法构建了一个后缀树的简化版本,之后转变成了S字串的真实后缀树。一串字符简化的后缀树,是一种从没有树边缘终止符存在,并消除了无标签边缘,以及没有满足关键特性,且子节点2个以下的节点的S$中得到的后缀树[10]。Ukkonen的算法是构成了每个S[1…i]前缀的Ti的简化后缀树,以T1开始,增加值到i,直到树Tm完整。完整的后缀树S是根据O(m)时间内的Tm而构造的[10](图2)。
3.2 BioJava
BioJava平台下的一个开源工程项目,旨在为处理和分析生物学数据提供程序库。BioJava项目的目的是推进生物信息学应用程序的发展[11]。这个项目使用了BioJavaAPI版本1.7.1。尽管从来没有BioJava库的版本,最新版本的数据库中没有本项目所必须的类别。Ukkonen后缀树和前缀树不属于BioJava更新的管理类别当中。
3.3 LibSea
概述:LibSea是由CAIDA团队开发的图表文件格式,从而以一种有弹性,可扩展并可以储存的方式去呈现大量的数据结构。通过这种格式,用户可以使用节点,边缘和路径链环元素等对需要的定向图的拓扑结构进行定义。在图表所有元素当中会有额外的数据,作为其属性特征。图表格式在可提供的属性数量上没有限制,且可以为这些属性接收不同的数据类型[17]。LibSea以图表扩展名形式储存为文本文件。图表文件的结构由5部分组成:元数据,结构数据,属性数据,可视化提示和界面提示[17]。
3.3.1 LibSea元数据
这个部分包含了关于图表的信息,比如图表名字,提供的描述,节点数量,边缘数量,路径数量,和路径链环数量等。每个节点,每个边缘和路径都含有指定的指标,这些指标可用于连接文件中的字符实体。编号以0开始,所以整个字符实体给定的下标也是从0到特定实体-1。
[Graph
{### metadata ###
@name = “OurSuffixTree”;
@description=”Description of the suffix tree”;
@numNodes=6;
@numLinks=5
@numPath=0;
@numPathLinks=0; ]
3.3.2 LibSea结构数据
结构数据定义了节点与图表之间的连接。图表的边缘必须总是定向的。这个连接包含从源到目的地的一个列表。线13的节点0到1的第一个链接的链接下标为0。
[{### structural data ###
@links=[
{ @source = 0; @destination=1; },
{ @source = 0; @destination=2; },
{ @source = 0; @destination=3; },
{ @source = 3; @destination=4; },
{ @source = 3; @destination=5; },
];
@paths=; ]
3.3.3 LibSea属性资料
属性可以提供图表字符实体的数据连接。图表节点,链接和路径可能保护任何属性数量。每个属性都可以表示为单一类型的一列。图表的所有属性都可以表现为以一个单一的属性定义表。列表的每个条目定义了这个属性的名称和类型。属性方块保护三个列表:节点值,链接值和路径值。这些列表使用{ id; value }配对,组成了从图表字符实体指标到列表中值的制图。
[@linkValues=[
{ @id=1; @value={ 1.0f; 0.0f; 0.0f; }; }
]; ]
这个线表明,红色映射到了指数为1的图表节点。
3.3.4 LibSea可视化提示和界面提示
这两个部分是作为辅助物为Walrus工具提供额外信息的。可视化和截面提示可以指定渲染选择器和过滤器或菜单,然后在图表被载入到渲染工具之后变得活跃起来。由于有些功能不支持,所以,这些部分并没有被用到这个项目当中来。
4 Walrus
Walrus是一个在三维空间里渲染大型定向图的独立工具。这个工具可以使用三维双曲线几何学来渲染一个范围内的图表。关于与屏幕的距离,每个节点都被放大了。这个物体距离屏幕越远,用户看到的也就越小。(图3)
5 实现
1)GUI.java构建了可以进行辅助性测试操作的框架。GUI等级构建了一个类别树的事例,并可以使用输入串序列来读取文本文件,和运行脚本类的主要功能,从而生成LibSea图表文件。
2) Record.java类别创建了一系列可以保存BioJavaUkkonen后缀树图表节点不可用参数的记录。
3)实现这个记录类别的原因是对母树节点和子树节点的信息进行维护,因为这些信息是无法从BioJava类别当中直接获取的。Ukkonen后缀树类存储了大部分关于被保护的子类实体的节点—SimpleNode的信息。Java遗传机制使得从这些被保护的实体当中获取和检索信息成为不可能的事情。从树种可以获得的关于节点的唯一信息是节点标签。记录类作为容器,实现了保存必要信息以维持后缀树结构的目的。这个类别有()集和获得()方法,且无法从构造器当中获得任何参数。
4) Script.java(脚本.java)使用从类别树种获得的信息创建一个记录列表,并使用记录中可用的信息生成LibSea格式的文件。
5)脚本类别为该项目的分类提供了必要的处理。脚本构造器需要两个参数:公共脚本public Script(树t,连成一列),脚本将从t代表的树种构建图表文件,然后就把获取文件的路径打印出来。这个类别有两种主要方法:运行脚本runScript()和编写图表writeGraph()。
6)公共voidrunScript() 方式可以从树中检索出一列节点,并创建一系列的记录列表。如之前所述,脚本必须创建一系列信息记录,以维护Ukkonen后缀树结构。
7)这个方法是以将树根加到数组当中为开端。再根源加到了数组当中之后,脚本可以确定每个节点的根源,并检测数组当中的元素。给定的当前节点n, 节点n-1是n的有效根源,如果:n的标签是以n-1的标签开始,n-1不需要终止符号$或它并不是树的根源。当这个迭代完成了runScript()方式就可以对脚本类别-writeGraph()所要求的第二个主要功能进行实现。
8)writeGraph()创建了一个BufferedWriter的实体,并编写了五个LibSea格式字符串到输出文件当中。字符串是通过援引辅助方法而生产的。每个辅助方法都可以创建LibSea图表文件所要求的五个部分的其中一个部分。
9) Tree.java从BioJavaorg和biojava符号程序包扩展为Ukkonen后缀树类别,并创建Ukkonen后缀树结构。这种扩展在获取隐藏在Ukkonen后缀树类别当中的所有节点AllNodes ()和标签Label(SuffixNoden) (后缀节点n)时是必要的。
6 结果
成熟的系统成功地为可视化工具Walrus构建了LibSea图表。
1)工具可以读取保护字符串序列的文件并使用Ukkonen算法构建后缀树。这个计算方法是由BioJava程序库为生物资讯v 1.7.1提供。
2)这个系统创建了可以保存关于节点和后缀树信息的记录列表。可以从Ukkonen后缀树被保护实体或直接用这个系统计算获取所需信息。
3)脚本类别恢复了记录列表中遗失的结构,并创建LibSea图表文件。
4)该系统与保护4个字母“ACGT”的序列一起操作。系统可以指定颜色属性到构建这个字母集的节点。
然后,LibSea图表构造时间受到制定后缀树制定节点和边缘属性数量的影响。我们从图表节点制定属性数量增加的长序列中,看到图表文件产生速度的明显变慢。
7 结束语
这个项目的目的在于寻找一种可以用三维结构展示后缀树的方法,这个方法可以给用户可视化地呈现不断增加的数据。
由于某些Walrus限制,这个系统并不支持后缀树所有功能。然而,这些优势的结合可以提供比二维后缀树应用更高的可视化水平[16-17]。
参考文献:
[1] Mehta D P, Sartaj Sahni.Handbook of Data Structures and Applications[M]. London: Chapman and Hall/CRC, 2004: 1387.
[2] Dan Gusfield. Algorithms on strings, trees, and sequences computer science and computational biology[M]. Cambridge University Press, 1997:110-140.
[3] Aluru, Srinivas. Handbook of Computational Molecular Biology[M]. London: Chapman and Hall/CRC, 2005:1104.
[4] Frank Klawonn. Introduction to Computer Graphics: Using Java 2D and 3D (Undergraduate Topics in Computer Science) [M]. Springer, 2008:286.
[5] BioJava - Open-Source libraries for bioinformatics[EB/OL]. Available: http://biojava.org/wiki/Main_Page.
[6] Gregg A Helt, Suzanna Lewis, Ann E Loraine, et al. BioViews: Java-Based Tools for Genomic Data Visualization[EB/OL].(1998)[2015-12-15].http://genome.cshlp.org/content/8/3/291.full#abstract-1.
[7] Ivan Herman,MaylisDelest,Guy Melancon.Tree Visualisation and Navigation Clues for Information Visualisation. (1998)[2015-12-14].http://onlinelibrary.wiley.com/doi/10.1111/1467-8659.00235/pdf.
[8] Alexandru Edgar Ghitza, Philippe-Antoine Warda. (.). Growing A Suffix Tree[EB/OL]. (2015-12-19)[2016-02-20]. http://pauillac.inria.fr/~quercia/documents-info/Luminy-98/albert/JAVA+html/SuffixTreeGrow.html.
[9] Bertini E. Eurographics/ IEEE-VGTC Symposium on Visualization[C].Computer graphics forum,2009,28(3).
[10] Ukkonen E. On-line construction of suffix trees[J]. Algorithmica,1995, 14(3).
[11] Havsiyevych I. Suffix Trees: Java Applet[EB/OL].(2009)[2016-03-19].http://illya-keeplearning.blogspot.hk/2009/06/suffix-trees-java-applet-sources.html.
[12] Holland R, Down T A, Pocock M, et al. BioJava: an open-source framework for bioinformatics[J]. Bioinformatics,2008, 24(18):2096-2097.
[13] Kay M.String Alignment Using Suffix Trees[D]. Stanford University, 2000.
[14] Munzner T.INTERACTIVE VISUALIZATION OF LARGE GRAPHS AND NETWORKS[EB/OL].(2000)[2016-03-19]. https://graphics.stanford.edu/papers/munzner_thesis/all.onscree n.pdf.
[15] Weiner P.Linear pattern matching algorithm[C]. 14th Annual IEEE Symposium on Switching Automata Theory, 1973:1.
[16] Shlomo Yona. ANSI C implementation of a Suffix Tree[EB/OL].(2002)[2016-02-19].http://biit.cs.ut.ee/~vilo/edu/2002-03/Tekstialgoritmid_I/Software/Loeng5_Suffix_Trees/Suffix_Trees/cs.haifa.ac.il/shlomo/suffix_tree/.
[17] CAIDA.Walrus - Graph Visualization Tool[EB/OL].(2005)[2016-03-05].http://www.caida.org/tools/visualization/walrus/.

