青藏高原蕨麻种质资源遗传多样性POD同工酶分析
刘贺贺,蒋红霞,富 贵,白世俊,包锦渊,韦梅琴,李军乔
(1.青海民族大学 化学化工学院,西宁 810007;2.青海省生物技术与分析测试重点实验室,西宁 810007;3.青海大学 农牧学院,西宁 810016)
青藏高原蕨麻种质资源遗传多样性POD同工酶分析
刘贺贺1,2,蒋红霞1,2,富贵1,2,白世俊1,2,包锦渊1,2,韦梅琴3,李军乔1,2
(1.青海民族大学 化学化工学院,西宁810007;2.青海省生物技术与分析测试重点实验室,西宁810007;3.青海大学 农牧学院,西宁810016)
摘要采用聚丙烯酰胺凝胶电泳技术分析75份蕨麻过氧化物同工酶,结果与形态学标记、细胞水平、分子标记研究的结果一致,显示蕨麻的遗传多样性丰富。证实蕨麻具有丰富遗传变异,为合理保护与科学利用蕨麻资源提供科学依据。检测显示,蕨麻叶片过氧化物同工酶电泳共出现14条酶带;酶带多态位点百分率为100%;NTSYSpc 2.1软件计算的遗传相似系数为0.214~1.000,平均值为0.681;以遗传相似系数为基础,采用非加权类平均法进行聚类分析,在相似系数为0.67时分为2大类,第1类为蕨麻,第2类为鹅绒委陵菜。蕨麻种质资源遗传多样性及遗传变异均较丰富。说明,蕨麻丰富的遗传多样性是在青藏高原复杂的地理环境中长期进化的结果。
关键词蕨麻;遗传多样性;过氧化物同工酶;聚丙烯酰胺凝胶电泳;相似系数;多态位点百分率
蕨麻(Potentillaanserina)属蔷薇科委陵菜属(Potentilla),是鹅绒委陵菜(PotentillaanserinaL.)的变种,以叶为锯齿状奇数羽状复叶,匍匐茎,单生小花,多年生草本植物为特征,因叶片被有白色绒毛而得名,生河岸、路边、山坡草地及草甸,海拔500~4 100 m,在甘肃、青海、西藏高寒地区,根部膨大,含丰富淀粉[1]。蕨麻是药食兼用的保健品,能够提高机体免疫能力,具有抗氧化、抗衰老、抗疲劳、抗菌、抗病毒、抗真菌、抑制肿瘤、保肝护肝、降低血脂及胆固醇等功效[2]。传统形态学分类上,块根膨大的为蕨麻,块根不膨大的为鹅绒委陵菜。
种质资源是遗传育种的物质基础,对种质资源遗传多样性和亲缘关系的正确评价是合理利用种质资源的前提。遗传多样性是确保物种延续和不断进化的关键,开展蕨麻遗传多样性研究,对蕨麻品种扩大遗传基础和选育新品种均有重要作用。2010年开始,蕨麻研究中心利用形态学标记、细胞学研究和分子标记的方法研究蕨麻种质资源的遗传多样性,表明蕨麻不仅具有丰富的遗传多样性,而且蕨麻的遗传变异较高。通常情况下,植物的表现型与基因型一致,3种方法分析显示大部分的蕨麻材料同样符合这一规律,少数蕨麻材料存在不一致的情况,为证实差异的存在,笔者应用生理生化标记的方法研究蕨麻种质资源的遗传多样性。
同工酶的酶谱同等位基因之间有明确的对应关系,因此成为一种十分有效的遗传多样性的检测标记[3-4]。 同工酶分析技术从蛋白质水平反映物种的遗传差异,且材料来源丰富、实验技术简单、结果易于比较,是一种十分有效的遗传标记[5]。丁玲等[6-7]、沈镝等[8]和易刚强等[9]采用同工酶的方法分析了菊花、芋和栀子的遗传多样性,得到理想的结果。本试验采用聚丙烯酰胺凝胶电泳技术,分析75份蕨麻种质资源进行过氧化物同工酶(POD)酶谱差异研究,从蛋白质水平上对遗传多样性进行分析,以期进一步证实蕨麻种质资源的遗传多样性及复杂的遗传变异,为合理保护与科学利用蕨麻资源提供科学依据。
1材料与方法
1.1试验材料
蕨麻分布广泛,生长区域地理气候条件差别较大,很难同时采集到不同地区同一个生长期内的野生蕨麻,课题组为了规避这一问题,从2008年开始逐步采集中国各地区的蕨麻进行移栽,目前已在青海省互助县、湟源县分别建立了2个蕨麻种质资源圃,共有种质资源128份。有些地区的蕨麻种质资源刚刚成活或植株仅有1~2株,故选择有一定区域代表性且生长旺盛的75份种质资源进行同工酶测定。选取的75份蕨麻种质资源材料详见表1。
取幼苗期生长健壮的健康完整植株,剪取幼嫩叶片1.0~2.0 g,使用蒸馏水简单冲洗叶片上的泥土和其他杂物,放入冰盒内低温保存。采集当天带回实验室,放入4℃冰箱内遮光保存,3 d内完成同工酶的提取。
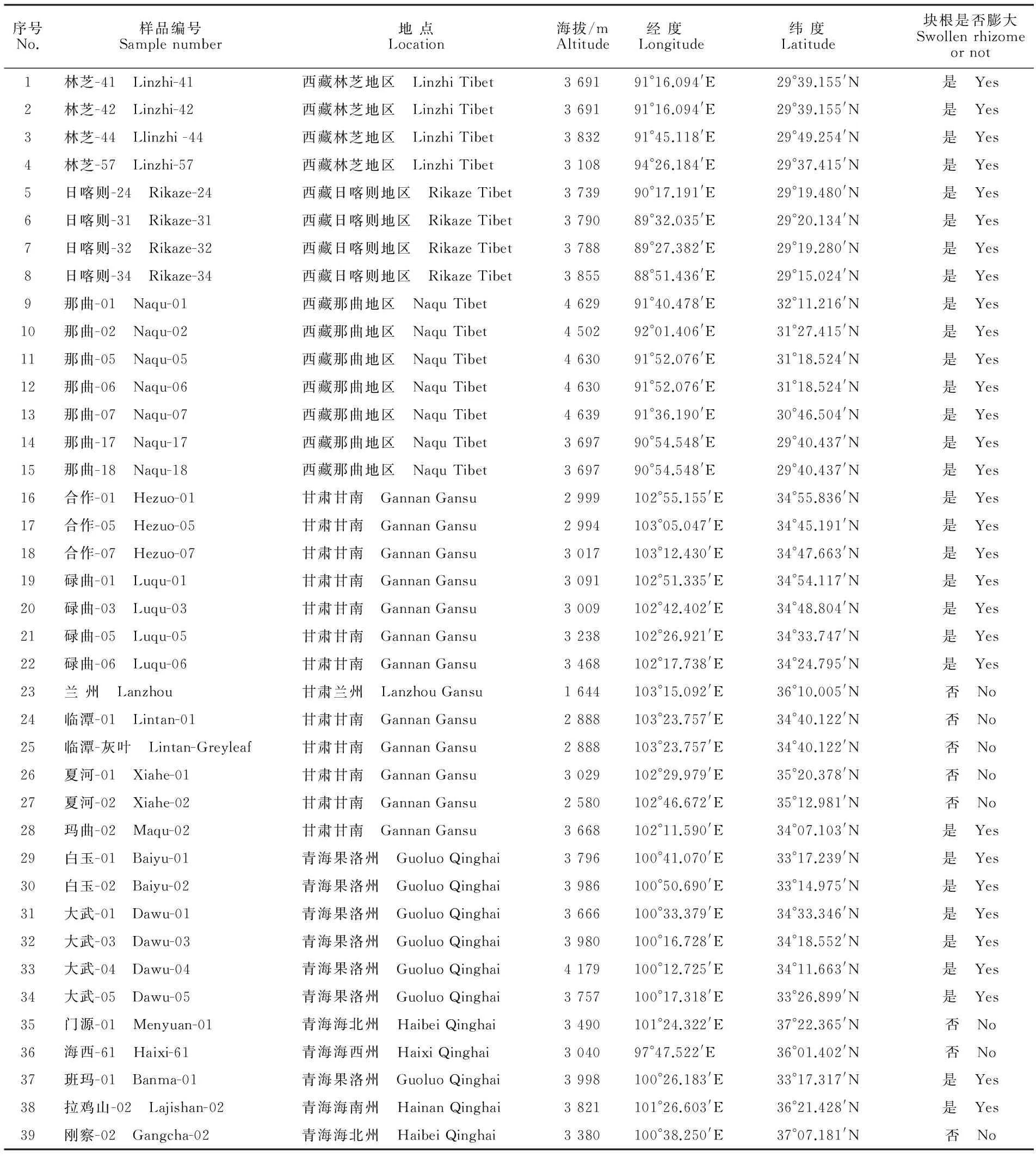
表1 供试蕨麻种质材料
(续表1Continued table 1)
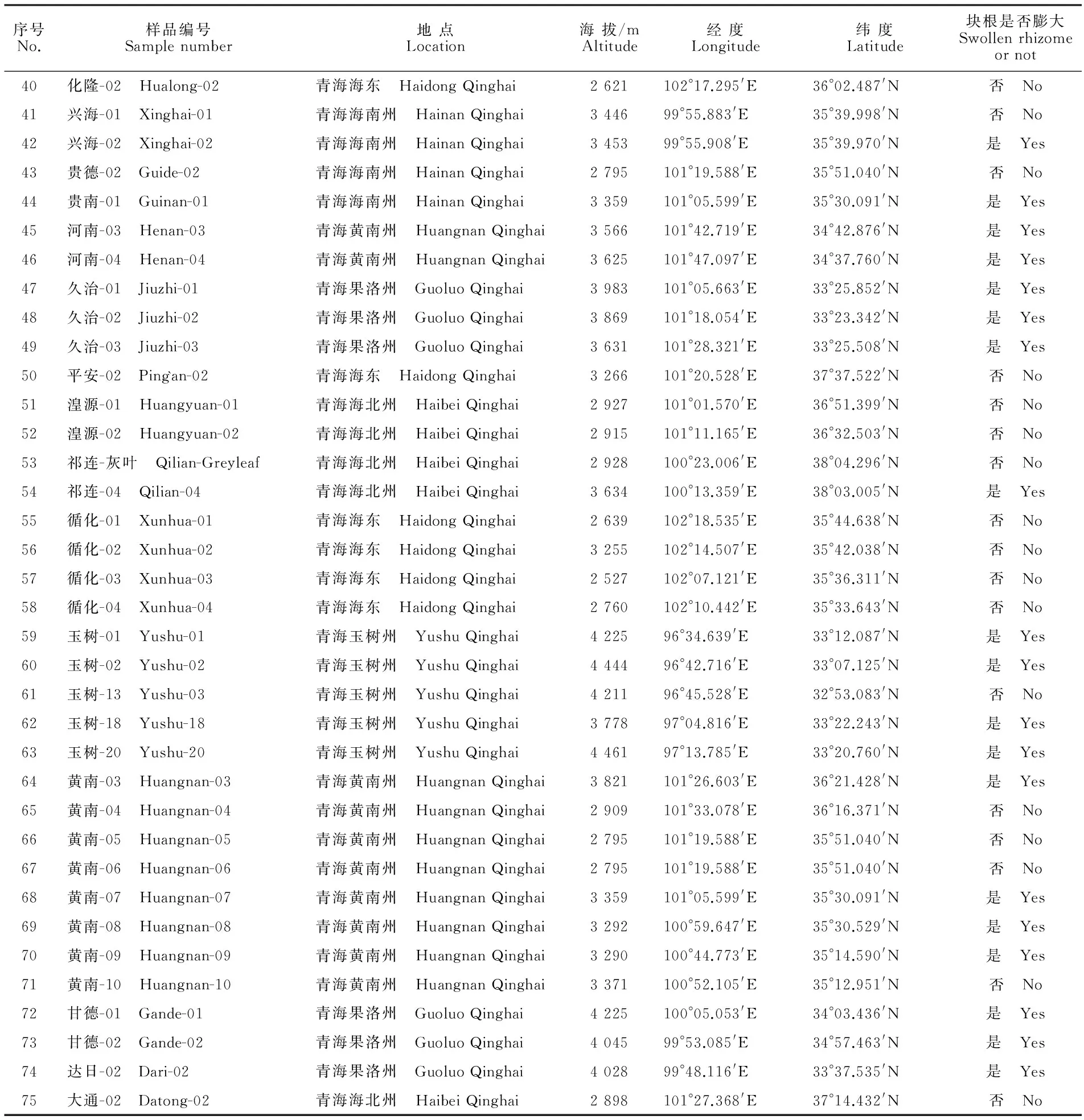
序号No.样品编号Samplenumber地点Location海拔/mAltitude经度Longitude纬度Latitude块根是否膨大Swollenrhizomeornot40化隆-02 Hualong-02青海海东 HaidongQinghai2621102°17.295'E36°02.487'N否 No41兴海-01 Xinghai-01青海海南州 HainanQinghai344699°55.883'E35°39.998'N否 No42兴海-02 Xinghai-02青海海南州 HainanQinghai345399°55.908'E35°39.970'N是 Yes43贵德-02 Guide-02青海海南州 HainanQinghai2795101°19.588'E35°51.040'N否 No44贵南-01 Guinan-01青海海南州 HainanQinghai3359101°05.599'E35°30.091'N是 Yes45河南-03 Henan-03青海黄南州 HuangnanQinghai3566101°42.719'E34°42.876'N是 Yes46河南-04 Henan-04青海黄南州 HuangnanQinghai3625101°47.097'E34°37.760'N是 Yes47久治-01 Jiuzhi-01青海果洛州 GuoluoQinghai3983101°05.663'E33°25.852'N是 Yes48久治-02 Jiuzhi-02青海果洛州 GuoluoQinghai3869101°18.054'E33°23.342'N是 Yes49久治-03 Jiuzhi-03青海果洛州 GuoluoQinghai3631101°28.321'E33°25.508'N是 Yes50平安-02 Ping`an-02青海海东 HaidongQinghai3266101°20.528'E37°37.522'N否 No51湟源-01 Huangyuan-01青海海北州 HaibeiQinghai2927101°01.570'E36°51.399'N否 No52湟源-02 Huangyuan-02青海海北州 HaibeiQinghai2915101°11.165'E36°32.503'N否 No53祁连-灰叶 Qilian-Greyleaf青海海北州 HaibeiQinghai2928100°23.006'E38°04.296'N否 No54祁连-04 Qilian-04青海海北州 HaibeiQinghai3634100°13.359'E38°03.005'N是 Yes55循化-01 Xunhua-01青海海东 HaidongQinghai2639102°18.535'E35°44.638'N否 No56循化-02 Xunhua-02青海海东 HaidongQinghai3255102°14.507'E35°42.038'N否 No57循化-03 Xunhua-03青海海东 HaidongQinghai2527102°07.121'E35°36.311'N否 No58循化-04 Xunhua-04青海海东 HaidongQinghai2760102°10.442'E35°33.643'N否 No59玉树-01 Yushu-01青海玉树州 YushuQinghai422596°34.639'E33°12.087'N是 Yes60玉树-02 Yushu-02青海玉树州 YushuQinghai444496°42.716'E33°07.125'N是 Yes61玉树-13 Yushu-03青海玉树州 YushuQinghai421196°45.528'E32°53.083'N否 No62玉树-18 Yushu-18青海玉树州 YushuQinghai377897°04.816'E33°22.243'N是 Yes63玉树-20 Yushu-20青海玉树州 YushuQinghai446197°13.785'E33°20.760'N是 Yes64黄南-03 Huangnan-03青海黄南州 HuangnanQinghai3821101°26.603'E36°21.428'N是 Yes65黄南-04 Huangnan-04青海黄南州 HuangnanQinghai2909101°33.078'E36°16.371'N否 No66黄南-05 Huangnan-05青海黄南州 HuangnanQinghai2795101°19.588'E35°51.040'N否 No67黄南-06 Huangnan-06青海黄南州 HuangnanQinghai2795101°19.588'E35°51.040'N否 No68黄南-07 Huangnan-07青海黄南州 HuangnanQinghai3359101°05.599'E35°30.091'N是 Yes69黄南-08 Huangnan-08青海黄南州 HuangnanQinghai3292100°59.647'E35°30.529'N是 Yes70黄南-09 Huangnan-09青海黄南州 HuangnanQinghai3290100°44.773'E35°14.590'N是 Yes71黄南-10 Huangnan-10青海黄南州 HuangnanQinghai3371100°52.105'E35°12.951'N否 No72甘德-01 Gande-01青海果洛州 GuoluoQinghai4225100°05.053'E34°03.436'N是 Yes73甘德-02 Gande-02青海果洛州 GuoluoQinghai404599°53.085'E34°57.463'N是 Yes74达日-02 Dari-02青海果洛州 GuoluoQinghai402899°48.116'E33°37.535'N是 Yes75大通-02 Datong-02青海海北州 HaibeiQinghai2898101°27.368'E37°14.432'N否 No
1.2试验方法
1.2.1酶液的制备称蕨麻叶片1.0 g,蒸馏水冲洗3次,滤纸吸干后剪碎。放入4 ℃预冷的研钵中,加入2 mL 0.1 mol/L 的Tris-HCl(pH 8.9)的酶液提取液,冰浴下研磨至匀浆。低温提取1 h,转移至离心管中离心,4 ℃下10 000 r/min 离心15 min。取上清液,加等体积质量分数为40%的蔗糖溶液,混匀后置4 ℃ 冰箱保存,备用。
1.2.2凝胶制备采用垂直板聚丙烯酰胺凝胶电泳,胶板厚度为1.5 mm。分离胶质量浓度70 g/L,凝胶母液[6](表2)按A∶C∶G∶水=4∶5∶8∶4.5(G储备液当天配制)的体积比混匀,混匀后从上部一侧缓缓加入凝胶,约占整个胶板的3/4,加入过程中防止气泡的产生。为保持胶面的平整,在上面加一层双蒸水。在日光灯下凝聚,1 h 后当水面与胶面出现明显界面时说明分离胶凝聚完成。用吸水纸吸去胶面上的水,加入浓缩胶,浓缩胶质量浓度为50 g/L,凝胶储备液按B∶D∶E=2∶3∶4的体积比混匀。加完浓缩胶后插上梳子,日光灯下凝聚,1 h后浓缩胶凝聚完成。
1.2.3电 泳待胶完全凝聚后,取下梳子,加样电泳。每样孔进样量20 μL,以溴酚蓝为指示剂。电泳槽内加入pH 8.3的Tris-Gly电极缓冲液约500 mL,4 ℃冰箱中恒温电泳。电泳初始电压150 V,溴酚蓝电泳至分离胶时稳压300 V。溴酚蓝电泳到分离胶底端1 cm处时,停止电泳。
1.2.4染 色POD同工酶显色参考胡能书等[11]和郭尧君[12]采用的醋酸联苯胺法,并进行改良。电泳完毕,小心剥取凝胶用蒸馏水漂洗数次,置醋酸联苯胺染液5~10 min,出现清晰过氧化物酶带后迅速倒出染液,蒸馏水洗数次,拍照、保存。染液配制:将1 g联苯胺溶于9 mL冰醋酸中,再加36 mL蒸馏水,混匀配成联苯胺溶液。使用时取5 mL联苯胺溶液,2 mL 30 g/L的H2O2,93 mL蒸馏水,混匀。
1.3数据处理
凝胶板上酶谱带颜色深浅反应同工酶活性的强弱。根据电泳图(图1),将酶活性分为强、较强、弱3个等级,绘制电泳模式图(图2)。分别计算酶带迁移率(Rf=酶带迁移距离/前沿指示剂距离)。根据电泳模式图将酶带分布情况转化为0,1二态性数值,有酶带记为“1”,无酶带记为“0”,建立POD同工酶谱带的相对迁移率和二态数据。
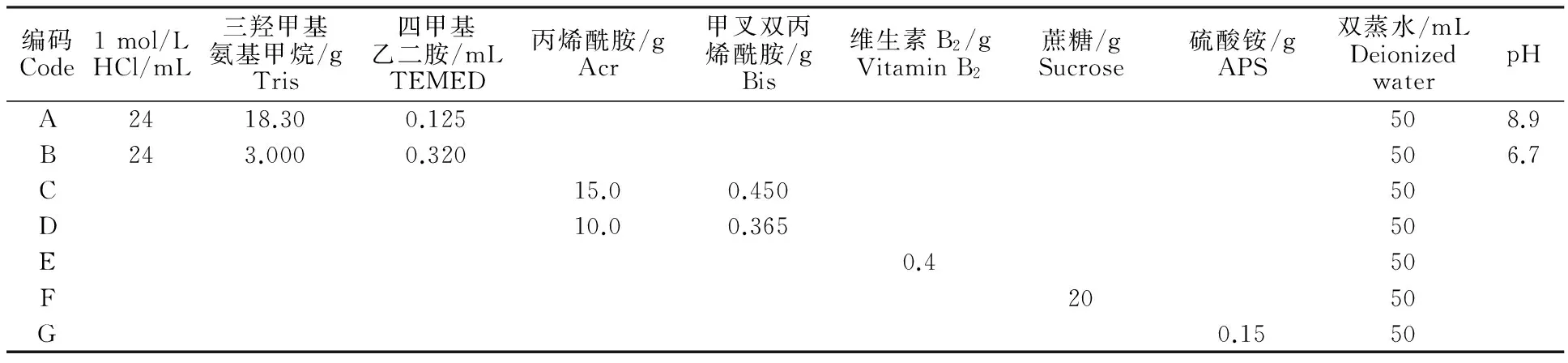
表2 凝胶储备液配方
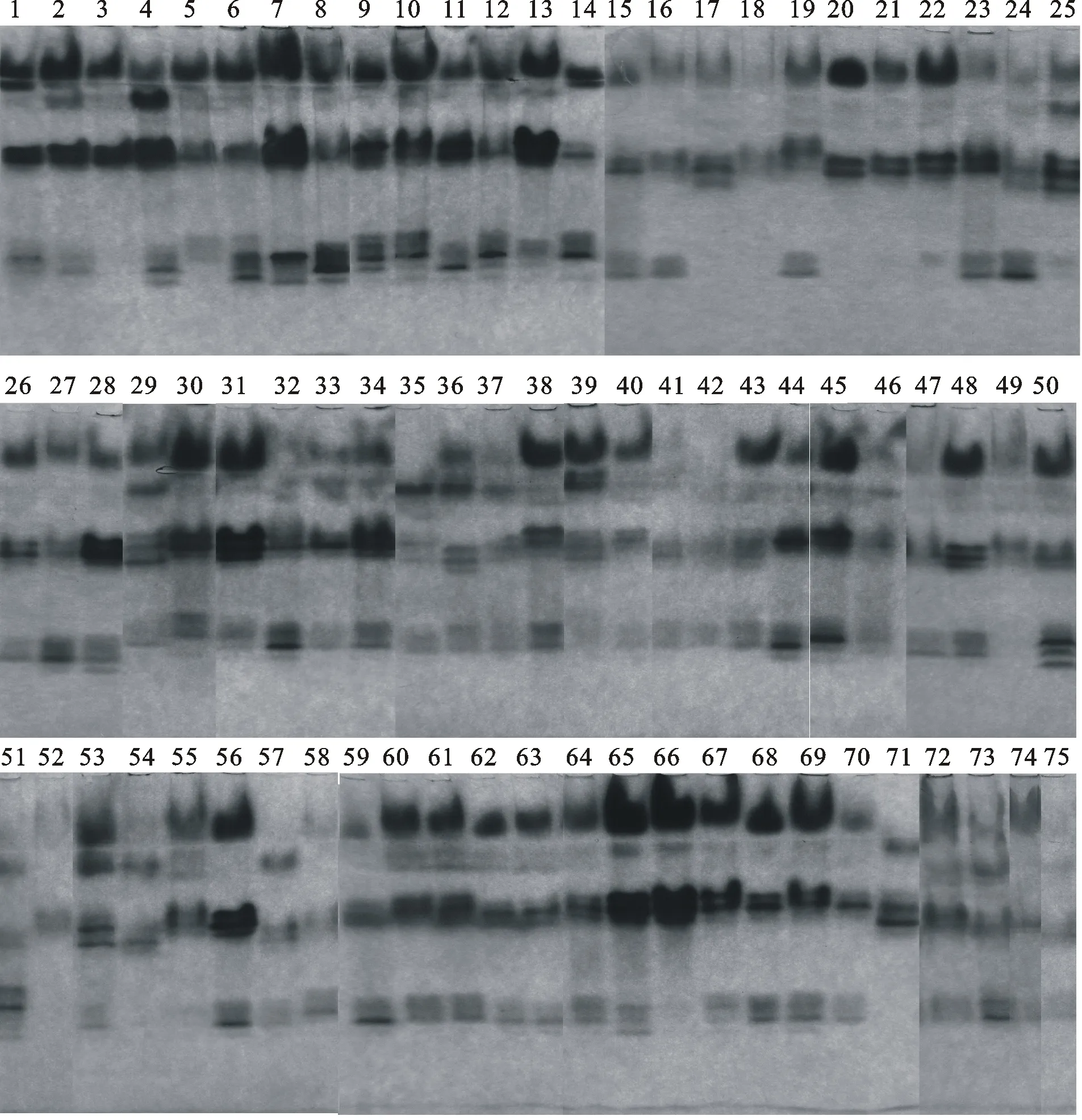
图1 75份蕨麻过氧化物同工酶酶谱

图2 蕨麻过氧化物同工酶模式
计算各酶的多态位点百分率[13](Percentage of Polymorphic Bands,PPB),PPB=NPB/TNB×100 %,式中,NPB(Number of Polymorphic Bands)为多态性条带数,TNB( Total Number of Bands)为总条带数。使用NTSYSpc 2.1计算POD酶谱带的遗传相似系数(Genetic Similarity,GS),计算公式[14]如下:GS=2a/(2a+b+c),其中,a为2个物种群共有的多态条带,b为X物种群特有条带数,c为Y物种群特有条带数。再用非加权组平均法(Unweighted Pair Group Method with Arithmetic Average,UPGMA)进行聚类分析并绘制相似系数树状聚类图。
2结果与分析
2.1POD同工酶分析
75份蕨麻种质资源叶片的POD电泳染色结果共出现14条酶带。不同蕨麻材料出现3~10条不同酶带,77%的材料含有6~9条酶带,酶带出现最多的是7号(日喀则-32)、9号(那曲-01)、10号(那曲-02)和69号(黄南-08)材料,有10条酶带;出现最少的是52号(湟源-02)和71号(甘德-01)材料,仅有3条酶带。
14条酶带分别用POD-1、POD-2、POD-3、POD-4……POD-14表示,Rf值为0.103~0.793。根据酶带的迁移率的大小,分为Ⅰ区、Ⅱ区、Ⅲ区。Ⅰ区(Rf<0.207)有4条酶带,包括POD-1~POD-4;Ⅱ区(0.328
酶带出现率为6.67%~96.00%,迁移率及出现频率见表3。酶带POD-1和POD-5的出现率低于10.00%,可作为特异性酶带,特异性的酶带是蕨麻材料的遗传特异性表现,具有较强的特异性。酶带出现率在80%以上的高频率有3条酶带,分别是POD-2(出现率为96%)、POD-7(出现率为93%)和POD-12(出现率为81%)。出现率在30%~80%的中频率酶带共6条,占条带总数的42.9%。出现率在30%以下的低频条代数为5条,占条带总数的35.7%,说明POD同工酶的多态性极高。
按照多态位点百分率公式,多态位点百分数为100%。有72份材料含有酶带POD-2,有70份材料出现酶带POD-7,75份蕨麻种质资源材料没有共有条带,14条酶带全部为多态性酶带,说明酶带POD-2和POD-7为蕨麻资源POD同工酶比较稳定遗传的酶带。多态性酶带数和百分率直接反映材料的多态性,间接反映酶谱的多样性信息含量,又可作为度量遗传变异水平高低的指标。
2.275份蕨麻种质资源的相似系数
NTSYSpc 2.1软件计算75份材料2 850对两两不同材料的相似系数,结果见表4。相似系数为0.214~1.000,平均值为0.681。
蕨麻材料7号(日喀则-32)与17号(合作-05)、71号(黄南-10)、75号(大通-02),4号(林芝-57)与54号(祁连-04),14号(那曲-17)与75(大通-02)号5组之间,相似系数最小(GS=0.214),亲缘关系最远。同一个地区的材料如1号(林芝-41)和2号(大武-04),虽同是来自自西藏林芝地区达孜县,采样点的不同,其同工酶的种类和酶带的活性也不完全一样,相似系数为0.929。66号(黄南-05)和67号(黄南-06)材料同样来自同一地点,其相似系数仅为0.786。说明,居群内存在着一定的遗传变异。
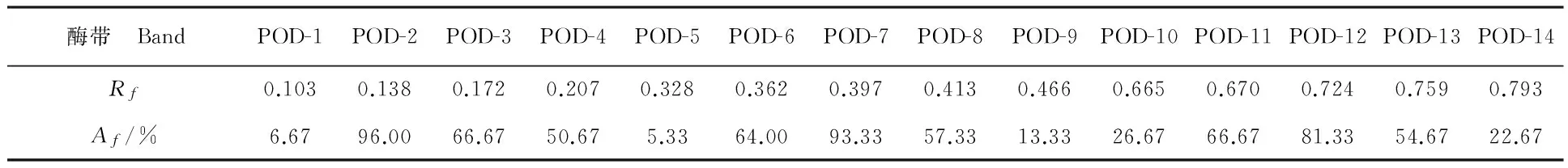
表3 蕨麻同工酶酶带迁移率(Rf)及出现频率(Af)

表4 蕨麻材料的相似系数(部分)
注:最上一行及最左侧一列的编号是样品编号。
Note:Numbers of top row and left-most column are sample number.
材料来源较近的8号(日喀则-34)与12号(那曲-06),20号(碌曲-03)、21号(碌曲-05)和49号(久治-03),41号(兴海-01)、72号(甘德-01)和73号(甘德-02),48号(久治-02)、60号(玉树-02)和61号(玉树-13),50号(平安-02)、59(玉树-01)号和62(玉树-18)号两两之间13组,地域相距较远的材料1号(林芝-41)与32号(大武-03),16号(合作-01)与44号(贵南-01),31号(大武-01)与45号(河南-03),34号(大武-05)与38号(拉鸡山-02)4组,共17组材料间的相似系数最大(GS=1.000),亲缘关系最近。可见,地域相距近和远的地区均存在变异小、亲缘关系近的蕨麻材料。
2.3蕨麻种质资源的聚类分析
使用NTSYSPC 2.1软件进行聚类分析,绘制树状图,见图3。结果表明,在相似系数为0.72时分为5类,第1类包括28份蕨麻材料,其中西藏有11份材料,甘肃有2份材料,青海有15份材料;第2类包括16份蕨麻材料,其中西藏有3份,甘肃有7份,青海有6份;第3类包括7份蕨麻材料,其中甘肃有1份,青海有6份;第4类包括14份蕨麻材料,其中西藏有1份,甘肃有3份,青海有10份;第5类包括10份蕨麻材料,全部来自青海,5份材料来自玉树,海东和黄南地区各2份材料,果洛地区1份材料;第1类与第2类在相似系数为0.70时聚为一类即第1大类,亲缘关系相对较近,44份材料中块根膨大的36份,为蕨麻。第3类与第4类在相似系数为0.67时聚为一类,并在相似系数为0.74时与第5类聚为第2大类,31份材料中块根不膨大的17份,为鹅绒委陵菜。
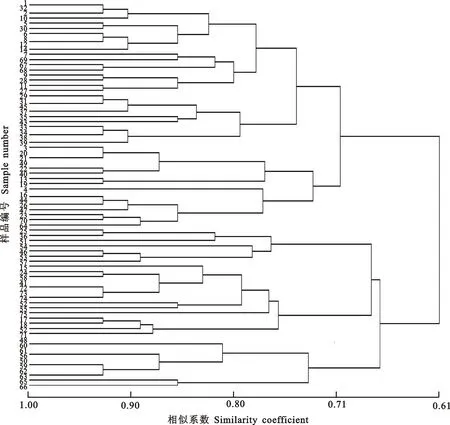
图3 75份蕨麻过氧化物同工酶聚类结果
西藏的15份蕨麻材料中有14份聚在第1大类,15号(那曲-18)聚类到第2大类。甘肃的13份材料有9份材料也聚在第1大类,其余4份材料聚类到第2大类,47份青海的蕨麻材料在第1大类中有20份,第2大类中有26份。可见,蕨麻的种质资源中遗传差异最大的是青海,其次是甘肃,最小是西藏。海拔3 000 m的18份材料,6份聚类到第1大类,12份聚类到第2大类,海拔3 000~4 000 m的材料和海拔4 000 m以上的材料也出现类似情况,且在第2大类中均有分布。
3结论与讨论
3.1蕨麻种质资源遗传多样性丰富
遗传多样性包括一个物种所有个体间遗传变异的总和,是居群生存和发展的前提[15]。遗传变异是种群长期适应环境变化的结果,遗传多样性丢失导致小种群对环境变化(如污染、气候变化)的响应能力非常有限[16]。蕨麻研究中心采用的形态学标记、细胞学研究和分子标记的方法研究表明蕨麻种质资源多样性丰富。多态性酶带数和多态位点百分率可以直接反映材料的多态性,间接反映酶谱的多样性信息含量,还可作为度量遗传变异水平高低的指标。相似系数可用来比较群体或个体间相似程度的度量参数,相似系数越高,说明相似程度越大,遗传背景相似性越强[17]。本试验使用生化标记的方法揭示蕨麻14条POD同工酶酶带全部为多态性酶带数和多态位点百分率达到最大100%,显示蕨麻种质遗传多样性丰富,与形态学标记、细胞学研究和分子标记的研究结果一致。
研究显示,75份蕨麻材料的14条POD同工酶酶带中无共同酶带,未检测到同工酶酶带及活性完全一致的材料。仅有部分材料,如1号(林芝-41)与32号(大武-03)等17组材料的相似系数为1.000,仅具有相同的酶带及酶带条数,酶带的活性并不一致,可见,蕨麻种质资源的丰富性使不同地区间的蕨麻材料存在一定差异,这与蕨麻广阔的生态适应性相一致。因而蕨麻在食用、药用及生态应用上的开发前景巨大,在研究、利用和保护野生蕨麻种质资源时,应注意不同地区和居群的代表性。
3.2蕨麻种质资源亲缘关系复杂
本试验聚类结果显示,50份蕨麻材料中仅有36份聚为蕨麻一类,剩余的14份聚到鹅绒委陵菜一类中,25份鹅绒委陵菜材料中17份聚到鹅绒委陵菜类中,此聚类结果与形态学标记和分子标记的聚类结果——不同比例的蕨麻材料聚到鹅绒委陵菜中的结果一致。在采样时发现同一地区的蕨麻性状也存在较大的差异,在甘肃临潭蕨麻原变种和灰叶蕨麻变种共同生长,在多数采样地,甚至同一株蕨麻,其块根的性状及色泽都不尽相同。表明蕨麻的遗传变异高、遗传关系复杂。蕨麻的表型是环境是基因决定还是由二者共同决定,需要进一步深入研究。
蕨麻这种遗传复杂的关系是长期在复杂的地理环境中适应的结果,使得蕨麻能在环境复杂的情况下正常生长发育繁殖。同样,人们能够获得不同品质的蕨麻资源,也为遗传育种提供了丰富变异。
目前研究表明,许多植物在冰期时存在避难所,而在最后一次大冰期后,不同的植物甚至同种植物的不同种群表现不同的进化反应(扩张或者不扩张),这些研究为植物分布区中心和边缘,种群繁殖分配策略的比较研究提供了基础[18-20]。蕨麻在青藏高原广泛分布,与其他许多植物一样在冰川时期存在避难所。本试验研究的蕨麻材料来自中国三大自然区之一的青藏高原,其自然环境的垂直变化和水平分异与低海拔区域迥然不同,具有独特的地生态现象及其空间格局。目前报道的36种高山植物的谱系地理分析显示,谱系地理模式主要表现为:一、冰期退却到高原边缘的避难所,冰后期回迁到高原面;二、地理隔离造成冰期存在多处避难所(含微型避难所),冰后期发生局域性扩张[21-22]。由蕨麻复杂的亲缘关系推测蕨麻的谱系地理模式为上述的第2种,即蕨麻在冰川时期就地寻找避难所,躲过了严酷的环境条件,并在冰川时期结束后逐渐分布青藏高原。生境变化较小的蕨麻发生较小甚至不发生变异,保持了较为原始生长类型,生境变化大的蕨麻材料发生了较大的变异。
青藏高原及其周围地区,由于特殊的环境条件使得蕨麻在青藏高原的分布一直处于野生状态,为了生存繁衍,蕨麻需要适应各种不同的微环境,环境的变化影响蕨麻同工酶基因的表达,恶劣的环境条件是导致蕨麻表现丰富的遗传多样性的重要原因。
参考文献Reference:
[1]中国科学院.中国植物志[M].北京:科学出版社,1985:283.
Chinese Academy of Sciences.Flora Reipublicae Popularis Sinicae[M].Beijing:Science Press,1985:283(in Chinese).
[2]张文娟,王庆伟,刘琳娜,等.藏药蕨麻的研究进展[J].中国药业,2010,19:1-3.
ZHANG W J,WANG Q W,LIU L N,etal,Research advances onPotentillaanserinaL[J].ChinaPharmaceuticals,2010,19:1-3(in Chinese with English abstract).
[3]葛颂.酶电泳资料和系统与进化植物学研究综述[J].武汉植物学研究,1994(1):71-84.
GE S.Electrophoretic data and studies of plant systematics and evolution [J].JournalofWuhanBotanicalResearch,1994(1):71-84(in Chinese) .
[4]王中仁.植物等位酶分析[M].北京:科学出版社,1996:29-34
WANG ZH R.Plant Allozyme Analysis[M].Beijing:Science Press,1996:29-34(in Chinese).
[5]尹春英,彭幼红,罗建勋,等.杨属遗传多样性研究进展[J].植物生态学报,2004,28(5):711-722.
YIN CH Y,PENG Y H,LUO J X,etal.Advances in research on genetic diversity in populus [J].ActaPhytoecologicaSinica,2004,28(5):711-722(in Chinese with English abstract).
[6]丁玲,陈发棣,滕年军,等.菊花品种间过氧化物酶、酯酶同工酶的遗传多样性分析[J].中国农业科学,2008(4):1142-1150.
DING L,CHEN F D,TENG N J,etal.Analysis of genetic diversity of cultivars in dendranthema grandiflorum based on POD and SOD isozyme [J].ScientiaAgriculturaSinica,2008(4):1142-1150(in Chinese with English abstract).
[7]丁玲,陈发棣,房伟民.菊属8个种27份材料遗传多样性的同工酶分析[J].西北植物学报,2007(2):249-256.
DING L,CHEN F D,FANG W M.Genetic diversity among 27 materials in 8 species of dendranthema by isozyme analysis[J].ActaBotanicaBoreali-OccidentaliaSinica,2007(2):249-256(in Chinese with English abstract).
[8]沈镝,朱德蔚,李锡香,等.云南芋种质资源遗传多样性的RAPD分析[J].植物遗传资源学报,2003(1):27-31.
SHEN D,ZHU D W,LI X X,etal.RAPD analysis of genetic diversity in taro in Yunnan province[J].JournalofPlantUeneticResources,2003(1):27-31(in Chinese with English abstract).
[9]易刚强,李云耀,崔培梧,等.栀子过氧化物酶、酯酶同工酶的遗传多样性分析[J].中南药学,2012(6):428-432.
YI G Q,LI Y Y,CUI P W,etal.Genetic diversity ofGardeniajasminoidesEllis based on POD and EST isoenzyme [J].CentralSouthPharmacy,2012(6):428-432(in Chinese with English abstract).
[10]杨海艳,罗中泽,王昆林,等.激光加电场处理水稻幼苗POD及CAT同工酶酶谱分析[J].安徽农业科学,2009(11):4892-4893,4914.
YANG H Y,LUO ZH Z,WANG K L,etal.Study on isoenzyn es patterns of ROD and CAT in rice seed lings treated by laser ligttt and electric field [J].JournalofAnhuiAgriculturalSciences,2009(11):4892-4893,4914(in Chinese with English abstract).
[11]胡能书,万贤国.同工酶技术及其应用[M].长沙:湖南科学技术出版社,1985:104-110.
HU N SH,WAN X G.Isozyme Technology and Its Application[M].Changsha:Hunan Science & Technology Press,1985:104-110(in Chinese).
[12]郭晓君.蛋白质电泳试验技术[M].北京:科学出版社,1999:95-100.
GUO X J.Protein Electrophoresis Experiment Technique[M].Beijing:Science Press,1999:95-100(in Chinese).
[13]陈立强,师尚礼,马春晖.野生及栽培苜蓿种质资源遗传多样性的同工酶分析[J].草业科学,2014(6):1070-1079.
CHEN L Q,SHI SH L,MA CH H.Genetic diversity analysis of wild and cultivated alfalfa germplasm resources by isozyme markers[J].PrataculturalScience,2014(6):1070-1079(in Chinese with English abstract).
[14]李强栋,孟林,毛培春,等.不同居群马蔺种质材料同工酶酶谱特征分析[J].草地学报,2012,20(1):116-124.
LI Q D,MENG L,MAO P CH,etal.Isozyme analysis of different chinese iris populations[J].ActaAgrestiaSinica,2012,20(1):116-124(in Chinese with English abstract).
[15]王霞,王静,蒋敬虎,等.观光木片断化居群的遗传多样性和交配系统[J].生物多样性,2012,20(6):676-684.
WANG X,WANG J,JIANG J H,etal.Genetic diversity and the mating system in a fragmented population ofTsoongiodendronodorum[J].BiodiversityScience,2012,20(6):676-684(in Chinese with English abstract).
[16]WILLI Y,VAN BUSKIRK J,HOFFMANN A A.Limits to the adaptive potential of small populations[J].AnnualReviewofEcology,Evolution,andSystematics,2006,37:433-458.
[17]NEI M.Estimation of average heterozygosity and genetic distance from a small number of individual[J].Genetic,1978,89:583-590.
[18]郑度,李炳元.青藏高原地理环境研究进展[J].地理科学,1999(4):295-302.
ZHENG D,LI B Y.Press in the study of the geographical environment of Qinghai-tibet plateau[J].ScientiaGeographicaSinica,1999(4):295-302(in Chinese with English abstract).
[19]刘志飞,王成善.新生代全球变冷与青藏高原隆升的关系[J].矿物岩石,1998(S1):137-141.
LIU ZH F,WANG CH SH.Relation between cenozolc global cooling and Qinghai-tibet plateau uplift [J].JournalofMineralogyandPetrology,1998(S1):137-141(in Chinese with English abstract).
[20]傅容珊,李力刚,黄建华,等.青藏高原隆升过程的三阶段模式[J].地球物理学报,1999(5):609-617.
FU R SH,LI L G,HUANG J H,etal.Three-step model of the Qinghai-Xizang plateau uplief[J].ChineseJournalofGeophysics,1999(5):609-617(in Chinese with English abstract).
[21]于海彬,张镱锂.青藏高原及其周边地区高山植物谱系地理学研究进展[J].西北植物学报,2013(6):1268-1278.
YU H B,ZHANG Y L.Advances in phylogeography of alpine plants in the tibetan plateau and adjacent regions[J].ActaBotanicaBoreali-OccidentaliaSinica,2013(6):1268-1278(in Chinese with English abstract).
[22]范广洲,程国栋.青藏高原隆升对西北地区降水量变化的影响[J].高原气象,2003(S1):67-74.
FAN G ZH,CHENG G D.Influence of the Qinghai Xizang plateau uplifting on precipitation change in northwest China [J].PlateauMeteorology,2003(S1):67-74(in Chinese with English abstract).
Received 2015-04-08Returned2015-06-24
Foundation itemTransformation Fund in Agricultural Sci-tech Achievement of Ministry of Science and Technology (No.2010GB2G00514); NNSF of China (No.30607026,No.30660019); Fund Project in Natural Science of Qinghai Province(No.2012-Z-907).
First authorLIU Hehe,male,master student.Research area:medicinal plant resourcesdevelopment and utilization.E-mail:516098384@qq.com
(责任编辑:潘学燕Responsible editor:PAN Xueyan)
Genetic Diversity ofPotentillaanserinain Qinghai-Tibetan Plateau of China Based on Peroxidase Isozyme
LIU Hehe1,2,JIANG Hongxia1,2,FU Gui1,2,BAI Shijun1,2,BAO Jinyuan1,2,WEI Meiqin3and LI Junqiao1,2
(1.College of Chemistry and Chemical Engineering,Qinghai University for Nationalities,Xining810007,China;2.Qinghai Provincial Biotechnology and Analytical Test Key Laboratory,Xining810007,China;3 Agriculture and Animal Husbandry College,Qinghai University,Xining810016,China)
AbstractWe used technique of polyacrylamide gel electrophoresto analyze the peroxidase isozyme in 75 Potentilla anserina. The results showed that biochemical markers were consistent with the results in morphological markers,cell level and molecular markers,and Potentilla anserinehad genetic diversity .By peroxidase isoenzyme electrophoresis,we found 14 enzyme bands of Potentilla anserina leaf;percentage of polymorphic bands was 100%;genetic similarity coefficient calculated by the software NTSYSpc 2.1 ranged from 0.214 to 1.000,and the average value was 0.681; Based on the genetic similarity coefficient,cluster analysis was done by method of unweighted pair group method with arithmetic mearns used Potentilla anserina material ,Potentilla anserina was divided into two categories when the genetic similarity was 0.67,the main one of the first category was Potentilla anserina material,the main one in the second category was Potentilla anserine material. In conclusion,rich genetic diversity and variations of Potentilla anserina was caused by evolution in the complicated geographical environment of the Qinghai Tibet Plateau in long time.
Key wordsPotentilla anserina;Genetic diversity;POD isozyme; Polyacryamide gel electrophoresis; Genetic similarity; Percentage of polymorphic bands
Corresponding authorLI Junqiao,female,Ph.D,professor.Research area:medicinal plant resources development and utilization.E-mail:ljqlily2002@126.com
中图分类号Q814.9
文献标志码A
文章编号1004-1389(2016)03-0413-10
通信作者:李军乔,女,博士,教授,研究方向为植物资源开发与利用。E-mail:ljqlily2002@126.com
基金项目:科技部农业科技成果转化基金(2010GB2G00514);国家自然科学基金(30607026,30660019);青海省自然科学基金(2012-Z-907)。
收稿日期:2015-04-08修回日期:2015-06-24
网络出版日期:2016-03-06
网络出版地址:http://www.cnki.net/kcms/detail/61.1220.S.20160306.1611.026.html
第一作者:刘贺贺,男,在读硕士,研究方向为药用植物资源开发与利用。E-mail:516098384@qq.com

