绵羊肺炎支原体贵州株P113基因的克隆与生物信息学分析
吴 燕,袁海文,王 琦,岳 筠,文 明,周碧君,程振涛
(1. 贵州大学 动物科学学院,贵阳 550025;2. 贵州省动物疫病与兽医公共卫生重点实验室,贵阳 550025;3. 贵州省动物疫病预防控制中心,贵阳 550008)
绵羊肺炎支原体贵州株P113基因的克隆与生物信息学分析
吴燕1,2,袁海文1,2,王琦1,2,岳筠3,文明1,2,周碧君1,2,程振涛1,2
(1. 贵州大学 动物科学学院,贵阳550025;2. 贵州省动物疫病与兽医公共卫生重点实验室,贵阳550025;3. 贵州省动物疫病预防控制中心,贵阳550008)
摘要为获得绵羊肺炎支原体贵州株 P113基因生物信息学特征,应用DNAStar、Mega 5.0、Protparam、Protscale、IEBD等工具对其(GZ-QX1株)P113蛋白特性、结构和功能进行预测分析。结果显示,绵羊肺炎支原体GZ-QX1株 P113基因序列大小为3 240 bp,编码1 079个氨基酸,与绵羊肺炎支原体Y98标准株、四川SC01株、猪肺炎支原体P97、丝状支原体山羊亚种、山羊支原体山羊亚种的核苷酸序列同源性分别为99.9%、81.9%、60.4%、3.9%和5.2%。P113蛋白是分子质量约119 ku的碱性蛋白,具有较多优势抗原表位;蛋白结构分析显示,P113蛋白无跨膜结构,有9个N糖基化位点,59个丝氨酸、16个苏氨酸的磷酸化位点,14种保守的特异性蛋白质激酶的结合位点;蛋白功能分析认为,P113可能是某信号传导通路的信号分子,也是一种具有良好抗原性的结构蛋白。
关键词绵羊肺炎支原体; P113基因;生物信息学分析
绵羊肺炎支原体(Mycoplasmaovipneumoniae,Mo)是引起山羊支原体肺炎的主要病原之一,也是目前影响养羊业发展的主要疫病病原[1-3]。支原体可粘附于宿主细胞表面,吸收细胞营养,通过细胞膜获得胆固醇等脂质,引起细胞损伤[4]。黏附素(Adhesin)是细菌表面的一类生物大分子,常是蛋白质或糖蛋白,研究显示,黏附素在支原体定殖过程中有关键作用[5]。黏附素亦是支原体重要的毒力因子之一,失去黏附素的支原体突变体致病作用也随之消失[6],因此,黏附素是支原体致病的重要因子,其与呼吸道黏膜纤毛的特异性黏附是引起肺炎的首要条件[7-8]。P113蛋白是Mo的一种重要黏附素和膜表面蛋白[9],是研究绵羊肺炎支原体致病性、基因工程疫苗、诊断试剂的重要靶蛋白。因此,本研究对Mo贵州分离株 P113基因进行体外扩增、克隆与测序,并对推导氨基酸序列进行生物信息学分析,以期为该蛋白体外表达可行性、蛋白应用可行性及致病性研究奠定基础。
1材料与方法
1.1材 料
1.1.1菌株Mo Y98标准株购自ATCC(29419TM),Mo贵州流行株(GZ-QX1株)由贵州省动物疫病与兽医公共卫生重点实验室分离并保存。
1.1.2主要试剂支原体基因组小量提取试剂盒购自Biomiga公司,2×TaqPCR MasterMix、200 bp DNA Marker 购自天根生化科技(北京)有限公司,pMD19-T载体购自TaKaRa公司,E.Z.N.A.TMGel Extraction Kit 胶回收、E.Z.N.A.TMPlasmid Kit 质粒提取试剂盒均购自Omega公司,DNA限制性内切酶BamHⅠ和HindⅢ均购自Thermo公司。
1.2方 法
1.2.1 P113基因克隆测序按照支原体基因组小量提取试剂盒说明书分别提取Y98标准株和Mo贵州流行株总DNA,参考GenBank登录的 SC01基因序列,设计合成 P113基因的分段引物(表1)进行PCR扩增,PCR产物用胶回收试剂盒进行目的基因回收与纯化,目的基因与pMD-19T载体连接转化宿主菌,重组质粒经PCR和双酶切鉴定,阳性重组质粒送至英潍捷基(上海)贸易有限公司测序。
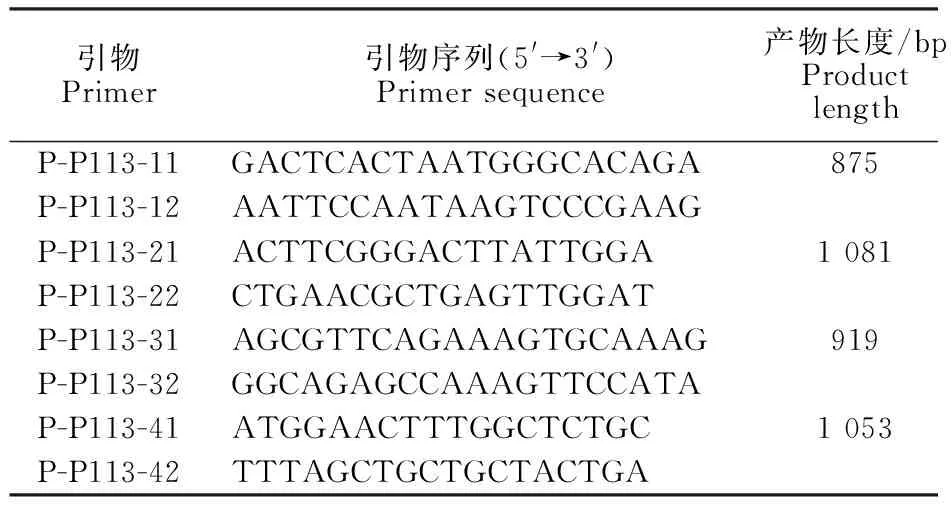
表1 试验所用引物信息
1.2.2Mo P113测序基因拼接和序列分析采用DNAStar 和Mega 5.0软件对Mo贵州流行株 P113基因序列进行氨基酸推导及进化关系分析。
1.2.3P113蛋白理化性质分析采用在线软件ExPASy(http://web.expasy.org/Compute PI/MW)和Protparam(http://web.expasy.org/protparam)分析预测编码蛋白质的理化性质及氨基酸组成。
1.2.4疏水性与亲水性的预测与分析采用在线软件Protscale(http://web.expasy.org/cgi-bin/protscale/)分析蛋白疏水性与亲水性。
1.2.5P113蛋白信号肽与跨膜区预测采用在线软件SignalP 4.0(http://www. cbs.dtu.dk/services/SignalP)和TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM/)分析蛋白信号肽以及预测蛋白质跨膜结构。
1.2.6P113蛋白修饰结构的预测 采用在线软件NetNGlyc1.0、NetPhos2.0和NetPhosk1.0(http://www.cbs.dtu.dk/services/)预测蛋白潜在N糖基化位点、潜在磷酸化位点以及保守的特异性蛋白激酶作用位点。
1.2.7P113蛋白二级和三级结构预测采用在线软件PHD sec(https://www.predictprotein.org/)和(Immune Epitope Database,IEDB)(http://www.iedb.org/)预测蛋白质的二级结构、溶剂可及性和三级结构。
1.2.8 P113蛋白质结构检验采用在线软件PDBsum Generate(http://www.ebi.ac.uk/thornton-srv/databases/pdbsum/Generate.html)检验蛋白质结构。
1.2.9P113蛋白B细胞抗原表位预测采用在线软件IEBD(http://www.iedb.org/)提供的蛋白氨基酸序列抗原表位在线预测工具进行B细胞抗原表位预测和分析。
2结果与分析
2.1Mo P113基因克隆与序列分析
对Mo贵州流行株 P113基因进行分段扩增发现,4对引物分别扩增出约875、1 081、919和1 053 bp的特异性片段(图1),与预期目的条带大小相符。应用pMD19-T载体对Mo贵州流行株 P113基因分段序列进行克隆并测序,测序序列经DNAStar 软件Seqman工具拼接后获得3 240 bp的 P113基因序列,推导氨基酸序列见图2。由图2可知,Mo GZ-QX1株 P113基因编码1 079个氨基酸,在推导氨基酸序列551位、621位、665位密码子为TGA,可作为终止密码子,但对 P113全基因推测不作为终止密码子,而是编码色氨酸(Trp)。其与Y98标准株、四川SC01株、猪肺炎支原体P97、丝状支原体山羊亚种(Mmc)和山羊支原体山羊亚种(Mccp)的相似性分别为99.9%、81.9%、60.4%、3.9%和5.2%,由进化分析结果可知,Mo GZ-QX1株与Y98标准株亲缘关系最近,与SC01株同处一个进化分支,与Mhp P97蛋白进化关系较其他种类支原体更近;而Mmc和Mcc分为一支,与Mo亲缘性较远(图3)。
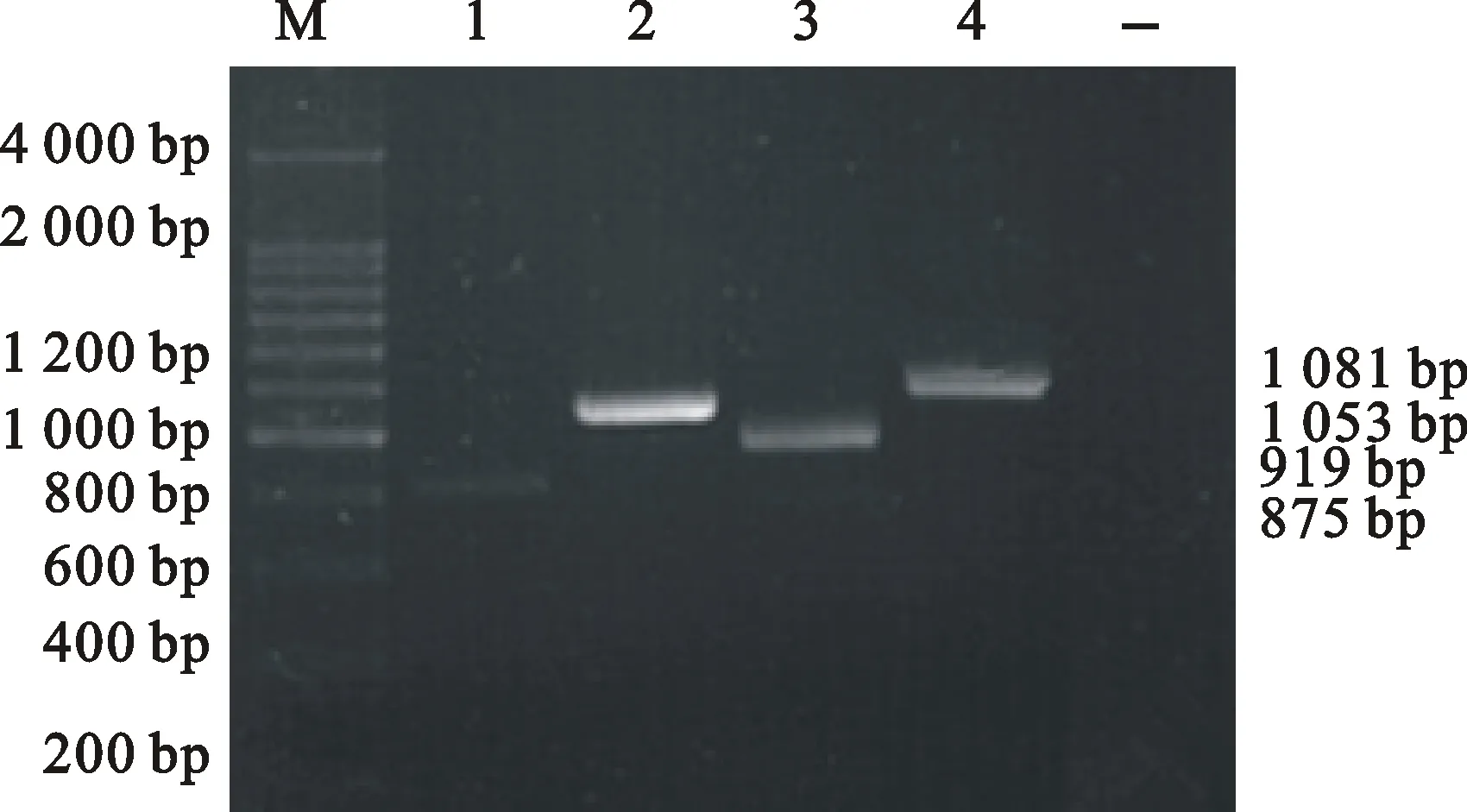
M.200 bp Marker;1~4.4段扩增产物Amplified products of four segments;-.阴性对照Negative control
图1PCR产物琼脂糖凝胶电泳
Fig.1Agarose gel electrophoresis pattern of PCR products

图2 Mo贵州株(GZ-QX1) P113基因推导的氨基酸序列
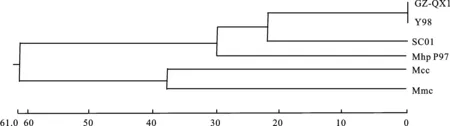
图3 各菌株基因系统进化树
2.2P113蛋白理化性质的分析
使用ExPASy(Compute PI/MW)、ProtParam 在线分析软件预测编码蛋白质的理化性质及氨基酸组成。P113蛋白分子质量为119.425 3 ku,分子结构式为C5273H8364N1430O1715S6,理论等电点(pI)为8.63。其氨基酸组成中Ser(S)和Lys(K)最多,均占到11.6%以上,不含Cys(C)。在该蛋白的氨基酸中,碱性氨基酸有3种(Arg、His、Lys),共156个,占14.5%;酸性氨基酸有2种(Asp、Glu),共144个,占13.3%;亲水性氨基酸有5种(Asn、Gly、Gln、Ser、Thr),共399个,占37.0%;疏水性氨基酸9种(Ala、Ile、Met、Phe、Pro、Trp、Tyr、Val、Leu),共383个,占35.5%,推测P113蛋白为碱性蛋白。该蛋白在水溶液中280 nm的摩尔消光系数为51 800 mol/(L·cm),蛋白质量浓度为1 g/L时的吸光系数(Abs)为0.434;该蛋白在哺乳动物网状细胞、酵母和埃希氏大肠杆菌中表达的半衰期分别大于 30、20和10 h,在溶液中的不稳定指数为28.18,低于阈值40,在溶液中性质稳定。
2.3疏水性与亲水性的预测与分析
利用Protscale在线预测Mo GZ-QX1株P113氨基酸序列的亲疏水性(图4)。图中纵坐标大于0的为疏水区,得分越高疏水性越强;反之,小于0的为亲水区。 P113基因编码的蛋白多肽链第491位具有最低得分,为-2.7(最强的亲水性),在第23位具有最高得分,为2.378(最强的疏水性)。由图4可知,除局部为疏水区域外,大部分均为亲水区域,因而整个多肽链表现为亲水性。此结果与ProtParam预测结果一致,同时较好的亲水性也使其可溶性随之增加。
2.4P113蛋白信号肽与跨膜区预测
利用在线软件SignalP 4.0和TMHMM 2.0分析此蛋白信号肽以及预测蛋白质跨膜结构。由图5可知,Mo GZ-QX1株 P113基因所编码氨基酸均不具有分泌信号肽的特征,因此进行蛋白原核表达前不需要对该蛋白基因进行修饰;由图6可知,该蛋白仅有1个较为明显的跨膜螺旋结构,其序列为17~33,处于基因序列开始,可通过部分基因表达消除其对P113蛋白体外表达效果的影响。
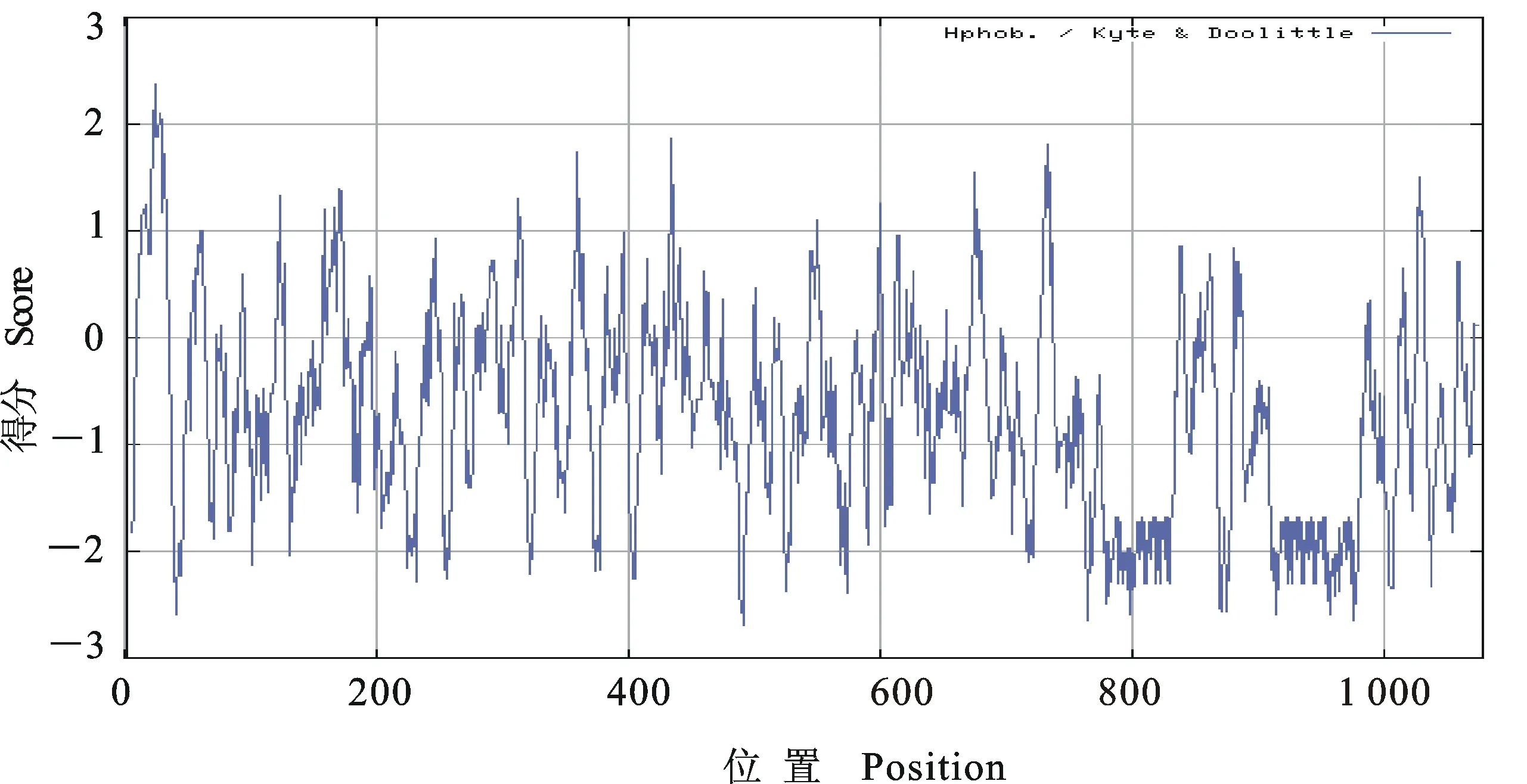
图4 Protscale程序分析Mo GZ-QX1株P113的疏水性
图5 Mo GZ-QX1株P113信号肽分析
2.5P113蛋白修饰结构的预测
利用在线软件NetNGlyc1.0、NetOGlyc3.1、NetPhos2.0和NetPhosk1.0分析预测Mo P113蛋白潜在N糖基化位点、潜在磷酸化位点以及保守的特异性蛋白激酶作用位点。由图7-A可知,P113蛋白第2、59、173、207、294、329、336、341和406个氨基酸处共存在9个N糖基化位点;由图7-B可知,P113蛋白有59个丝氨酸、16个苏氨酸可能被磷酸化。上述磷酸化位点所对应的磷酸激酶预测结果显示,在P113蛋白上可能有PKC、PKA、cdc2、CKⅡ、DNAPK、PKG、ATM、p38MAPK、CKⅠ、INSR、SRC、RSK、EGFR和cdk5共14种保守的特异性蛋白质激酶的结合位点(表2),其中,在604处PKC得分最高,为0.84。因此推测,P113蛋白功能的发挥可能与激酶磷酸化有关,说明P113蛋白在翻译后发生相关修饰,从而对其生物学功能进行调控。
图7 Mo GZ-QX1株P113蛋白修饰结构预测
项目ItemPKCPKAcdc2CKⅡDNAPKPKGATMp38MAPKCKⅠINSRSRCRSKEGFRcdk541085212322497109109253372275253906962338698718957209063053723253943384252752293494407976094403941551050508568574325305581453711326位点5976046185608956851017Position6336897026098997131019898902906694900715104010191023105510427711043105610661050103810441069
2.6P113蛋白二级和三级结构预测
利用在线软件PHD sec和SWISSMODEL预测蛋白质的二级结构、溶剂可及性和三级结构(图8和图9)。结果显示,Sec预测组成P113蛋白多肽链二级结构的3种类型分别是α-螺旋(H)(图9-A)、β-折叠(S)(图9-B)和无规则卷曲(L),比例分别是15.57%、14.18%和70.25%,以无规则卷曲为主,β-折叠较少;而其溶剂可及性[10-11]分别是暴露70.99%、埋藏23.54%以及中间5.47%,因此,P113氨基酸残基主要是暴露的,也证明其具有亲水性。分析P113蛋白的三级结构可知,其主要以无规则卷曲和转角散在存在,充分与周围的极性环境相接触,为抗原表位的形成提供有利条件(图9)。
2.7P113蛋白质结构检验
利用PDBsum Generate在线软件进行蛋白质结构检验(图10)。根据构象的稳定性,Ramachandran图预测结果显示红色、棕色2个区的比例分别为93.9%和6.1%,其余的都为0%(图10-A)。蛋白结构G-factor得分总平均值为0.33(图10-B),说明该结构在正常范围内。其二级结构和拓扑结构信息 ( 图10-C)、以及蛋白质结合位点三维图和预测蛋白结构表明,其表面可能存在催化口袋及结合位点( 图 10-D),体积最大前4位分别为980.44、946.27、946.89和635.34,对应的R比值依次是 1.04、0.00、0.00和0.00,可接近顶点依次为 60.22、63.35、60.52和67.37,深埋顶点依次为8.55、8.95、7.45和11.26。
图8 Mo GZ-QX1株P113二级结构与溶剂可及性预测结果
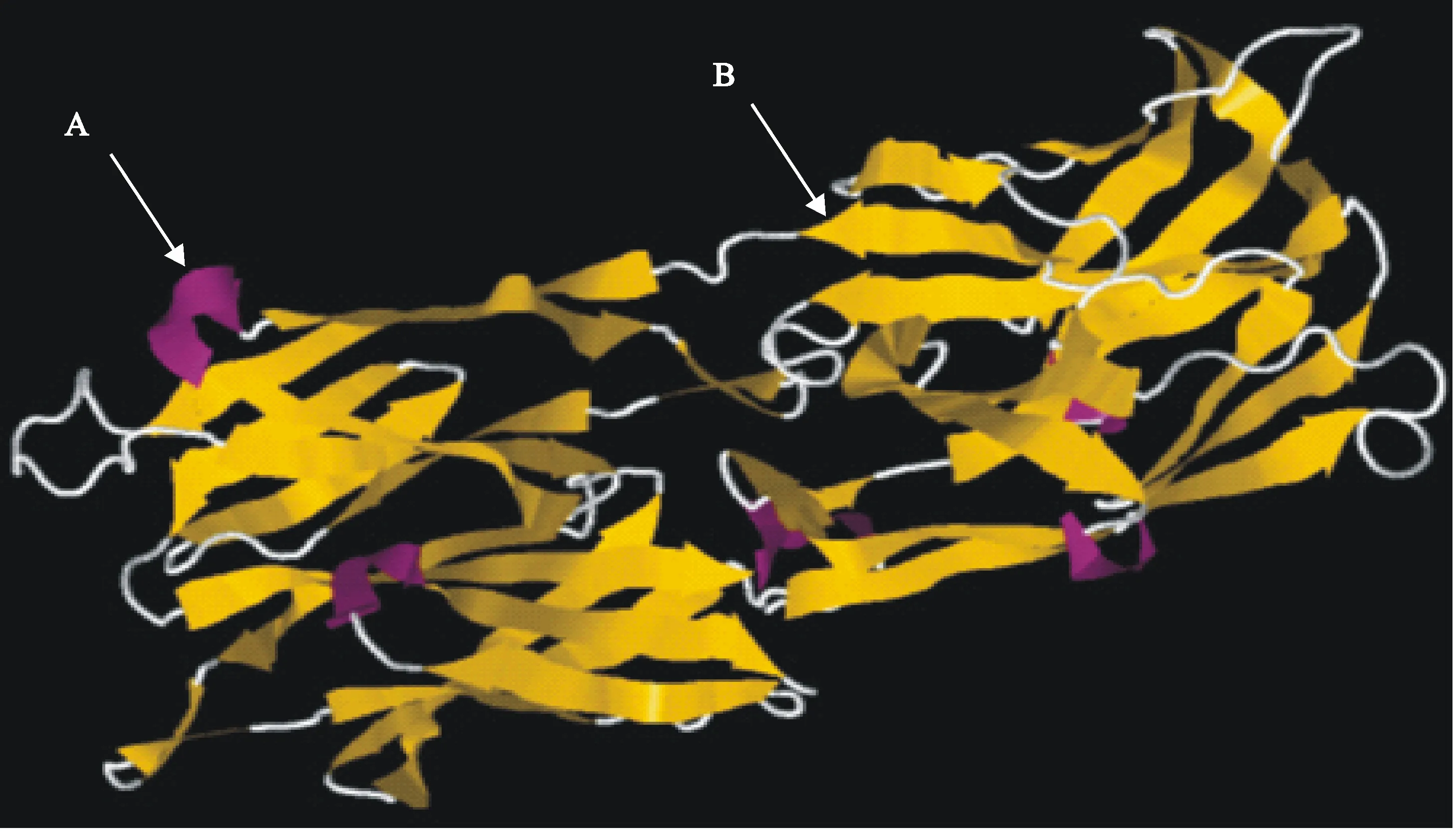
A.α-螺旋 α-Helix;B.β-折叠 β-Strand
2.8P113蛋白B细胞抗原表位预测
采用Emini、Kolaskar-Tongaonkar、Parker、Karplus-Schulz以及Bepipred方法分别对P113蛋白的表面可及性、抗原指数、亲水性、柔韧性和线性表位进行预测(图11)。表面可及性程度(默认阈值为1.0)预测结果表明,P113蛋白共有19个表面可及性程度高的肽段,其中有7个肽段的表面可及性程度最高(图11-A);抗原性(默认阈值为1.0)预测结果显示,共获得20个具有抗原性的肽段,且抗原性的强弱不同,其中有5个肽段的抗原性最强(图11-B);柔韧性(默认阈值为1.0)和亲水性(默认阈值为1.0)等预测结果显示,高于阈值的肽段部分可能参与P113蛋白线性B细胞表位的功能体现(图11-C和图11-D);预测的线性B细胞表位(默认阈值为0.35)结果显示,共获得21条线性B细胞表位,其中2、5、10号和19号肽段的氨基酸均小于7个,不符合B细胞表位的要求。综合上述预测结果,最终确定P113蛋白的B细胞表位(图11-E)。说明,Mo GZ-QX1株P113蛋白含有较多的优势抗原表位结构,预测其具有较好的抗原性,体外表达产物可应用于诊断、检测方法建立,也可作为免疫防控靶标蛋白使用。
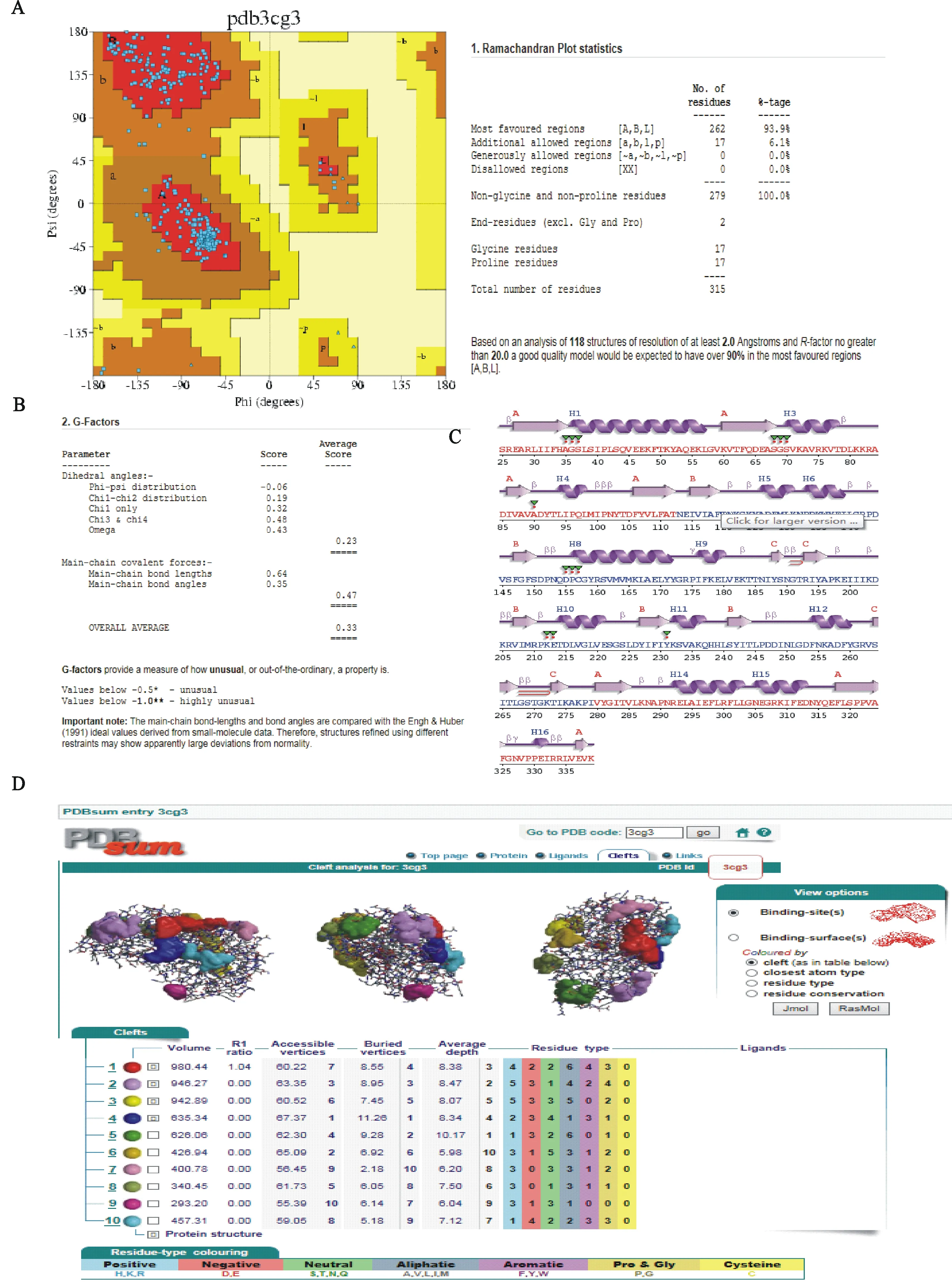
A. Procheck预测蛋白Ramachandran图及检验数据The protein Ramachandran diagram and structural test data of Ramachandran prediction;B. Procheck结构检验G-factor数据The protein structural test G-factor data of Ramachandran prediction;C. PDBsum Generate预测模型蛋白质结构信息PDBsum Generate information on protein structure prediction models;D. PDBsum Generate预测蛋白表面可能存在的催化口袋及结合位点Prediction of the protein surface binding sites and the catalytic pocket with PDBsum Generate
图10Mo GZ-QX1株P113蛋白质结构检验
Fig.10Verification of Mo GZ-QX1 P113 protein structure
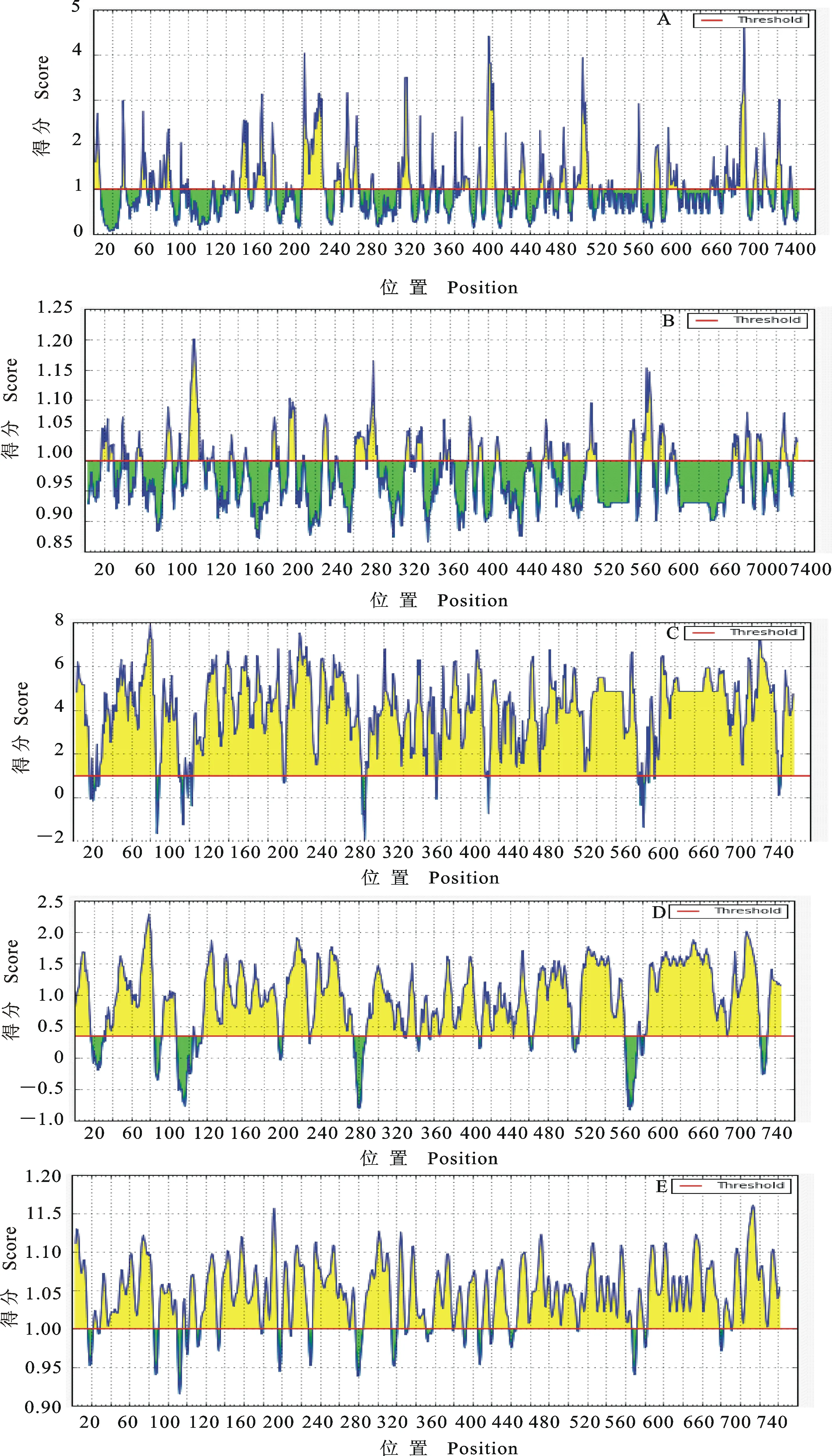
A. Emini表面可及性预测图Emini surface accessibility prediction; B. Kolaskar-Tongaonkar抗原指数预测图Kolaskar & Tongaonkar antigenicity prediction; C. Parker亲水性预测图Parker hydrophilicity prediction; D. Karplus-Schulz柔韧性预测图Karplus & Schulz flexibility prediction;E. B细胞线性表位肽预测 Bepipred linear epitope prediction
图11P113蛋白B细胞抗原表位预测结果
Fig.11Bepipred cell antigen epitope prediction results of P113 protein
3讨 论
生物信息学(Bioinformatics)是在生命科学研究中以计算机为工具,对生物信息进行储存、检索和分析的科学,其目的在于认识生命的起源、进化、遗传和发育的本质,破译隐藏在DNA序列中的遗传信息,为疾病的诊断、预防和治疗提供最合理的和有效的方法或途径[12]。P113蛋白序列生物信息学分析结果显示,P113蛋白是分子质量约为119 ku的强亲水性碱性蛋白;该蛋白在哺乳动物、酵母细胞和埃希氏大肠杆菌中有较长的半衰期,且蛋白质特性稳定,因此可在原核和真核细胞中进行重组表达[13-14]。螺旋和折叠化学键能的高低是能否牢固维持蛋白高级结构的条件之一,化学键能越高越牢固,因此,二级结构是蛋白功能分析的重要指标之一[15]。P113蛋白二级结构预测表明,该蛋白包含螺旋、折叠和无规卷曲,且以无规卷曲为主。蛋白质结构检验是采用ExPASy提供的同源建模、线串法和从头算法3种生物信息学方法及工具对P113蛋白质结构数据进行初步模拟[16],预测结果显示,P113蛋白表面可能存在催化口袋及结合位点,其对DNA与蛋白质间的相互作用在基因调节方面起着重要作用。
蛋白表达是指用模式生物,如细菌、酵母、动物细胞或者植物细胞表达外源基因蛋白的一种分子生物学技术,在基因工程技术中占有核心地位[17]。本研究根据GenBank公布的四川SC01株绵羊肺炎支原体 P113基因序列,设计特异性引物,从GZ-QX1贵州株和Y98标准株中克隆该基因,经酶切试验及DNA测序验证克隆成功。分析GZ-QX1贵州株 P113基因编码的氨基酸序列可知,该基因全长3 240 bp,编码1 079个氨基酸,且在3个氨基酸处表现与其他支原体相同的编码特性,TGA编码色氨酸且不作为终止密码子;然而目前的生物技术尚不能直接对该基因进行原核表达,需通过PCR方法进行该基因的定点突变,使TGA突变为TGG后再进行表达,或选择避开这几个位点且抗原性较好的肽段进行表达[18-19]。在对Mo GZ-QX1株 P113基因推导的氨基酸序列进行跨膜结构预测发现,P113蛋白是一种具有1个跨膜结构的膜蛋白,在进行表达时应避开其所处位置,减少其对表达效果的影响。
磷酸化、去磷酸化是信号传导通路中最简便快捷的方式,其磷酸基团的增加或减少对很多生物反应起着决定性作用,通常表现为磷酸化激活和去磷酸化失活[20]。NetPhos2.0 预测发现,P113蛋白有59个丝氨酸、16个苏氨酸以及8个酪氨酸可能作为蛋白激酶磷酸化潜在作用位点而参与细胞的信号传导;其可能有14种保守的特异性蛋白质激酶的结合位点,其中在604位点处PKC得分最高,为0.84,说明P113蛋白可能是某信号传导通路中的信号分子,但仍需进一步试验研究和验证。
抗原表位是抗原分子中决定抗原特异性的特殊化学基团,而B细胞表位是可被B细胞表面受体或抗体特异性识别并相互结合的片段[21]。有研究显示,可通过氨基酸性质(表面可及性、亲水性、柔韧性以及抗原指数)和隐形马尔可夫模型预测线性表位[22]。抗原的柔韧性高说明其易于结合B细胞受体;表面可及性的大小代表抗原中的B细胞表位与B细胞受体识别并接近的难易程度且呈正相关;抗原亲水性分子的大小决定抗原B细胞表位的多少[23]。本研究应用生物信息学预测方法对结构蛋白的潜在B细胞抗原表位进行预测和分析,根据不同参数综合评价结构蛋白可能存在的抗原优势表位,预测结果显示,P113蛋白中共有19个表面可及性程度高的肽段,同时获得20个具有抗原性的肽段和21条线性B细胞表位,表明Mo GZ-QX1株P113蛋白含有较多的优势抗原表位结构,可作为一种免疫原性蛋白初步应用于免疫学相关研究。通过DNAStar软件对Mo GZ-QX1 P113蛋白序列与Y98标准株、四川SC01株及猪肺炎支原体P97、丝状支原体山羊亚种(Mmc)和山羊支原体山羊亚种(Mccp)的相似性进行比对,发现其相似性分别达到99.9%、81.9%、60.4%、3.9%和5.2%;同时通过对推导的氨基酸序列构建进化树,发现Mo P113蛋白与Mhp p97蛋白存在交叉抗原,与丝状支原体山羊亚种和山羊支原体山羊亚种的交叉抗原相关系数较低,也证实目前市场商业化羊传染性胸膜肺炎的疫苗不能防控Mo的感染与流行,需尽快制备有效防控Mo的疫苗[24-25]。本研究的分析结果将为Mo P113基因工程疫苗和体外表达的应用研究提供依据。
参考文献Reference:
[1]LONGLEY E O.A study of the disease in Nigeria [J].VetSciAnimalHusbandry,1951,7:4-27.
[2]BALIKCI E,KIZIL O,KARAPINAR T,etal.Efficacy of marbofloxacin for naturally occurring contagious caprine pleuropneumonia [J].Small Ruminant Research,2008,77(1):75-79.
[3]刘文青,胡明丽,孙红岩,等.绵羊肺炎支原体P30-HSP70C融合蛋白的表达及免疫原性研究[J].中国预防兽医学报,2014,36(11):894-897.
LIU W Q,HU M L,SUN H Y,etal.The prokaryotic expression and immunogenicity of P30-HSP70C fusion protein ofMycoplasmaovipneumoniae[J].ChineseJournalofPreventiveVeterinaryMedicine,2014,36(11):894-897(in Chinese with English abstract).
[4]NAKANE D,ADAN-KUBO J,KENR T,etal.Isolation and characterization of P1 adhesin,a leg protein of the gliding bacteriumMycoplasmapneumoniae[J].JournalofBacteriology,2011,193(3):715-722.
[5]DEBEY M C,ROSS R F.Ciliostasis and loss of cilia induced byMycoplasmahyopneumoniaein porcine tracheal organ cultures[J].InfectionandImmunity,1994,62(12):5312-5318.
[6]KRAUSE D C,LEITH D K,BASEMAN J B.Reacquisition of specific proteins confers virulence inMycoplasmapneumoniae[J].InfectionandImmunity,1983,39(2):830-836.
[7]SIMIONATTO S,MARCHIORO S B,GALLI V,etal.Immunological characterization ofMycoplasmahyopneumoniaerecombinant proteins[J].ComparativeImmunology,MicrobiologyandInfectiousDiseases,2012,35(2):209-216.
[8]张贤宇,杨发龙,冯旭飞,等.绵羊肺炎支原体P113蛋白N端原核表达及其抗原性分析[J].中国兽医杂志,2013,49(7):7-10.
ZHANG X Y,YANG F L,FENG X F,etal.Prokaryotic expression and antigenicity analysis of N-terminal ofMycoplasmaovipneumoniaeP113 protein [J].ChineseJournalofVeterinaryMedicine,2013,49(7):7-10(in Chinese with English abstract).
[9]杨发龙,张焕容,汤承,等.绵羊肺炎支原体 P113基因的序列分析及功能预测[J].华南农业大学学报,2013,34(1):117-121.
YANG F L,ZHANG H R,TANG CH,etal.Sequence analysis and functional prediction ofMycoplasmaovipneumoniaeP113 gene[J].JournalofSouthChinaAgriculturalUniversity,2013,34(1):117-121(in Chinese with English abstract).
[10]ROST B,SANDER C.Conservation and prediction of solvent accessibility in protein families[J].Proteins,1994,20(3):216-226.
[11]MESHKIN A,GHAFURI H.Prediction of relative solvent accessibility by support vector regression and best-first method [J].ExcliJournal,2010,9:29-38.
[12]黄科,曹家树,吴秋云,等.生物信息学[J].情报学报,2002,4(21):491-496.
HUANG K,CAO J SH,WU Q Y,etal.Development of bioinformatics[J].JournaloftheChinaSocietyforScientificandTechnicalInformation,2002 ,4(21):491-496(in Chinese with English abstract).
[13]MARCHLER-BAUER A,ANDERSON J B,CHITSAZ F,etal.CDD:specific functional annotation with the conserved domain database[J].NucleicAcidsResearch,2009,37(suppl 1):205-210.
[14]MARCHLER-BAUER A,LU S,ANDERSON J B,etal.CDD:a Conserved Domain Database for the functional annotation of proteins [J].NucleicAcidsResearch,2011,39(suppl 1):225-229.
[15]唐嫒,李春花,张瑗,等.蛋白质的二级结构预测研究进展[J].现代生物医学进展,2013,13(26):5180-5182.
TANG Y,LI CH H,ZHANG Y,etal.Advanced studies on protein secondary structure prediction[J].ProgressinModernBiomedicine,2013,13(26):5180-5182(in Chinese with English abstract).
[16]薛庆中.DNA和蛋白质序列数据分析工具[M].第3版.北京:科学出版社,2012:71-116.
XU Q ZH.Tools for Analysis of DNA and Protein Sequence Data[M].3rd Edition. Beijing:Science Press,2012:71-116(in Chinese).
[17]HUANG C J,LIN H,YANG X.Industrial production of recombinant therapeutics inEscherichiacoliand its recent advancements[J].JournalofIndustrialMicrobiology&Biotechnology,2012,39(3):383-399.
[18]刘劼,吴移谋.支原体基因组学研究进展[J].中国人畜共患病学报,2006,22(11):1073-1077.
LIU J,WU Y M.Research progress ofMycoplasmagenomics[J].ChineseJournalofZoonoses,2006,22(11):1073-1077(in Chinese with English abstract).
[19]张玲,郝永清,徐春光,等.丝状支原体山羊亚种脂蛋白LppA基因的定点突变及原核表达[J].畜牧与兽医,2013,45(10):74-76.
ZHANG L,HAO Y Q,XU CH G,etal.Site-directed mutations and prokaryotic expression ofMycoplasmamycoidesLppAlipoprotein gene[J].AnimalHusbandry&VeterinaryMedicine,2013,45(10):74-76(in Chinese).
[20]张春香,郭云雁,白元生,等.绵阳 GPx5基因克隆、生物信息学分析及表达特性的研究[J].畜牧兽医学报,2014,45(7):1075-1083.
ZHANG CH X,GUO Y Y,BAI Y SH,etal.Clone,bioinformatics analysis and expression characteristics of ovine GPx5[J].ChineseJournalofAnimalandVeterinarySciences,2014,45(7):1075-1083(in Chinese with English abstract).
[21]SUN P P,JU H X,LIU ZH B,etal.Bioinformatics resources and tools for conformational B-cell epitope prediction[J].ComputationalandMathematicalMethodsinMedicine,2013,2013:943636.
[22]BAIROCH A,APWEILER R,WU C H,etal.The universal protein resource (UniProt)[J].NucleicAcidsResearch,2005,33(Suppl 1):154-159.
[23]刘争春,王颖,王树军.人卵巢癌相关抗原OVA66的B细胞表位预测[J].实用医学杂志,2009,25(4):513-516.
LIU ZH CH,WANG Y,WANG SH J.Prediction of B-cell epitopes on human ovarian cancer-associated antigen OVA66[J].TheJournalofPracticalMedicine,2009,25(4):513-516(in Chinese with English abstract).
[24]赵东,马春骥,李敏,等.绵羊肺炎支原体与猪肺炎支原体交叉抗原研究[J].黑龙江畜牧兽医,2011(15):155-156.
ZHAO D,MA CH J,LI M,etal.The antigens study ofMycoplasmaovipneumoniaeandMycoplasmapneumoniae[J].HeilongjiangAnimalScienceandVeterinaryMedicine,2011(15):155-156(in Chinese).
[25]张双翔,程振涛,唐宇,等.绵羊肺炎支原体热休克蛋白Hsp70的生物信息学分析[J].中国畜牧兽医,2014,41(7):74-80.
ZHANG SH X,CHENG ZH T,TANG Y,etal.Bioinformatics analysis of Hsp70 gene ofMycoplasmaovipneumoniae[J].ChinaAnimalHusbandry&VeterinaryMedicine,2014,41(7):74-80(in Chinese with English abstract).
Received 2015-09-09Returned2015-10-22
Foundation itemThe Scientific and Technological Cooperation Projects with Development of Grass to Keep Sheep Industry in Qianxinan State(No. The Scientific and Technological Cooperation in Qianxinan[2012]5); Science Foundation of Guizhou Province (No. The Scientific and Technological Cooperation Section J of Guizhou [2015]2082); Agricultural Research Project in Guizhou Province (No. The Scientific and Technological Cooperation of Guizhou NY[2014]3042); Science and Technology Innovation Team Project in Guizhou Province (No. The Scientific and Technological Cooperation Team of Guizhou [2015]4016); Graduate Student Innovation Fund Project in Guizhou University (No. School Fund Agriculture,2015029).
First author WU Yan,female,master student. Research area:animal disease etiology. E-mail: 491537385@qq.com
(责任编辑:郭柏寿Responsible editor:GUO Baishou)
Clone and Bioinformatics Analysis of P113 Gene ofMycoplasmaovipneumoniaein Guizhou Province
WU Yan1,2,YUAN Haiwen1,2,WANG Qi1,2,YUE Jun3,WEN Ming1,2,ZHOU Bijun1,2and CHENG Zhentao1,2
(1. College of Animal Science,Guizhou University,Guiyang550025,China; 2.Key Laboratory of Animal Disease and Veterinary Public Health of Guizhou Province,Guiyang550025,China; 3. Animal Disease Prevention and Control Center in Guizhou Province,Guiyang550008,China)
AbstractThe assay was aimed to access the biological characteristics of P113 protein in Mycoplasma ovipneumoniae(Mo) Guizhou strain (GZ-QX1). The protein structure,characteristics and function of GZ-QX P113 protein were predicted and analyzed by DNASTAR,MEGA 5.0 softwares,Protparam,Protscale and IEBD online tools in this study. The results showed that Mo GZ-QX1 P113 gene predicted is 3 240 bp,encoding 1 079 amino acid(AA),and sharing nucleotides sequence identity 99.9% with Mo Y98 standard strains 81.9% with SC01 Sichuan strain,60.4% with Mycoplasma hyopneumoniae P97,3.9% with Mycoplasma mycoides and 5.2% with Mycoplasma capripneumoniae. The molecular weight of P113 protein is 119 ku,with more advantages antigen epitope protein structure. Protein structure analysis showed that the protein had no transmembrane domain. There were 9 N-glycosylation sites,59 serine,16 threonine may be phosphorylated predicted and 14 conserved and specific phosphkinase binding sites. Protein functional analysis showed that P113 may function as a mediator in signaling pathway,but also a kind of good antigenic structure of proteins.
Key wordsMycoplasma ovipneumoniae; P113 gene; Bioinformatics analysis
Corresponding authorCHENG Zhentao,male,associate professor,master supervisor.Research area:animal studies on molecular etiology.E-mail: chengzhentao@sohu.com
中图分类号S852.62
文献标志码A
文章编号1004-1389(2016)03-0317-11
通信作者:程振涛,男,副教授,硕士生导师,从事动物分子病原学研究。E-mail:chengzhentao@sohu.com
基金项目:黔西南州种草养羊产业发展省州科技合作专项(黔西南科合[2012]5号);贵州省自然科学技术基金(黔科合J字 [2015]2082号);贵州省农业攻关(黔科合NY[2014]3042);贵州省科技创新人才团队项目(黔科合人才团队[2015]4016号);贵州大学研究生创新基金(校基金研农2015029号)。
收稿日期:2015-09-09修回日期:2015-10-22
网络出版日期:2016-03-06
网络出版地址:http://www.cnki.net/kcms/detail/61.1220.S.20160306.1610.002.html
第一作者:吴燕,女,硕士,研究方向为动物疫病病原学。E-mail:491537385@qq.com

