癌症基因表达数据的集成分类器设计与分析
宋年丰
摘 要:基因表达水平对癌症诊断起到重要的作用。文章提出了一种基于SVM(Support Vector Machine)的集成分类算法,从基因表达水平的微阵列数据中对癌症和正常群体进行分类。文章提出了一种结合Adaboost算法和递归特征消除(Recursive Feature Elimination,RFE)算法,选取最显著的特征并设计与之适合的分类器。该方法已应用于癌症病人的基因表达微阵列数据的分类中,其分类结果在成功率方面有极大的提升。
关键词:SVM;集成方法;ROC;微阵列;基因表达
基因的表达模式对于疾病的诊断具有特征性。到目前为止,在机器学习领域,有众多分类或预测方法已经被提出来,其中许多已被应用到基于基因表达水平的微阵列数据的癌症分类。但是,由于高维微阵列数据容易带来过拟合、性能差和效率低等缺陷,因此,将传统的学习算法应用到这类数据中将会遇到极大的挑战。为了降低这种所谓的“高维小样本”的问题所带来的不足,近年来,一些改进和综合比较的算法已经被提出来。这些算法都已表现出有效性和成为可行的解决方案。尽管很多研究人员对癌症分类已经做了很多研究,但是鲜有研究者关注基于支持向量机的综合集成方法来处理这类问题和特征如何影响分类器的性能。
本文试图引入综合递归特征消除(RFE)算法连同基于SVM的Adaboost算法作为学习算法来极为显著地改善樣本分类的准确性和鲁棒性。结合分类器的特征选择可以利用样本的更多信息同时移除分类中的特征噪声。通过使用集成支持向量机,本文能够更有效地结合这些特征并改善结果的稳定性和鲁棒性。
1 方法与数据
1.1 实验流程
基于基因表达微阵列数据的预处理和标准化。用RFE算法选择特征,基于选择的特征、训练和建立一个基于SVM的集成分类器作为学习算法。最后,通过竞争性的集成算法,鲁棒性大大改善。这里,本文用多数表决来结合Adaboost算法中的结果。所有处理框架的完成都通过MATLAB来实现。
1.2 数据描述
在本研究中,采用了2个来自不同群组的基因表达微阵列数据集。这2个数据集有不同的特性(其中一个数据集可以线性地分开,而另一个则不行)。第一个数据集来自患白血病的癌症病人(急性髓细胞性白血病-AML和急性淋巴细胞白血病-ALL)。这个数据集有两个子集,训练集包含38个骨髓样本,测试集包含34个样本(其中20个All样本,14个AML样本)。所有样本共7129个特征,对应一些从微阵列图像中提取出来被标准化的基因表达水平值。
第二个数据将来自正常的和癌变的乳腺组织。这个数据集包含295个样本,8141个特征。病人有217个样本,正常人只有78个样本,为了数据的均匀化,本文从第一类中抽取了61个样本,从第二类中抽取65个样本作为训练集。在第一类中抽取27个样本在第二类中抽取26个样本作为测试集。
2 结果与讨论
2.1 分类器的分类性能
将SVM和基于SVM的集成算法应用于乳腺癌数据中。尽管核函数为线性的SVM比核函数为RBF的效果更好,达96.23%,但前者需要更多的特征和时间来运行程序。SVM-RBF的成功率只有90.566%,但集成方法的成功率是94.3396%,且只需要更少的特征数量,所以可以得出结论基于SVM的集成算法可以改进分类器的性能。当基因数量为34时,训练集和测试集的成功率最高,这些基因被称为与分类最相关的标记基因(见图1)。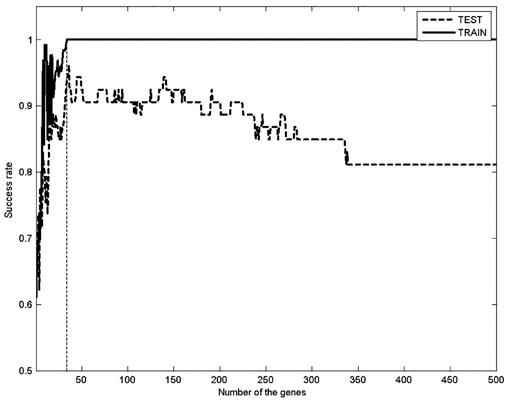
2.2 不同参数对分类器性能的影响
2.2.1 核函数选择的重要性
首先,本文应用SVM算法(核函数为RBF)的白血病数据集,但分类结果很差,成功率只有58.8235%。如果本文设置内核函数为线性核函数类型时,分类成功率得到了极大的提高,达到82.3529%。同样的情况也发生在乳腺癌的数据中。当应用SVM算法的基因表达数据集进行分类,核函数的选择对分类结果有着重要的影响(见表1)。
2.2.2 特征选择的重要性
本文将SVM或者基于SVM的集成算法应用到白血病数据集,结果如表2所示。在选择特征之前,测试集的成功率很低。当使用了重要的特征,无论核函数是否合适,成功率都改善了很多,因此特征选择是实验的关键因素。由于数据特征维数比数据集的样本维数更多,会导致过拟合。这些特征中可能包含了噪声,也会对分类有影响。实验中,本文发现集成方法对于白血病数据没那么有效。原因是当只使用了SVM时,已经获得了91.1764%的结果。如果SVM已表现出很好的性能,那么集成方法将失去它的优势。
3 结语
本文应用特征选择改善Adaboost算法,通过RFE方法选取基因,得出结论:(1)集成方法在某种程度上改善了SVM分类器的性能。(2)如何选择和提取特征子集对基因分类有至关重要的影响。(3)如果支持向量基的效果在某些数据集上更好,那集成将变得没有作用。本文将在今后继续探究导致基于支持向量基的集成算法变得无效的因素。
[参考文献]
[1]Boulesteix AL,Strobl C,Augustin T,et al. Evaluating microarray-based classifiers:an overview[J].Cancer Informatics,2008(6):77-97.
[2]Liu H,Sun J,Liu L,et al. Feature selection with dynamic mutual information[J].Pattern Recognition,2009(42):1330-1339.
[3]Freund Y,Schapire RE.A decision-theoretic generalization of on-line learning and an application to boosting[J]. Journal of computer and system sciences,1997(55):119-139.
[4]Golub TR,Slonim DK,Tamayo P,et al.Molecular classification of cancer: class discovery and class prediction by gene expression monitoring[J].Science,1999(286):531-537.
Design and Analysis of Ensemble Classifier for Gene Expression Data of Cancer
Song Nianfeng
(Automation Department, Xiamen University, Xiamen 361005, China)
Abstract: Gene expression levels are important for disease, such as, Cancer diagnosis. This paper proposed a SVM-based ensemble classifier to classify the control and cancer groups based on gene expression levels from microarray data. A combinational Recursive Feature Elimination in conjunction with the Adaboost algorithm was developed to select significant features and design the proper classifier. The method is applied to microarray data of cancer patients, and the results show improvements on the success rate.
Key words: SVM; ensemble methods; ROC; microarray; gene expression

