可可核心种质遗传多样性及果实性状与SSR标记关联分析
李付鹏 秦晓威 郝朝运 闫林 伍宝朵 赖剑雄

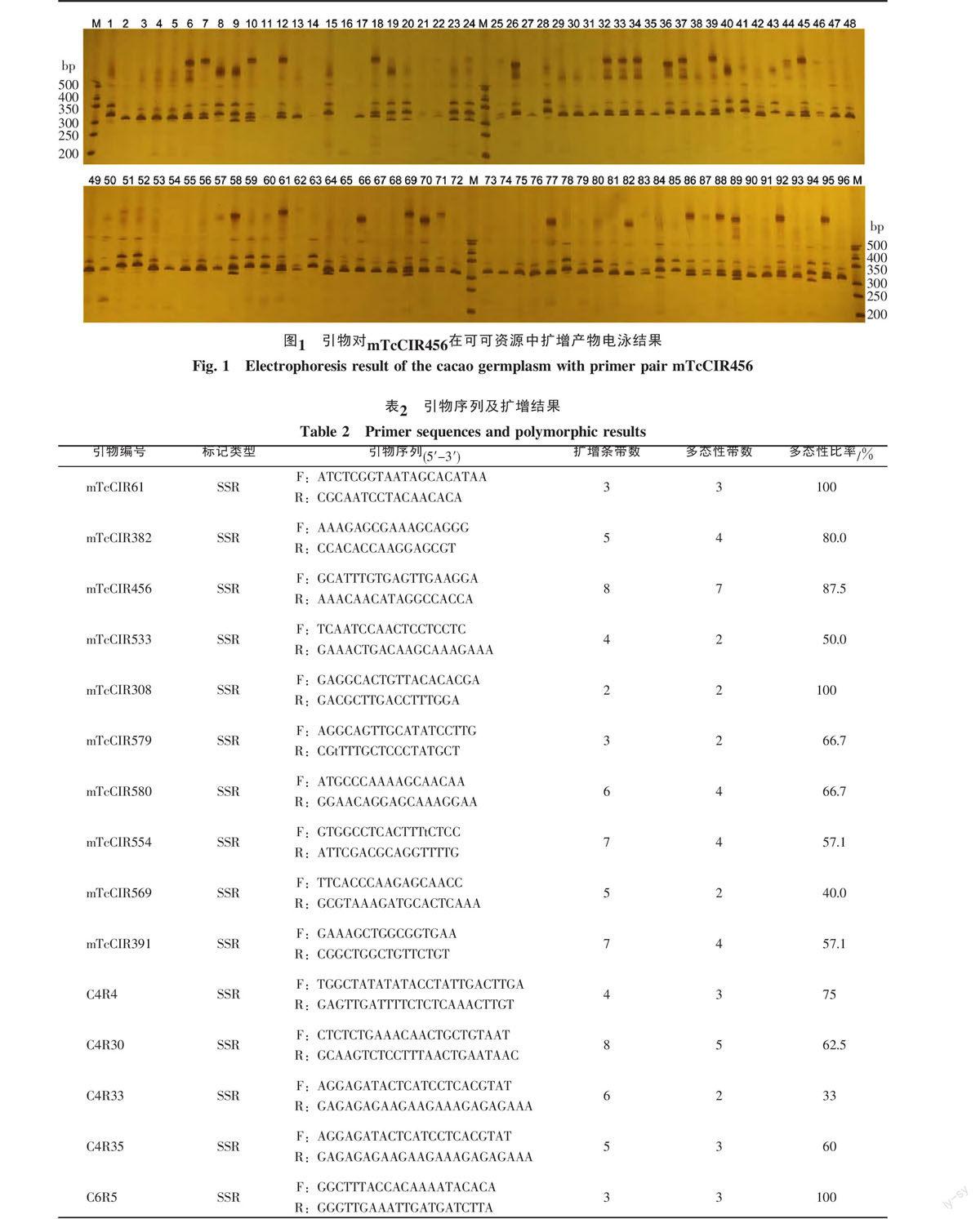

摘 要 利用15对SSR引物,分析具有广泛来源的70份可可资源的遗传多样性。结果表明,15对引物共扩增出76个条带,其中50个多态性条带,占总带数的65.8%。70份可可资源间遗传相似系数(SM)在0.341~0.943之间,平均值为0.625,说明可可资源具有丰富的遗传多样性。在相似系数为0.65水平上,可将70份可可资源分成10类。利用Structure 2.3.2软件分析群体结构,结合可可果实相关性状的表型数据,采用Tassel 2.1的一般线性模型(General linear model,GLM)进行关联分析;结果表明18个位点与果重、果壳重、果长、果径围、果壳厚显著相关(p<0.05),各位点对表型变异贡献率为5.5%~13.1%。
关键词 可可;SSR;遗传多样性;关联分析
中图分类号 S571.3 文献标识码 A
Genetic Diversity and Association Analysis of
Cacao Germplasm Using SSR Markers
LI Fupeng1,2,3, QIN Xiaowei1,2,3, HAO Chaoyun1,2,3, YAN Lin1,2,3,
WU Baoduo1,2,3, LAI Jianxiong1,2,3 *
1 Spice and Beverage Research Institute, Chinese Academy of Tropical Agricultural Sciences, Wanning, Hainan 571533, China
2 Tropical Spice and Beverage Germplasm Repository, Ministry of Agriculture, Wanning, Hainan 571533, China
3 Ministry of Agriculture Key Laboratory of Genetic Resources Utilization of Spice and Beverage Crops, Wanning, Hainan 571533, China
Abstract Fifteen SSR primers were used to study the genetic diversity among wide original 70 cacao germplasm resources. Seventy-six loci were detected by the 15 primers, of which 50 were polymorphic and its polymorphic ratio was amounted for 65.8%. The genetic similarity was ranged from 0.341 to 0.943 among the accessions, with an average of 0.625. The results indicated that the cacao accessions were rich in genetic diversity. Based on the dendrogram of UPGMA, 70 accessions could be divided into ten groups at the level of 0.65. Based on the analysis of population structure,an association analysis between SSR markers and pod agronomic traits were performed using the Tassel 2.1 GLM(general linear model)program. Of the SSR loci, 18 were significantly associated with pod weight, pod shell weight, pod length, pod equatorial circumference, and pod shell thickness under GLM program, and the ratio of explanation on the phenotype variation of related markers ranged from 5.5%-13.1%.
Key words Theobroma cacao L.; SSR marker; Genetic diversity; Associate analysis
doi 10.3969/j.issn.1000-2561.2016.02.003
可可(Theobroma cacao L.)与咖啡、茶并称世界三大饮料作物,全世界热带地区均有栽培[1]。2014年世界可可收获面积为1.01×107 hm2,总产量4.30×106 t,预计未来种植面积将继续扩大[2]。可可是中国新兴的热带特色经济作物,适宜在海南岛东南部推广种植,栽培管理简单,是典型的“懒人作物”,有巨大的发展潜力[3-4]。
可可以经济利用价值高在世界农业发展中占有重要地位,种质资源的多样性一直备受研究人员关注。自Pound[5]首次在亚马逊河流域开展可可抗病种质资源调查以来,世界可可主产国相继开展了资源普查与收集工作,现已建立了10多个国家级和3个国际级种质资源保存中心[6]。传统上,人们依据地理起源和形态特征将可可分成Criollo,Forastero和Trinitario(Criollo×Forastero)3大遗传类群。Motamayor等[7]和Utro等[8]分别利用SSR标记和基因组序列分析可可种质遗传多样性,将可可划分成10大遗传类群:Amelonado,Contamana,Curaray,Guiana,Iquitos,Maraňón,Nanay,Purús,Criollo和Nacional,为可可种质的遗传多样性研究提供了有益参考。
中国热带农业科学院香料饮料研究所从20世纪60年代开始,从世界可可主产区收集三大类可可遗传资源近300份。可可为异花授粉,遗传背景复杂,早期引进的可可种质经过自然杂交又创制出大量表型特异的新种质。Hansen等[9]首次将全基因组关联分析用于植物后,现已广泛应用于小麦[10]、马铃薯[11-12]、木薯[13]等作物中。国外相继在可可中开展了关联研究,Schnell等[14]以149份可可资源为自然群体,利用46对SSR标记的扩增多态性与群体材料种苗丰产性进行关联,筛选出17个与丰产性显著相关的位点。Marcano等[15]利用自然群体分析果重和粒重的QTLs,鉴定出5个与果重和粒重性状关联的区间。但是,关于可可果壳重、果长、果径围、果壳厚等性状与SSR标记关联分析少见报道。
为明确中国可可资源遗传差异,本研究分析了70份可可核心种质遗传多样性,明确其遗传背景,为种质准确鉴定及资源创新提供可靠分子依据;通过挖掘与果实性状显著关联的SSR标记,从而为可可等位基因发掘以及分子标记辅助选择育种提供一些有意义的参考。
1 材料与方法
1.1 材料
供试材料来自中国热带农业科学院香料饮料研究所农业部热带香料饮料作物种质资源圃的70份可可资源,其中印尼18份(INA-)、厄瓜多尔7份(ECU-)、马来西亚7份(MAS-)、泰国6份(THA-)、越南5份(VN-)、委内瑞拉5份(WEN-)、科特迪瓦4份(CIV-)、巴新3份(PNG-)、科摩罗2份(COM-),名称中有“SBRI-”的13份种质为20世纪60~80年代香饮所在引进基础上创制的资源,详见表1。2013年2~4月期间,考察70份可可资源材料的果重/g、果壳重/g、果长/cm、果径围/cm、果壳厚/cm表型值。采集供试材料的嫩叶后,在实验室用液氮速冻研磨,保存于-80 ℃冰箱中备用。
1.2 方法
1.2.1 总DNA提取 取出各研磨粉碎的可可叶片样品,利用基因组DNA提取试剂盒(购自Omega公司,D3471-01)抽提总DNA。1.2%琼脂糖凝胶电泳检测抽提DNA质量,UV-2310II型紫外可见分光光度计测定DNA浓度,稀释为20 ng/μL,备用。
1.2.2 SSR引物开发及遗传多样性分析 利用可可产量与品质相关QTLs信息[16-17],分析可可全基因组(http://www.cacaogenomedb.org/),在90条系列中获得65个微卫星。采用Primer 5.0软件设计特异引物,再挑选均匀分布于可可10条连锁群上的SSR引物53对,由上海生工生物工程公司合成。选用果重、果色、粒重等表型差异较大、不同来源的4份可可(VN-o1、SBRI-e7、PNG-tn13、WEN-ev1),使用Agilent SureCycler 8800 PCR仪进行SSR反应,反应体系为20 μL,包含50 ng模板DNA,10 μmol/L的左右引物各0.5 μL,2 μL Buffer(10×),1.6 μL Mg2+(25 mmol/L),0.2 μL Taq(MBI Fermentas),补ddH2O至20 μL。PCR程序:94 ℃ 3 min;94 ℃ 45 s,56 ℃ 1 min,72 ℃ 1 min,10个循环,退火温度每个循环下降1 ℃;94 ℃ 45 s,50 ℃ 1 min,72 ℃ 1 min,30个循环;72 ℃ 10 min,4 ℃保温。PCR反应产物用8%的聚丙烯酰胺凝胶(PAGE)检测,电泳在君意东方JY-JX5型电泳仪上进行,时间为90 min,电压设定为150 V,电泳后银染显影。
1.3 数据处理
采用人工方法统计引物多态性,相同迁移位置,清晰度好的扩增条带赋值“1”,无条带或肉眼不易分辨的弱带赋值“0”。利用PopGen32软件计算Nei's基因多样性和Shannon信息指数;用NTSYS-pc2.1软件,按SAHN邻接法(neighbor-joining method,NJ)对供试资源进行无权重配对算术平均数法(Unweighted pair group method using arithmetic averages,UPGMA)遗传相似性聚类,并绘制树状聚类图。
运用Structure 2.3.2软件进行群体结构分析,群体数目K值的取值范围为2~12,每个K值重复运行10次,参照Evanno等[18]的方法,依据LnP(D)计算ΔK最小且逐渐趋于稳定时的K值,为原始群体的亚群数,并计算所有材料相应的Q值(第i材料其基因组变异源于第k群体的概率)。采用Tassel 2.1软件基于一般线性模型(General linear model,GLM)的关联分析,将各性状的表型数据对标记逐一进行回归分析,并计算各标记对表型变异的解释率。
2 结果与分析
2.1 引物扩增筛选及多态性
118对SSR引物中,有51对引物能扩增出清晰、易于辨识的条带,其中15对具有稳定的多态性(图1);15对SSR引物在70份资源中共扩增出76个条带,平均每对引物5.07个;得到50个多态性条带,占总带数的65.8%,不同引物对揭示的等位基因为2~7个,平均每对引物3.33个(表2)。70份资源的Nei's基因多样性指数平均为0.328,Shannon信息指数平均为0.496。可见,上述可可资源遗传多态性丰富,也表明其遗传背景的复杂性与基因的多样性。
2.2 聚类分析
根据扩增条带的数据矩阵,分析参试材料间遗传相似系数,结果表明,70份可可资源间遗传相似系数(SM)介于0.341~0.943之间,平均值为0.625,从分子水平说明参试资源间存在丰富的遗传差异。其中SBRI-s19与VEN-e12之间的遗传相似系数最低(0.341),亲缘关系最远;THA-t16与MAS-tr17之间的遗传相似系数最高(0.943),2份种质分别来自泰国和马来西亚,不仅来源地相近,分子水平上的亲缘关系也非常紧密。
综合SSR引物扩增的50个多态性位点进行系统聚类分析,得到70份可可资源的聚类分析树状图。从图2可见,在遗传相似系数为0.65水平上可将70份可可资源分成10类。其中SBRI-sv18(B)、VEN-tn5(C)、ECU-sv8(E)分别单独聚成一类,A、F、G类有2份资源。D类和H类均有17份资源,I类有12份资源,J类有15份资源,各自类群的种质来源地不一,表明可可扩散传播区域比较广泛。D、H、I、J类群资源的果重表型分别为552.51、603.84、584.87、628.75 g,类群间存在一定的差异性。在实践中,可以有目的地选用不同类群间的种质材料配制杂交组合,一方面可以创制新种质,扩大和丰富可可的遗传背景,另一方面筛选表型优异的株系,培育可可新品种。
2.3 果实性状与SSR标记关联分析
Structure 2.3.2软件分析表明,LnP(D)值随假定亚群数K值的增大而呈现增大趋势,由图3可知,在K等于5时,LnP(D)值和ΔK均出现拐点,因此推测该群体材料可被分为5个亚群。
将K=5时,70份材料对应的Q值作为协变量,基于GLM模型将SSR分子标记变异与果重、果壳重、果长、果径围、果壳厚等表型进行回归分析,寻找关联的标记,确定表型变异解释率。共检测到18位点与果重、果壳重、果长、果径围、果壳厚在p<0.05水平上相关。其中有1个位点与果长形状的相关性呈极显著(p<0.01);其余17个位点中,1个与果重相关,3个与果壳重相关,4个与果长相关,6个与果径围相关,3个与果壳厚相关。各位点对表型贡献率为5.5%~13.1%;对表型贡献率最大的是mTcCIR391-3(13.1%),它与果长极显著相关。值得一提的是,mTcCIR554-1标记位点同时与果壳重、果长、果径围相关,mTcCIR61-1标记位点同时与果重、果壳重、果径围、果壳厚相关(表3)。
3 讨论与结论
DNA分子标记技术,可以从根本上揭示不同种质间的遗传差异,极大方便了种质资源亲缘关系研究,有助于提高育种效率。近年来,国外研究人员利用RFLP[19]、SSR[20-21]、SNP[22]等分子标记分析了原产地和主产国可可种质的遗传多样性,其中利用SSR标记开展的研究较多。SSR标记能揭示多态性高且比较稳定,本研究利用SSR标记分析了中国收集保存可可种质遗传多样性,资源间Nei's基因多样性指数平均为0.328,遗传变异明显,接近可可原产地的水平[23]。结果表明,中国收集保存的可可资源在分子水平上表现出高度的遗传差异性,与其在果实和种子性状表现出丰富的多样性一致。
可可花结构极为特殊,雄蕊被花瓣囊所包裹,主要依靠蚊蠓、苍蝇、蚂蚁等为其传粉[24-25]。可可异花授粉更易结实[26],有性生殖后代遗传背景高度杂合,因此种质间表现出高水平的遗传多样性。70份参试资源在遗传相似系数为0.65水平上,聚成10类,受限于参试资源的数量,类群间所包含的资源份数有较大差异。资源份数较多的I、J、D、H类群内,其果重和单粒重表型也表现出广泛的多样性,也说明可可资源遗传丰富。先前的研究结果表明,果重与种子产量呈显著的正相关,但与果实经济系数呈极显著负相关;果径围、果长均与种子产量呈极显著的正相关[27]。结合种质的遗传多样性和性状表型,在育种实践中可以挑选MAS-tr7、MAS-tr18、CIV-f20、SBRI-fv17、ECU-sv8、PNG-tn13等资源作为亲本,配制杂交组合,在杂交后代中筛选携带双亲优良表型的单株,培育新品种。
关联分析以自然群体为材料,省去构建作图群体所需的巨大工作量和时间,目前己发展成解析作物复杂数量性状,继而发掘优异基因/QTL的有效方法之一。本研究利用15个SSR标记对5个果实相关性状进行了关联分析,共找到18个与果重、果壳重、果长、果径围、果壳厚相关的SSR位点,分别位于3、4、6、8、10号染色体。本研究定位的与果重性状相关的位点mTcCIR61-1位于第10染色体,Marcano等[15]在第10连锁群上72 cM处定位出一个与果重性状相关的位点,表型解释率为10.8%,两者位置相近。由于中国可可种质均由国外引进,来源广泛,引进资源多通过种子扩繁,完全了解可可的系谱关系存在相当大的困难,所以本研究对所选资源进行种群结构分析时只能依据遗传差异进行分类,各类群种质的地域来源或者地理生态型不如Motamayor等[7]研究的清晰。以后,可将中国保存的可可资源的分子标记多态性或基因组序列,与国际上谱系来源清晰的资源进行比对,以明晰中国可可种质传播路径与遗传背景;进而可对自然群体材料进行全基因组SNP位点的关联分析,检测优良变异的等位基因,辅助分子设计育种。
参考文献
[1] Iwaro A D, Bekele F L, Butler D R. Evaluation and utilisation of cacao(Theobroma cacao L.)germplasm at the International Cocoa Genebank, Trinidad[J]. Euphytica, 2003, 130(2): 207-221.
[2] 国际可可组织[DB]. http: //www.icco.org/.2014/2015年鉴.
[3] 朱自慧. 世界可可业概况与发展海南可可业的建议[J]. 热带农业科学, 2003, 23(3): 28-33.
[4] 赵溪竹, 朱自慧, 王 华, 等. 世界可可生产贸易现状[J]. 热带农业科学, 2012, 32(9): 76-81.
[5] Pound F J. Cacao and witches' broom disease of South America, with notes on the other species of Theobroma[M]// Report on a visit to Ecuador, the Amazon Valley and Colombia, April 1937-April 1938. Port-of-Spain, Trinidad and Tobago, 1938: 58.
[6] 秦晓威, 郝朝运, 吴 刚, 等. 可可种质资源多样性与创新利用研究进展[J]. 热带作物学报, 2014, 35(1): 188-194.
[7] Motamayor J C, Lachenaud P, da Silva e Mota J W, et al. Geographic and genetic population differentiation of the Amazonian chocolate tree(Theobroma cacao L.)[J]. PLoS One, 2008, 3(10): e3 311.
[8] Utro F, Cornejo O E, Livingstone D, et al. ARG-based genome-wide analysis of cacao cultivars[J]. BMC Bioinformatics, 2012, 13(Suppl 19): S17.
[9] Hansen M, Kraft T, Gaaaestam S, et al. Linkage disequilibrium mapping of the bolting gene in sea beet using AFLP markers[J]. Genet Res, 2001, 77(1): 61-66.
[10] Maccaferri M, Saaguineti M C, Enrico N, et al. Population structure and long-range linkage disequilibrium in a durum wheat elite collection[J]. Mol Breeding, 2005, 15(3): 271-290.
[11] Gebhardt C, Ballvora A, Walkemeier B, et al. Assessing genetic potential in germplasm collections of crop plants by marker-trait association: a case study for potatoes with quantitative variation of resistance to late blight and maturity type[J]. Mol Breeding, 2004, 13(1): 93-102.
[12] Simko I, Costanzo S, Haynes K G, et al. Linkage disequilibrium mapping of a Verticillium dahliae resistance quantitative trait locus in tetraploid potato(Solanum tuberosum)through a candidate gene approach[J]. Theor Appl Genet, 2004, 108(2): 217-224.
[13] 何 静, 王文泉, 郑永清, 等. 木薯栽培品种农艺性状与分子标记的关联分析[J]. 热带作物学报, 2010, 31(5): 693-700.
[14] Schnell R J, Olano C T, Brown J S, et al. Retrospective determination of the parental population of superior cacao (Theobroma cacao L)seedlings and association of microsatellite alleles with productivity[J]. J Amer Soc Hort Sci, 2005, 130(2): 181-190.
[15] Marcano M, Pugh T, Cros E, et al. Adding value to cocoa (Theobroma cacao L.)germplasm information with domestication history and admixture mapping[J]. Theor Appl Genet, 2007, 114(5): 877-884.
[16] Clement D, Risterucci A M, Motamayor J C, et al. Mapping quantitative trait loci for bean traits and ovule number in Theobroma cacao L.[J]. Genome, 2003, 46(1): 103-111.
[17] Araújo I S, de Souza Filho G A, Pereira M G, et al. Mapping of quantitative trait loci for butter content and hardness in cocoa beans(Theobroma cacao L.)[J]. Plant Mol Biol Rep, 2009, 27(2): 177-183.
[18] Evanno G, Regnant S, Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study[J]. Mol Ecol, 2005, 14(8): 2 611-2 620.
[19] N'Goran J A K, Laurent V, Risterucci A M, et al. The genetic structure of cocoa populations(Theobroma cacao L.)revealed by RFLP analysis[J]. Euphytica, 2000, 115(2): 83-90.
[20] Saunders J A, Mischke S, Leamy E A, et al. Selection of international molecular standards for DNA fingerprinting of Theobroma cacao[J]. Theor Appl Genet, 2004, 110(1): 41-47.
[21] Efombagn I B M, Motamayor J C, Sounigo O, et al. Genetic diversity and structure of farm and GenBank accessions of cacao(Theobroma cacao L.)in Cameroon revealed by microsatellite markers[J]. Tree Genet Genomes, 2008, 4(4): 821-831.
[22] Ji K, Zhang D P, Motilal L, et al. Genetic diversity and parentage in farmer varieties of cacao(Theobroma cacao L.)from Honduras and Nicaragua as revealed by single nucleotide polymorphism(SNP)markers[J]. Genet Resour Crop Ev, 2013, 60(2): 441-453.
[23] Sereno M L, Albuquerque P S B, Vencovsky R, et al. Genetic diversity and natural population structure of cacao(Theobroma cacao L.)from the Brazilian Amazon evaluated by microsatellite markers[J]. Conserv Genet, 2006, 7(1): 13-24.
[24] Silva C R, Albuquerque P S, Ervedosa F R, et al. Understanding the genetic diversity, spatial genetic structure and mating system at the hierarchical levels of fruits and individuals of a continuous Theobroma cacao population from the Brazilian Amazon[J]. Heredity, 2011, 106(6): 973-985.
[25] Thomas E, van Zonneveld M, Loo J, et al. Present spatial diversity patterns of Theobroma cacao L. in the neotropics reflect genetic differentiation in pleistocene refugia followed by Human-influenced dispersal[J]. PLoS One, 2012, 7(10): e47 676.
[26] Chumacero de Schawe C, Durka W, Tscharntke T, et al. Gene flow and genetic diversity in cultivated and wild cacao(Theobroma cacao)in Bolivia[J]. Am J Bot, 2013, 100(11): 2 271-2 279.
[27] 李付鹏, 王 华, 伍宝朵, 等. 可可果实主要农艺性状相关性及产量因素的通径分析[J]. 热带作物学报, 2014, 35(3): 448-453.

