基于SYBR Green检测小麦黄花叶病毒的两步法qPCR
刘成 铃木孝子 佐久间俊 宋健民 李豪圣 刘爱峰 曹新有 韩冉

摘要:小麦黄花叶病毒病是其常发病,常造成严重减产。为建立可用于定量检测小麦黄花叶病毒(WYMV)拷贝数的qPCR方法,本研究对不同来源的WYMV小种序列进行比对分析,在序列保守区域设计引物11对,对其退火温度进行优化,进而建立各引物的qPCR标准曲线。结果发现,11对引物的扩增效率在50.60%~116.04%之间,仅2对引物可用于后续研究,扩增效率分别为97.21%(WY-05和WY-06)和91.85%(WY-F2和WY-R2)。利用其中的WY-F2和WY-R2对高抗和高感WYMV的小麦品种进行分析,结果显示,抗感小麦品种WYMV拷贝数差异极显著。对抗感分离群体266个单株进行分析,发现该qPCR方法灵敏度显著高于酶联免疫吸附剂测定法,更适用于抗感杂交分离群体的表型鉴定,因而可以用于抗WYMV基因的遗传定位与图位克隆工作。
关键词:小麦黄花叶病毒;两步法qPCR;酶联免疫吸附剂测定法
中图分类号:S435.121.4+9文献标识号:A文章编号:1001-4942(2016)07-0118-07
小麦黄花叶病毒(Wheat yellow mosaic virus,WYMV)是一种由禾谷多黏菌(Polymyxa graminis)介导才能侵染寄主的土传病原微生物,最早在日本发现[1],后在中国和加拿大等国发现[2,3]。感病小麦表现为根系发育差、植株松散[2]、叶片发黄、生长发育迟缓甚至矮化[2,4],分蘖力、成穗率和产量降低[2,5],对小麦品质也有不利影响[6]。我国小麦黄花叶病毒病分布范围广,包括北部冬麦区的胶东沿海副区、黄淮冬麦区大部、长江中下游麦区和西南麦区部分地区等[7],该病害受寄主感病程度、病毒毒力以及气候因子特别是温度影响较大[5]。近10年来,我国小麦黄花叶病毒病呈逐年加重之势[2],仅2006年,就有9个省爆发该病,受灾面积达到200万公顷,造成小麦减产15亿千克[8]。
WYMV及其载体在土壤中的潜伏期很长。研究显示,有WYMV发病土地,即使10年不种植小麦,10年后仍然对小麦具有较强的致病毒力[9]。所以,一旦某一麦区暴发该病将很难根除,对小麦生产威胁较大。因此,研究清楚抗小麦黄花叶病毒病品种的抗病机制对小麦分子育种具有重要意义,定位并图位克隆抗WYMV基因进而进行基因功能验证是研究抗WYMV机制的基础。基于此,我们开展了抗WYMV基因的精细定位与图位克隆工作(待发表)。而精确鉴定分离群体抗病性则为能否成功精细定位并图位克隆到基因的关键,因此,材料抗病性鉴定方法就成为问题的重要一环。目前,对WYMV的鉴定方法有形态学调查[10]、蛋白水平检测[11,12]和分子水平检测[8,13]等。然而,上述方法均是检测WYMV的定性分析方法,不能定量获得植株感染病毒拷贝数进而对植株抗病程度进行精确界定。为了解决这一问题,本研究拟通过建立一种基于SYBR Green检测WYMV的两步法实时定量PCR(qPCR),并对抗感分离群体进行病毒拷贝数定量分析,确认可应用于WYMV病毒数鉴定的方法。
1材料与方法
1.1试验材料
高抗WYMV的美国小麦品种Madsen由日本国家研究中心提供。日本小麦品种(系)Hokushin(感WYMV)、TK-2(抗WYMV)和Shinchunaga(未鉴定WYMV抗性)由日本北海道中央农业试验站提供,其中TK-2是Hokushin/Madsen的BC5F4近等基因系。编号为#001~#266的植株为Hokushin/TK-2的F2抗感WYMV分离群体。除Shinchunaga外,其余试验材料全部种植于富含WYMV的日本北海道中央试验农场编号为Minami-Mareppu 40的区域地块里,当年秋季播种,次年4月中旬待植株充分发病后取样进行病毒数检测。所有参试材料均测定来自同一地块不同位点的三个不同单株(取旗叶下的第二叶)的病毒拷贝数,减少因种植过于集中且土壤含病毒不均匀或病毒感染上传不充分造成的误差。除此之外,Madsen、Hokushin和Shinchunaga还被种植于日本国立农业生物资源研究所(筑波)不含WYMV的试验农场中,作为前述种植于含WYMV地块中样品的阴性对照。
DNA酶Ⅰ购自TaKaRa公司,RNA纯化及反转录所用试剂耗材、连接载体均购自Invitrogen公司。 qPCR所需试剂购自日本东洋纺公司,48孔反应板及高透光封口膜购自ABI公司。 灭菌DEPC移液枪头和RNase AWAY分别购自QSP和MBP公司,ELASI试剂盒购自WAKO公司。
1.2试验方法
1.2.1总RNA提取、检测与纯化取200 mg 鲜叶片放于加入氧化锆珠的2 mL 离心管中,利用Retsch MM300混合型碾磨仪在25 Hz下震荡1.5 min;加入Trizol 500 μL,室温放置5 min;接着加入氯仿100 μL,室温放置3 min,4℃、10 000×g条件下离心15 min;转移上清液到另一新离心管中,加入异丙醇300 μL轻轻混匀,室温放置10 min,4℃、10 000×g条件下离心10 min;弃上清,加入预冷的75%乙醇清洗,4℃、7 000×g条件下离心5 min,重复两次;待RNA干燥后加入50 μL DEPC水溶解后保存于-20℃冰箱备用。
RNA的完整性在琼脂糖凝胶中检测,完整性较好的RNA浓度检测在Thermo Scientific NanoDrop 2000分光光度计下进行。RNA中DNA污染的去除:50 μL的总反应体系包含50 μg总RNA、10 U的DNA酶Ⅰ、20 U的RNA酶抑制剂和DEPC水,轻轻混匀,37℃条件下处理2 h;处理完毕后,加入3 mol/L醋酸钠(pH=5.2) 5 μL,轻轻混匀后加入异丙醇56 μL,混匀后在4℃、10 000×g条件下离心10 min;弃上清,用预冷的75%乙醇清洗,4℃、7 000×g条件下离心5 min,重复两次;待RNA干燥后加入50 μL DEPC水溶解。
1.2.2高质量RNA的选择与反转录纯化后的RNA再次利用琼脂糖凝胶检测其完整性,完整性较好的RNA浓度再次利用Thermo Scientific NanoDrop 2000分光光度计进行检测。分光光度计下A260/A280值在1.9~2.1范围内且A260/A230值在2.0~2.4范围内的用于后续反转录试验。
反转录体系:包含总RNA 1 μg、2.5 mmol/L dNTPs 4 μL、Oligo(dT)12~18引物0.5 μL,DEPC水补至10 μL,65℃条件下处理5 min,迅速置于冰上;随后,加入5×第一链缓合成缓冲液4 μL、0.1 mol/L二硫苏糖醇(DTT) 1 μL、20 U的RNA酶抑制剂和25 U的SuperScriptTMⅢ 反转录酶,最后用DEPC水补足到20 μL后轻轻混匀。反应条件:25℃反应5 min,然后,50℃反应3 h,紧接着70℃反应15 min。反应完成后离心机瞬时离心,加入DEPC水80 μL,轻轻混匀使得cDNA终浓度为10 ng/μL,保存于-20℃冰箱备用。
1.2.3序列比对与引物设计对NCBI数据库(http://www.ncbi.nlm.nih.gov/)中注册的多条WYMV序列进行比对,在序列保守区域进行引物设计。序列比对在软件DNAMAN上进行,引物设计利用在线软件Primer 3(http://frodo.wi.mit.edu/cgi-bin/primer3/primer3_www.cgi)进行。根据序列比对结果共设计7条引物,包含左引物3条(WY-05、WY-F1和WY-F2),右引物4条(WY-R1、WY-R2、WY-06和WY-16),序列分别如下:WY-05: 5′-ACCAATGCCGCCACCAAAG-3′;WY-F1: 5′-TCGAACACGACAACTCCATC-3′;WY-F2: 5′-CCGTCAACGCAGGACTAAA-3′; WY-R1: 5′-CAGAGCGATGGAGTTGTCG-3′;WY-R2: 5′-TGGTGATGCCAAGACTGGT-3′;WY-06: 5′-GCATGGTCTGGCTTTCTGC-3′;WY-16: 5′-GCCCATTTTGTGACAATCTTGTG-3′。上述左引物和右引物分别组合(表1),用于qPCR最适引物筛选;其中WY-05和WY-16还用于小麦黄花叶病毒序列克隆。用作内参的Actin基因特异引物DN182500F425和DN182500R533的序列参照文献[14],所有WYMV拷贝数均用相应Actin拷贝数进行标准化。
1.2.4qPCR标准曲线建立将克隆的WYMV序列与内参Actin基因序列分别连接到pCR4-TOPO载体上,分别用引物WY-05和WY-16、DN182500F425和DN182500R533进行阳性克隆的筛选。阳性克隆在含氨苄青霉素的LB液体培养基里培养,提取质粒。利用上述序列克隆引物对获得质粒进行测序,确认连接序列的正确性。将含WYMV序列与内参Actin基因序列的质粒DNA从107拷贝等比稀释到10个拷贝(共7个浓度梯度),用于计算每对引物的扩增效率及后续绝对定量分析。样品设置3个生物学重复及2个技术重复,平均log拷贝数及对应Ct值用于标准曲线的建立。
1.2.5qPCR反应与酶联免疫吸附剂测定qPCR总体积为20 μL,包括TBSYBR混合液10 μL,3 mmol/L左右引物各2 μL,10 ng/μL cDNA 2 μL,参考染料0.4 μL,灭菌双蒸水3.6 μL。反应程序为95℃ 1 min;主反应为35个循环,95℃ 15 s,60~65℃ 45 s(依据引物反应的优化温度);最后熔解曲线建立为1个循环,95℃ 15 s,55~65℃ 45 s(依据引物),95℃ 15 s。酶联免疫吸附剂测定法(ELISA)参照文献[11]。
1.2.6小麦黄花叶病毒数绝对量计算按照如下步骤计算:(1)质粒DNA的长度为空质粒3 956 bp(未连接小麦黄花叶病毒序列)+本研究所用小麦黄花叶病毒序列长度440 bp=4 396 bp;(2)质粒DNA分子量=质粒DNA长度(bp)×碱基平均分子量330道尔顿(D)×2(碱基/bp)=2 901 360 D(D=g/mol);(3)1个拷贝的DNA重量=质粒DNA分子量(g/mol)÷(6.023×1023分子/mol)(阿佛加德罗常数)=481 713×10-23g/分子≈4.82×10-18 g/分子;(4)1 ng RNA中含病毒拷贝数=1 ng÷1个拷贝DNA重量(g/分子)=2.07×108分子(拷贝)/ng RNA。利用log(拷贝数/ng RNA)数值与ELISA数值进行方法学比较分析。
2结果与分析
2.1qPCR引物的选择及标准曲线的建立
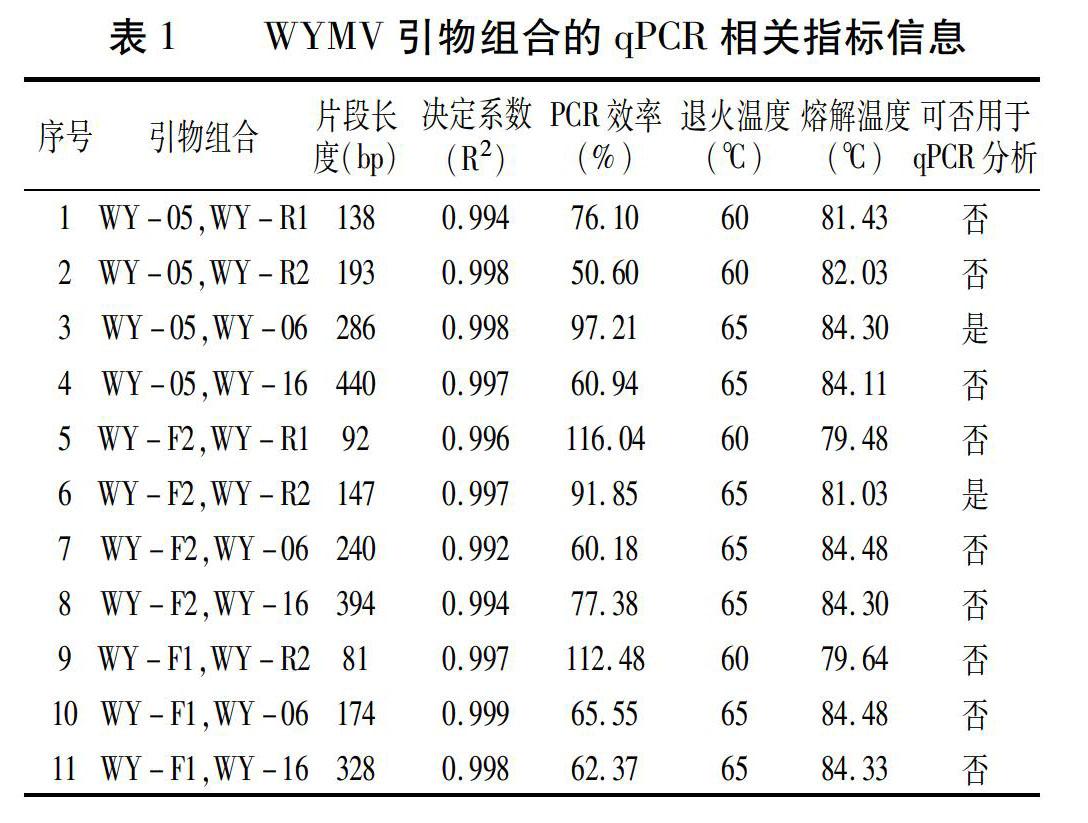
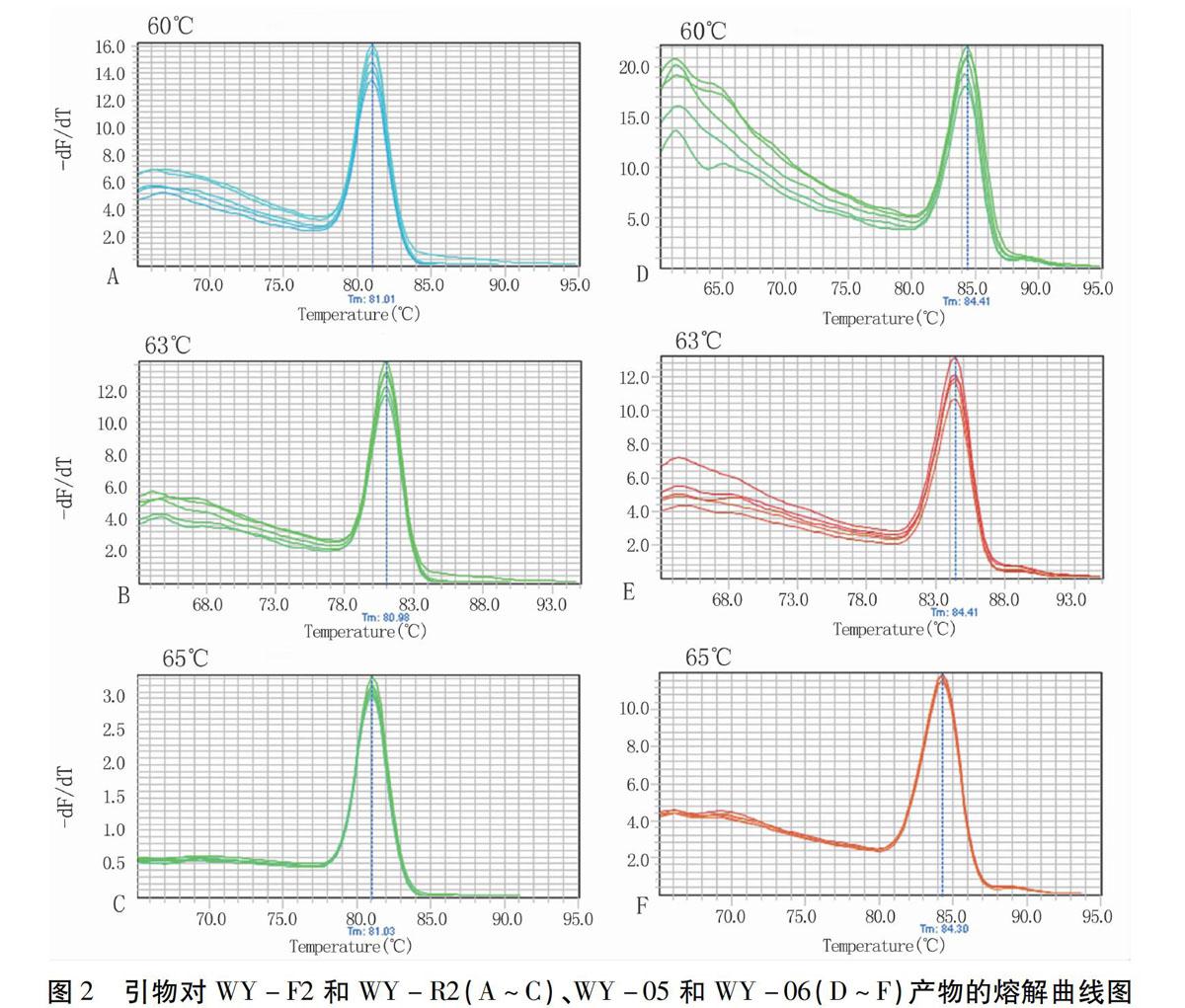
设计的qPCR引物在克隆的440 bp WYMV序列上的位置如图1所示。左引物和右引物分别组合,获得11个引物对(表1),用于qPCR最适引物的选择。各引物对进行最适退火温度优化,最适温度的确定用扩增产物熔解曲线结果判定,其中引物对WY-F2和WY-R2、WY-05和WY-06在60℃、63℃和65℃条件下的产物熔解曲线如图2所示。两对引物在60℃和63℃条件下产物不单一或出现双峰(图2A、B、D和E),而65℃条件下两引物对产物熔解曲线峰值单一(图2 C和F),因此,该两对引物的最适退火温度均为65℃。各引物对的扩增片段长度、PCR效率和退火温度等信息如表1所示。
利用合成的11对引物在其最适退火温度下分别建立标准曲线。通过对qPCR决定系数和引物PCR效率等指标进行分析,结果显示,所有引物对的qPCR决定系数均在0.99以上(表1),但仅引物对WY-05和WY-06、WY-F2和WY-R2的扩增效率(97.21%和91.85%)在可接受范围90%~110%之内,可用于后续qPCR试验。二者对应计算公式分别为y=-3.3782x+41.081和y=-3.3933x+38.580,其中,y为Ct值,而x为log拷贝数。利用内参基因特异引物DN182500F425和DN182500R533建立标准曲线(引物间序列长度为130 bp),获得计算公式为y=-3.2379x+40.159。
2.2原始亲本的qPCR检测
对种植于含WYMV地块的Madsen和Hokushin,种植于未感染WYMV地块的Madsen、Hokushin和Shinchunaga的叶片、茎秆、根和麦穗分别提取RNA,反转录成cDNA。用扩增片段较短的WY-F2和WY-R2(相对于引物对WY05和WY06)对上述样品以及绝对阴性对照ddH2O进行qPCR分析,结果发现,种植于含WYMV地块的Hokushin含病毒数高达几十万个拷贝/ng RNA(log拷贝数值在5~6之间,图3),而种植于不含WYMV地块的Madsen、Hokushin和Shinchunaga各器官以及ddH2O含病毒数均为10个左右(log拷贝数值约为1,图3,Madsen和Hokushin结果未列出),因此,种植于不含WYMV的植株以及种植于含WYMV地块Madsen中的病毒数是由于试验背景造成的。种植于含WYMV地块的Madsen和Hokushin病毒数量差异极显著,种植于不含WYMV地块的植株病毒数一致极低,说明以WY-F2和WY-R2为引物进行的两步法qPCR结果是准确可信的。
2.3抗感分离群体的qPCR和ELISA检测
利用WY-F2和WY-R2对Hokushin/TK-2的F2抗感WYMV分离群体共266个单株进行qPCR分析,结果显示,log(拷贝数/ng RNA)值在0~1(区间最大值的植株数包含在下一区间,下同)区间的有29株;在1~2区间的植株数最多,为186株;在2~3区间的有8株;在3~4区间的有15株;在4~5区间的有28株(图4A)。原始杂交亲本Madsen和Hokushin的log(拷贝数/ng RNA)值分别在0~1和5~6区间,即Hokushin/TK-2的F2单株的log(拷贝数/ng RNA)值均在两原始亲本log(拷贝数/ng RNA)范围内。
利用ELISA对该群体进行分析,结果发现,ELISA值在0~0.5的植株最多,为220株;在0.5~1.0区间的仅有1株;在1.0~1.5区间的有3株;在1.5~2.0区间的仅有1株;未检测到ELISA值在2.0~2.5区间的植株;ELISA值在2.5~3.0区间的有4株;在3.0~3.5区间的有37株(图4B)。原始杂交亲本Madsen和Hokushin的ELISA值分别在0~0.5和3.0~3.5之间,因此,Hokushin/TK-2的F2单株的ELISA值也均在两原始亲本ELISA值范围内。
为了清晰地比较两种方法,笔者对266个单株中编号为#228~#266植株以及原始杂交亲本Madsen和Hokushin的log(拷贝数/ng RNA)值和ELISA值进行散点分析,结果发现,ELISA可以把供试植株分为ELISA值小于1(本研究中定义为抗病)和大于1.5(感病)两类;qPCR也可以把供试植株分为log(拷贝数/ng RNA)值小于2.5(抗病)和大于3.5(感病)两类(图5A)。两种方法均能把供试材料分成抗病和感病两类,但是,编号为#243、#262和#266植株的ELISA值均低于1(图5B),被认定为抗WYMV,但是三者的log(拷贝数/ng RNA)值却高于3.5(图5A、C),属于感病类型,这表明本研究建立的两步法qPCR较ELISA灵敏度高。
3讨论与结论
迄今,检测或鉴定WYMV的方法有肉眼鉴定法[10]、酶联免疫吸附剂测定法[11]、反转录聚合酶链式反应法[8]、蛋白免疫印迹法[12]和环介导等温扩增技术[13],但是,这些方法仅能获得植株是否感染WYMV信息,不能对植株感染病毒拷贝数进行精确鉴定。qPCR因为具有操作简单、检测方便和可以定量分析等优势,已经在植物研究中得到广泛应用[15]。Liu等[16]建立了依据TaqMan检测小麦WYMV的一步法RT-qPCR,该方法对鉴定小麦WYMV拷贝数非常有效,然而,其价格相对于SYBR Green qPCR成本高3~5倍,且一步法RT-qPCR是将反转录和qPCR一次性完成,一旦试验结果出现问题,将无法返回检测。完成小麦抗病基因的图位克隆往往需要筛选几千甚至是上万个单株才能实现获得所需重组体[17,18],因此,成本问题往往是需要考虑在内的。本研究建立的基于SYBR Green的两步法qPCR能够弥补上述一步法的不足之处,对大群体或较多样本WYMV拷贝数鉴定可以优先选用。
因为WYMV需通过真菌感染小麦根部,进而向上进行转运复制,最终在小麦叶片上表现出来[19],两步法qPCR在种植于无WYMV土壤中的Houkushin 的根部、茎秆、叶片、麦穗和绝对阴性对照ddH2O中均检测到10个拷贝的WYMV,说明这10个拷贝是试验背景造成的,该背景的无法消除性说明该方法在材料绝对拷贝数的检测进而研究抗WYMV机理方面可能具有一定局限性。然而,10个拷贝的背景相对于感病植株几十万个WYMV拷贝差异极其显著,因而,完全可以应用于抗感分离群体的表型鉴定。
qPCR能够检测到ELISA无法检测到的感病植株,表明前者在对植株表型鉴定上具有明显优势。之前,Takeuchi等利用ELISA对Hokushin/Madsen重组自交系进行研究,认为来自Madsen的WYMV抗性可能受1个单显性核基因控制[11]。近期,该课题组对这一结论进行了修正,认为来自Madsen的WYMV抗性受来自2DL和3BS染色体上的两个主效QTL控制[20]。本研究利用qPCR对Hokushin/TK-2的F2 266个分离群体单株进行检测,结果发现,这些F2单株的log(拷贝数/ng RNA)值不是完全正态分布的,而是分布于0~5的每个区间,说明该群体的WYMV抗性是一个数量性状,而不是由单个基因控制的,与文献[20]的研究结果相互印证。虽然文献[20]依据ELISA对小麦表型鉴定的研究结论与本研究两步法qPCR结果类似,但是依据本研究中两种方法的比较结果,在条件允许的情况下,应优先推荐使用两步法qPCR。该方法不仅可以应用于遗传群体的抗WYMV表型鉴定,还有望被应用于抗WYMV小麦品种或种质的鉴定工作。
致谢:感谢日本国立农业生物资源研究所李超博士后、默罕默德博士后、王宁博士后和宁顺宗博士的有益讨论及小山晴美女士的技术支持。
参考文献:
[1]Sawada E. Control of wheat yellow mosaic virus [J]. J. Plant Prot., 1927, 14:444-449.
[2]赵玖华,徐德坤,尚佑芬,等. 山东省小麦黄花叶病的突发与防控措施 [J]. 山东农业科学,2012,44(10):95-97,100.
[3]Slykhuis J T. Evidence of soil-borne mosaic of wheat in Ontario [J]. Can. J. Plant Dis. Surv., 1960, 40:43.
[4]Liu W H, He Z T, Geng B, et al. Identification of resistance to yellow mosaic disease of wheat and analysis for its inheritance of some varieties [J]. Acta Phytopathol. Sin., 2004, 34:542-547.
[5]Clover G, Henry C. Detection and discrimination of wheat spindle streak mosaic virus and wheat yellow mosaic virus using multiplex RT-PCR [J]. European J. Plant Pathol., 1999, 105: 891-896.
[6]Kühne T. Soil-borne viruses affecting cereals-known for long but still a threat [J]. Virus Research, 2009, 141:174-183.
[7]阮义理,邹皖和,王卉. 我国真菌传大小麦病毒病的地理分布 [J]. 植物保护学报,1997,24(1):35-38.
[8]岳红妮,吴云锋,李毅然,等. 小麦3 种病毒病BSMV、BYDV-PAV、WYMV及WBD植原体病害的多重PCR同步检测 [J]. 中国农业科学,2008,41(9):2663-2669.
[9]Chen J P. Research status and prospect of cereal viruses transmitted by Polymyxa graminis in China [J]. Proc. Natural Sci., 2005, 15:524-533.
[10]Liu W H, Nie H, Wang S B, et al. Mapping a resistance gene in wheat cultivar Yangfu 9311 to yellow mosaic virus, using microsatellite markers [J]. Theor. Appl. Genet., 2005, 111: 651-657.
[11]Takeuchi T, Munekata S, Suzuki T, et al. Breeding wheat lines resistant to wheat yellow mosaic virus and localization of the resistance gene (YmMD) derived from wheat cultivar ‘Madsen [J]. Breed. Res., 2010, 12: 1-8.
[12]Xing Y, Su N, Li D, et al. Over-expression of 72 kDa protein of wheat yellow mosaic virus in E. coli and preparation of its antiserum [J]. Chin. Sci. Bull., 2000, 45(6):525-528.
[13]Zhang Z Y, Liu X J, Li D W, et al. Rapid detection of wheat yellow mosaic virus by reverse transcription loop-mediated isothermal amplification [J]. Virology J., 2011, 8:550.
[14]Ning S Z, Wang N, Sakuma S, et al. Structure, transcription and post-transcriptional regulation of the bread wheat orthologs of the barley cleistogamy gene Cly1 [J]. Theor. Appl. Genet., 2013, 126:1273-1283.
[15]Lyons R, Kutluk Yilmaz N D, Powers S, et al. Characterization of two unusual features of resistance to soilborne cereal mosaic virus in hexaploid wheat: leakiness and gradual elimination of viral coat protein from infected root tissues [J]. Mol. Plant-Micro. Inter., 2009, 22:560-574.
[16]Liu W W, Zhao X J, Zhang P, et al. A one step real-time RT-PCR assay for the quantitation of wheat yellow mosaic virus (WYMV) [J]. Virology Journal, 2013, 10:173.
[17]Fu D L, Uauy C, Distelfeld A, et al. A kinase-START gene confers temperature-dependent resistance to wheat stripe rust [J]. Science, 2009, 323: 1357-1359.
[18]Periyannan S, Moore J, Ayliffe M, et al. The gene Sr33, an ortholog of barley Mla genes, encodes resistance to wheat stem rust race Ug99 [J]. Science, 2013, 341: 786-788.
[19]Soosaar J L M, Burch-Smith T M, Dinesh-Kumar S P. Mechanisms of plant resistance to viruses [J]. Nature Rev. Microbio.,2005, 3:789-798.
[20]Suzuki T, Murai M N, Hayashi T, et al. Resistance to wheat yellow mosaic virus in Madsen wheat is controlled by two major complementary QTLs [J]. Theor. Appl. Genet., 2015,128:1569-1578.

