基于节点关联性的关键蛋白质识别算法研究
许睿,李琳芳,白林峰
(河南科技学院,河南新乡453003)
基于节点关联性的关键蛋白质识别算法研究
许睿,李琳芳,白林峰
(河南科技学院,河南新乡453003)
为提高蛋白质网络中关键蛋白质识别的效率,提出一种基于节点关联性的关键蛋白质识别方法.方法在保持蛋白质网络拓扑结构完整性的基础上,兼顾了蛋白质网络局部特性,从蛋白质节点关联性的角度,具体衡量蛋白质节点重要性,并采用节点失效法进行关键蛋白质的识别.实验分析证明了方法的有效性.
节点关联性;蛋白质网络;关键蛋白质;节点重要性
在蛋白质网络中,蛋白质节点之间往往联系密切,且关键蛋白质不是唯一的,他们构成一个集合,共同完成某种生命活动.随着关键蛋白质研究的深入,人们发现各种生物个体的蛋白质网络中,存在着一些共性,如生物体有自我修复的机制,少量的关键蛋白质的缺失可能会致病,经过细胞的自我修复可以恢复部分功能,但是大量的关键蛋白质的缺失则后果严重,导致生物体致死或者致残,无法恢复.可见,在蛋白质网络中,任何连通的蛋白质节点之间都存在着相互依赖的关系.这种依赖关系普遍存在,但是有强弱之分,比如相邻的蛋白质节点之间依赖关系会比中心节点和边缘节点之间依赖关系强很多,随着蛋白质节点对之间的路径的增加,这种依赖关系逐渐减弱.因此,这种依赖关系最直接地反映在直接相连的邻居蛋白质节点之间.
在复杂网络领域中,对于网络中节点重要性的评价标准,主要是从两个方面进行考虑:一是从局部特性的角度,主要的依据是节点的连接属性,常用的测度有度中心性、聚集系数等;二是从全局特性的角度,主要的依据是节点的位置特性,比较典型的测度有特征向量中心性、信息中心性、介数中心性等.本文提出一个新的算法,利用蛋白质节点之间的关联性,通过兼顾局部和全局两个方面的特性,进行关键蛋白质的识别.
1 相关定义说明
1.1 度中心性
节点的度中心性(Degree Centrality,DC)也就是节点的连接度,表示与该节点直接相邻的其他节点的数目[1].

式(1)中,Cij表示蛋白质网络邻接矩阵中节点j到节点i的对应元素值.节点的度是最简单、最直观的中心性测度,当节点度越大时,反映出该节点参与的相互作用越多,则该节点对于整个蛋白质网络产生的影响也越大.
1.2 接近度中心性
节点的接近度中心性(Closeness Centrality,CC)通过衡量该节点对于其他节点的依赖程度,反映该节点在整个网络中所处的中心程度[2],接近度中心性定义如下

1.3 介数中心性
节点的介数中心性(Betweenness Centrality,BC)指的是蛋白质网络中经过某个节点v的最短路径在所有最短路径数中所占有的比例[3],公式如下


1.4 特征向量中心性


2 节点重要性评估及算法描述
2.1 节点重要性评估
在蛋白质网络中,可以作这样的假设,分化前细胞中的蛋白质的部分功能会被分化后细胞中的蛋白质继承过去,会造成前者的重要性的部分减弱,后者的重要性相应的增强.在具体网络中,后者的重要性之和可以用它们之间连线的多少来衡量,如果这些蛋白质节点对之间的部分直接连接,则前者按照比例减少部分重要性;如果所有节点对之间都存在着直接连接,则可以完全替代前者的作用,前者的重要性会降到零.因此在蛋白质网络中,节点的重要性可以用公式(5)来表示.

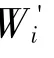
2.2 算法描述

算法描述如下:

3 实验及结果分析
实验选取酵母蛋白质网络作为测试对象,该测试数据集包含2 361个节点和7 182对相互作用[5],将Essential Proteins数据集作为关键蛋白质标准数据集,该标准数据集是收集和整理4个常用的生物数据库(MIPS[6]、SGD[7]、DEG[8]、SGDP[9])的数据得来的,包含有1 285个关键蛋白质.首先对Yeast数据集进行预处理,去除数据集中蛋白质的自相互作用和冗余相互作用,得到该网络的实验数据.之后,使用4种测度算法(度中心性DC、接近度中心CC、介数中心性BC和特征向量中心性EC)和本算法分别对于实验所选取的Yeast数据集进行关键蛋白质识别.5种不同的算法得到不同的蛋白质节点的重要性排序,每一个算法得到的结果按照降序排列,选择不同规模的蛋白质节点作为预测关键蛋白质,与关键蛋白质标准数据集Essential Proteins进行对比,从而发掘该算法在不同规模下的预测准确率.
5种不同算法分别针对Yeast数据集进行节点重要性评价,选取TOP 1%的蛋白质节点,即前24个蛋白质节点,其编号如表1所示.
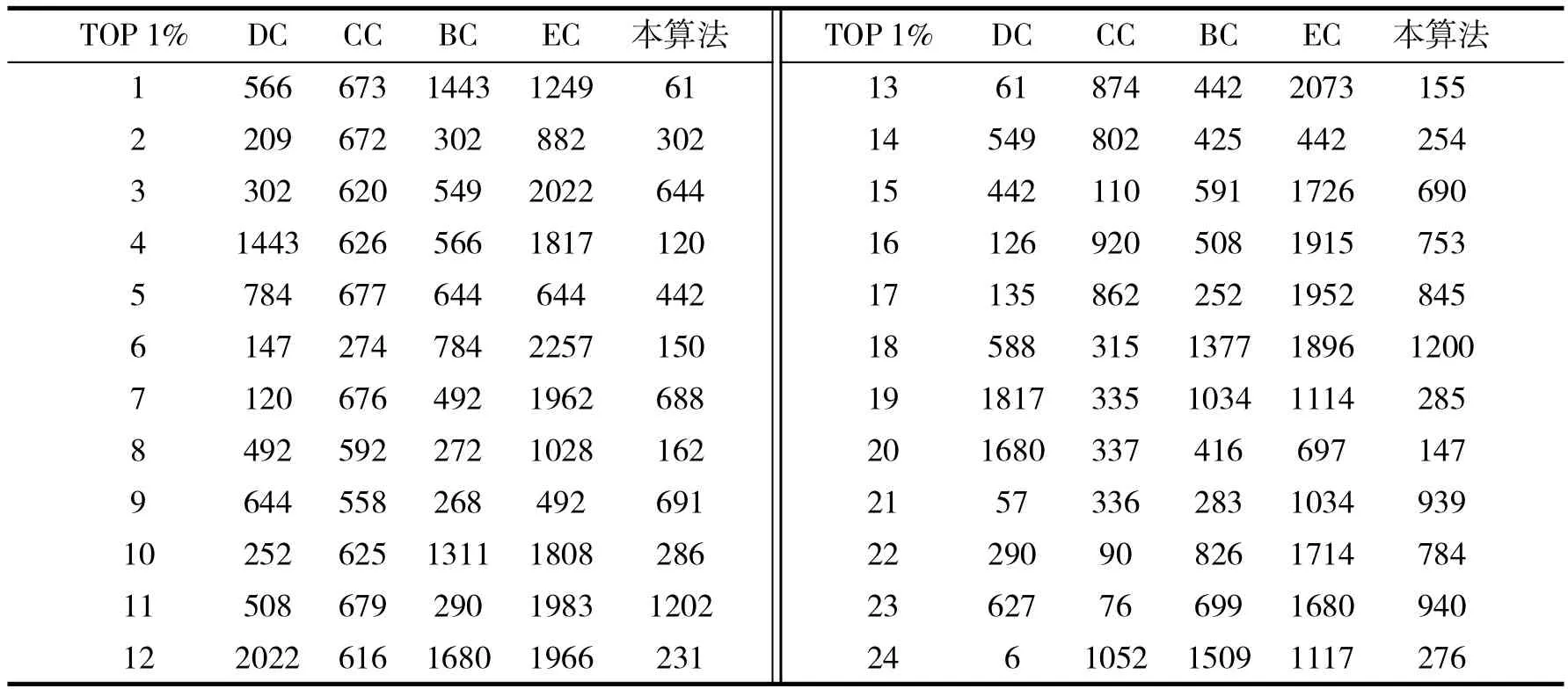
表1 DC、CC、BC、EC和本算法预测得到的TOP 1%蛋白质节点编号Tab.1 Top 1%protein nodes predicted by DC,CC,BC,EC and the algorithm
在表1中,将5种不同算法得到的前24个蛋白质节点编号与关键蛋白质标准集进行匹配,得到5种算法的预测准确率分别为62.5%、41.67%、50%、33.3%、50%,本算法的预测准确率略低于DC,和BC的准确率相同,同时高于CC、EC.5种不同算法预测得到的前100个关键蛋白质的交集见表2.

表2 依据不同算法得到的前100个预测关键蛋白质交集的预测准确率Tab.2 The intersection of Top 100 essential proteins predicted by different algorithms%
由表2可知,5种算法预测的前100个蛋白质与标准关键蛋白质集进行比较,预测准确率分别为52%、48%、48%、43%、56%.本算法预测准确率高于其他4个算法,分别高出了4、8、8、13个百分点.且本算法的初步设想是兼顾蛋白质节点在蛋白质网络中表现出的局部和全局两方面的特性进行关键蛋白质识别,根据蛋白质网络局部特性识别关键蛋白质的诸多算法中,DC、BC算法是基于局部特性的经典算法,DC算法度量的是节点参与相互作用的数量,BC算法度量的是节点保持网络紧密连接的能力,并反映该蛋白质节点是否处在蛋白质网络的枢纽位置.在表2中,可以发现本算法与DC、BC算法得到蛋白质的重合率有所提高,反映出本算法在兼顾蛋白质网络的局部特性方面有所提高,完成了本实验的初始设想.5种算法分别得到的蛋白质个数见图1,纵坐标表示各个算法识别出的关键蛋白质的个数,图1-a~图1-e表示由各个算法得到前24、120、240、360、480个蛋白质.

图1 由DC、CC、BC、EC和本算法分别得到的关键蛋白质个数Fig.1 The number of essential proteins detected by DC,CC,BC,EC and the Algorithm
由图1可知,本算法在以上5个规模的样本上预测准确率分别为50%、55.8%、52.9%、54.1%、53.1%.在TOP 5%、TOP 10%、TOP 15%、TOP 20%数据中,本算法的预测准确率均高于其他4种算法.一些研究表明有效的挖掘重要性前20%的数据对于准确预测关键蛋白质是非常重要的[10],可见本算法在识别关键蛋白质方面是可行的.
4 小结
本文在蛋白质节点失效算法的基础上,结合蛋白质网络节点的关联性,提出基于节点关联性的关键蛋白质识别算法.在保存蛋白质网络拓扑结构完整性基础上,兼顾了蛋白质网络局部特性,改进了网络中蛋白质节点重要性的评价标准,该标准反映了生物细胞分化时蛋白质节点的功能传递这一过程.实验结果表明本算法对于关键蛋白质的识别具有比较好的效果.
[1]Jeong H,Mason S P,Barabási A L,et al.Lethality and centrality in protein networks[J].Nature,2001,411(6833):41-42.
[2]Wuchty S,Stadler P F.Centers of complex networks[J].Journal of Theoretical Biology,2003,223(1):45-53.
[3]Wuchty S.Interaction and domain networks of yeast[J].Proteomics,2002,2(12):1715-1723.
[4]Bonacich P.Power and centrality:A family of measures[J].The American Journal of Sociology,1987,92(5):1170-1182.
[5]Pajek.Protein-protein interaction network in budding yeast[DB/OL].(2003-07-25)[2015-12-12].http://vlado.fmf.uni-lj. si/pub/networks/data/bio/Yeast/Yeast.htm
[6]Mewes H W,Frishman D,Mayer K F X,et al.MIPS:analysis and annotation of proteins from whole genomes in 2005[J].Nucleic Acids Research,2006,34(Database issue):D169-D172.
[7]Cherry J M,Adler C,Ball C,et al.SGD:Saccharomyces Genome Database[J].Nucleic Acids Research,1998,26(1):73-79.
[8]Zhang R,Lin Y.DEG 5.0,a data base of essential genes in both prokaryotes and eukaryotes[J].Nucleic Acids Research,2009,37(Database issue):D455-D458.
[9]Saccharomyces genome deletion project[DB/OL].[2015-12-12].http://www-sequence.stanford.edu/group/yeast_deletion_project.
[10]Estrada E.Virtual identification of essential proteins within the protein interaction network of yeast[J].Proteomics,2006,6(1):35-40.
(责任编辑:卢奇)
Identification of essential proteins based on network node correlation
XU Rui,LI Linfang,BAI Linfeng
(Henan Institute of Science and Technology,Xinxiang 453003,China)
To improve the efficiency of identification of essential proteins in protein networks,a new method for identification of essential proteins was proposed based on network node correlation.This method considers the local characteristics of protein networks on the basis of maintaining the topological structure of protein network,measure the importance of protein nodes from the perspective of protein node correlation and use the invalidation of protein node method to identify essential proteins in protein networks.Finally,the validity of the proposed method was verified by experiments.
node correlation;protein networks;essential protein;node importance
TP301.6
A
1008-7516(2016)02-0068-05
10.3969/j.issn.1008-7516.2016.02.016
2016-01-09
许睿(1987—),男,河南新乡人,硕士,助教.主要从事数据挖掘和生物信息学研究.

