基因组测序技术及其临床应用研究进展
梁振山综述,曾令兵,万腊根审校(1、南昌大学医学院,江西南昌0006;2、江西省儿童医院检验科,江西南昌0006;、南昌大学第一附属医院检验科,江西南昌0006)
基因组测序技术及其临床应用研究进展
梁振山1,2综述,曾令兵3,万腊根3审校
(1、南昌大学医学院,江西南昌330006;2、江西省儿童医院检验科,江西南昌330006;3、南昌大学第一附属医院检验科,江西南昌330006)
摘要:目前,随着全基因组测序技术的进一步发展,基因组测序技术在临床与科研中越来越发挥着重要作用。近几年来,基因组测序技术迅速发展,其在愈来愈受到学者们的关注。本文旨在对基因组及、基因组测序技术及其在临床诊断中的应用做一综述。
关键词:基因组;基因序列;基因测序技术
1 基因组及基因测序技术的进展
从分子生物学角度来讲,双链螺旋结构的DNA分子控制每个生物个体的遗传特性。基因组在生物学中的定义,是指包含该生物体中所有的DNA(有些病毒是RNA)中的全部遗传信息,也即是一个生物体中完整的染色体DNA序列。顾名思义,微生物的基因组,即是指微生物染色体中的所有DNA序列。基因组学,也即是研究基因组构成的一门学科,它兴起于上世纪90年代初的人类基因组计划。在人类基因组计划实施的同时,人们也启动了微生物的基因组研究计划。1995年,Fleischmann研究小组率先发布了第一株微生物的全基因图谱。他们采用全基因组随机测序法(即wholegenome shotgun sequencing,简称“鸟枪测序法”),对流感嗜血杆菌(Haemophilus influenzae)的全基因组进行了较为全面的分析,获得了第一株微生物的全基因组数据,这在细菌基因组测序的历史上属首次[1]。此后,随着测序技术的发展,越来越多的临床微生物(或者称为病原微生物)、模式微生物、环境微生物、工业微生物等都被测序,并获得全基因组数据[2-7]。近年来,完成的基因组(包括微生物基因组在内)呈现几何级数增长,这不仅为探索基因本质提供了大量的数据,也为后基因组时代的到来奠定了坚实的基础。
2 基因组测序技术的研究进展
Sanger测序是最早的基因测序技术,被称为第一代测序技术,它是由Fredrick Sanger在1975年发明的。该测序方法又称为“双脱氧链终止法”[8]。其基本原理是在聚合酶链反应(Polymerase Chain Reaction,PCR)的过程中,不断地在引物的3’端加入dNTP,使得模板得以延伸,最终得到新的模板。一旦在合成模板链的过程中掺和进的是双脱氧核苷酸(ddNTP)。由于ddNTP的3’端缺少羟基,无法再继续延伸,因此PCR反应至此位点终止。最终,PCR的产物将形成一系列由5’-3’ddNTP组成的、长短不一的DNA片段。采用变性的聚丙烯酰胺凝胶电泳,分离这些DNA片段。并借助放射自显影技术检测这些DNA片段,就可以获得相应的序列信息。除Sanger测序外,这个时期还出现了一系列其他的测序方法,如焦磷酸测序法(二代测序技术中被Roche公司所采用)、链接酶法等[9-11]。但是,他们的共同核心都是借助了Sanger的可中断PCR反应的ddNTP作为底物。
Sanger测序法的优势是其测序读长能够达到1,000bp,并且测序的准确率高达99.999%。但是缺点是测序成本高、通量低,会严重影响全基因组测序的效率,非常有必要开发新的全基因组测序技术。
二代测序技术主要由Roche、Illumina和ABI三个公司主导分别推出了454、Solexa和Solid测序平台。二代测序技术是在Sanger测序的基础上进行改良[12]。首先进行预扩增:未标记的dNTP和普通Taq酶进行固相桥式PCR扩增,单链待测片段通过PCR生成双链片段。高温变性后解链得到互补的单链,固定在附近的固相表面,经过PCR循环后,可以得到大量的待测序单链片段。常用的固相有磁珠,带有待测片段的磁珠在PTP的平板上反应,PTP平板上有许多孔,每个孔只能容下单个磁珠反应,然后在反应环境中加入四种不同颜色荧光标记的dNTP以及聚合酶进行PCR扩增,通过捕捉四种不同荧光信号,换算成信号峰值强度,再通过计算机软件计算测序数值。相对于Sanger测序,二代测序技术最大的特点是实现了高通量,同时高通量测序一次可以读取10~100万条序列。在很大程度上降低了测序的成本,获得大规模的基因组数据就成为了可能。但是,二代测序技术也存在较多的缺陷,其最大的一个不足就是测序片段较短,从而会对全基因组的后续分析造成较大的困难,导致测序之后出现较多的contigs,很难在短时间内拼接完成。对于一些GC含量较为极端的物种,就会出现大量contigs,导致难以拼接至染色体上,进而严重影响测序的进度。而解决这种困难的途径,就需要构建一系列的DNA文库,进行重测序工作,这同时也增大了工作量。见表1。
近两三年以来,基因组测序技术取得了非常大的进步。其中以PacificBio公司的SMRT和OxfordNanopore technologies的纳米孔技术最为突出,也被称之为第三代测序技术[13,14]。第三代测序技术又称之为“单分子测序技术”,与二代测序技术相比,其不需要经过PCR扩增,并且可以实现超长读长。第三代基因组测序的基本原理是:基因组模板与DNA聚合酶结合之后,标有4种不同颜色荧光的dNTP,在碱基配对阶段不断地加入,检测器可以通过检测不同的荧光来判断碱基序列。而与基因组模板结合的DNA聚合酶是实现三代测序超长读长的重要因素之一。DNA聚合酶的活性直接影响到最终测序的读长。而聚合酶的活性受到众多因素的影响,包括测序前样本的处理(其中是否含有酶的螯合剂、蛋白、RNA等)、上机测试是激光对酶活性的影响等等。因此,第三代测序技术对于基因组样本质量要求较高。在基因组样本制备的过程中,不能含有过高的蛋白,同时也不能含有聚合酶的抑制剂等污染物。通常,三代测序前都需要对基因组样本质量进行测定,以确保后续实验能够顺利地进行。第三代测序技术最大的优势,在于其超长的读长(有时可以超过3,000bp),但是其最大的缺陷是测序的错误率较高。测序速度较快的SMRT技术的错误率可以达到15%,但好在错误率的出现是随机性的,并不像二代测序技术具有偏向性。
综上所述,每一种全基因组测序技术都有其优势及缺陷。当今,为了快速而准确地获得全基因组数据,我们就需要结合不同的测序方法,发挥各种测序技术的优势,规避劣势;从而能够准确而快速的完成测序工作。
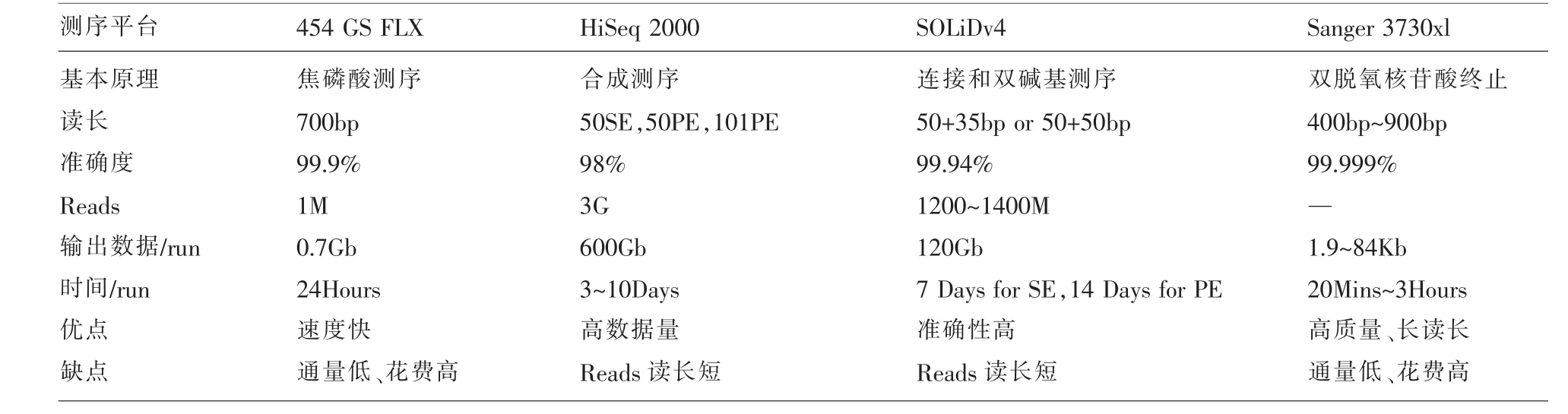
表1 各种二代测序方法及平台的比较
3 基因组测序技术在临床中的应用
3.1临床微生物诊断以及微生物耐药监测传统的微生物培养加药敏通常需耗时一周左右,基因组测序可以在短时间内完成微生物的鉴定及药敏,为临床及时提供用药指导,另外还可以克服药敏鉴定仪种数据库不完整和过时的缺点,避免误诊。
此外,基因组测序技术还可以为临床微生物提供可靠的流行病学资料,对于医院院内感染的控制和预防具有非常重要的作用。2012年,在The New England Journal of Medicine上发表了一篇关于新生儿感染MRSA(耐甲氧西林金黄色葡萄球菌)的全基因组报道[3]。
基因组测序技术不仅可以运用在临床微生物的鉴定,同时也可以用于临床微生物的药敏结果检测。例如多重耐药的肺结核(简称“MTB”)和人类免疫缺陷病毒(简称“HIV”)的药敏检测,已经能够通过基因组测序技术进行快速、准确地检测。2013年,Jacqueline等在New England Journal发表了通过全基因组测序的方法,鉴定多重耐药肺结核分枝杆菌的耐药情况[15]。他们完成全基因组测序之后,发现分离得到的肺结核分枝杆菌基因组中共包含了39个耐药基因,而标准化实验室只报道了对其中的9种药物耐药。后期的表型实验证实,该株肺结核分枝杆菌对amikacin,capreomycin,clofazimine和linezolid敏感。基因组数据与这一表型完全符合。此外,通过基因组测序,该研究小组还在amithiozone,gatifloxacin,levofloxacin,rifapentine 和rifabutin的作用位点中发现了突变,而这几类药物并没有包含在标准化实验室的报告中。因此,借助基因组测序技术,可以更加完整、快速、准确地了解临床微生物的耐药情况,为临床治疗选择合适的药物提供重要的支持[16],很多报道将全基因组测序技术运用到临床微生物的诊断[3,4,7,17,18],为临床微生物的诊断和治疗提供重要依据。
3.2基因组测序技术在遗传病和产前诊断中的应用。染色体异常检测[19-21]:在基因测序出现之前,临床上针对遗传病主要是染色体培养,镜检染色带型来诊断。染色体培养耗时长,效率低,检测范围有限。在测序技术出现后,2007年Korbel[22]等提出一种新的高通量分析方法,基因组配对对比法,首先将染色体打碎成基因片段,PCR扩增后进行基因组测序,得到整个基因组图谱后与人类基因组参考图片配对比对,可发现例如倒置,方向错误,序列长度不等等1000多个结构变异。在临床中,染色体结构变异的常见类型有缺失、重复、倒位和易位4种,如猫叫综合征,女性习惯性流产,可以导致慢性粒细胞白血病等;同时在临床中还有许多遗传病是因为基因点突变导致的。而通过传统遗传学染色异常来诊断是很受限制的,例如β-地中海贫血,其基因含1个41~42位点突变基因杂合子,传统遗传学方法敏感性很低,但是通过高通量测序则可以逐一检测出异常位点,及时做出准确的诊断。畸形胎儿一直困扰广大孕妇和医生,虽然可以通过脐带血或者羊水的染色体带型来诊断一部分,但是这种传统方法非常局限,而且具有较大风险,采用新一代高通量基因测序技术的基因芯片可准确检测胎儿遗传异常。翁惠男,梁嘉颖等[23]回顾了1800多例孕期12周以上的孕妇,发现无创产前基因测序高危孕妇,再通过脐带血或者羊水细胞培养染色体分型(21-三体综合征、18-三体综合征、13-三体综合征)。发现21-三体综合征的敏感性为100%,准确性为92.9%。18-三体综合征和13-三体综合征的敏感性及准确性均为100%。无创产前胎儿基因检测可提高产前诊断效率,其敏感性准确性与染色体带型分析技术高度一致,可减少畸形胎儿的出生,且安全,较介入性产前诊断易于接受,具有较高的临床实际应用价值[24]。
3.3基因测序技术与个性化医疗。个性化医疗即指精准医疗—基于个人基因组信息,为患者量身制定治疗方案,是让效果最大化而副作用最小化的最佳选择。1.药物基因组学,根据基因组来选择最佳药物种类和剂量,在保证安全的前提下达到最佳治疗效果[25-28]。范宇飞等[29],对25例肿瘤患者进行基因检测,执行个体化医疗,对比对照组发现实施个体化治可提高有效率,延长患者生存期。2.肿瘤及肿瘤遗传学,目前对肿瘤的诊断,治疗效果评价以及预后检测都是通过测定血清中肿瘤标志物[21]。包括甲胎蛋白、癌胚抗原、同工酶、激素(人绒毛膜促性腺激素)、组织特异性抗原(前列腺特异性抗原、游离前列腺特异抗原)、黏蛋白、糖蛋白、糖脂(CAl25)的等。然而最新的基因测序技术可以捕获肿瘤基因[30],及时做出诊断,甚至预判肿瘤的发生,及时作出预防和早期干预。好莱坞知名女星安吉丽娜?朱莉通过测序技术确定带有乳腺癌基因1号(BRCA1号)基因[21,32],估测分别有87%和50%的概率患上乳腺癌和卵巢癌,结合她母亲50岁左右因乳腺癌去世的家族病史,她接受了预防性的切除术以降低罹癌风险。基于基因测序技术的个性化医疗可精准诊断,降低分险,提供更加安全高效的治疗方案。
4 展望
随着全基因组测序技术的进一步发展,测序成本的进一步降低,将全基因组测序引入临床诊断也将逐渐成为现实。一方面,不仅可以快速地诊断出一些常见及不常见的疾病;还可以预测许多疾病,通过早期干预和预防,降低发病率。另一方面,随着技术的愈发成熟,测序技术从实验室慢慢走向临床,风靡全球,市场潜力巨大,但是政府部门却缺少有效监管和测序实验室的准入制度,甚至没有质量控制体系,2014年2月9号,中国国家食品药品监管总局、国家卫生与计划生育委员会联合发布(25号文件)“临床使用基因测序相关产品和技术管理的通知”,要求停止一切商用基因测序产品[33]。近期,政府已出台基因测序相关政策,未来在政府有效监管和质量监控、技术准入制度的进一步完善下,基因测序技术会飞速发展,为临床提供不可限量的帮助。
参考文献
[1]Fleischmann RD,Adams MD,White O,et al.Whole-genome random sequencing and assembly of Haemophilus influenzae Rd[J]. Science,1995,269(5223):496-512.
[2]Koser CU,Ellington MJ,Cartwrigh EJP,et al.Routine use of microbial whole genome sequencing in diagnostic and public health microbiology[J].PLos pathogens,2012,8(8):464-470.
[3]Koser CU,Holden MTG,Ellington MJ,et al.Rapid whole-genome sequencing for investigation of a neonatal MRSA outbreak[J].New England journal ofmedicine,2012,366(24):2267-2275.
[4]Rasko DA,Webster DR,Sahl JW,et al.Origins of the E.coli strain causing an outbreak of hemolytic-uremic syndrome in Germany[J]. New England journal ofmedicine,2011,365(8):709-717.
[5]Rasko DA,Worsham PL,Abshire TG,et al.Bacillus anthracis comparative genome analysis in supportof the Amerithrax investigation [J].Proceedings of the National Academy of Sciences,2011,108 (12):5027-5032.
[6]Snitkin ES,Zelazny AM,Thomas PJ,et al.Tracking a hospital outbreak of carbapenem-resistant Klebsiella pneumoniae with wholegenome sequencing[J].Science translational medicine,2012,4 (148):148ra116-148ra116.
[7]Gardy JL,Johnston JC,Sui SJH,et al.Whole-genome sequencing and social-network analysis of a tuberculosis outbreak[J].New England journal ofmedicine,2011,364(8):730-739.
[8]Sanger F,Nicklen S,Coulson AR.DNA Sequencing with Chain-Terminating Inhibitors[J].Biotechnology,1992,24(12):104-108.
[9]Mardis ER.Next-generation DNA sequencingmethods[J].Annual Review of Genomics&Human Genetics,2008,9(9):387-402
[10]Shendure J,Ji H.Next-generation DNA sequencing[J].Nature Biotechnology,2008,26(10):1135-1145.
[11]Metzker ML.Sequencing technologies-the next generation[J].Nature Reviews Genetics,2010,11(11):1-13.
[12]刘莉杨,杨学工.高通量测序技术在临床医学中的应用进展[J].医学综述,2013,19(16):2971-2973.
[13]Rothberg JM,Wolfgang H,Rearick TM,et al.An integrated semiconductor device enabling non-optical genome sequencing[J].Nature,2011,475(7356):348-352.
[14]Levene MJ,Korlach J,Turner SW,et al.Zero-mode waveguides for single-molecule analysis at high concentrations[J].Science,2003,299(5607):682-686.
[15]Koser CU,Bryant JM,Becq J,et al.Whole-genome sequencing for rapid susceptibility testing of M.tuberculosis[J].New England journal ofmedicine,2013,369(3):290-292.
[16]吴琴,邹义春,罗卓跃,等.铜绿假单胞菌耐药基因的检测与分析[J].实验与检验医学,2009,27(05):478-480
[17]Hall N.Advanced sequencing technologies and their wider impact in microbiology[J].The Journal of experimental biology,2007,210 (9):1518-1525.
[18]Medini D,Serruto D,Parkhill J,et al.Microbiology in the postgenomic era[J].Nature reviewsMicrobiology,2008,6(6):419-430.
[19]陈丹,刘根焰,徐建,等.基因测序及时及临床应用[J].中华实验室管理电子杂志,2014,2[4]:19-23.
[20]姚婷,向萍霞,胡雁婻,等.孕妇血浆游离胎儿DNA检测在产前诊断中的应用[J].实用医学杂志,2014,30(8):1248-1250.
[21]Chiu RWK,Lo YMD.Clinical applications ofmaternal plasma fetal DNA analysis translating the fruits of 15 years of research[J]. Clinical Chemistry&Laboratory Medicine Cclm,2013,51(1):197-204.
[22]Korbel JO,Urban AE,Affourtit JP,et al.Paired-end mapping reveals extensive structural variation in the human genome[J].Science,2007,19(5849):420-426.
[23]翁慧男,梁嘉颖,曾伟宏,等.无创产前基因测序在胎儿染色体非整倍体基因检测中的临床应用[J].国际医学检验杂志,2015,(16):2386-2388
[24]杨昕,廖灿,李东至,等.无创性产前诊断技术在产前诊断中的应用前景预测-233例染色体病胎儿病例回顾分析[J].中国产前诊断杂志(电子版),2012,4(04):6-9.
[25]于磊,张阳德.合成生物学与个性化肿瘤治疗[J].中国现代医学杂志,2013,(14):58-62.
[26]李晓芬,袁瑛.基因检测指导下的晚期结直肠癌的个体化治疗[J].肿瘤防治研究,2015,42,(09).
[27]朱铁峰,崔贵,景凯,等.ERCC1、RRM1、TYMS、TUBB3表达对晚期非小细胞肺癌个体化治疗的应用分析[J].中国临床研究,2015,28(06):736-739.
[28]周宏灏,刘洁.抗肿瘤药物的基因导向性个体化治疗[J].肿瘤药学,2011,(1):6-11.
[29]范宇飞,任东,秦苑,等.基于基因检测的个体化化疗方案治疗晚期肿瘤[J].肿瘤研究与临床,2014,11(26):763-766.
[30]韩露,马培,周新.基因诊断在乳腺癌个体化医疗中的应用[J].中国医药导报,2015,(12):39-42.
[31]Li SY.The Human Cancer Genome Laboeratory Analysis and Clinical Application[J].Laboratory Medicine,2014,29(5):414-427.
[32]Borzekowski DL,Guan Y,Smith KC,et al.The Angelina effect:immediate reach,grasp,and impactof going public[J].Genetics in Medicine Official Journal of the American College of Medical Genetics,2014,16(7):516-521.
[33]国家食品药品监督管理总局,国家卫生和计划计生育委员会.关于加强临床使用基因测序相关产品和技术管理的通知.2014-02-09.
·讲座·
(收稿日期2015-11-30;修回日期2016-01-10)
通信作者:万腊根,男,1964年12月生,主任技师,研究方向:从事临床检验诊断及分子生物学,邮箱:wlgme196412@126.com
作者简介:梁振山,男,1984年8月生,在读硕士研究生,检验师,研究方向:从事临床检验诊断及分子生物学。
DOI:10.3969/j.issn.1674-1129.2016.01.016
中图分类号:R446.62,Q751
文献标识码:A
文章编号:1674-1129(2016)01-0048-04

