棘白菌素B脱酰基酶工程菌的构建及酶学性质研究
邹树平,廖思行,牛 坤,郑裕国,沈寅初
(浙江工业大学 生物与环境工程学院,浙江 杭州 310014)
棘白菌素B脱酰基酶工程菌的构建及酶学性质研究
邹树平,廖思行,牛坤,郑裕国,沈寅初
(浙江工业大学 生物与环境工程学院,浙江 杭州 310014)
摘要:棘白菌素B(ECB)脱酰基酶能选择性催化水解脱去ECB酰基侧链,得到ECB母核,可用于合成新型抗真菌药物阿尼芬净.从实验室保藏的犹他游动放线菌(Actinoplanes utahensis)ZJB-0801基因组中克隆到ECB脱酰基酶基因,并将其构建入表达质粒pSET152,通过接合转移的方法将表达质粒导入到天蓝色链霉菌(Streptomyces coelicolor)A3中,构建可以高效表达ECB脱酰基酶的工程菌.对重组ECB脱酰基酶的酶学性质进行了考察,结果表明:重组酶的最适反应温度为45 ℃,最适pH为7.5.Zn2+,Mg2+,Mn2+,Fe2+对重组酶有显著的激活作用,而Cu2+,Co2+具有显著地抑制作用.动力学参数分析表明:该酶对底物ECB的Km和Vmax分别为0.356 mmol/L和397 μmol/(min·mg).
关键词:棘白菌素B脱酰基酶;天蓝色链霉菌;重组表达;酶学性质
近年来,由于免疫低下患者逐年增多,真菌感染发病率明显升高,尤其是深部真菌感染的发病率和病死率显著增加[1-2].棘白菌素(Echinocandins)类抗真菌药物能够非竞争性地抑制真菌细胞壁β-1,3-葡聚糖合成酶的活性,是临床治疗系统性真菌感染的新一代抗真菌药物[3].与传统抗真菌药物相比,该类药物具有作用机制独特,毒副作用低,且对一些唑类和两性霉素B耐药的真菌具有很强的抗菌活性,是目前临床治疗深部真菌感染的“王牌抗生素”[4].目前,FDA已批准的棘白菌素类新药有卡泊芬净(默沙东,2001年)、米卡芬净(藤泽药业,2005年)和阿尼芬净(辉瑞,2006年)[5].
棘白菌素B(Echinocandins B, ECB)是由构巢曲霉(Aspergillusnidulans)发酵产生的一种次级代谢产物,是合成抗真菌药物阿尼芬净的前体化合物.ECB脱酰基酶能选择性催化水解ECB侧链酰胺键生成ECB母核,是阿尼芬净合成过程的关键酶[6].目前,已报道的ECB脱酰基酶主要来源于犹他游动放线菌及链霉菌属[7-10],但野生酶普遍存在表达产量低,活性不高的问题.为了满足日益增长的生产需求,有关ECB脱酰基酶高效表达系统的构建逐渐引起了人们的关注.Kreuzman等[11]将源于游动犹他放线菌的ECB脱酰基酶基因在S.lividansTK24中重组表达,获得的重组酶活性比野生酶提高了4.1倍;Satoshi等[12]将FR901379脱酰基酶基因在S.lividans1236中重组表达后,酶活提高了250倍;Inokoshi等[13]比较了不同的链霉菌表达系统对ECB脱酰基酶表达的影响,发现不同表达系统中酶的表达量及活性均存在显著差异.天蓝色链霉菌作为链霉菌表达系统中现已研究最完善的宿主菌之一,具有遗传稳定性高,胞外分泌能力强,能够正确表达高GC%的基因片段等优点,是理想的链霉菌表达宿主[14].根据数据库序列设计引物,从实验室保存的犹他游动放线菌基因组DNA中克隆获得了ECB脱酰基酶基因,构建了该酶的天蓝色链霉菌工程菌,并进一步分析了其酶学性质,旨在为ECB脱酰基酶高效表达及产业化生产提供理论指导.
1材料与方法
1.1 材 料
游动犹他放线菌ActinoplanesutahensisZJB-0801,天蓝色链霉菌StreptomycescoelicolorA3,链霉菌质粒pSET152菌来自于实验室保藏.Primix和DNA标准分子量为Takara公司产品,限制性内切酶EcoRⅠ,XbaⅠ,T4DNA连接酶均为Thermo公司产品,质粒提取试剂盒为大连宝生工生物技术有限公司产品,基因组提取试剂盒为MP公司产品.BCA蛋白质定量试剂盒为北京康为生物科技有限公司产品.PCR仪(Techne公司);多功能酶标仪(美国分子仪器公司);高效液相色谱仪LC-10TSPD-10A(日本岛津公司生产).
1.2 重组质粒pSET152-ecbD的构建
编码ECB脱酰基酶基因片段通过下列引物由犹他游动放线菌中扩增获得.正向引物为5′-AAAACTAGATCTTGTTGGTCGAGAGGAAAG
ATC-3′;反向引物为5′-AAACCGCGGTGGCAGGTCCATGGGC-3′(Xha Ⅰ和EcoR Ⅰ酶切位点用下划线标注).50 μL反应体系中含有50 ng犹他游动放线菌A.utahensisZJB-0801基因组,引物各200 ng,Primix 25 μL.PCR扩增程序如下:98 ℃预变性5 min,30次扩增循环(98 ℃×15 s,58 ℃×30 s,72 ℃×5 min),72 ℃延伸10 min.琼脂糖凝胶电泳回收扩增片段,用XhaⅠ和EcoRⅠ双酶切.酶切产物与双酶切的pSET152载体通过T4连接酶进行连接,将连接产物转化到大肠杆菌JM109中,阳性克隆序列分析由上海桑尼生物科技有限公司完成.序列正确的质粒即获得表达质粒pSET152-ecbD.
1.3 重组S. coelicolorA3/pSET152-ecbD的构建
将重组表达质粒pSET152-ecbD导入E.coliET12567/pUZ8002,E.coliET12567/pUZ8002/pSET152-ecbD作为供体菌,培养至OD600为0.4~0.6,离心收集菌体,用新鲜的2×YT培养基悬浮备用.刮取新鲜的S.coelicolorA3孢子,用2×YT打散悬浮,50 ℃水浴热激处理10 min,置于37 ℃的摇床,预萌发3 h直至长出芽管(或不处理),将处理好的大肠杆菌和孢子悬液混合,稀释100倍后涂布于含有10 mmol/L的MgCl2的MS平板,28 ℃培养,待培养至24 h,用含有50 μg/mL萘啶酮酸和安普霉素的1 mL水溶液均匀覆盖平板表面进行抗生素覆盖.将平板继续置于28 ℃培养7~10 d,直至菌落面积稳定.
1.4 重组S.coelicolorA3/pSET152-ecbD的复选
用切割器将选择性平板上的转化子菌落切成直径为1 cm的菌块,转接到带抗性的MS平板上,30 ℃培养7~10 d,定时观察并记录生长直径.从中选出生长速率快、菌落直径大的单菌落转化子.筛选得到的阳性接合转移子接种于含有抗生素的液体TSBY培养基(TSB质量分数为3%,酵母提取物质量分数为0.25%,蔗糖质量分数为37%)中培养48 h,收集菌丝体,提取总DNA作为模板,以pSET152-ecbD质粒为阳性对照,以S.coelicolorA3的总DNA为阴性对照,利用1.2的引物,进行PCR扩增验证.PCR条件反应程序为98 ℃预变性5 min,30次扩增循环(98 ℃×15 s,58 ℃×30 s,72 ℃×255 s),72 ℃延伸10 min.
1.5 重组ECB脱酰基酶在S. coelicolorA3中表达
将重组菌株S.coelicolorA3/pSET152-ecbD链霉菌孢子接种于50 mL含有安普霉素(50 μg/mL)的种子培养基TSBY中,于30 ℃摇床(转速200 r/min)培养24 h后,按5%的接种量转接50 mL链霉菌发酵培养基:葡萄糖2.5%,黄豆饼粉1%,CaCO30.3%,NaCl 0.3%,(50 μg/mL安普霉素),30 ℃摇床(转速200 r/min),培养3 d.
1.6 ECB脱酰基酶酶活检测
离心取发酵上清液,加入ECB(200 μg/mL),KH2PO4(0.05 mol/L,pH=6.5),助溶剂乙醇(体积比为5%),35 ℃,200 r/min反应1 h,反应结束后加入乙酸(体积比2%)终止反应.取样1 mL,HPLC检测产物ECB母核生成量.酶活的定义为一个酶活力单位为在标准反应条件下每分钟内由底物ECB生成1 μmol产物ECB母核所需的酶量.用BAC总蛋白定量试剂盒检测蛋白质量分数.
1.7 HPLC检测条件
采用高效液相色谱仪LC-10TSPD-10A(日本岛津公司生产),依利特C18柱Hypersil ODS2 5 μm(4.6 mm×250 mm);检测波长220 nm,流速1 mL/min,柱温40 ℃,进样量:20 μL,流动相:V(乙腈)∶V(质量比0.2%的乙酸铵水溶液)=5∶95,出峰时间:10.7 min.
2结果与分析
2.1 重组S. coelicolorA3/pSET152-ecbD的构建
重组质粒pSET152-ecbD大小共9.8 kb,含有大小4.3 kb的ECB脱酰基酶基因,其中包含An抗性基因,促进异源基因在链霉菌中表达的ABC-转运系统基因和ermE*P强启动子(图1).该重组质粒通过大肠杆菌-链霉菌接合转移,导入S.coelicolorA3中.按照1.3的方法进行E.coliET12567/pUZ8002接合转移S.coelicolorA3,链霉菌孢子作为宿主.孢子经50 ℃萌发处理后,大肠杆菌和链霉菌孢子混合液涂布的MS平板上,菌落长出较为密集,在稀释100倍的平板上长出的菌落个数为257个,且生长速度较快,在培养2 d后有菌落长出,待培养8 d,菌落大小基本不变;而未经萌发处理的孢子,在稀释100倍的MS平板上长出的菌落为46个,在培养13 d后菌落生长缓慢,大小基本不变,说明虽然萌发处理孢子并非必须步骤,但是能够节省约5 d的培养时间,并提将转化效率提高5倍左右.

图1 含有ECB脱酰基酶基因的pSET152-ecbD质粒的构建Fig.1 Construction of expression plasmid pSET152-ecbD
2.2 转化子复筛
参照1.4实验方法,在MS培养基上对转化子进行复筛,从中筛选出4株生长速度较快,菌落直径较大,均在0.5 cm以上的菌落(表1).依次编号为1~4.经菌落PCR验证,从4株转化子的基因组中均可以扩增得到大小约4.3 kb的条带(图2),说明目的基因已经导入链霉菌宿主细胞中.
表1天蓝色链霉菌转化子在MS培养基上的生长情况
Table 1The growth of S. coelicolortransformants on MS screening media

转化子参数MS平板上的生长情况1)平均直径2)/cm0.1~0.30.3~0.50.5~0.8菌落数量/个102514比例/%39.719.81.6
注:1) 以黄豆饼粉为唯一碳源的制备培养基;2) 测量菌落直径,重复三次取平均值.
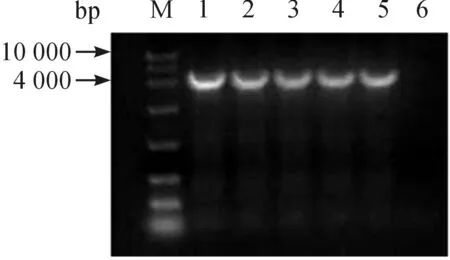
M—marker;1~4—转化子基因组中PCR获得的条带;5—质粒pSET152-ecbD中PCR获得的条带;6—S. coelicolor基因组中PCR获得的条带图2 天蓝色链霉菌中ECB脱酰基酶基因RCB检测Fig.2 Electrophoresis of ECB deacylase gene with 4.3 kb obtained by PCR from the chromosomal DNA of recombinant S.coelicolor
2.3 ECB脱酰基酶基因在天蓝色链霉菌中的表达
将复筛得到的4株转化子经种子培养,发酵培养.根据1.6的方法检测发酵液上清液酶活(图3).结果显示:筛选得到的4株转化子菌株显示了不同的ECB脱酰基酶活性,其中编号3菌株的酶活最高,酶活为336 mU/mL,与原始菌株相比,活性提高了2.8倍.Inokoshi等[10]将游动放线菌ECB脱酰基酶在S.lividansJT46中表达后,重组酶酶活力仅为31 mU/mL;Kreuzman等[11]构建的S.lividansTK24重组菌的酶活为360 mU/mL;Ueda等[13]将链霉菌来源的FR901379脱酰基酶基因在S.lividans1236中重组表达,酶活为14.7 U/mL.可以看出:S.coelicolorA3作为宿主菌,表达的ECB脱酰基酶是不同于其他宿主菌的.异源表达的重组蛋白活性往往受到多种因素的影响[15-17].

图3 不同天蓝色链霉菌转化子产重组ECB脱酰基酶活力比较Fig.3 Echinocandins B deacylase activity of different transformants
2.4 重组ECB脱酰基酶酶学性质研究
2.4.1重组ECB脱酰基酶的最适温度
在酶促反应中,温度对催化剂的活性和稳定性都会产生影响,同时温度还会对反应的平衡产生影响.测定了ECB脱酰基酶在20~70 ℃下转化ECB的活力.由图4可知:随着温度的升高,ECB脱酰基酶酶活逐渐升高,当温度达到45 ℃时,显示了最高酶活力,继续升高温度,酶活迅速降低,因此,确定其最适反应温度为45 ℃.Inokoshi等[13]将犹他游动放线菌来源的ECB脱酰基酶基因在S.lividansJT46中重组表达后,重组酶最适温度为60 ℃,Kreuzman等[11]构建的S.lividansTK24重组菌酶催化反应的最适温度为65 ℃;常青等[5]构建的S.lividans重组菌产ECB脱酰基酶,催化反应所选的温度为30 ℃;Satoshi等[12]将FR901379脱酰基酶基因在S.lividans1236中重组表达,催化反应所选的最适温度为50 ℃,说明不同宿主菌对表达ECB脱酰基酶有重要的影响.

图4 ECB脱酰基酶的最适温度和温度稳定性Fig.4 Optimal reaction temperature of echinocandins B deacylase
2.4.2重组ECB脱酰基酶的最适pH
在酶促反应中,相缓冲液的pH值不仅改变了反应中底物和带有电荷的酶的离子状态,还会对酶的催化活性产生影响[10].在pH为4.5~10.0的缓冲液中反应,测ECB脱酰基酶在不同pH下反应的酶活.由图5可知:随着pH的升高,ECB脱酰基酶酶活逐渐升高,当pH=7.5时,显示了最高酶活力,继续升高pH,酶活迅速降低,因此,该酶最适反应pH=7.5,Kreuzman等[11]报道的重组ECB脱酰基酶最适为pH=6.5;Satoshi等[12]报道的链霉菌来源的FR901379脱酰基酶最适pH为8~9;Inokoshi等[13]构建的重组S.lividansJT46,酶催化反应时的pH=7.通过比较可以发现:S.coelicolorA3-ecbD产的ECB脱酰基酶最适pH与现已报道的重组菌最适pH均不同.

图5 ECB脱酰基酶的最适pHFig.5 Optimal reaction pH of echinocandins B deacylase
2.4.3金属离子对重组ECB脱酰基酶活力的影响
据报道,某些一价、二价的金属离子对重组ECB脱酰基酶催化反应有促进作用[11].在1.5的反应体系中,加入常见的Ca2+,Mg2+,K+,Mn2+,Fe2+,Cu2+,Co2+,使其终浓度为0.25~0.5 mol/L,反应体系pH=7.5,在45 ℃,200 r/min转化1 h,反应结束后加入2%乙酸终止反应,测定不同金属离子作用下催化反应的酶活.结果表明(表2):Ca2+,Mg2+,K+,Mn2+,Fe2+对催化反应均有不同程度的促进作用,分别提高了36.2%,21.1%,43.6%,2.4%,31.9%,K+的促进作用最强;而Cu2+和Co2+对该酶有抑制作用,其中Co2+的抑制最强,相对酶活下降到了15.8%.Kreuzman等[11]研究也发现:K+对ECB脱酰基酶催化活性有显著的激活作用,当添加0.6 mol/L的K+时,其酶活提高约1.5倍.

表2 不同金属离子对催化反应的影响
2.4.4重组ECB脱酰基酶催化ECB水解反应的动力学参数
以ECB为底物,缓冲液为磷酸钠缓冲液(pH=7.5,0.05 mol/L),底物浓度分别为0.25,0.050,1.00,1.50,2.00,2.50,3.00,3.50,4.00,4.50,5.00 g/L.总反应体积都为20 mL.在45 ℃下反应60 min,加入200 μL乙酸溶液(0. 5 mol /L)终止反应,测定酶活.求得ECB脱酰基酶催化ECB水解的Km和Vmax分别为0.356 mmol/L和397 μmol/(min·mg).
3结论
从A.utahensisZJB-0801从克隆到ECB脱酰基酶基因,以pSET152作为表达质粒,构建重组天蓝色链霉菌基因工程菌,能够高效表达ECB脱酰基酶,酶活330 mU/mL,是目前已报道ECB脱酰基酶活性的较高水平.重组酶催化反应的最适温度为45 ℃,最适pH=7.5.Zn2+,Mg2+,Mn2+和Fe2+对ECB脱酰基酶有激活作用,而Cu2+和Co2+对其有抑制作用.催化动力学参数分析表明:该酶对底物ECB的Km和Vmax分别为0.356 mmol/L和397 μmol/(min·mg).
参考文献:
[1]莫智菲.新型抗真菌药物研究进展[J].中国处方药,2014,12(12):150-151.
[2]LOEFHER J, STEVENS D A. Antifungal drug resistance[J]. Clinical infectious diseases,2003,36(Suppl I):3-41.
[3]刘晓环,张朝晖.抗真菌药物作用靶点机理及新药研发进展[J].药物分析杂志,2015,35(2):193-202.
[4]EMRI T, LASZLO M, VIKTORIA T, et al. Echinocandins:production and applications[J]. Applied microbiology and biotechnology,2013(7):67-3284.
[5]常青,邵雷,李继安,等.酰胺水解酶基因工程菌的构建及其对棘白菌素B的转化[J].工业微生物,2012,41(2):11-15.
[6]丁彦博,可爱兵,蔡超靖,等.来源棘白霉素B脱酞基酶产生菌的筛选和转化条件优化[J].中国抗生素杂志,2012,37(6):449-454.
[7]BOECK L D, FUKUDA D S, ABBOTT B J, et. alDeacylation of A21978C, an acidic lipopeptide antibiotic complex, byActinoplanesutahensis[J]. Journal of antibiotics,1988,41(8):1085-1092.
[8]UEDA S, SHIBATA T, ITO K, et al. Cloning and expression of the FR901379 acylase gene fromStreptomycessp. no. 6907[J]. Journal of antibiotics,2011,64(2):169-175.
[9]HIMABINDU M, RAVICHANDRA P, ANNAPURNA J. Enhancement of gentamicin production by mutagenesis and non-nutritional stress conditions inMicromonosporaechinospora[J]. Preparative biochemistry and biotechnology,2007,42:1352-1356.
[10]SHAO Lei, LI Jian, LIU Aijuan, et al. Efficient bioconversion of echinocandin B to its nucleus by overexpression of deacylase genes in different host strains[J]. Applied and environment microbiology,2013,79(4):1126-1133.
[11]KREUZMAN A J, HODGES R L, SWARTLING J R, et al. Membrane-associated echinocandin B deacylase ofActinoplanesutahensis: purification, characterization, heterologous cloning and enzymatic deacylation reaction[J]. Journal of industrial microbiology and biotechnology,2000,41(3):173-178.
[12]SATOSHI U, KAZUNPRI S, NOBUTAKA O, et al. Screening an charaterization of microorganisms with FR901379 acylase activity[J]. Journal of antibiotics,2009,63:65-70.
[13]INOKOSHI J, TAKESHIMA H, TAKESHIMA H, et al. Efficient production of aculeacin A acylase in recombinantStreptomycesstrains[J]. Applied biochemistry and biotechnology,1993,39:532-536.
[14]RICHARD H, BALTZ.StreptomycesandSaccharopolysporahosts for heterologous expression of secondary metabolite gene clusters[J]. Journal of industrial microbiology and biotechnology,2010,37:759-777.
[15]朱廷恒,方三余,严宏波,等.重组蛋白Hd3a在大肠杆菌中的表达、纯化及活性研究[J].浙江工大学学报,2013,41(6):601-604.
[16]林陈水,陈微微,分泌型水蛭素Ⅱ基因工程菌的构建[J].浙江工业大学学报,2014,42(1):50-53.
[17]洪敏,赵春,张正波,等.L—天冬氨酸仅一脱羧酶工程菌株的构建及其培养条件研究[J].浙江工业大学学报,2011,39(3):252-260.
(责任编辑:陈石平)
Heterologous expression and enzymatic properties of echinocandins B deacylase inStreptomycescoelicolor
ZOU Shuping, LIAO Sixing, NIU Kun, ZHENG Yuguo, SHEN Yinchu
(College of Biological and Environmental Engineering, Zhejiang University of Technology, Hangzhou 310014, China)
Abstract:Echinocandin B (ECB) deacylase can catalyze echinocandin B. The side chain amide bond was removed. In this way, ECB nucleus was acquired. In the new antifungal, the chemical-enzymatic synthesis has important applications in the preparation of anidulafungin. Clone of ECB dealyse gene from Actinoplanes utahensis ZJB-0801, and linked into the overexpression vector pSET152.Then the plasmid was transformed into Streptomyces coelicolorA3.This genetically engineered strain could transform ECB into ECB nucleusand. Investigating its enzymatic properties, the results showed that the optimum temperature for the recombinant enzyme is 45 ℃, the optimum pH is 7.5. Zn2+, Mg2+, Mn2+, Fe2+are activated on the recombinant enzyme. The Cu2+and Co2+are inhibition on the recombinant enzyme. Enzyme kinetic parameters Kmand Vmaxare 0.183 mmol / L and 0.085 μmol / min·mg, respectively.
Keywords:echinocandin B deacylase; Streptomyces coelicolor; recombinantion and expression; enzymatic properties
中图分类号:Q814
文献标志码:A
文章编号:1006-4303(2016)01-0099-05
作者简介:邹树平(1980—),男,湖南常德人,副教授,主要从事生物催化与微生物发酵制药研究,E-mail:zousp@zjut.edu.cn.
基金项目:国家自然科学基金资助项目(21176224);教育部博士点新教师基金资助项目(2012331712004)
收稿日期:2015-04-01

