基于线粒体COI序列探讨安徽长江流域黄鳝群体遗传分化
胡玉婷,江 河,潘庭双,凌 俊,段国庆(安徽省农业科学院水产研究所,合肥 230031)
基于线粒体COI序列探讨安徽长江流域黄鳝群体遗传分化
胡玉婷,江河*,潘庭双,凌俊,段国庆
(安徽省农业科学院水产研究所,合肥230031)
摘要:探讨安徽长江流域黄鳝群体遗传结构,采用线粒体COI的DNA条形码序列研究该区6个黄鳝野生群体(当涂、无为、繁昌、贵池、怀宁和望江)遗传分化。180个样本COI片段(665 bp)中共检出52个变异位点(变异率7.8%)、40种单倍型。序列中A、T、G、C碱基平均含量分别为24.7%、28.2%、17.1%、30.0%,A+T含量大于G+C含量。AMOVA分析中,高达68.45%遗传变异来自群体间,说明黄鳝群体间已产生显著遗传分化。群体间系统进化树显示:6个群体形成两大类群(当涂和繁昌群体为一类群,其余群体为另一类群),分析表明结果与各群体地理位置密切相关。同时由NCBI下载合鳃鱼科几个物种的COI同源序列,构建系统进化树,显示黄鳝与同属的山黄鳝聚为一支,并与不同属的合鳃鱼和穴栖蛇胸鳝聚为一支。研究表明,基于COI的DNA条形码序列适于黄鳝群体遗传结构研究。
关键词:黄鳝;COI条形码;遗传分化;长江
胡玉婷,江河,潘庭双,等.基于线粒体COI序列探讨安徽长江流域黄鳝群体遗传分化[J].东北农业大学学报, 2016, 47(2): 74-80. Hu Yuting, Jiang He, Pan Tingshuang, et al. Genetic differentiation of Monopterus albus populations from Anhui Province in Yangtze River basin based on mitochondrial COI barcode sequence[J]. Journal of Northeast Agricultural University, 2016, 47 (2): 74-80. (in Chinese with English abstract)
黄鳝(Monopterus albus)隶属合鳃鱼目(Synbranchiformes)合鳃鱼科(Synbranchidae)黄鳝属,适宜生长在泥塘、沟渠和稻田等缓流浅水中,广泛分布于我国中东部地区淡水水域。黄鳝具有重要营养和药用价值,市场需求大,是我国重要淡水经济鱼类之一,人工养殖已在长江中下游地区广泛开展。段国庆研究表明,随养殖规模扩大和环境污染加剧,黄鳝野生资源越来越少,且出现种质退化和遗传多样性降低等问题[1]。
贺顺连研究表明,由于我国各地黄鳝相互流通,东南亚黄鳝及山黄鳝Monopterus cuchia、穴黄鳝Monopterus fossorius涌入我国[2],加剧本土黄鳝种质污染风险。目前,通过我国黄鳝不同地理群体RAPD、微卫星和线粒体序列(Cytb、D-loop)等遗传多样性研究,发现我国黄鳝形成遗传变异不同的地方群体,在一些小尺度区域内存在较大遗传差异[3-7],显示我国黄鳝群体具有较高遗传多样性和较大遗传分化。目前尚无采用线粒体COI序列研究黄鳝群体遗传结构的相关报道。
线粒体细胞色素c氧化酶亚基I(Cytochrome c oxidase subunit I,COI)基因具有引物通用性高、长度适宜、进化速率适中等特点,既可作为动物种属系统进化研究的良好标记[8-12],又可广泛应用于群体遗传学研究,是理想的动物DNA条形码[13-14]。研究采集安徽长江流域6个地理群体的黄鳝,通过COI基因DNA条形码序列分析,探讨黄鳝群体间遗传分化水平,可为丰富黄鳝遗传结构研究内容、黄鳝种质资源保护和合理开发利用提供理论依据。
1 材料与方法
1.1材料
试验用野生黄鳝样品分别采集于安徽长江流域6个群体(当涂县31.45°N、118.61°E;无为县31.18°N、117.99°E;繁昌县31.02°N、118.32°E;贵池县30.77°N、117.64°E;怀宁县30.72°N、116.96°E和望江县30.40°N、116.83°E),各30尾。样品采集后活体运回,立即解剖取其背部肌肉保存于无水乙醇中备用。
1.2方法
1.2.1基因组DNA提取、PCR扩增及测序
采用天根生化科技(北京)有限公司动物基因组提取试剂盒,按照说明书提取黄鳝基因组DNA。下载NCBI中黄鳝线粒体基因组序列(登录号:NC003192),Primer 5.0软件设计COI扩增引物,委托生工生物工程(上海)股份有限公司合成。
引物序列为MF1:AACCACAAAGACATTG GCACC;MR1:CCAAAGCCTGGAAGAATCAAG。反应体系总体积50 μL,其中:10×PCR Buffer (MgCl2)5.0 μL,10 mmol·L-1dNTP 4.0 μL,10 mmol·L-1引物各1.5 μL,50 ng·L-1模板DNA 2 μL,5 U·μL-1的Taq DNA酶0.3 μL,灭菌超纯水35.7 μL。上述试剂购自宝生物工程(大连)有限公司。反应条件为:94℃预变性5 min;94℃变性30 s、52℃退火35 s、72℃延伸45 s,35个循环;72℃延伸7 min。PCR产物经1%的琼脂糖凝胶电泳检测后,送生物公司纯化并采用ABI3730XL测序仪双向测序,测序引物为MF1和MR1。
1.2.2数据统计与分析
利用Clustal X[15]和Seaview[16]软件排序并辅以人工校正。采用DNAsp 5.0[17]软件计算多态位点和单倍型数目、单倍型多样性(Haplotype diversity,Hd)、核苷酸多样性(Nucleotide diversity,Pi)。使用Mega 4.0[18]软件计算序列碱基组成、变异率、群体和个体间平均遗传距离(Kumara2-parameter距离模型)并构建相关分子系统树。软件Network 4.600(http:// www.fluxus-engineering.com/sharenet.htm)绘制单倍型进化网络图。由Arlequin 3.5软件[19]计算群体遗传分化系数(F-statistics,Fst)和分子变异分析(Analysis of molecular variance,AMOVA),并用排列测验法(Permutation test)检验Fst的显著性水平(重复1 000次)。
2结果与分析
2.1序列变异分析
经比对排序后,获得黄鳝线粒体COI同源序列665 bp,未发现碱基插入和缺失。其中,有变异位点52个(变异率7.8%),包括简约信息位点42个,单突变位点10个。平均A、T、G、C含量分别为24.7%、28.2%、17.1%和30.0%,A+T含量较高(52.9%),G+C含量(47.1%)较低,表现出较为明显的碱基组成偏倚性。序列变异主要发生在三联密码子的第3个碱基上,符合蛋白质编码基因第3位点进化最快一般规律。4种碱基在密码子第1、2、3位点的平均含量具有较大差异,密码子第3位点上G含量偏低尤为明显。
6个群体共180条COI序列有40种单倍型(见表1),单倍型间最大变异率为4.06%。各群体单倍型差异较大,各群体独有单倍型较多(29个),其中仅包含1个样品的单倍型19个。群体间共享单倍型仅11个,单倍型Hap4为5个群体共有,分布最广泛,包含43个个体(出现频率23.9%);另有2种单倍型(Hap5,Hap12)为3个群体共有、8个单倍型(Hap2-3,10,13,15,27-28,31)为2个群体共有。繁昌与其他群体间均无共享单倍型。
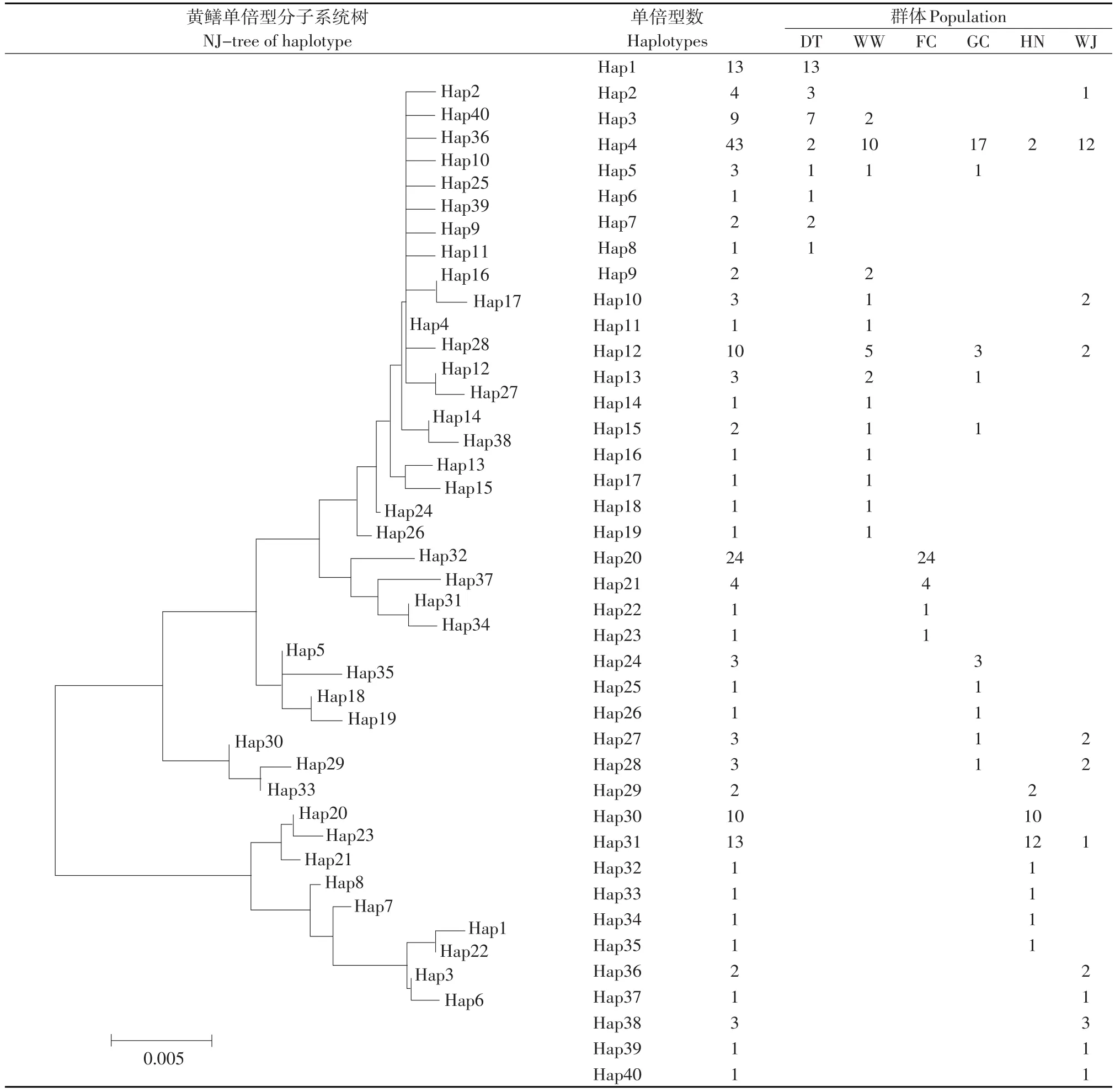
表1黄鳝COI基因单倍型系统进化树及在不同群体中的分布Table 1 NJ-tree of and distributions of haplotypes of M. albus based on COI sequences
黄鳝单倍型在群体间分布和简约进化网络图分别见表1、图1。结果显示:黄鳝单倍型的群体分布非随机,存在一定地理结构,形成两大类群:类群I主要包括当涂、繁昌群体,且各自有一定独立性;其余群体组成类群II,其中怀宁群体主要介于其他三群体与类群I之间。黄鳝单倍型分子系统树显示40种单倍型属于两遗传谱系(见表1),两谱系单倍型间的最小碱基变异数为11个,序列差异率为1.65%(见图1),最小遗传距离为0.01994;两谱系单倍型间最大遗传距离为0.04054。
黄鳝各群体单倍型多样性普遍较高(0.352~ 0.867),而所有群体核苷酸多样性则偏低(0.00126~ 0.01321)。其中,无为(WW)群体单倍型多样性最高;当涂群体核苷酸多样性最高;繁昌(FC)群体单倍型多样性和核苷酸多样性均最低(见表2)。

图1基于线粒体COI序列的黄鳝单倍型简约进化网络Fig. 1 Statistical parsimony network of M. albus haplotypes based on mtDNA COI sequences
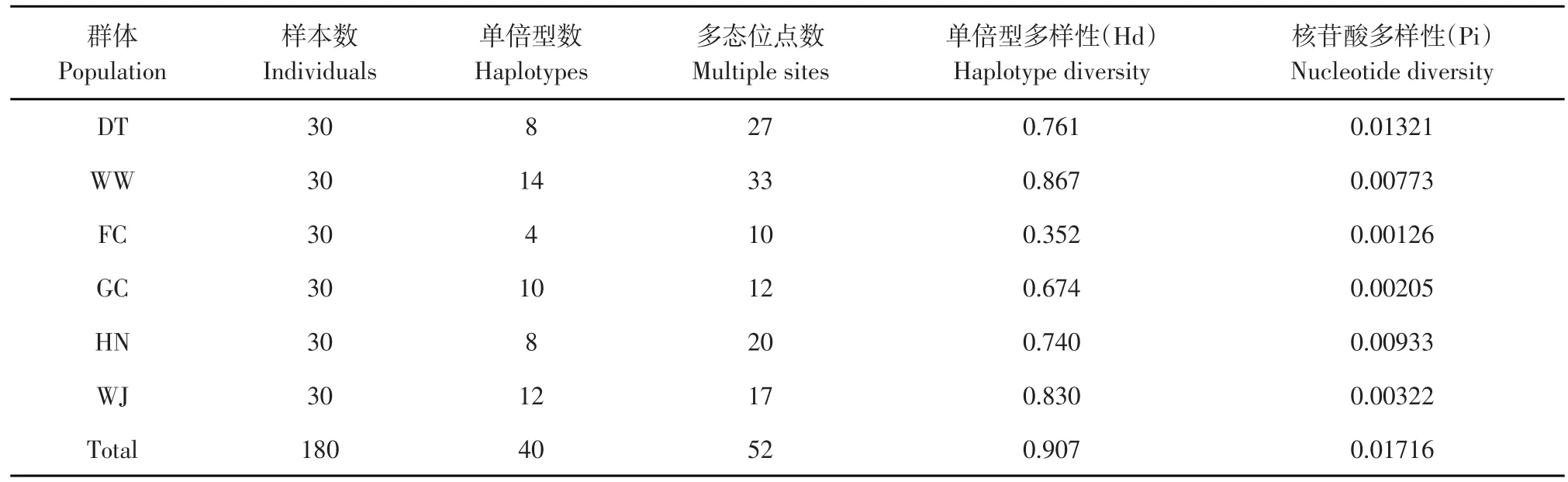
表2黄鳝群体遗传多样性参数Table 2 Parameter of genetic diversity of M. albus populations based on COI sequences
2.2群体遗传结构与分化
黄鳝群体间K-2P遗传距离见表3。群体间平均遗传距离较大(0.00268~0.03027),其中繁昌群体与所有群体间的遗传距离均较大(0.01519~0.03027),其中繁昌与望江群体间遗传距离最大。由于不同群体间遗传距离反映遗传组成分化程度,上述结果显示黄鳝群体间存在较高遗传差异。群体间遗传分化系数Fst值显示(见表3):除三对群体间(GC、WW、WJ),其他群体间Fst值(0.359~0.926)均明显大于0.05,且有极显著差异(P<0.001)。
分子变异分析(AMOVA)结果显示:来自群体间遗传变异高达68.45%,群体内变异为31.55%(P< 0.001),表明黄鳝6个群体间产生明显遗传分化(见表4)。

表3黄鳝群体间遗传距离(对角线上)和遗传分化系数Fst值(对角线下)Table 3 Paired genetic distances between populations (above diagonal) and genetic differentiation index (Fst) (below diagonal) of M. albus based on COI sequences

表4黄鳝群体间分子变异分析(AMOVA)Table 4 Analysis of molecular variance (AMOVA) among populations of M. albus
以群体间K-2P遗传距离矩阵构建的群体间系统树(NJ tree)如图2,与单倍型系统树、单倍型进化网络图结果一致:黄鳝6个地理群体聚为两大进化枝,而当涂(DT)、繁昌(FC)和怀宁(HN)群体在各自进化枝内具一定的独立性。

图2基于线粒体COI条形码序列的黄鳝群体间系统树Fig. 2 NJ tree of M. albus populations based on K-2P distances
2.3合鳃鱼科系统发育分析从NCBI下载合鳃鱼科三属三种的线粒体COI序列,即黄鳝属山黄鳝(Monopterus cuchia)、蛇胸鳝属穴栖蛇胸鳝(Ophisternon aenigmaticum)和合鳃鱼属合鳃鱼(Synbranchus marmoratus),登录号分别为:FJ459509、EU751878、AP004439。结合上文
中黄鳝COI同源序列片段,采用MEGA4.0软件K-2P模型计算4种黄鳝间平均遗传距离(见表5)。
基于表5遗传距离构建合鳃鱼科间4种鱼类的系统发育树(NJ法)(见图3)。分子系统树显示,黄鳝与同属的山黄鳝聚为一支,并与穴栖蛇胸鳝和合鳃鱼聚为一支。

表5 4种合鳃鱼科鱼类平均遗传距离Table 5 Average genetic distance among four Synbranchidae fishes in the study
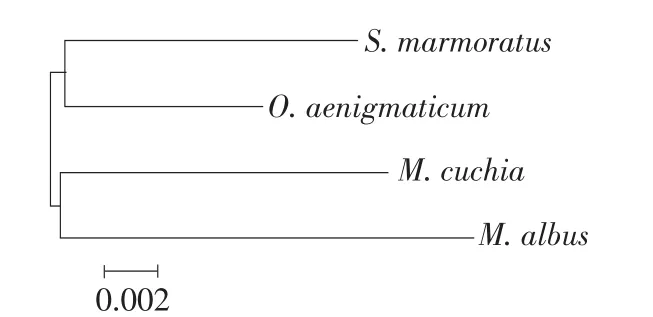
图3合鳃鱼科4种鱼类系统发育树Fig. 3 Molecular phylogenetic tree (NJ-tree) of four Synbranchidae fishes based on COI sequences
3讨论与结论
3.1黄鳝群体遗传分化
遗传分化系数(Fst)是表观群体间遗传分化程度的重要参数,Fst值越大群体间分化程度越高,Fst=0~0.05时为无分化,0.05~0.15为中度分化,0.15~0.25为高度分化[20]。本研究中6个群体间总Fst值为0.6845,表明黄鳝群体间存在显著遗传分化。由图1可见,6个黄鳝群体分为两大类群,其中类群Ⅰ两群体间Fst=0.511;类群Ⅱ仅怀宁群体与其他三群体间有遗传分化,即Fst值(0.359、0.476、0.513)大于0.05。单倍型分子系统树中40种单倍型分属于两遗传谱系,构成两类群间主要遗传差异,其中两谱系单倍型间最小序列差异率为1.65%,最大高达4.06%。两谱系单倍型间最小遗传距离0.01994、最大遗传距离0.04054,而6个群体两两间平均遗传距离为0.00268~0.03027。均高于一般淡水鱼类COI序列的地理群体间遗传差异,甚至高于一些鱼类近缘种间的COI差异[21-23]。如毛云涛等选择线粒体COI基因作为分子标记研究沙鳅亚科鱼类(Botiinae)的分子系统发育关系[24],结果发现沙鳅亚科鱼类种内平均遗传距离为0.002±0.000,种间平均遗传距离为0.148±0.008。说明该区黄鳝具有较高的遗传异质性。
群体分化是物种分化的初期表现,研究群体分化对于理解物种分化过程与机制具有重要作用。隔离是群体分化重要影响因素之一。研究中6个黄鳝群体间地理距离比较短,通过长江及其支流相通,显示遗传差异大,与各群体所处小流域有关。类群Ⅰ中两群体分属于长江南岸支流青衣江和漳河,而类群2则距长江干流较近(除怀宁群体外其余3群体的具体采样地均距长江干流< 5 km),除贵池外均位于长江北岸(贵池采样点距长江干流直线距离<1 km),由于怀宁群体采样地距长江干流较远(>20 km)、受菜子河水系影响且地处其余3群体中间位置,形成既与其他三群体遗传距离较近又有区别的遗传分布格局。由于黄鳝营穴居性生活、迁移能力较弱,致使不同群体间难以基因交流,长期地理隔离导致黄鳝群体间积累较大遗传差异,形成显著遗传分化。
3.2 COI基因适于黄鳝群体遗传学研究
线粒体基因组不同区域进化速率存在差异,遗传变异分析能力也不同,因此适合不同水平的进化研究。COI基因是鱼类线粒体13个蛋白质编码基因之一,具中度保守性,既能保证足够变异又易被通用引物扩增,且基本不存在插入与缺失。许多研究表明,COI基因一般适于研究鱼类种间及种以上阶元的分析,作为DNA条形码广泛用于种属间的系统发育关系和物种鉴定[11-12, 21-24]。但在一些鱼类类群中COI序列也可为同种种群间遗传多样性和遗传结构分析提供信息[13, 25-26]。
就种下水平研究而言,COI基因蕴含的信息量通常小于线粒体Cytb基因和D-loop区。文中测序结果显示黄鳝Cytb基因变异率仅约为COI基因的1.2倍[7],表明COI序列较适合黄鳝群体水平的遗传研究。刘连为等基于线粒体COI和Cytb基因序列研究北太平洋柔鱼种群遗传结构的结果显示[27],其Cytb变异率是COI的1.6倍;而太湖新银鱼Cytb基因的变异率约为COI基因的3倍[25]。利用线粒体COI序列检测到的鱼类群体遗传多样性水平显著高于基于线粒体Cytb的序列分析结果,原因是不同物种的线粒体同一区域进化速度不同,由于选择COI和Cytb序列为部分序列,而同一基因序列内不同片段的进化速率存在差异,因此同一基因的片段大小及位置影响变异率的检测。由于所选分子标记不同导致检测多样性水平不同,影响鱼类种质资源状况评价准确性。因此,研究比较选择线粒体DNA不同区域多个基因,反映遗传差异水平。
合鳃鱼科3属4种鱼类的系统发育分析结果显示,4种鱼类间的遗传距离为0.1833~0.2783,可见其间遗传距离较小。其中,同属的黄鳝与山黄鳝、穴栖蛇胸鳝和合鳃鱼分别聚为一支,然后两属鱼类再聚为一支。但黄鳝与山黄鳝间遗传距离却最大(0.2783),大于各自与其他两属鱼类的遗传距离,即出现种间遗传距离大于属间遗传距离现象。这说明合鳃鱼属与穴栖蛇胸鳝属间的亲缘关系很近,而黄鳝属鱼类遗传变异大。由于仅选择一个基因、少数个体难以反映真正的遗传关系,其真实亲缘关系尚需结合更多分子标记、样本和群体开展深入研究。
[参考文献]
[ 1 ]段国庆,江河,胡王,等.洗涤剂对黄鳝幼鱼急性毒性的研究[J].东北农业大学学报, 2014, 45(8): 84-89.
[ 2 ]贺顺连,刘学文,郭照良,等.3种黄鳝遗传多样性分析[J].湖南农业大学学报:自然科学版, 2004, 30(2): 145-147.
[ 3 ]蔡欣,张海容.川渝部分地区野生黄鳝RAPD引物筛选及其在绵阳群体的验证[J].西南科技大学学报, 2010, 25(4): 96-99.
[ 4 ]杨太有,温树红,郝艳军,等.黄河流域黄鳝自然群体遗传多样性和遗传结构的研究[J].水生生物学报, 2011, 35(3): 532-537.
[ 5 ]周宇芳,胡杭娇,张龙韬,等.长江中下游黄鳝遗传多样性的微卫星分析[J].生物技术通报, 2011, 11: 187-192.
[ 6 ] Cai X, Yu S M, Mipam T, et al. Phylogenetic lineages of Monopterus albus (Synbranchiformes: Synbranchidae) in China inferred from mitochondrial control region[J]. Journal of Zoological Systematics and Evolutionary Research, 2013, 51(1): 38-44.
[ 7 ]胡玉婷,江河,胡王,等.安徽长江流域黄鳝六个地理种群的遗传变异研究[J].四川动物, 2015, 34(1): 21-28.
[ 8 ] Hebert P D N, Cywinska A, Ball S L, et al. Biological identification through DNA barcodes[J]. Proc R Soc Lond B Biol Sci, 2003, 270: 313-321.
[ 9 ] Vences M, Thomas M, Bonett R M, et al. Deciphering amphibian diversity through DNA barcoding: chances and challenges[J]. Phil Trans R Soc B, 2005, 360: 1859-1868.
[10] Ward R D, Zemlak T S, Innes B H, et al. DNA barcoding Australia's fish species[J]. Phil Trans R Soc B, 2005, 360: 1847-1857.
[11] Cui Z X, Liu Y, Liu J, et al. Molecular identification of Pampusfishes (Perciformes, Stromateidae) [J]. Ichthyological Research, 2010, 57(1): 32-39.
[12] Smith P J, Steinke D, Dettai A, et al. DNA barcodes and species identifications in Ross Sea and Southern Ocean fishes[J]. Polar Biology, 2012, 35(9): 1297-1310.
[13] Sun P, Shi Z H, Yin F, et al. Genetic variation analysis of Mugil cephalus in China sea based on mitochondrial COI gene sequences [J]. Biochemical Genetics, 2012, 50(3-4): 180-191.
[14]裴男才,陈步峰.生物DNA条形码:十年发展历程、研究尺度和功能[J].生物多样性, 2013, 21(5): 616-627.
[15] Thompson J D, Gibson T J, Plewniak F. The Clustal X windows interface: flexible strategies for multiple sequences alignment aided by quality analysis tools[J]. Nucleic Acids Res, 1997, 25(4): 4876-4882.
[16] Galtier N, Gouy M, Gautier C. SeaView and Phylo_win: two graphic tools for sequence alignment and molecular phylogeny[J]. Computer Applications in the Biosciences, 1996, 12: 543-548.
[17] Librado P, Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25: 1451-1452.
[18] Tamura K, Dudley J, Nei M, et al. MEGA 4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24: 1596-1599.
[19] Excoffier L, Lischer H E L. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10: 564-567.
[20] Wright S. Evolution and the genetics of populations[C]. Chicago: University of Chicago Press, 1978: 580.
[21]彭居俐,王绪祯,王丁,等.基于线粒体CO1序列的DNA条形码在鲤科鲌属鱼类物种鉴定中的应用[J].水生生物学报, 2009, 33 (2): 271-276.
[22]杨天燕,孟玮,马燕武,等.基于线粒体COI和Cytb基因序列对新疆两水系斑重唇鱼遗传结构的分析[J].淡水渔业, 2014, 44 (4): 41-47.
[23]于亚男,宋超,侯俊利,等.基于线粒体COI基因部分序列的长江口虾虎鱼科鱼类系统分类[J].淡水渔业, 2014, 44(5): 3-8.
[24]毛云涛,甘小妮,王绪祯.基于线粒体COI基因的沙鳅亚科鱼类DNA条形码及其分子系统发育研究[J].水生生物学报, 2014, 38 (4): 737-744.
[25]张迪,雷光春,龚成,等.基于COI基因序列的太湖新银鱼遗传多样性[J].湖泊科学, 2012, 24(2): 299-306.
[26] Sun X X, Xu D D, Lou B, et al. Genetic diversity and population structure of Eleutheronema rhadinumin the East and South China Seas revealed in mitochondrial COI sequences[J]. Chinese Journal of Oceanology and Limnology, 2013, 31(6): 1276-1283.
[27]刘连为,许强华,陈新军.基于线粒体COI和Cytb基因序列的北太平洋柔鱼种群遗传结构研究[J].水产学报, 2012, 36(11): 1675-1684.
Genetic differentiation of Monopterus albus populations from Anhui Province in Yangtze River basin based on mitochondrial COI barcode sequence
HU Yuting, JIANG He, PAN Tingshuang, LING Jun, DUAN Guoqing(Fisheries Research Institute of Anhui Academy of Agricultural Sciences, Hefei 230031, China)
Abstract:In the study, partial sequences of the mitochondrial DNA cytochrome c oxidase subunit I gene (COI) of six Monopterus albus populations, which were collected from Yangtze River basin in Anhui Province, were amplified by PCR to explore genetic differentiation. The length of COI sequences of 180 M. albus individuals were all 665 bp, 40 haplotypes were identified and 52 mutation sites were detected. The nucleotide composition of A, T, G, C was 24.7%, 28.2%, 17.1%, 30.0%, respectively; A+T content was higher than G+C content. Analysis of Molecular Variance (AMOVA) indicated significant genetic differentiation among populations. The six populations could be divided into two groups based on the genetic distance and phylogenetic analysis of COI sequences, which were closely related to geographical location of each population. The molecular phylogenetic tree (NJ-tree) of Synbranchidae showed that M. albus were more closely related to Monopterus cuchia than Ophisternon aenigmaticuma and Synbranchus marmoratus. The results indicated that mitochondrial COI barcode sequence wasbook=75,ebook=80suited for the study of population genetics study.
Key words:Monopterus albus; COI barcode; genetic differentiation; Yangtze River
*通讯作者:江河,教授级高级工程师,研究方向为淡水水产养殖。E-mail: hfjianghe@sohu. com
作者简介:胡玉婷(1986-),女,助理研究员,博士,研究方向为鱼类生态学。E-mail: huyuting1021@126. com
基金项目:农业部行业(农业)科研专项(201003076);安徽省农业科学院院长青年创新基金项目(13B0529);安徽省农业科学院黄鳝创新团队项目(12c0501);安徽省科技攻关项目(12010302056)
收稿日期:2015-08-11
中图分类号:S917.4;Q347
文献标志码:A
文章编号:1005-9369(2016)02-0074-07

