基于ITS2序列的禹州漏芦和漏芦药材基因识别*
陈江平,侯典云,严绪华,胡志强,魏 蒙,胡志刚,5**,汪乐原**
(1. 湖北中医药大学药学院 武汉 430065;2. 河南科技大学农学院 洛阳 471023;3. 武汉市中医医院 武汉 430010; 4. 武汉市第一医院 武汉 430022;5. 中国中医科学院中药研究所 北京 100700)
基于ITS2序列的禹州漏芦和漏芦药材基因识别*
陈江平1,侯典云2,严绪华3,胡志强4,魏 蒙1,胡志刚1,5**,汪乐原1**
(1. 湖北中医药大学药学院 武汉 430065;2. 河南科技大学农学院 洛阳 471023;3. 武汉市中医医院 武汉 430010; 4. 武汉市第一医院 武汉 430022;5. 中国中医科学院中药研究所 北京 100700)
目的:应用ITS2序列鉴别禹州漏芦和漏芦药材的真伪,为临床准确用药提供可靠的基因识别方法。方法:收集多基原药材禹州漏芦基原物种蓝刺头和华东蓝刺头共14份样品和漏芦药材基原物种祁州漏芦8份样品,经实验获取ITS2序列,结合GenBank中20条相关近缘混伪品序列,建立以上物种和混伪品的DNA条形码鉴定方法;将从市场上收集的11份禹州漏芦和20份漏芦药材的ITS2序列与以上序列进行比对分析和构建系统NJ树。结果:禹州漏芦两个基原物种的序列主导单倍型相同;禹州漏芦和漏芦药材基原物种最大K2P距离均小于其与混伪品的种间最小K2P距离;系统NJ树分析均能准确区分禹州漏芦和漏芦药材与各自混伪品。药材检测结果表明,11份禹州漏芦中10份为正品,1份为伪品;20份漏芦中2份为正品,18份为伪品。结论:本研究建立了基于ITS2序列的禹州漏芦和漏芦药材DNA条形码鉴定方法,可用于快速鉴别禹州漏芦和漏芦药材真伪。
禹州漏芦 漏芦 ITS2序列 基因识别 近缘混伪品
禹州漏芦为菊科蓝刺头属植物蓝刺头Echinops latifolius Tausch.或华东蓝刺头Echinops grijisii Hance的干燥根[1]。这两个种为中国特有物种,分布于辽宁南部、浙江、山东、安徽、江苏、福建等地区[2,3]。漏芦始载于《神农本草经》,为菊科漏芦属植物祁州漏芦Rhaponticum uniflorum (L.) DC.的干燥根。在中国分布的漏芦属仅有2个物种,为祁州漏芦和鹿草R. carthamoides (Willd.) Dittrich[4,5]。禹州漏芦曾与漏芦均作为漏芦药材收载于《中华人民共和国药典》,近代研究表明两者的化学成分及疗效各有不同,禹州漏芦具有良好的抗炎消肿、抗肿瘤、抗病毒作用;漏芦具有较好的抗动脉粥样硬化、抗脂质过氧化、免疫促进、改善脑功能和抗衰老作用[6-8]。因此,《中华人民共和国药典》1995年版开始将祁州漏芦单独作为漏芦药材使用,故禹州漏芦和漏芦药材需区别使用[9]。历代本草所述漏芦药材的基原物种不尽相同,民间混用、误用情况普遍,漏芦的混伪品涉及8个科约30种植物,严重影响了临床用药的准确性和安全性[2]。因此,寻找一种快速、简便、可靠的禹州漏芦和漏芦药材及其混伪品鉴定方法尤为重要。
DNA条形码技术(DNA Barcoding)是指利用基因组中一段公认的、相对较短的DNA片段来进行物种鉴定的分子诊断新技术。陈士林等[10]提出的以ITS2为主,psbA-trnH为辅的药用植物条形码鉴定体系已得到广泛认可[11],目前已成功应用于功劳叶、牡丹皮、葶苈子、苍耳子等大量药材的物种鉴定[12-15]。本研究采用ITS2序列鉴定禹州漏芦和漏芦药材及其近缘混伪品,为其快速准确鉴定、临床用药安全及资源开发利用提供可靠的基因识别方法。
1 材料
本研究实验材料包括禹州漏芦基原物种蓝刺头8份、华东蓝刺头6份,漏芦基原物种祁州漏芦8份以及祁州漏芦同属物种鹿草1份。所有样品经中国医学科学院药用植物研究所林余霖研究员鉴定,凭证标本保存于中国中医科学院中药研究所。同时,从GenBank数据库下载20条相关近缘混伪品序列,所有序列经查阅文献验证,确保准确可靠[10,16-18]。禹州漏芦和漏芦药材基原物种及其近缘混伪品的ITS2序列信息详情见表1。

表1 禹州漏芦和漏芦药材基原物种及其近缘混伪品ITS2序列信息
2 方法
2.1 DNA提取、PCR扩增及测序和数据处理
用75%乙醇擦拭实验样品表面,取禹州漏芦基原物种蓝刺头、华东蓝刺头和漏芦基原物种祁州漏芦叶片各30 mg,鹿草根茎50 mg。用球磨仪(德国Retsch MM400公司)研磨2 min(30次/s),然后用改良的CTAB法提取植物总DNA。采用通用引物、PCR反应条件及扩增程序对样品总DNA进行ITS2序列扩增[19]。扩增产物经琼脂糖凝胶电泳检测,对条带单一且清晰的PCR产物进行双向测序。测序峰图用CodonCode Aligner V4.2.4(美国CodonCode Co公司)进行校对拼接,基于隐马尔可夫模型HMMer注释除去低质量端,获得ITS2序列,并提交至NCBI得到GenBank登录号。
2.2 基原物种和市场收集的药材鉴定分析
利用MEGA(Molecular Evolutionary Genetics Analysis)6.0对拼接后的禹州漏芦和漏芦药材基原物种及其混伪品ITS2序列进行比对分析,计 算 种 内 和 种 间K2P(Kimura 2-parameter)距离,并将序列提交到中药材DNA条形码鉴定系统(http://www.tcmbarcode.cn/china/),用 于 后 续研究。将从市场上收集的11份禹州漏芦药材(编号:YC0004YP01-11)和20份漏芦药材(编号:YC0003YP01-20)的ITS2序列导入到中药材DNA条形码鉴定系统进行最大相似度比对鉴定,并结合两种药材基原物种及其混伪品的ITS2序列共同构建系统NJ(Neighbor Joining)树,以检测两种药材的真伪。
3 基原物种鉴定结果与分析
3.1 ITS2序列变异位点分析
禹州漏芦基原物种蓝刺头和华东蓝刺头共14条序列,序列比对后长度为220 bp,有2个单倍型。由于华东蓝刺头的6条序列均为A1型,且两个物种的主导单倍型相同,故以禹州漏芦的两个基原物种为整体进行后续分析,其变异位点见表2。两个基原物种和同属混伪品16条ITS2序列中共产生55个变异位点。
漏芦基原物种祁州漏芦共11条序列,序列比对后长度为222 bp,有7个单倍型,产生5个变异位点,见表3。祁州漏芦与同属物种鹿草的2条ITS2序列共产生10个变异位点。
3.2 K2P距离分析
基于MEGA6.0分析两种药材基原物种的种内K2P距离及其与混伪品的种间K2P距离,详见表4。由结果可知,禹州漏芦药材最大K2P距离(0.004 6)小于其与混伪品的最小K2P距离(0.014 0),平均K2P距离(0.000 7)小于其与混伪品的种间平均K2P距离(0.146 9);漏芦药材最大K2P距离(0.018 5)小于其与混伪品的最小K2P距离(0.023 3),平均K2P距离(0.007 5)小于其与混伪品的种间平均K2P距离(0.250 2),这说明K2P距离可以将禹州漏芦和漏芦药材与其混伪品区分开。
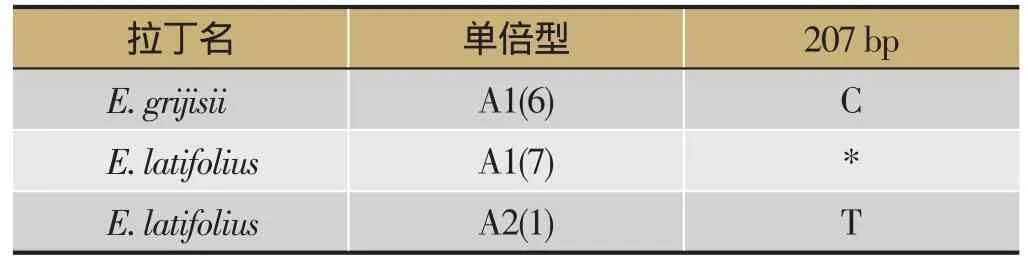
表2 蓝刺头和华东蓝刺头ITS2序列变异位点
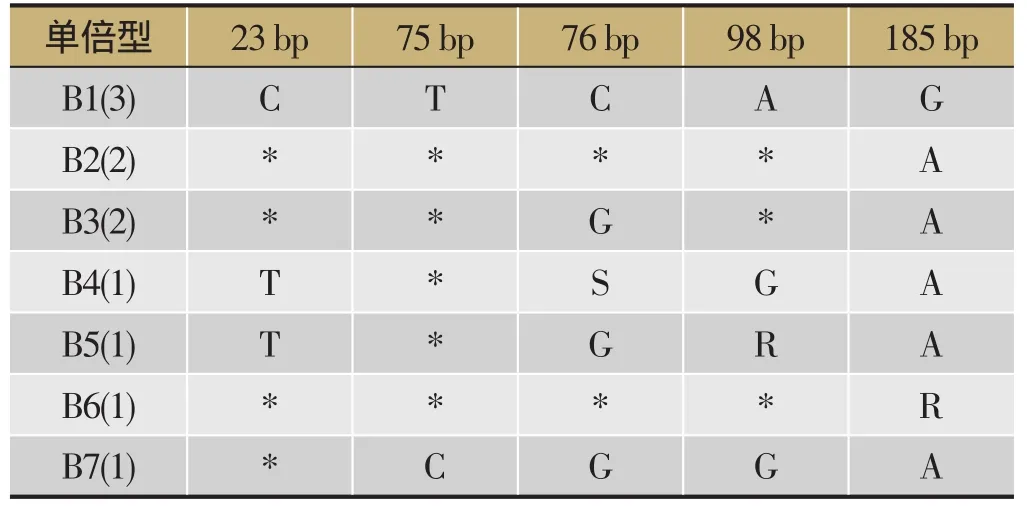
表3 祁州漏芦ITS2序列变异位点

表4 禹州漏芦和漏芦药材及其混伪品K2P距离
4 市场收集药材结果鉴定分析
4.1 序列比对鉴定结果
禹州漏芦药材(编号:YC0004YP01-11)和漏芦药材(编号:YC0003YP01-20)ITS2序列的最大相似度比对鉴定结果中,10份禹州漏芦药材可比对到其基原物种蓝刺头或华东蓝刺头,1份比对到其它科属物种;20份漏芦药材中,2份可比对到其基原物种祁州漏芦,18份比对到其它科属物种。比对结果详见表5。
4.2 NJ树鉴定结果
基于ITS2序列构建的系统NJ树中,禹州漏芦的基原物种蓝刺头和华东蓝刺头聚为一支,漏芦的基原物种祁州漏芦单独聚为一支,且均能与它们的混伪品区分开。11份禹州漏芦药材中有10份与基原物种聚为一支,1份在其它支上;20份漏芦药材中只有2份聚到基原物种的支上,18份分散在其它支上。
5 讨论
5.1 ITS2序列鉴定禹州漏芦和漏芦药材基原物种的准确性和可靠性
本研究表明,基于ITS2序列可将禹州漏芦的基原物种蓝刺头和华东蓝刺头与漏芦的基原物种祁州漏芦区分开,且均能与各自的混伪品区分开;蓝刺头和华东蓝刺头在NJ树上聚为一支,且主导单倍型相同,故将其作为整体进行分析。ITS2序列变异分析可知,两个药材的基原物种与同属混伪品种间变异位点数均明显大于其种内变异位点数。K2P距离表明,禹州漏芦和漏芦的种内最大(平均)K2P距离均小于它们与各自混伪品的种间最小(平均)K2P距离。从构建的NJ树可知,两种药材的基原物种均单独聚为一支。综上可知,ITS2序列能将两种药材基原物种与各自的混伪品区分开。

表5 禹州漏芦和漏芦药材ITS2序列比对鉴定结果
5.2 ITS2序列检测市场收集的禹州漏芦和漏芦药材真伪的可行性
基于序列相似性比对和系统NJ树对两种药材真伪鉴定的结果均表明,11份禹州漏芦药材中,10份为正品,1份为混伪品;20份漏芦药材中,只有2份为正品,其余均为混伪品。需要说明的是,本研究目的是检测药材真伪,而不侧重对药材混伪品进行基原物种溯源;故对于ITS2序列比对结果为其它科属物种的市场收集的药材样品,本研究只认为其为混伪品,而无需明确其基原物种,因此该方法适用于禹州漏芦和漏芦药材的真伪检测。随着中药材DNA条形码鉴定系统数据库的不断完善,对于药材检测的物种鉴定结果将更加稳定可靠。
5.3 中药材DNA提取方法的选择
DNA提取是中药材DNA条形码鉴定的首要环节,能否获得符合要求的高质量DNA是决定DNA条形码鉴定能否成功的关键因素[20]。常用的动植物总DNA提取方法有试剂盒法和CTAB法。试剂盒法具有快速、方便、避免有机溶剂污染等优点,但是成本偏高;且根据曹琳等[21]和罗焜等[22]等的研究,试剂盒对DNA降解严重的中药材不太适用。本研究采用改良的CTAB法配制DNA提取试剂,用于提取禹州漏芦和漏芦两种根茎类药材及其叶片的DNA,序列扩增成功率为100%,测序峰图质量高,这表明改良的CTAB法适用于根茎类药材及其叶片的DNA提取,且大大降低了获取高质量DNA的成本。随着DNA条形码鉴定中药材技术的不断普及,运用改良的CTAB法提取药材DNA的方法将实现标准化流程。

图2 基于ITS2序列的市场收集的禹州漏芦和漏芦药材及其混伪品NJ树
5.4 DNA条形码技术对市售药材进行监管的可行性
本研究对市场流通的禹州漏芦和漏芦药材真伪鉴定结果表明,两种药材均存在混伪情况,其中漏芦药材尤为严重。市售药材品种混乱给中药安全和准确使用造成极大隐患,传统鉴定方法已经难以满足药店、医院、企业等各行业对药材进行快速、标准化鉴定的需求,建立新的检测方法对市售药材真伪进行监管十分必要和迫切[23,24]。DNA条形码技术在物种鉴定上具有快速、准确、高效和通用性等特点,并且不受药材生长年限、药用部位、植物性状和研究者鉴别经验等因素的影响,基于通用的标准化技术流程可以实现对中药材真伪的快速、准确鉴定,非常适用于对市售药材的真伪进行监管。中药材DNA条形码分子鉴定指导原则已经被纳入《中华人民共和国药典》,为中药材基原物种的鉴定提供了强有力的技术支撑[25,26]。目前,DNA条形码技术已成功用于枸杞子、羌活、红景天、矮地茶和老鹳草等药材的真伪检测[27-29]。随着中药材DNA条形码鉴定系统的推广应用,DNA条形码技术将满足不同行业人员对中药材快速、标准化真伪鉴定的需求,实现对市售药材的快速、准确鉴定,为保障市售药材质量和临床用药安全提供技术支持。致谢:感谢新疆维吾尔自治区中药民族药研究所贾晓光研究员和卿德刚研究员为本研究提供部分实验材料!
1 国家药典委员会.中华人民共和国药典(一部).北京:中国医药科技出版社, 2015∶ 260-261.
2 金文荣,翟海斌.华东南刺头化学成分及其生物活性研究.杭州:浙江大学硕士学位论文, 2008∶ 1-60.
3 帕尔哈提·柔孜,哈勒木别克·木哈买提江,波拉提·马卡比力,等.我国蓝刺头属植物分布介绍.中国民族医药杂志, 2015, 4∶ 35-37.
4 国家药典委员会.中华人民共和国药典(一部).北京:中国医药科技出版社, 2015∶ 370-371.
5 帕尔哈提·柔孜,波拉提·马卡比力,吾古力汗·努尔哈别克,等.蓝刺头属植物在我国传统医药中的应用与研究概况.中国中药杂志, 2014, 39(19)∶ 3865-3869.
6 包小妹,罗素琴.祁州漏芦化学成分和药理学研究进展.亚太传统医药, 2011, 7(9)∶ 176-177.
7 李喜凤,王优湑,安硕,等.禹州漏芦抗炎有效部位的谱效关系研究.中国实验方剂学杂, 2014, 20(18)∶ 137-141.
8 杨美珍,王晓琴,刘勇,等.祁州漏芦化学成分与药理活性研究.中成药, 2015, 37(3)∶ 611-618.
9 汪毅,李铣,张鹏.禹州漏芦化学成分及药理活性的研究进展.中草药, 2005, 36(2)∶ 309-311.
10 Chen S L, Yao H, Han J P, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plants species. PLoS One, 2010, 5(1)∶ e8613.
11 Yao H, Song J Y, Liu C, et al. Use of ITS2 region as the universal DNA barcode for plants and animals. PLoS One, 2010, 5(10)∶ e13102.
12 夏叶,周红,向丽,等.ITS2序列鉴定“功劳叶”药材及其近缘混伪品.世界科学技术-中医药现代化, 2014, 16(11)∶ 2361-2365.
13 魏蒙,邬兰,凃媛,等.基于ITS2序列鉴定牡丹皮药材及其混伪品.中国中药杂志, 2014, 39(12)∶ 2180-2183.
14 凃媛,赵博,温放,等.基于ITS2序列鉴定南北葶苈子药材及其混伪品.世界科学技术-中医药现化, 2014, 16(2)∶ 288-294.
15 王俊,刘霞,张雅琴,等.苍耳子药材及其混伪品ITS2序列鉴定研究.世界科学技术-中医药现代化, 2014, 16(2)∶ 329-334.
16 Garnatje T, Vallès J, Garcia S, et al. Genome size in Echinops L.and related genera (Asteraceae, Cardueae):karyological, ecological and phylogenetic implications. Biol Cell, 2004, 96(2)∶ 117-124.
17 Noor M A, Kliman R M, Machado C A. Evolutionary history of microsatellites in the obscura group of Drosophila. Mol Biol Evol, 2001, 18(4)∶ 551-556.
18 Hidalgo O, Garcia-Jacas N, Garnatje T, et al. Phylogeny of Rhaponticum (Asteraceae, Cardueae-Centaureinae) and related genera inferred from nuclear and chloroplast DNA sequence data:taxonomic and biogeographic implications. Ann Bot, 2006, 97(5)∶ 705-714.
19 陈士林,庞晓慧,姚辉,等.中药DNA条形码鉴定体系及研究方向.世界科学技术-中医药现代化, 2011, 13(5)∶ 747-754.
20 Gao T, Yao H, Song J Y, et al. Identification of medicinal plants in the family Fabaceae using a potential DNA barcode ITS2. J Ethnopharmacol, 2011, 130(1)∶ 116-121.
21 曹琳,刘忠权,郝明干,等.不同种类中药材的DNA提取方法.中国现代应用药学杂志, 2004, 21(6)∶ 465-468.
22 罗焜,马培,姚辉,等.中药DNA条形码鉴定中的DNA提取方法研究.世界科学技术-中医药现代化, 2012, 14(2)∶ 2011-1439.
23 陈士林,苏钢强,邹健强,等.中国中药资源可持续发展体系构建.中国中药杂志, 2005, 30(15)∶ 1141-1146.
24 陈士林,郭宝林,张贵军,等.中药鉴定学新技术新方法研究进展.中国中药杂志, 2012, 37(8)∶ 1043-1055.
25 陈士林,姚辉,韩建萍,等.中药材DNA条形码分子鉴定指导原则.中国中药杂志, 2013, 38(2)∶ 141-148.
26 辛天怡,雷美艳,宋经元.中药材DNA条形码鉴定研究进展.中国现代中药, 2015, 17(2)∶ 170-176.
27 辛天怡.《中国药典》中药材DNA条形码分子鉴定研究—以羌活、枸杞子、红景天为例.北京:北京协和医院硕士学位论文, 2014∶1-83.
28 答国政,张秀桥,姚辉,等.基于ITS2序列的矮地茶药材基因识别.中药材, 2015, 38(11)∶ 2277-2280.
29 贺海波,熊超,郭力城,等.ITS2序列鉴定多基原药材老鹳草.中国药学杂志, 2015, 50(17)∶ 1505-1511.
Identification of Echinopsis Radix and Rhapontici Radix Using ITS2 Sequences
Chen Jiangping1, Hou Dianyun2, Yan Xuhua3, Hu Zhiqiang4, Wei Meng1, Hu Zhigang1,5, Wang Leyuan1
(1. College of Pharmacy, Hubei University of Chinese Medicine, Wuhan 430065, China; 2. College of Agricultural, Henan University of Science and Technology, Luoyang 471023, China; 3. Wuhan City Chinese Medicine Hospital, Wuhan 430010, China; 4. First Hospital of Wuhan, Wuhan 430022, China; 5. Institute of Chinese Materia Medica, China Academy of Chinese Medical Sciences, Beijing 100700, China)
This study aimed to identify Echinopsis Radix and Rhapontici Radix using ITS2 sequences, in order to provide a reliable method for clinical accurate medication by gene recognition. Fourteen samples of Echinops latifolius Tausch and E. grijisii Hance from the origin-species of multi origin traditional Chinese medicine Echinopsis Radix, and eight samples of Rhaponticum uniflorum (L.) DC from the origin-species of Rhapontici Radix, were collected. ITS2 sequences were acquired based on standard experiments, while DNA barcoding system for the materials was established through combining twenty sequences of relative adulterants from GenBank. Based on DNA barcoding system, we detected eleven samples of Echinopsis Radix and twenty samples of Rhapontici Radix from the market. As a result, the sequences of two origin-species of Echinopsis Radix shared the same dominant haplotype. The maximum intra-specific K2P genetic distance between Echinopsis Radix and Rhapontici Radix was less than the minimum inter-specific K2P genetic distance. The analysis of intra-specific and genetic divergence and NJ tree showed that Echinopsis Radix and Rhapontici Radix were separated from their adulterants obviously. Among eleven samples of Echinopsis Radix, ten of them were genuine while the remaining one was fake. However, among twenty samples of Rhapontici Radix, two were genuine and the remaining eighteen were fake. It was concluded that, in this study, the DNA barcoding technique for identification of Echinopsis Radix and Rhapontici Radix was successfully established on the basis of ITS2 sequences, which was a powerful tool in distinguishing Chinese medicines in the marketplaces.
Echinopsis Radix, Rhapontici Radix, ITS2 sequences, gene recognition, adulterants
10.11842/wst.2016.02.010
R282.5
A
(责任编辑:马雅静 张志华,责任译审:朱黎婷 王 晶)
2015-11-29
修回日期:2015-12-07
* 科学技术部国家重大新药创制科技专项子课题(2014ZX09304307001-021):中药新药安全性检测技术与标准研究-中药原料药混伪品分子基因鉴定技术与标准,负责人:胡志刚;湖北省科技支撑计划(研发与示范类)(2015BCA275):茯苓等8种湖北优势中药材DNA条形码分子鉴定体系构建,负责人:胡志刚。
** 通讯作者:胡志刚,博士,副教授,主要研究方向:中药资源与分子鉴定;汪乐原,教授,主要研究方向:中药资源与品质研究。

