兰州鲇G-SSR和EST-SSR在鲇形目和鲤形目鱼类中的通用性研究
杨忠礼,连总强,吴旭东,3,俞兆曦,王 燕,肖 伟,赛清云(.甘肃农业大学 动物科学技术学院,甘肃 兰州730070; .宁夏回族自治区水产研究所,宁夏 银川75000; 3.宁夏渔业工程技术研究中心,宁夏 银川75000)
兰州鲇G-SSR和EST-SSR在鲇形目和鲤形目鱼类中的通用性研究
杨忠礼1,2,连总强2,3,吴旭东1,2,3*,俞兆曦1,王 燕2,肖 伟2,3,赛清云2,3
(1.甘肃农业大学 动物科学技术学院,甘肃 兰州730070; 2.宁夏回族自治区水产研究所,宁夏 银川750001; 3.宁夏渔业工程技术研究中心,宁夏 银川750001)
为研究兰州鲇G-SSR和EST-SSR在鲇形目和鲤形目鱼类中的通用性,以12种不同鱼类为试验材料,分别采用改进的酚-氯仿法结合DNA产物纯化试剂盒法、传统酚-氯仿法、试剂盒法提取其尾鳍基因组DNA,对提取的DNA进行琼脂糖凝胶电泳、紫外分光光度计检测。结果表明,改进的酚-氯仿法结合DNA产物纯化试剂盒法提取的鱼类基因组DNA 在纯度和稳定性方面明显优于传统酚-氯仿法、试剂盒法,且电泳条带清晰、整齐、明亮,操作简单,耗时短,便于快速、批量提取。利用兰州鲇14对G-SSR引物和21对EST-SSR引物在12种鱼类中进行跨目通用性分析,结果显示,兰州鲇G-SSR和EST-SSR在鲇形目、鲤形目鱼类的通用率分别为63.10%、32.14%和61.11%、44.44%,表明随着物种间亲缘关系变远,其通用性随之降低。
基因组DNA; 兰州鲇; 鲇形目; 鲤形目; G-SSR; EST-SSR; 通用性
随着分子生物学的快速发展,基于PCR技术从核酸水平对鱼类的起源进化、种质资源鉴定、遗传多样性评估等研究,是目前鱼类分子生物学研究的有效手段。获得高质量的DNA是这些分子生物学研究的基础。目前,关于鱼类基因组DNA提取已有较多报道[1-5],主要有3种常见方法:传统的酚-氯仿抽提法[6-7]、高盐沉淀法[8-9]、离心柱型试剂盒法[10]。传统的酚-氯仿抽提法消化时间长,操作步骤繁琐且稳定性差;高盐沉淀法操作简便快捷,但所提取的DNA纯度及稳定性较差[11];离心柱型试剂盒提取效果较好,可以最大限度地除去蛋白质、脂肪、及其他有机化合物对DNA的污染,但由于试剂的限制(保存、运输、安全等因素),DNA提取量较少,且价格较贵[12]。一些对DNA质量要求高的试验,如构建基因组文库、简化基因组测序、Southern杂交等,传统酚-氯仿法和高盐法提取的DNA往往达不到其质量要求。基于以上3种方法的缺点和科研的需求,急需开发一种快捷、廉价且稳定性高的DNA提取方法。
微卫星(microsatellites)又称简单重复序列(simple sequence repeats,SSR),是由1~6个碱基串联重复排列的DNA序列。根据其来源分为基因组SSR(genomic SSR,G-SSR)和表达序列标签SSR(expressed sequence tag,EST-SSR)。微卫星标记因其具有共显性遗传、重复性好、多态性高等优点,而广泛应用于鱼类遗传多样性分析、亲缘关系鉴定和遗传连锁图谱构建等方面的研究[13-14]。研究表明,微卫星的侧翼序列在近缘物种之间较为保守,具有通用性,根据这一特性,已有不少科研工作者利用近缘物种的微卫星开发出目标物种的微卫星标记[14-16]。此外,有报道称某些微卫星位点在亲缘关系较远的物种间也具有一定通用性[17]。关于同一物种的G-SSR和EST-SSR在其他物种中的通用性报道较少,缺乏系统性比较研究。为此,本研究以12种不同鱼类的尾鳍为试验材料,用改进的酚-氯仿法抽提其基因组DNA,对兰州鲇(Siluruslanzhouensis)的G-SSR和EST-SSR在亲缘关系较近的鲇形目(Siluriformes)鱼类和亲缘关系较远的鲤形目(Cypriniformes)鱼类中进行通用性研究,旨在研发一种操作简单、提取效果好、稳定性高、便于批量提取的DNA提取方法,同时为兰州鲇G-SSR和EST-SSR在其他物种间的通用性研究奠定基础。
1 材料和方法
1.1 材料
试验所用12种鱼分属2个目,5个科,10个属(表1)。将活鱼带回实验室处死,剪取部分尾鳍组织浸泡在无水乙醇中,于-20 ℃保存。
主要试剂:组织裂解液(10 mmol/L Tris-HCl、 pH值8.0的0.1mol/L EDTA、pH值8.0的1% SDS)、20 mg/mL蛋白酶K、普通DNA产物纯化试剂盒及海洋动物基因组DNA提取试剂盒等均购自天根生化试剂公司;3 mol/L NaAc、Tris-饱和酚、氯仿、异戊醇等均为国产分析纯。
1.2 基因组DNA提取
分别采用改进的酚-氯仿法结合DNA产物纯化试剂盒法、传统酚-氯仿法、试剂盒法提取其尾鳍基因组DNA。
消化:将保存在无水乙醇中的兰州鲇尾鳍组织取出,剪取30 mg左右,用干净的滤纸将乙醇吸干后放置于1.5 mL离心管中,将组织剪碎后加入350 μL组织裂解液和8 μL蛋白酶K,充分混匀后放置在56 ℃水浴锅中消化3~4 h,期间每隔30 min将样品颠倒混匀1次,直至组织完全溶解并澄清。
苯酚-氯仿抽提:消化完成后加入1/5体积(约70 μL)的3 mol/L NaAc、等体积(约500 μL)的Tris-苯酚︰氯仿︰异戊醇混合液(25∶24∶1),上下颠倒充分混匀2~5 min,使核蛋白彻底变性裂解,DNA充分溶解。充分混匀后于室温下12 000 r/min离心10 min,吸取上清液200~250 μL至另一新的离心管中。
采用DNA纯化试剂盒进一步纯化:按照普通DNA产物纯化试剂盒说明对上述酚-氯仿法初步抽提的DNA进一步纯化,得到高纯度的DNA。由于DNA提取量较大,在洗脱步骤时,可以适当加大洗脱液用量,提高DNA得率。
用上述方法提取20尾兰州鲇基因组DNA并验证其稳定性,用同样的方法提取大口鲇、小口鲇、革胡子鲇、斑点叉尾鮰、黄颡、鲤鱼、草鱼、鲫鱼、白鲢、花鲢、大鼻吻鮈基因组DNA。此外,对4尾兰州鲇分别采用传统的酚-氯仿法[2]和海洋动物基因组DNA提取试剂盒提取其基因组DNA。
表1 试验用12种鱼类的分类及采样地

种名属名科名目名样品数量/尾采样地兰州鲇(S.lanzhouensis)鲇属(SilurusLinnaeus)鲇科(Siluridae)鲇形目(Siluriformes)20宁夏水产研究所科研基地大口鲇(S.meridionalis)鲇属(SilurusLinnaeus)鲇科(Siluridae)鲇形目(Siluriformes)1当地水产市场小口鲇(S.asotus)鲇属(SilurusLinnaeus)鲇科(Siluridae)鲇形目(Siluriformes)1当地水产市场革胡子鲇(C.fuscus)胡子鲇属(ClariasScopoli)胡子鲇科(Siluridae)鲇形目(Siluriformes)1当地水产市场斑点叉尾鮰(I.Punetaus)鮰属(Ictalurus)鮰科(Ictaluridae)鲇形目(Siluriformes)1宁夏水产研究所科研基地黄颡(P.fulvidraco)黄颡鱼属(Pelteobagrus)鲿科(Bagridae)鲇形目(Siluriformes)1当地水产市场鲤鱼(C.carpio)鲤属(Cyprinus)鲤科(Cyprinidae)鲤形目(Cypriniformes)1宁夏水产研究所科研基地草鱼(C.idellus)草鱼属(Ctenopharyngodon)鲤科(Cyprinidae)鲤形目(Cypriniformes)1宁夏水产研究所科研基地鲫鱼C.auratus)鲫属(Carassius)鲤科(Cyprinidae)鲤形目(Cypriniformes)1宁夏水产研究所科研基地白鲢(H.molitrix)鲢属(Hypophthalmichthys)鲤科(Cyprinidae)鲤形目(Cypriniformes)1宁夏水产研究所科研基地花鲢(A.nobilis)鳙属(Aristichthys)鲤科(Cyprinidae)鲤形目(Cypriniformes)1宁夏水产研究所科研基地大鼻吻鮈(R.nasutus)吻鮈属(Rhinogobio)鲤科(Cyprinidae)鲤形目(Cypriniformes)1内蒙古澄口
1.3 DNA提取结果检测
用1%琼脂糖凝胶电泳检测3种方法所提取的鱼类基因组DNA,电压120 V,室温下电泳30 min,用凝胶成像系统检测电泳结果,并拍照保存。DNA样品用紫外分光光度计检测其含量并计算A260/A280比值。检测完后根据需要将DNA稀释,并于-20 ℃保存备用。通过电泳图谱、DNA含量及其A260/A280比值判断不同提取方法的效果。
1.4 兰州鲇G-SSR和EST-SSR通用性检测
将采用改进方法提取的12种不同鱼类DNA稀释至50 ng/μL作为底物。14对兰州鲇G-SSR引物来自魏大为等[18]公布的序列,21对EST-SSR引物由笔者所在实验室积累,引物序列见表2。引物由生工生物工程(上海)股份有限公司合成。PCR反应体系20 μL:上、下游引物(10 μmol/L)各1 μL,2×PCR Master Mix 10 μL,模板DNA(50 ng/μL)2 μL,去离子水6 μL。PCR程序:94 ℃预变性 3 min;94 ℃变性 30 s,退火30 s,退火温度依引物而定,72 ℃延伸 1 min,34 个循环;72 ℃延伸5 min。扩增产物进行1.2%的琼脂糖凝胶电泳检测,凝胶成像系统拍照保存。统计每对引物在其他12种鱼类中的扩增情况,根据统计结果计算每对引物的有效扩增率,以及G-SSR型和EST-SSR型微卫星的平均有效扩增率。
表2 21对兰州鲇EST-SSR分子标记引物序列

位点引物序列(5′—3′)退火温度/℃JZ845705 F:TGTGCGAAAGGTGAAAGATTA R:AACTTGAATGGGAAGGAGGTG58JZ845090 F:CATAAAGTCTCCGTCAACC R:GTCATGGGAAATCAACAAT53JZ845784 F:ATCAATAAGCGTGGTGTCA R:GCTGTTTGCCTAACCTGA57JZ845762 F:TGTGAGTGATTGGTATGTGGT R:TTAATGCTGGGTCATGTAGAG59JZ845190 F:ACCTCCACTGAATCACAACA R:AAGACAGAAAAGAAAGAAACG56JZ845743 F:AGTGACTGCTAGTCCCAGAG R:GAGACGCAACATACAGAACC61JZ845100 F:ATCCCTGTATTTACCAGTCTCAC R:GCTTCACTTGGGTTCCTTTGT58JZ845703 F:CCAAATAGCGTGTAGTAGT R:CACTAAACTGTGAAGAAAGA55JZ845726 F:TATGAATGACCGTGGCAGAG R:CCAAGCGTAAGTTGAGGAA56JZ845789 F:ACGCAGACCAGACACCACC R:GCCAGCGGAGAAAGCAGT63JZ905272 F:GTAAGAGCGTCAGGGACTG R:GAATGTTGGTATGCTGGAAT62JZ905276 F:TCAAGCGTTCTGTGGGGTAT R:AAGTCGCATCCGTTCGTG59JZ905300 F:AGTATGTGAGTGCGAGA R:TAACGTAATGCAGGTAA53JZ905308 F:GGAAGTGATGTCGGTGTC R:CTGCTCGATGTAAAGAATGT59JZ905309 F:CCTGCTCCTTCCATCCTTT R:CGTTTACGCTCCGACTCTTAT59JZ905311 F:CCAGAATCCACCTTTGCCTAT R:GTCCTCCTGCTCCTTCCATC63JZ905314 F:TGACAGTGGAGGAGTATTGAGG R:ACAGGAAACCGAAGTGGAGT59JZ905328 F:TACATATTATGGCACTCGC R:AGATTGGTGAAACAGGCTA56JZ905334 F:CGCTCATTCCACATCCTT R:ACCACATCACCCAGTCCA61JZ905338 F:CAGGAAATGATGTGGGAGAA R:GAGACGAAGATGGGAAAGAGTA58JZ905348 F:TGTAAAGGCTGCGGTTCTC R:CCAATTTGTTGATGCTTAATGTA55
2 结果与分析
2.1 基因组DNA质量检测
由图1—3可见,酚-氯仿法和试剂盒法提取的DNA主条带模糊,有拖尾现象,表明DNA含量较低,降解严重。与传统的酚-氯仿法和试剂盒法相比,改进方法提取的基因组DNA质量较好,DNA主条带清晰、明亮、整齐、无拖尾,DNA含量高,纯度好,蛋白质、RNA和杂质污染少,且DNA降解程度低。
纯净的DNA样品 A260/A280介于1.8~2.0,小于1.8表明有蛋白质污染,大于2.0表明严重降解或含有大量RNA。由表3可以看出,传统酚-氯仿法、试剂盒提取的DNA其A260/A280比值偏大,表明DNA降解比较严重,与电泳检测结果一致。改进方法提取的DNA其A260/A280比值介于1.78~1.91、接近1.8,表明提取的DNA纯度较好。
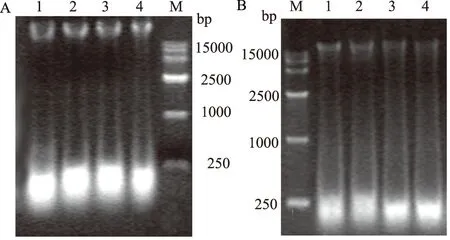
M:DL15000 Marker;1—4:兰州鲇基因组DNA; A:酚-氯仿法; B:试剂盒法
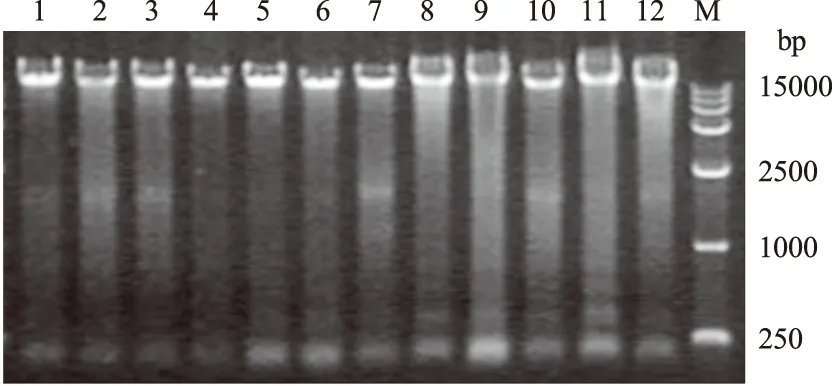
M:DL15000 Marker; 1—12:分别为兰州鲇、大口鲇、小口鲇、胡子鲇、斑点叉尾鮰、黄桑、鲤鱼、草鱼、鲫鱼、白鲢、花鲢、大鼻吻鮈的基因组DNA
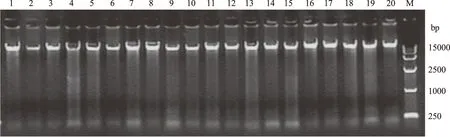
M:DL15000 Marker; 1—20:兰州鲇基因组DNA图3 改进方法提取的20尾兰州鲇基因组DNA结果
表3 不同方法提取的鱼类基因组DNA纯度及浓度

方法A260/A280平均含量/(ng/μL)含量范围/(ng/μL)酚-氯仿法2.02~2.14404.52±53.54349.4~468.8试剂盒法1.94~2.0689.67±13.4274.7~104.1改进的方法1.78~1.91216.96±71.3289.7~353.5
2.2 兰州鲇G-SSR和EST-SSR通用性
2.2.1 兰州鲇G-SSR和EST-SSR在12种鱼类中的扩增结果 由表4可以看出,14对G-SSR引物和21对EST-SSR引物均可在兰州鲇个体中扩增出特异性片段。兰州鲇G-SSR和EST-SSR在鲇形目鱼类中的通用率相近,EST-SSR在鲤形目鱼类中的通用率明显高于G-SSR;6个EST-SSR型位点JZ845705、JZ905272、JZ905276、Z905309、JZ905311、JZ905314在12种鱼类中均可获得特异性扩增,且扩增片段大小基本一致,在G-SSR型位点中不曾发现这种现象。2种类型的微卫星均表现出随物种间遗传距离的增加,其通用性下降(表5)。
表4 14个G-SSR和21个EST-SSR在12种鱼类的扩增结果
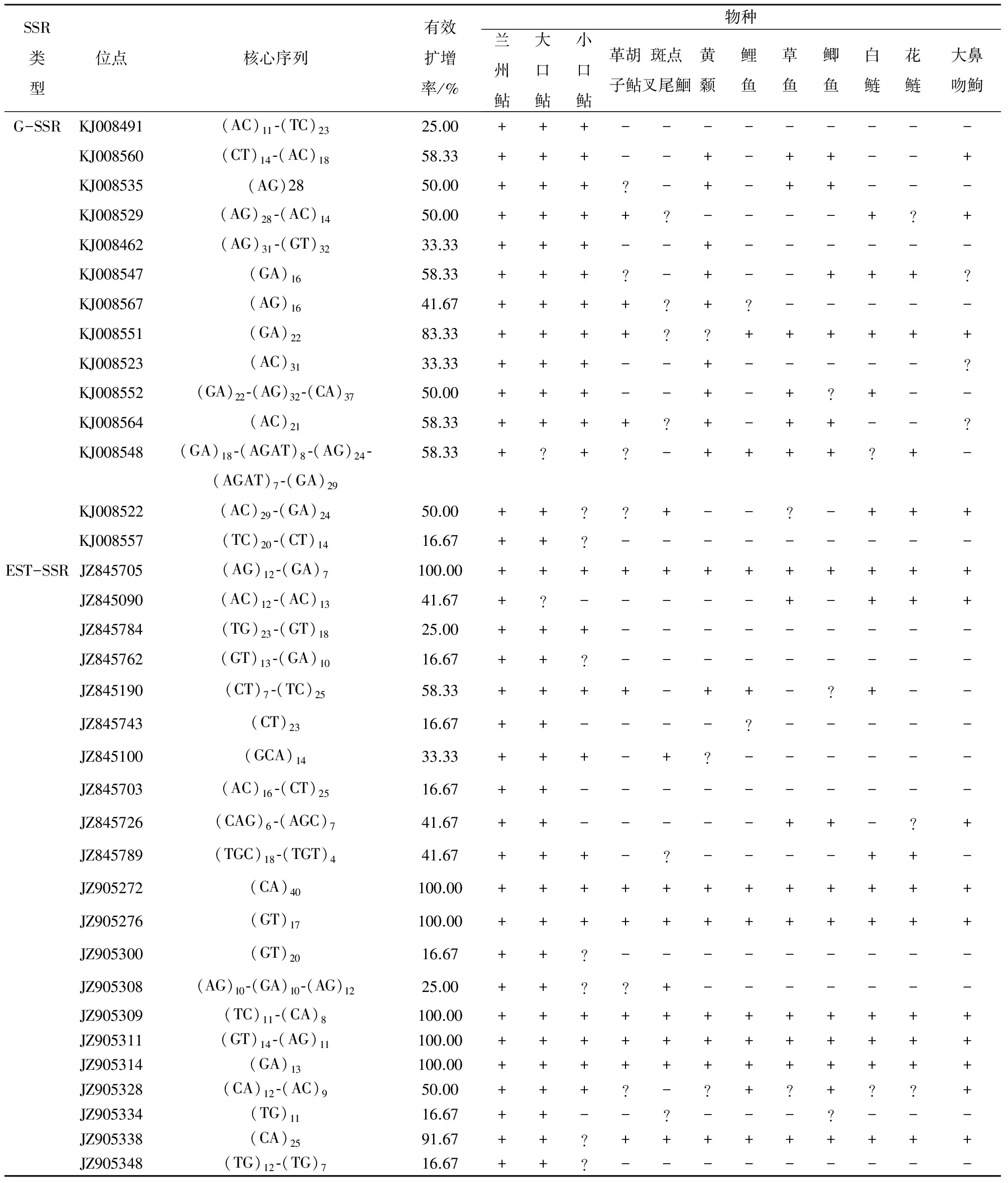
SSR类型位点核心序列有效扩增率/%物种兰州鲇大口鲇小口鲇革胡子鲇斑点叉尾鮰黄颡鲤鱼草鱼鲫鱼白鲢花鲢大鼻吻鮈G-SSRKJ008491(AC)11⁃(TC)2325.00+++---------KJ008560(CT)14⁃(AC)1858.33+++--+-++--+KJ008535(AG)2850.00+++?-+-++---KJ008529(AG)28⁃(AC)1450.00++++?----+?+KJ008462(AG)31⁃(GT)3233.33+++--+------KJ008547(GA)1658.33+++?-+--+++?KJ008567(AG)1641.67++++?+?-----KJ008551(GA)2283.33++++??++++++KJ008523(AC)3133.33+++--+-----?KJ008552(GA)22⁃(AG)32⁃(CA)3750.00+++--+-+?+--KJ008564(AC)2158.33++++?+-++--?KJ008548(GA)18⁃(AGAT)8⁃(AG)24⁃58.33+?+?-++++?+-(AGAT)7⁃(GA)29KJ008522(AC)29⁃(GA)2450.00++??+--?-+++KJ008557(TC)20⁃(CT)1416.67++?---------EST-SSRJZ845705(AG)12⁃(GA)7100.00++++++++++++JZ845090(AC)12⁃(AC)1341.67+?-----+-+++JZ845784(TG)23⁃(GT)1825.00+++---------JZ845762(GT)13⁃(GA)1016.67++?---------JZ845190(CT)7⁃(TC)2558.33++++-++-?+--JZ845743(CT)2316.67++----?-----JZ845100(GCA)1433.33+++-+?------JZ845703(AC)16⁃(CT)2516.67++----------JZ845726(CAG)6⁃(AGC)741.67++-----++-?+JZ845789(TGC)18⁃(TGT)441.67+++-?----++-JZ905272(CA)40100.00++++++++++++JZ905276(GT)17100.00++++++++++++JZ905300(GT)2016.67++?---------JZ905308(AG)10⁃(GA)10⁃(AG)1225.00++??+-------JZ905309(TC)11⁃(CA)8100.00++++++++++++JZ905311(GT)14⁃(AG)11100.00++++++++++++JZ905314(GA)13100.00++++++++++++JZ905328(CA)12⁃(AC)950.00+++?-?+?+??+JZ905334(TG)1116.67++--?---?---JZ905338(CA)2591.67++?+++++++++JZ905348(TG)12⁃(TG)716.67++?---------
注:“+”表示有特异性扩增条带;“-”表示无特异性扩增条带;“?”表示非特异性扩增或扩增条带很弱难以判断。
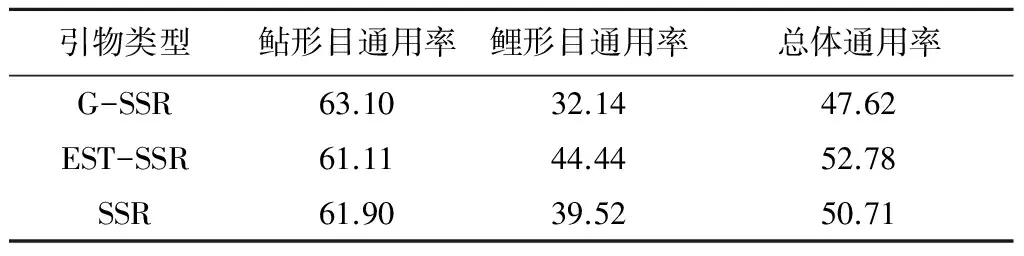
表5 兰州鲇G-SSR和EST-SSR在鲇形目鱼类和鲤形目鱼类中的通用率 %
2.2.2 G-SSR和EST-SSR扩增图谱分析 根据兰州鲇G-SSR和EST-SSR在12种不同鱼类的扩增图谱(图4)发现,G-SSR比EST-SSR在不同物种间具有更高长度变异性,如G-SSR型微卫星位点KJ008547在白鲢和花鲢中的扩增条带明显偏大。
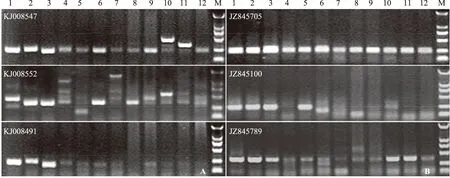
M:DL2000 Marker; 1—12:分别为以兰州鲇、大口鲇、小口鲇、革胡子鲇、斑点叉尾鮰、黄颡、鲤鱼、草鱼、鲫鱼、白鲢、花鲢、大鼻吻鮈基因组DNA为底物的PCR产物图4 部分兰州鲇G-SSR(A)和EST-SSR(B)PCR产物电泳检测结果
3 结论与讨论
高质量的DNA提取有4个关键:对组织和细胞进行有效破碎,使核蛋白释放出来;核蛋白充分变性,释放出DNA;使DNA酶失去活性,防止DNA被其酶解;尽量除去蛋白质、多糖等杂质污染。为此,在DNA提取过程中应尽量做到以上几点以确保提取到高质量的基因组DNA。传统的酚-氯仿法是提取动物基因组DNA最常用的方法,此方法为了最大限度除去蛋白质等杂质需多次抽提,每次抽提为了不吸到中间相杂质,需留下较多的上清液,抽提次数越多,DNA的损失量就越大;此外吸取上清对试验操作者的技能要求较高,对初学者是一种挑战;因整个试验流程较长,出错的可能性较大,造成提取效果不稳定。
本试验参考Turtinen等[19]和乐小亮等[5]的方法对传统的酚-氯仿法进行改进,在组织消化完毕后,加入高浓度的NaAc,有利于DNA的充分溶解,蛋白质变性析出。同时加入等体积的苯酚∶氯仿∶异戊醇混合物(25∶24∶1)对裂解液抽提1次,得到大量低纯度的基因组DNA,然后用基于吸附材料的离心柱型DNA纯化试剂盒对其纯化,得到高纯度的DNA。这种DNA提取方法减少了抽提次数,DNA的损失量大大减少,DNA得率较高,并且可最大限度地除去蛋白质等杂质。改进的方法中DNA纯化步骤主要采用离心的方式进行,便于批量化操作,且比传统酚-氯仿法操作简单,耗时少。与传统的酚-氯仿法相比较,改进方法成本有所提高,但提取效果却明显提高。与专业的DNA提取试剂盒相比较,成本大幅降低,并且提取效果优于试剂盒。此外,改进方法还具有需要样品量小、稳定性高、便于批量提取等优点。
关于微卫星的通用性在水生生物中的研究,前人已有报道[20-21]。本研究利用兰州鲇14对G-SSR引物和21对EST-SSR引物对来自鲇形目和鲤形目的12种鱼类进行通用性分析,结果显示:兰州鲇G-SSR和EST-SSR在鲇形目、鲤形目鱼类中的通用率分别为63.10%、32.14%和61.11%、44.44%,表明兰州鲇G-SSR和EST-SSR在近缘物种中有很高的通用性,但随着亲缘关系拉大,微卫星的通用性随之降低。
6个EST-SSR型位点JZ845705、JZ905272、JZ905276、Z905309、JZ905311、JZ905314在12种鱼类中均可获得特异性扩增,且扩增片段大小基本一致,在G-SSR型位点中不曾发现这种现象。此外,根据二者的扩增图谱发现,G-SSR比EST-SSR在不同物种间具有更高长度变异性,可见,基因组中编码序列要比非编码序列更为保守。本研究表明,兰州鲇G-SSR和EST-SSR在鲇形目鱼类中的通用率相近,但EST-SSR在鲤形目鱼类中的通用率明显高于G-SSR,这表明EST-SSR比G-SSR在物种间通用性好,这一结果与Liewlaksaneeyanawin等[22]和宿俊吉等[23]的报道结果一致。兰州鲇G-SSR和EST-SSR在12种鱼类的通用率分别为47.62%和52.78%,具有较高的通用性,当缺乏微卫星标记时,借用近缘物种的微卫星是一种更为简单、快捷、廉价的方法[15]。
本研究利用酚-氯仿法结合DNA纯化试剂盒提取鱼类基因组DNA效果较好,具有稳定性高、操作简单、节约时间和成本低等优点,非常适合一些样品珍贵、需批量提取的科学研究试验。此外,本研究利用兰州鲇微卫星对来自鲇形目和鲤形目的12种鱼类进行跨目通用性分析,结果显示,随着物种间亲缘关系变大,其通用性逐渐降低,但部分微卫星在亲缘关系较远的物种间仍具有很好的通用性。这些研究结果为微卫星在跨目物种间的通用性研究及利用微卫星开展比较基因组学等方面的研究提供基础资料。
[1] 吴旭东,张奇,侯玉霞,等.黄河鲶基因组DNA提取与纯化方法的研究[J].淡水渔业,2006,36(1):19-21.
[2] 马洪雨,郭金峰,岳永生.用改进的酚-氯仿法提取鱼类基因组DNA效果的分析[J].家畜生态学报,2006,27(2):85-87.
[3] 范武江,王晓清,杨品红,等.鳙鱼不同组织基因组DNA提取方法的探讨[J].南方水产,2007,3(1):44-47.
[4] Aljanabi S M,Martinez I.Universal and rapid salt-extraction of high quality genomic DNA for PCR-based techniques[J].Nucleic Acids Research,1997,25(22):4692-4693.
[5] 乐小亮,章群,赵爽,等.一种高效快速的鱼类标本基因组DNA提取方法[J].生物技术通报,2010(2):202-204.
[6] Sambrook J,Fritsch E F,Maniatis T.分子克隆实验指南[M].2版.金冬雁,黎孟枫,译.北京:科学出版社, 1992:463-469.
[7] Poncz M,Solowiejczyk D,Harpel B,etal.Construction of human gene libraries from small amounts of peripheral blood:Analysis of β-Like globin genes[J].Hemoglobin, 2009,6(1):27-36.
[8] Miller S A,Dykes D D,Polesky H F.A simple salting out procedure for extracting DNA from human nucleated cells[J].Nucleic Acids Research,1988,16(3):1215.
[9] Salazar L A,Hirata M H,Cavalli S A,etal.Optimized procedure for DNA isolation from fresh and cryopreserved clotted human blood useful in clinical molecular testing[J].Clinical Chemistry,1998,44(1):1748-1750.
[10] 李进波,盛婧,李想,等.五种DNA提取方法对鱼加工制品DNA提取效果的比较[J].生物技术通报,2014(4):43-49.
[11] 鲍毅新,孙波,张龙龙,等.对动物组织DNA提取方法的改进及PCR检测[J].浙江师范大学学报(自然科学版),2009,32(3):317-321.
[12] 刘哲,康鹏天,柴文琼,等.鱼类血液基因组DNA提取方法优化[J].水生态学杂志,2009,2(6):102-106.
[13] Liu Z J,Cordes J F.DNA marker technologies and their applications in aquaculture genetics[J].Aquaculture,2004, 238(1):1-37.
[14] 林能锋,苏永全,丁少雄,等.大黄鱼微卫星标记引物在石首鱼科几个近缘种中的通用性研究[J].中国水产科学,2008,15(2):237-243.
[15] 吴旭东,连总强,侯玉霞,等.大口鲇微卫星标记在三个鲇形目鱼类种群间适用性研究[J].水生生物学报, 2011,35(4):638-645.
[16] 吴雪萍,马海涛,冯艳微,等.缢蛏(Sinonovaculaconstricta)微卫星标记的分离及近缘物种通用性[J].海洋与湖沼,2014,45(6):1330-1337.
[17] Rico C,Rico I,Hewitt G.470 million years of conservation of microsatellite loci among fish species[J].Proceedings of the Royal Society B(Biological Sciences),1996,263:549-557.
[18] 魏大为,连总强,吴旭东,等.磁珠富集法筛选兰州鲇微卫星分子标记[J].水生生物学报,2014,38(4):791-796.
[19] Turtinen L W,Juran B D.Protein salting-out method applied to genomic DNA isolation from fish whole blood[J].Biotechniques,1998,24:238-239.
[20] Varshney R K,Sigmund R,Börner A,etal.Interspecific transferability and comparative mapping of barley EST-SSR markers in wheat, rye and rice[J].Plant Science,2005,168(1):195-202.
[21] 董迎辉,吴国星,姚韩韩,等.泥蚶34个EST-SSR标记的开发及在格粗饰蚶中的通用性检测[J].水产学报,2013,37(1):70-77.
[22] Liewlaksaneeyanawin C,Ritland C E,El-Kassaby Y A,etal.Single-copy, species-transferable microsatellite markers developed from loblolly pine ESTs[J].Theoretical and Applied Genetics,2004,109(2):361-369.
[23] 宿俊吉,柴守诚,刘伟华,等.普通小麦SSR和EST-SSR引物对冰草通用性的比较分析[J].西北植物学报,2007,27(7):1311-1316.
Transferability Analysis ofSiluruslanzhouensisG-SSR and EST-SSR Markers in Siluriformes and Cypriniformes Fishes
YANG Zhongli1,2,LIAN Zongqiang2,3,WU Xudong1,2,3*,YU Zhaoxi1,WANG Yan2,XIAO Wei2,3,SAI Qingyun2,3
(1.College of Animal Science and Technology,Gansu Agricultural University,Lanzhou 730070,China; 2.Ningxia Fisheries Research Institute,Yinchuan 750001,China; 3.Ningxia Engineering Research Center for Fisheries,Yinchuan 750001,China)
In order to investigate the transferability ofSiluruslanzhouensisG-SSR and EST-SSR in Siluriformes and Cypriniformes,12 different fishes were used as research objectives to extract genomic DNA by using improved phenol-chloroform method,which combined with DNA purification kit,traditional phenol-chloroform method and DNA extraction kit,respectively.The genomic DNA was detected by agarose gel electrophoresis and ultraviolet spectrophotometer.The results showed that the genomic DNA extracted by the improved phenol-chloroform method was obviously superior to the other two methods in purity and stability,and the electrophoresis bands were very clear,neat and bright.The method had the advantages of simple manipulating,short time-consuming,speediness and batch extraction.Furthermore,14 G-SSRs and 21 EST-SSRs ofSiluruslanzhouensiswere selected to analyze the transferability of 12 Siluriformes and Cypriniformes fishes.The results indicated that the transferable rates of 14 G-SSRs and 21 EST-SSRs in Siluriformes and Cypriniformes fishes were 63.10%,32.14% and 61.11%,44.44% respectively,revealing that the transferability decreased with the increase of genetic relationship among species.
genomic DNA;Siluruslanzhouensis; Siluriformes; Cypriniformes; G-SSR; EST-SSR; transferability
2015-12-28
国家自然科学基金项目(31360633);宁夏对外科技合作项目;国家科技支撑计划项目(2012BAD25B09)
杨忠礼(1990-),男,甘肃合水人,在读硕士研究生,研究方向:淡水经济动物生物学及增养殖。 E-mail:yang2009191048@163.com
*通讯作者:吴旭东(1967-),男,宁夏银川人,研究员,博士,主要从事水生动物分子生物学及种质资源保护与增养殖研究。E-mail:amy95@126.com
Q346+.5
A
1004-3268(2016)06-0130-07

