转cry1Ie基因抗虫玉米对土壤中细菌群落结构的影响
叶慧香, 崔跃原, 宋新元, 马 娟, 万 虎, 李建洪*
1华中农业大学植物科学与技术学院,湖北 武汉 430070; 2吉林省农业科学院,
农业部转基因植物环境安全监督检验测试中心,吉林 长春 130124
转cry1Ie基因抗虫玉米对土壤中细菌群落结构的影响
叶慧香1, 崔跃原1, 宋新元2, 马娟1, 万虎1, 李建洪1*
1华中农业大学植物科学与技术学院,湖北 武汉 430070;2吉林省农业科学院,
农业部转基因植物环境安全监督检验测试中心,吉林 长春 130124
摘要:【背景】土壤微生物是维持土壤生态系统中生物活性的重要组成部分,土壤细菌作为土壤中数量最丰富、分布最广泛的微生物类群,其结构多样性和动态分布对土壤生态系统的稳定具有重要意义。因此,评价转基因作物的安全性必须考虑转基因产物对土壤细菌的影响。【方法】在大田条件下,连续2年以转cry1Ie基因抗虫玉米和受体对照玉米为研究材料,采用变性梯度凝胶电泳(PCR-DGGE)和磷脂脂肪酸(PLFA)研究转cry1Ie基因抗虫玉米和受体对照玉米不同生育期根际土壤中细菌群落结构的变化。【结果】PLFA分析结果显示,2012年,转cry1Ie基因抗虫玉米和受体对照玉米间微生物总量在拔节期有显著性差异,其他时期无显著性差异;然而,土壤中细菌微生物总量和革兰氏阳性菌与革兰氏阴性菌微生物量比值在检测的4个时期均无显著性差异;其中,革兰氏阳性菌与革兰氏阴性菌比值均大于1。2013年的整个生育期内,转cry1Ie基因抗虫玉米和受体对照的微生物总量、细菌微生物量、革兰氏阳性菌与革兰氏阴性菌微生物量比值均无显著性差异。DGGE结果显示,2012年和2013年玉米的4个生育时期内,玉米根际土壤细菌群落结构相对较稳定,同一生育期转cry1Ie基因抗虫玉米和受体对照玉米间无显著性差异,且根际细菌群落结构相似性均达到较高水平,与PLFA检测结果大体一致。【结论与意义】2年的数据均表明,转cry1Ie基因抗虫玉米对土壤中细菌群落结构无显著性差异,可为转cry1Ie基因抗虫玉米安全性评价提供理论依据。
关键词:磷脂脂肪酸分析; 变性梯度凝胶电泳; 根际; 细菌总量; 革兰氏阳性菌与革兰氏阴性菌生物量比值
自20世纪70年代重组DNA技术建立并于1983年首次获得转基因植物以来(Lal & Lal,1991; Zambryske,1983),转基因作物的开发和应用取得了突飞猛进的发展。根据国际农业生物技术应用服务组织(ISAAA)最新报道,2013年全球转基因作物种植面积达1.75亿hm2,较2012年增长3%,在转基因作物商业化种植的18年间,其种植面积增长了100倍(James,2013)。其中转基因玉米种植面积达5800万hm2,转Bt玉米种植面积占1/4。随着转Bt玉米的长期种植,其中的Bt蛋白可通过根系分泌物、秸秆还田、残茬分解及花粉飘落等方式在土壤中积累和富集,可能影响土壤微生物区系的组成和结构,改变微生物的多样性(Fanetal.,2001; Gaughey & Whalon,1992)。研究发现,Bt蛋白进入土壤后,可与土壤粘粒和腐殖酸迅速结合且不易分离,并可抵抗土壤微生物的降解(Tapp,1994; Tapp & stotzky,1998、1999)。
微生物种类多、生长繁殖快、代谢能力强、对环境影响敏感、与其他生物具有同一套遗传密码,上述特性决定了微生物是环境中最敏感的指示生物之一(昌艳萍等,2011),其活性和群落结构的变化能反映土壤生态系统的质量和健康状况(黎宁等,2006; Knightetal.,1997; Zelles,1999)。因此,研究转基因作物对土壤微生物的影响对转基因作物的安全性具有重要意义。一些学者提出并研究了转基因植物环境释放后土壤微生物群落的变化(Angle,1994; Jepsonetal.,1994),Fangetal.(2005)通过变性梯度凝胶电泳(Denaturing gradient gel electrophoresis,DGGE)和Biolog分子生物学方法研究发现,转Bt基因玉米显著影响土壤微生物群落结构。Liuetal. (2008)应用DGGE和末端限制性片段长度多态性(Terminal restriction fragment length polymorphism,T-PRLP)技术,通过3年的田间试验,检测了转Bt基因和非转基因水稻根际细菌、真菌群落多样性,结果表明,水稻生育期显著影响根际微生物多样性,但转cry1Ab基因水稻对土壤中细菌、真菌无显著影响。刘丽等(2010)通过随机扩增多态性DNA(Random amplified polymorphic DNA,RAPD)分子标记技术结合传统的平板计数培养等方法研究发现,转抗菌肽D烟草可能抑制病原细菌及其根围相关的微生物的繁殖,但不影响微生物的遗传多样性。李长林等(2008)通过PCR、T/A克隆、RFLP等技术发现,转基因棉花对根际土壤微生物多样性无影响,棉花根际土壤微生物区系差异主要受生育期影响。
目前,国内外研究转Bt玉米对土壤微生物的影响多数在温室内或人工培养的条件下进行,田间研究相对较少,难以真实反映转Bt玉米与根际微生物间的关系。本研究通过连续2年田间试验,以转cry1Ie基因抗虫玉米和受体对照玉米为研究材料,分析和比较了2种玉米在同1个生育期内以及整个生育期内对土壤细菌的影响,为研究转cry1Ie基因抗虫玉米对土壤细菌群落结构影响提供理论依据。
1材料与方法
1.1 材料
转cry1Ie基因抗虫玉米和对照玉米由中国农业科学院提供,于2012年和2013年连续2年种植。
1.2 试验小区设计
试验地设在中国农业科学院河北省廊坊转基因植物环境安全检测试验基地。基地地势平坦,小区面积225 m2(15 m×15 m),随机区组排列,小区间设有1 m走道和3 m保护行。试验设3次重复。常规耕作管理,整个生育期内不使用杀虫剂。
1.3 样品采集
在玉米的播种前期、拔节期、抽丝期和成熟期分别采集土壤样品。播种前取地表下15~20 cm土壤;其他各时期以抖落法取根际土壤(Watrud & Seidler,1998)。每个小区采用S型取样法选取6点,剔除土表杂物,每点分别取土样约300 g(若因单株的根系抖落法难以取到300 g,可在每点同时取几棵植株的混合土壤作为样本)。取样后立即分样放入冰盒中并储存于实验室。土样平均分成2份,一份储存于4 ℃冰箱用于PCR-DGGE检测,另一份储存于-80 ℃用于测定PLFA。
1.4 土壤总DNA的提取
采用Soil DNA Kit试剂盒(Omega公司),分别称取混合好的土样0.5 g,按照操作说明书进行提取,土壤DNA经1.0%琼脂糖凝胶电泳检测样品质量后置于-20 ℃保存备用。
1.5 土壤总DNA的聚合酶链式反应(PCR)
采用巢式PCR扩增细菌16S rRNA V3区,第1次PCR所用引物P0(5′-GAGAGTTTGATCCTGGCTCAG-3′)和反向引物P6(5′-CTACGGCTACCTTGTTACGA-3′),片段长度1500 bp,第2次PCR所用引物338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和反向引物518R(5′-ATTACCGCGGCTG5′CTGG-3′),在正向引物的5′端添加“GC-clamp”(5′-CGCCCGCCGCGCGCGCGGCGGGCGGGGCGGGGGCCCGGGGG-3′)(Hareetal.,2005; Thompsonetal.,2002),片段长度230 bp。50 μL的PCR反应体系:0.25 μL Ex Tag polymerase,5 μL 10×PCR Buffer(Mg2+Plus),4 μL dNTP Mixture(各2.5 mmol·L-1),引物(20 μmol·L-1)各1 μL,第1次PCR所用模板为DNA 1 μL,第2次PCR所用模板为第1次PCR产物1 μL,无菌超纯水补足50 μL。PCR采用touch-down扩增程序:94 ℃预变性 5 min,94 ℃变性1 min,68 ℃退火1 min,依次每个循环降1 ℃,72 ℃延伸2 min,共10个循环;94 ℃变性1 min,58 ℃退火1 min,72 ℃延伸2 min,共20个循环,72 ℃延伸10 min。
1.6 16S rDNA V3区DGGE检测
采用Bio-Rad DGGE电泳系统,配制聚丙烯酰胺(37.5∶1)浓度8%、变性剂浓度分别为0%和100%的胶,借助旋转进样器配制成梯度浓度为40%~60%的胶,并插入梳子,凝胶3~4 h(可在空调下加热加速凝胶),同时预热缓冲液1×TAE(Tris-Ace-tata-EDTA)到60 ℃以上(周小奇等,2007)。将纯化后的16S rDNA V3区PCR产物20 μL点样,电泳温度60 ℃,200 V电泳5 min,待样品进入凝胶后60 V电泳16 h。电泳结束后,凝胶加入固定液(10%乙醇,0.5%冰乙酸)固定15 min,蒸馏水漂洗3次;加入染色液(0.2%硝酸银,0.1%甲醛)银染15 min,蒸馏水漂洗3次;加入显色液(15%氢氧化钠,0.5%甲醛)显色10 min并摄像(梁宏伟等,2008)得到DGGE图谱。
1.7 PLFA分析
取8 g土样,利用FAMEs法进行提取,将含有磷脂的提取液过柱分析,并将得到的磷脂甲酯化处理,以酯化C19∶0为内标,Agilent6890气相色谱仪检测分析,PLFA的鉴定采用Sherlock MIS 4.5系统(Bligh & Dyer,1959; Bossioetal.,1998; Frostegårdetal.,2010)。
1.8 数据分析
采用Sigmaplot 12.0和 SPSS19.0软件对试验数据进行方差分析(LSD法)。DGGE指纹图谱应用Quantity One软件分析,构建聚类树状图。应用SAS 18进行主成分分析。
2结果与分析
2.1 玉米根际土壤微生物的磷脂脂肪酸分析
2.1.1玉米根际微生物磷脂脂肪酸含量及组成对2年的玉米根际微生物总磷脂脂肪酸分析表明,2012年共分离了27种碳链长度在14~22的PLFAs,转cry1Ie基因抗虫玉米和对照根际PLFA总量均呈现先降低后升高再降低的趋势,在播前期、抽丝期和成熟期,转cry1Ie基因抗虫玉米和对照玉米土壤总微生物量无显著差异,但在拔节期显著低于对照31.56%(图1A)。2013年共分离出了51种碳链长度在14~22的PLFAs,转cry1Ie基因抗虫玉米和对照玉米的主峰相似,在整个生育期内,两者的间根际土壤总PLFA含量变化趋势与2012年一致,但在拔节期,两者无显著差异,且转cry1Ie基因抗虫玉米PLFA含量均高于对照玉米(图1B)。
2.1.2土壤细菌PLFA总量分析2012年细菌微生物量变化趋势与总微生物量变化趋势一致,最低值均出现在拔节期,且拔节期的转cry1Ie基因抗虫玉米细菌生物量低于对照,但差异不显著。选取的代表细菌生物量指标:14∶0 iso,15∶1 iso G,15∶0 iso,15∶0 anteiso,15∶00,16∶1 iso G,C16 N alcohol,16∶0 iso,16∶1 ω9c,16∶1 ω7c,16∶1 ω5c,16∶00,iso 17∶1G,17∶0 iso,17∶0 anteiso,17∶1 ω8c,17∶0 cyclo,16∶1 2OH,18∶1 ω5c,18∶00,19∶1(ω8?)alcohol,19∶0 cyclo c1-12,21∶1 ω3c。革兰氏阳性菌通过PLFA总含量估算:14∶0 iso,15∶1 iso G,15∶0 iso,15∶0 anteiso,16∶1 iso G,16∶0 iso, iso 17∶1G,17∶0 iso,17∶0 anteiso。革兰氏阴性菌通过以下指标估算:C 16N alcohol,16∶1 ω5c,16∶1 ω9c,16∶1 ω7c,17∶1 ω8c,17∶0 cyclo,16∶1 2OH,18∶1 ω5c,19∶1(ω8?) alcohol,19∶0 cyclo c1-12,21∶1 ω3c(Fangetal.,2000; Yuetal.,2009)。在除拔节期外的整个生育期内,转cry1Ie基因抗虫玉米的细菌生物量高于对照,但无显著差异(图2A)。在拔节期,转cry1Ie基因玉米根际土壤中革兰氏阳性细菌的比例较低,在抽丝期,转cry1Ie基因玉米G+/G-比值较高,但差异未达到显著水平(图3A)。2013年转cry1Ie基因玉米和对照根际土壤细菌生物量以及G+/G-比值见图2B和图3B。2013年的细菌生物量通过12∶00,13∶0 iso,14∶1 iso E,12∶0 3OH,14∶1 ω5c,14∶00,15∶1 iso G,15∶1 anteiso A,5∶0 iso,15∶0 anteiso,15∶00,16∶0 N alcohol,16∶0 iso,16∶0 anteiso,16∶1 ω5c,16∶00,15∶0 iso 3OH,17∶0 iso,17∶0 anteiso,17∶1 ω8c,17∶0 cyclo,17∶00,16∶0 2OH,18∶1 ω5c,18∶00,19∶0 iso,19∶0 cyclo ω8c,18∶1 2OH,20∶1 ω9c,20∶00,16∶1 ω6c,17∶1 iso I,18∶1 ω8c来估算。革兰氏阳性菌通过以下PLFA总含量估算:12∶00,13∶0 iso,14∶1 iso E,14∶0 iso,16∶0 iso,16∶0 anteiso,16∶00,17∶0 iso,17∶0 anteiso,17∶00,18∶00,19∶0 iso,20∶00,17∶1 iso I。革兰氏阴性菌通过以下PLFA的总含量估算:12∶0 3OH,14∶1 ω5c,16∶0 N alcohol,16∶1 ω5c,15∶0 iso 3OH,17∶1 ω8c,17∶0 cyclo,16∶0 2OH,18∶1 ω5c,19∶0 cyclo ω8c,18∶1 2OH,20∶1 ω9c,16∶1 ω6c,18∶1 ω6c(Fangetal.,2000; Yuetal.,2009)。2013年玉米的细菌PLFA总含量和G+/G-比值变化趋势与2012年变化趋势相一致,均为先减少后升高再减少,细菌PLFA总含量在拔节期最低,G+/G-比值在成熟期最低(图2B)。在播前期和拔节期,转cry1Ie基因玉米的比值均小于对照,但在抽丝期和成熟期,转cry1Ie基因玉米的比值高于对照,但二者也无显著差异(图3B)。

图1 2年中转cry1Ie基因抗虫玉米和对照对根际土壤磷脂脂肪酸(PLFA)的影响
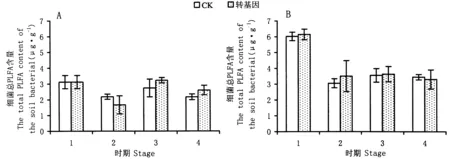
图2 2年转cry1Ie基因抗虫玉米和对照对根际土壤细菌微生物量的变化

图32年转cry1Ie基因抗虫玉米和对照对根际土壤革兰氏阳性菌/革兰氏阴性菌动态变化
Fig. 3Temporal variation of soil G+/G- in transgeniccry1Iegene insect-resistant maize and control maize fields in 2012 and 2013
A.2012年;B.2013年。1:播前期;2:拔节期;3:抽丝期;4:成熟期。
A. 2012; B. 2013. 1: Pre-seed stage; 2: Joint stage; 3: Ladder stage; 4: Mature stage.
2.1.3土壤微生物群落结构2012年PCA分析结果表明,与微生物PLFA生物标记多样性相关的主成分1和主成分2分别解释变量方差的73.51%和16.96%(图4)。不同生育期的PLFA在图中各占不同的空间,表明不同生育期玉米根际土壤微生物群落组成差异较大。同一时期,转cry1Ie基因抗虫玉米和对照分布较集中,说明2种玉米的根际土壤微生物群落结构较相似(图4)。2013年主成分1解释变量方差的69.37%,主成分2解释变量方差的15.84%,播前期玉米的PLFA在图中与其他3个时期相距较远,表明播前期玉米根际土壤微生物群落组成与其他3个时期相差较大,同一时期,转cry1Ie基因抗虫玉米和对照在图中分布较集中,说明两者的根际土壤微生物群落结构无显著差异(图5)。
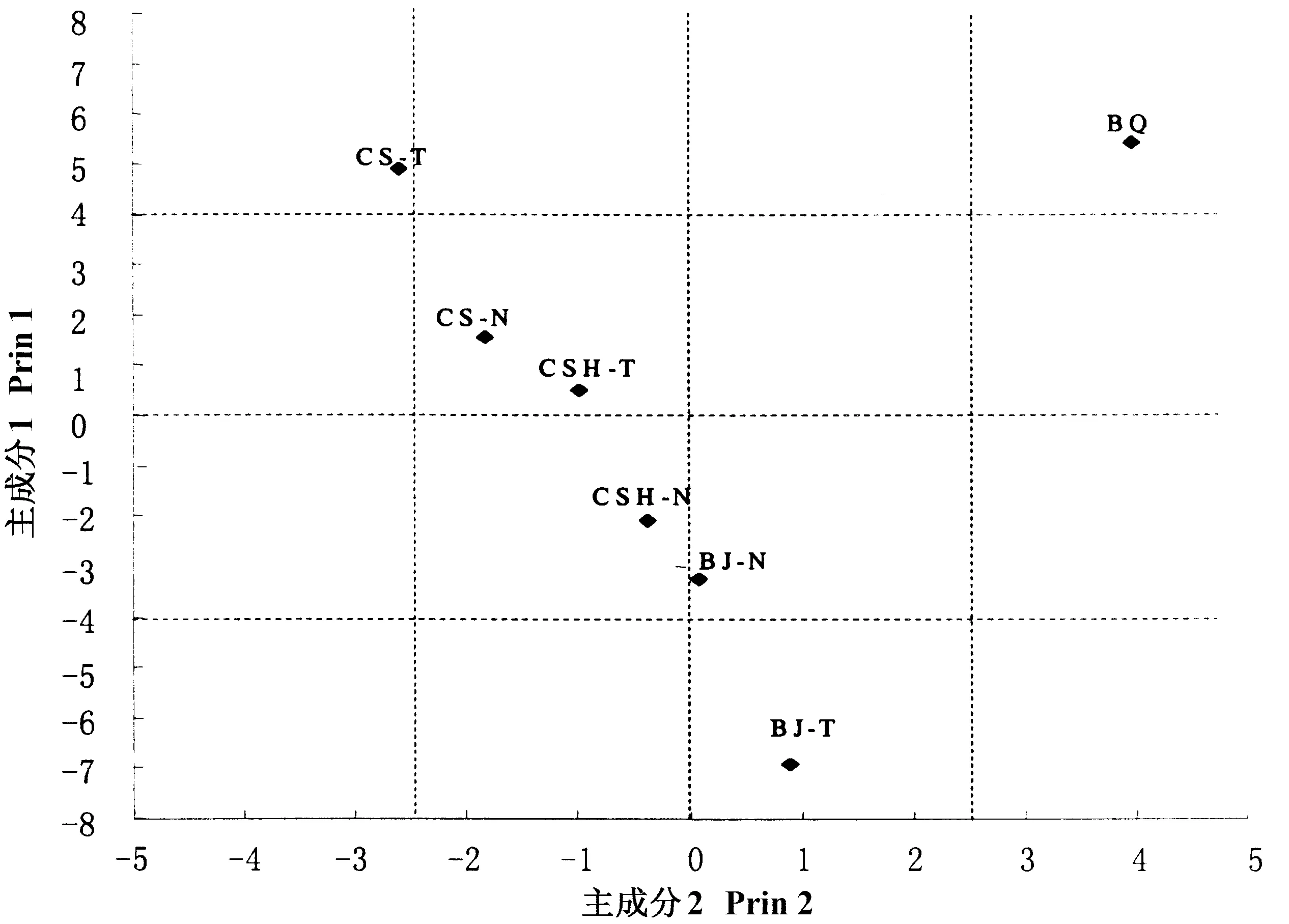
图4 2012年转cry1Ie基因抗虫玉米和对照对根际土壤磷脂脂肪酸(PLFA)主成分分析
2.2 玉米根际土壤微生物DGGE分析
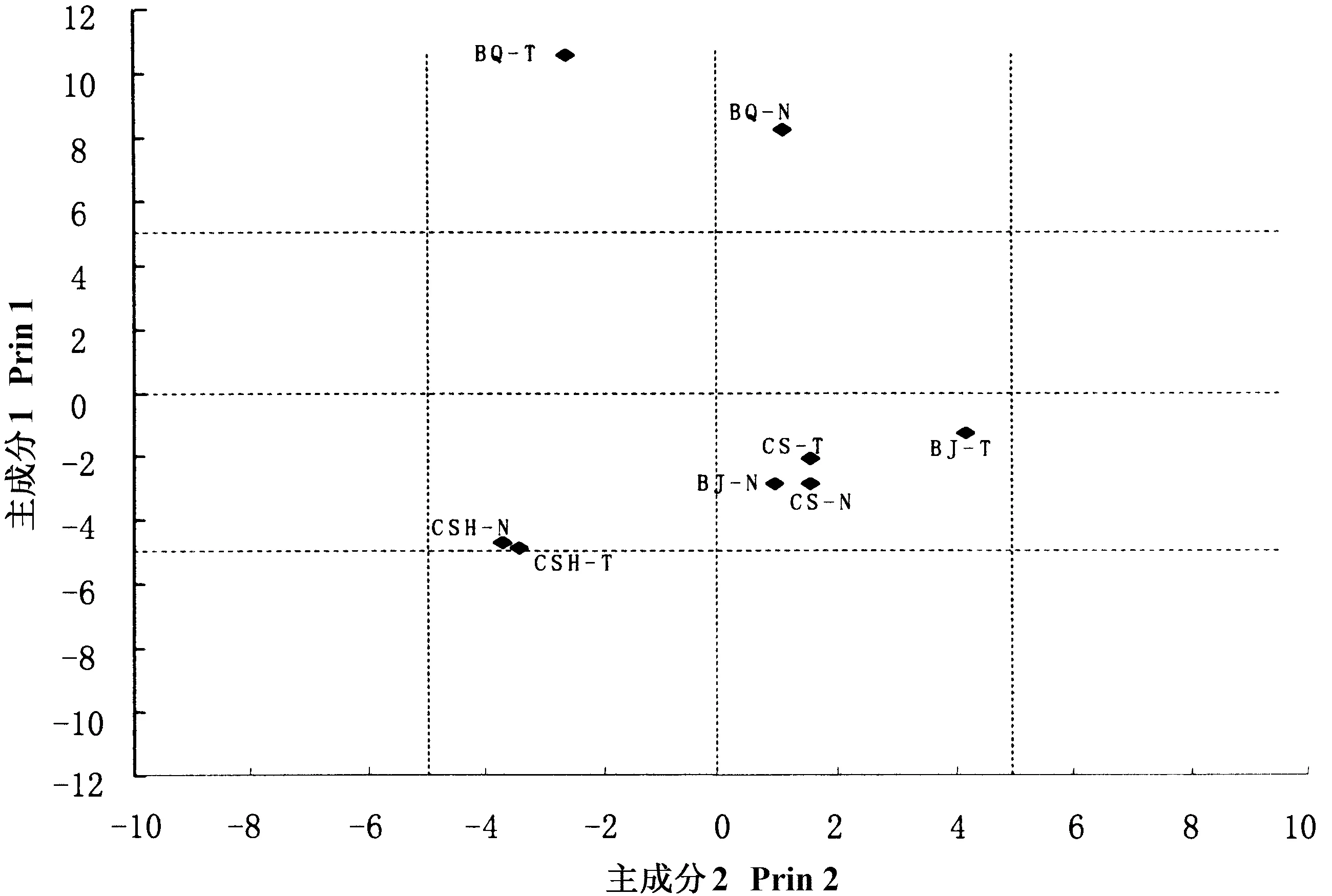
图 52013年转cry1Ie基因抗虫玉米和对照对根际土壤磷脂脂肪酸(PLFA)主成分分析
Fig.5The principal component analysis of transgeniccry1Iegene insect-resistant maize and control maize in 2013
BQ:播前期;BJ-T:拔节期转基因玉米;BJ-N:拔节期非转基因玉米;CS-T:抽丝期转基因玉米;CS-N:抽丝期非转基因玉米;
CSH-T:成熟期转基因玉米;CSH-N:成熟期非转基因玉米。
BQ: Pre-seed stage; BJ-T: Transgenic maize in joint stage; BJ-N: Non-transgenic maize in joint stage; CS-T: Transgenic maize in ladder stage;
CS-N: Non-transgenic maize in ladder stage; CSH-T: Transgenic maize in mature stage; CSH-N: Non-transgenic maize in mature stage.
16S rDNA V3区DGGE图谱显示了转cry1Ie基因抗虫玉米和对照在不同时期根部土壤的细菌群落结构组成(图6~7)。2012年,播前期二者相似性高达100%,拔节期86%,抽丝期83%,成熟期86%(图6);2013年,播前期转cry1Ie基因玉米和对照根际细菌群落结构相似性为99%,拔节期95%,抽丝期和成熟期均高达96%(图7)。因此,在同一时期内二者的土壤样品细菌群落结构组成相似性较高,在整个生育期内,细菌群落结构组成比较稳定。2年结果均显示土壤中细菌菌群类别丰富,且同一年的玉米整个生育期内所有优势种群均未发生较大变化。
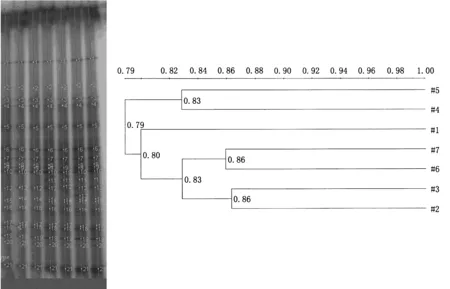
图6 2012年转cry1Ie基因抗虫玉米和对照DGGE图谱分析结果
3讨论
土壤微生物是维持土壤生态系统中生物活性的重要组成部分(Wardleetal.,2004),土壤细菌作为土壤中数量最丰富、分布最广泛的微生物类群,其结构多样性和动态分布对土壤生态系统的稳定具有重要意义。因此,评价转基因作物的安全性必须考虑转基因作物对土壤细菌的影响。
在本研究中,2012年,除了在拔节期转cry1Ie基因抗虫玉米土壤PLFA总量较对照玉米显著降低以外,在其他同一时期二者均无显著差异。出现差异的可能原因是在第1年玉米种植时,有些植物残体进入土壤生态系统,在拔节期造成土壤微生物群体结构产生一些变化,造成二者之间出现差异,还有待进一步深入研究。细菌微生物量以及G+/G-比值在同一生育期内,转cry1Ie基因抗虫玉米和对照玉米均未出现显著差异。2013年的研究结果表明,同一时期内,转cry1Ie基因抗虫玉米和对照玉米的总PLFA量、细菌微生物量以及G+/G-比值均无显著差异。且2年的结果均表明,在不同生育期内玉米根际土壤微生物群落结构差异也不显著。Kaietal.(2005)利用PLFA技术,以转Bt基因玉米和转Bt基因马铃薯为材料进行研究,结果表明,转基因作物提高了细菌PLFA含量,但二者之间差异不显著,G+/G-比值也无显著差异。Kimetal.(2008)研究2个抗草甘膦转基因水稻品系Iksan 483 和 Milyang 204对根部土壤细菌群落影响,发现转基因水稻和非转基因水稻根部细菌群落也无明显差异。刘微等(2011)研究表明,各个生育期,转Bt基因水稻的种植没有造成根际土壤细菌、真菌、放线菌磷脂脂肪酸含量发生显著改变。
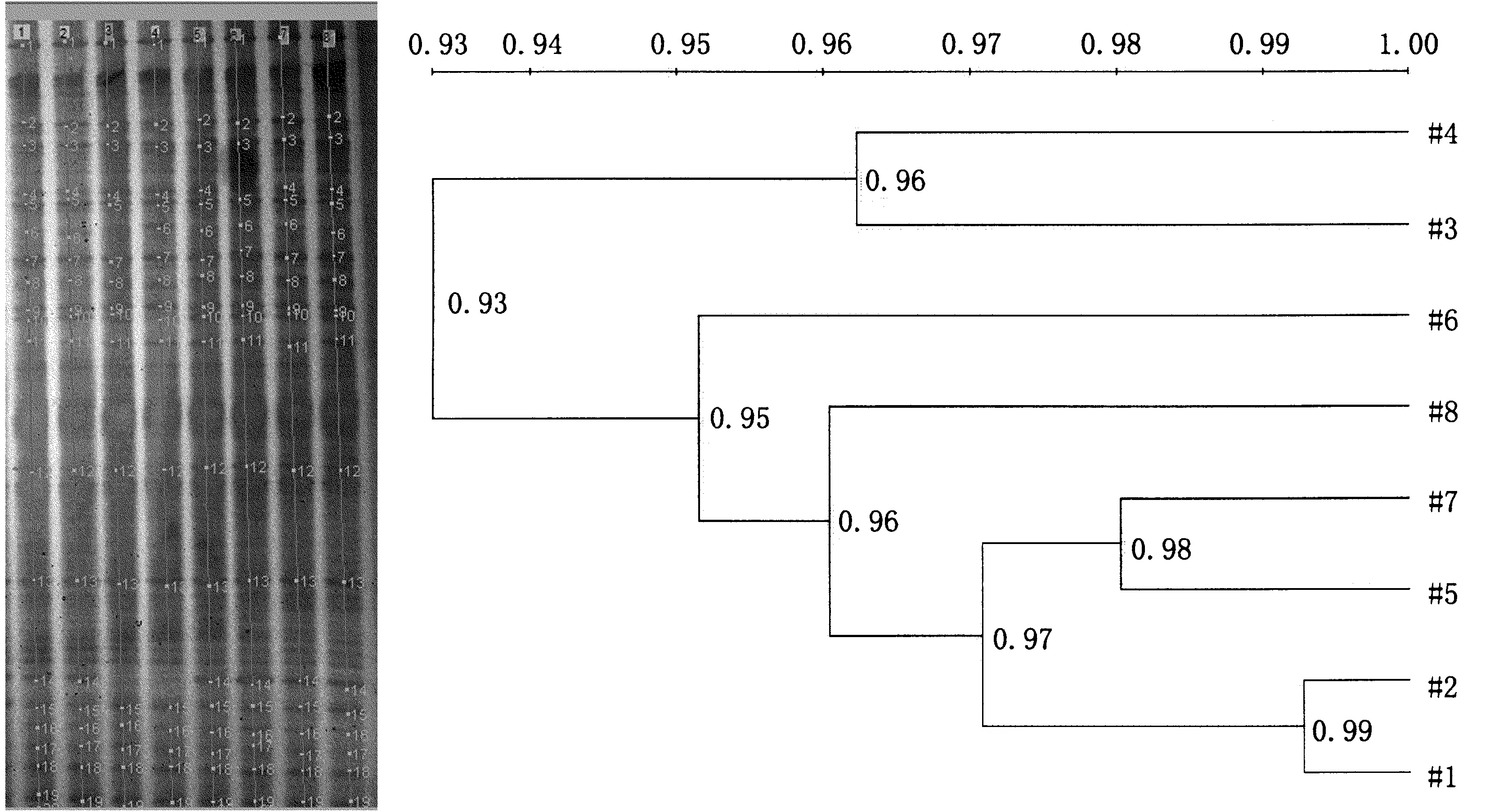
图7 2013年转cry1Ie基因抗虫玉米和对照玉米DGGE图谱分析结果
关于PLFA方法也存在一些争议,如环境温度以及营养元素可能会影响结果,另外土壤中的植物组织也可能含有与微生物相同的脂肪酸(张洪勋等,2003)。本文同时采用PCR-DGGE技术来探讨土壤细菌微生物群落结构以揭示转cry1Ie基因抗虫玉米对根际微生物的影响。2年的DGGE结果均显示,在同一玉米生育期,转cry1Ie基因抗虫玉米和对照玉米细菌群落结构相似性均很高。邹雨坤等(2011)和刘玲等(2012)利用DGGE技术分析转Bt基因玉米对土壤细菌群落的影响,结果无显著差异;Liuetal.(2008)用PCR-DGGE在分析转Bt基因水稻对酶活性和微生物区系组成影响时指出,转Bt基因水稻与亲本之间无显著性差异,不同生育期土壤细菌群落结构变化也较稳定。本研究以PLFA法和DGGE法分析了转cry1Ie基因抗虫玉米对土壤微生物群落结构的影响,与PLFA研究结果基本一致,2种方法均表明转cry1Ie基因抗虫玉米对土壤微生物群落结构无明显影响。
参考文献
昌艳萍, 杨亮, 李春青, 王华芳. 2011. 转基因植物对环境微生物的影响及其检测技术的研究进展. 河北大学学报:自然科学版, 31(3): 331-336.
樊龙江, 周雪平, 胡秉民, 石春海, 吴建国. 2001. 转基因植物的基因漂流风险. 应用生态学报, 12(4): 630-632.
李长林, 张欣, 吴建波, 刘惠芬. 2008. 转基因棉花对根际土壤微生物多样性的影响. 农业环境科学学报, 27(5):1857-1859.
梁宏伟, 长忠, 李忠, 罗相忠, 邹桂伟. 2008. 聚丙烯酰胺凝胶快速、高效银染方法的建立. 遗传, 30(10): 1379-1382.
黎宁, 李华兴, 朱凤娇, 刘远金, 邝培锐. 2006. 菜园土壤微生物生态特征与土壤理化性质的关系. 应用生态学报, 17(2): 285- 290.
刘丽, 鲁国东, 唐乐尘, 周杰珑, 王宗华. 2010. 转抗菌肽D烟草对土壤微生物群落的影响. 生态学报, 30(2): 364-370.
刘微, 王树涛, 陈英旭, 吴伟祥, 王璟. 2011. 转Bt基因水稻根际土壤微生物多样性的磷脂脂肪酸(PLFAs)表征. 应用生态学报, 22(3): 727-733.
刘玲, 赵建宁, 李刚, 杨殿林, 王生荣, 娜布其, 娜日苏. 2012. 转Bt玉米对土壤酶活性及速效养分的影响. 土壤, 44(1): 167-171.
张洪勋, 王晓谊, 齐鸿雁. 2003. 微生物生态学研究方法进展. 生态学报, 23(5): 988-995.
周小奇, 王艳芬, 蔡莹, 黄祥忠, 郝彦宾, 田建卿, 柴团耀. 2007. 内蒙古典型草原细菌群落结构的PCR-DGGE检测. 生态学报, 27(5): 1684-1689.
邹雨坤, 张静妮 ,杨殿林, 陈秀蓉. 2011.转Bt基因玉米对根际土壤细菌群落结构的影响. 生态学杂志, 30(1): 98-105.
Angle J S. 1994. Release of transgenic plants: biodiversity and population level considerations.MolecularEcology, 3: 45-50.
Bligh E G and Dyer W J. 1959. A rapid method of total lipid extraction and purification.CanadianJournalofBiochemistryandPhysiology, 37: 911-917.
Bossio D A, Scow K M and Gunapala N. 1998. Determinants of soil microbial communities:effects of agricultural management, season, and soil type on Phospholipid Fatty Acid Profiles.MicrobiologyEcology, 36: 1-12.
Fang M, Kremer R J, Motavalli P P and Davis G. 2005. Bacterial diversity in rhizospheres of nontransgenic and transgenic corn.AppliedandEnvironmentalMicrobiology, 71: 4132-4136.
Fang J, Barcelona M J and Alvarez P J. 2000. A direct comparison between fatty acid and intact phospholipid profiling for microbial identification.OrganicGeochemistry, 31: 881-887.
Frostegård Å, Tunlid A and Bååth E. 2010. Use and misuse of PLFA measurements in soils.SoilBiologyandBiochemistry, 10: 1-5.
Hare C E, Demir E and Coyne K J. 2005. A bacterium that inhibits the growth ofPfiesteriapiscicidaand other dinflagellates.HarmfulAlgae, 4: 221-234.
James C. 2013. Global status of commercialized Biotech/GM crops.ISAAABriefNo. 42, ISAAA: Ithaca, NY.
Jepson P C, Croft B A and Prattg E. 1994. Test systems to determine the ecological risks posed by toxin release fromBacillusthuringiensisGenes in crop plants.MolecularEcology, 3: 81-89.
Kim M C, Ahn J H, Shin H C, Kim T, Ryu T H, Kim D H, Song H G, Lee G H and Ka J O. 2008. Molecular analysis of bacterial community structures in paddy soils for environmental risk assessment with two varieties of genetically modified rice, Iksan 483 and Milyang 204.JournalofMicrobiolBiotechnol, 18: 207-218.
Knight B P, McGrath S P and Chaudri A M. 1997. Biomass carbon measurements and substrate utilization patterns of microbial populations from soil a mended wish cadmium, copper, or zinc.AppliedandEnvironmentalMicrobiology, 63: 39-43.
Lal R and Lal S. 1993.GeneticEngineeringofPlantsforCropImprovement. Boca Raton, FL: CRC Press.
Liu W, Lu H H, Wu W X, Wei Q K, Chen Y X and Thies J E. 2008. TransgenicBtrice does not affect enzyme activities and microbial composition in the rhizosphere during crop development.SoilBiologyandBio-chemistry, 40: 475-486.
McGaughey W H and Whalon M E. 1992. Managing insect resistance toBacillusthuringiensistoxin.Science, 258: 1451-1455.
Tapp H, Calamai L and Stotzky G. 1994. Adsorption and binding of the insecticidal proteins fromBacillusthuringiensissubsp.kurstaki and subsp.tenbriones on clay minerals.SoilBiologyandBiochemistry, 26: 663-679.
Tapp H and Stotzky G. 1999. Insecticidal activity of the toxins fromBacillusthuringiensissubspecies kurstaki and tenebrionis adsorbed and bound on pure and soil clays.AppliedandEnvironmentalMicrobiology, 61: 1786-1790.
Tapp H and Stotzky G. 1998. Persistence of the insecticidal toxin fromBacillusthuringiensissubsp. Kurstaki in soil.SoilBiologyandBiochemistry, 30: 471-476.
Thompson J R, Marcelino L A and Polzm F. 2002. Heteroduplexes in mixed-template amplifications: formation, consequence and elimination by reconditioning PCR.NucleicAcidsResearch, 30: 2083-2088.
Watrud L S and Seidler R J. 1998. Nontarget ecological effects of plant, microbial, and chemical introductions to terrestrial systems∥SoilChemistryandEcosystemHealthSpecialPublication52. Soil Science Society of America Madison: Wisconsin, 313-340.
Wardle D A, Bardgett R D, Klironomos J N, Setala H, Putten W H and Wall D H. 2004. Ecological linkages between aboveground and belowground biota.Science, 304: 1629-1633.
Xue K, Luo H F, Qi H Y and Zhang H X. 2005. Changes in soil microbial community structure associated with two types of genetically engineered plants analyzing by PLFA.EnvironmentScience, 17: 130-134.
Yu M, Zeng G M and Chen Y N. 2009. Microbial communities responsible for the solid-state fermentation of rice straw estimated by phospholipid fatty acid(PLFA)analysis.ProcessBiochemistry, 44: 17-22.
Zambryske P. Tiplasmid vector for the introduction of DNA into plant cells without alteration of their normal regulation capacity.1983.EMBOJournal, 2: 21-431.
Zelles L. 1999. Fatty acid patterns of phospholipids and lipopolysac charides in the characterization of microbial communities in soil: A review.BiologyandFertilityofSoils, 29: 111-129.
(责任编辑:郭莹)
The effect of transgenic maize withcry1Iegene on rhizosphere bacteria community structure
Hui-xiang YE1, Yue-yuan CUI1, Xin-yuan SONG2, Juan MA1, Hu WAN1, Jian-hong LI1*
1CollegeofPlantScienceandTechnology,HuazhongAgriculturalUniversity,Wuhan,Hubei430070,China;
2NationalCenterforEnvironmentalSafetyInspectionofTransgenicPlants,MinistryofAgriculture,
JilinAcademyofAgriculturalSciences,Changchun,Jilin130124,China
Abstract:【Background】 Soil microbes are an important part for maintaining biological activities. Soil bacteria are diverse, widely distributed and their structural diversity and dynamic distribution are important for the stability of the lithosphere. In this study, the safety of genetically modified crops on soil bacteria was examined. 【Method】 The community structure of the rhizosphere in fields of transgenic cry1Ie maize and its parental non-Bt maize (the control) were measured at different growth stages. This was assessed using culture-independent technique polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE) and Phospholipids fatty acid (PLFA) assays. 【Result】 PLFA analysis showed that the bacteria biomass and G+/G- of transgenic cry1Ie maize and maize receptors control of 2012 had no significant difference at the same growth stage, but significant differences were detected for microbial biomass at different following stages. The values of G+/G- were greater than one, suggesting that at each growth stage, microbial biomass of Gram-positive bacteria was greater than Gram-negative bacteria. During the entire growing period, insect-resistant corn with cry1Ie receptor gene control was not a significant different than control corn. Tt was no significant difference in 2013 in the total amount of microbial and bacterial biomass and the ratio of Gram-positive bacteria and gram-negative bacteria between both corn types. The cluster analysis results showed no remarkable difference in DGGE profile of bacteria at the same growth stage. Corn rhizosphere bacterial community structure was relatively stable and similar in both 2012 and 2013. 【Conclusion and significance】 Two years of data showed that insect-resistant cry1Ie transgenic maize had no significant effects on soil bacterial community structure. The study can provide a theoretical basis to evaluate the safety of transgenic maize with cry1Ie gene.
Key words:phospholipids fatty acid assay; polymerase chain reaction-denaturing gradient gel electrophoresis; rhizosphere; soil bacterial; the value of G+/G-
DOI:10.3969/j.issn.2095-1787.2015.01.012
通讯作者*(Author for correspondence), E-mail: XL6479@163.com
作者简介:张雁, 女, 硕士研究生。 研究方向: 植物检疫病害。 E-mail: 568779683@qq.com
基金项目:国家质检公益性行业科研专项(201310091)
收稿日期(Received): 2014-12-14接受日期(Accepted): 2015-01-13

