集胞藻6803基因组DNA提取方法的比较
陈雪峰, 焦文冬, 刘 宁, 马文锦, 孙玉姣
(陕西科技大学 食品与生物工程学院, 陕西 西安 710021)
集胞藻6803基因组DNA提取方法的比较
陈雪峰, 焦文冬, 刘宁, 马文锦, 孙玉姣
(陕西科技大学 食品与生物工程学院, 陕西 西安710021)
摘要:比较了PVP法、CTAB法和SDS法对集胞藻6803(Synechocystis sp.PCC6803)基因组DNA的提取效果,并采用紫外-可见分光光度计、琼脂糖凝胶电泳及PCR扩增检测基因组DNA质量.结果表明,三种方法均能提取到符合分子生物学要求的基因组DNA,其中SDS法提取的DNA综合质量最高.以提取的基因组DNA为模板,以sll0853引物进行PCR扩增,三种方法均能扩增出目的条带,其中CTAB法及SDS法的条带更为清晰.
关键词:集胞藻6803; 基因组DNA提取; PCR扩增
0引言
集胞藻属于蓝藻门、蓝藻纲、色球藻目、色球藻科集胞藻属,是一种生活在淡水中的单细胞蓝藻.其光合作用与高等植物类似,即吸收光能转化成CO2和水,生成有机化合物并释放氧气,也能利用葡萄糖等物质进行异养生长[1].
近年来,随着分子生物学技术的快速发展,集胞藻等一些研究较多的蓝藻已成为分子生物学和基因工程研究中的重要模式生物,广泛应用于异形胞的发育、光合作用、固氮作用和外源基因的表达等方面的研究,并且越来越多的基因在蓝藻中都得到了表达[2-5].集胞藻作为光合作用的模式生物,具有很高的光合效率.发状念珠藻是一种具有很高营养价值和经济价值的可食用丝状蓝藻,若将集胞藻的高光合作用系统转入食用发状念珠藻中并有效表达,得到具有高固碳功能的发状念珠藻,对于发状念珠藻人工培养、荒漠生态恢复、改善土壤性质和沙漠治理发挥着积极作用.
简单、快速地分离提取高质量的基因组DNA是进行分子生物学研究的基础,藻类基因组DNA的提取方法多与植物基因组DNA提取方法相同,但不同的藻类细胞中所含次生代谢产物的种类、含量不同,适合的提取方法也不同.
董学卫等[6]采用高盐法、CTAB法、PVP法和石英砂法4种方法对聚球藻7002基因组DNA进行提取并进行PCR扩增,结果表明4种方法均能提取到符合分子生物学要求的基因组DNA,其中PVP法质量最高,石英砂法浓度最高,4种方法提取到的基因组DNA均能扩增出理想条带;邰丽华等[7]比较并摸索了超声波和溶菌酶两种不同破壁方法对螺旋藻基因组DNA的提取效果的影响,结果表明两种破壁方法均可得到高质量、高得率的螺旋藻基因组DNA,其中超声波法破壁不易掌控,而适当浓度的溶菌酶法可获得较理想的结果;杨泽民等[8]对金藻基因组DNA的提取方法进行了深入研究,结果表明,就球形棕囊藻而言,玻璃粉法是4种方法中最好的DNA提取方法,就绿色巴夫藻而言,从经济和方便考虑,以高盐法为最好.由于集胞藻细胞壁外有一层由多糖所组成的透明胶质,同时细胞内也含有多种蛋白及色素[9],高质量的藻类基因组DNA较难提取,目前有关藻类基因组DNA的提取的文章报道较少,集胞藻基因组DNA的提取的文章还未见报道.
本实验选取PVP法、SDS法和CTAB法3种常规提取植物基因组DNA的方法[10]分别用于集胞藻6803基因组DNA的提取,并采用紫外-可见分光光度计、琼脂糖凝胶电泳及PCR扩增检测基因组DNA质量.通过检测结果的比较,以期筛选出一种能够得到高质量、高产量的集胞藻6803基因组DNA的提取方法,为集胞藻分子生物学水平的进一步研究提供理论基础和技术支持.
1材料与方法
1.1 供试菌株、主要试剂及仪器
(1)供试菌株:集胞藻6803(Synechocystis sp.PCC6803),购自中科院典型培养物保藏委员会淡水藻种库(FACHB).藻种采用BG-11培养基进行培养,培养温度为25 ℃,光照强度为2 500 lx,光暗比为12 h∶12 h,培养时间为10 d.
(2)试剂:PCR所用试剂均购自天根生化科技(北京)有限公司,引物购自上海生物工程技术服务有限公司.
(3)仪器:Eppendorf 5430小型高速离心机(艾本德中国有限公司),CYCP-21DN型电泳仪(北京六一仪器厂),UV-1100紫外-可见分光光度计(上海美谱达有限公司),MyCycler Thermal Cycler型PCR仪(北京伯乐生命科学发展有限公司),FR-980A生物电泳成像分析系统(上海复日科技有限公司).
1.2 DNA提取方法
取冷冻干燥后的集胞藻藻粉,于液氮中冷冻研磨后称取0.1 g置于离心管中,分别采用以下3种方法提取DNA.提取的DNA均溶于100μL TE缓冲液中,并于-20 ℃保藏以备后续使用.
1.2.1SDS法
参考王景雪[11]的方法.先加入800μL预热(65 ℃)的SDS提取液至装有藻粉的离心管,65 ℃水浴30 min,不时颠倒混匀;再加入1/3体积KAC(5 M)混匀,冰浴20 min后离心20 min(10 000 r/min,4 ℃);取上清液加入等体积预冷的氯仿∶异丙醇(24∶1),颠倒混匀,离心5 min(12 000 r/min,4 ℃);取上清液加入等体积预冷的异丙醇,颠倒混匀至见丝状沉淀,-20℃静置30 min沉淀DNA;离心10 min(12 000 r/min,25 ℃)后,用70%乙醇清洗沉淀两次,自然干燥.
SDS法提取液:100 mM Tris-HCl(pH 8.0),500 mM NaCl,50 mM EDTA(pH8.0),20% SDS,10 mM β-巯基乙醇.
1.2.2PVP法
参考C.S.Kim[12]的方法.液氮冷冻研磨时加入适量β-巯基乙醇(1% V/V),加入800μL PVP提取液至装有藻粉的离心管,充分混匀并于室温下温育1 h,期间上下颠倒数次;再依次加入PVP(终浓度达到6%)及1/2倍体积NH3AC(7.5 M)并充分混匀,冰浴30 min后离心10 min(10 000 r/min,4 ℃);取上清液加入等体积预冷的异丙醇,于-20 ℃下静置30 min沉淀DNA;离心10 min(10 000 r/min,4 ℃)后去上清,沉淀自然干燥后重悬于500μL TE缓冲液中,加入2μL Rnase(1 mg/mL)并于37 ℃温育15 min;然后加入等体积氯仿∶异戊醇(24∶1)轻摇至乳白色,离心5 min(10 000 r/min,4 ℃)后将上清液转移至新离心管中,重复至无蛋白质层出现;最后取上清液加入等体积预冷异丙醇,-20 ℃下静置10 min后离心,用80%的乙醇清洗沉淀,自然干燥.
PVP法提取液:200 mM Tris-HCl(pH 8.0),250 mM NaCl,25 mM EDTA(pH8.0),0.5% SDS.
1.2.3CTAB法
参考杨泽民[8]的方法.先加入800μL CTAB提取液(65 ℃预热)至装有藻粉的离心管,充分混匀并于65 ℃水浴1 h,期间上下颠倒数次;冷却至室温后加入预冷的氯仿∶异丙醇(24∶1),旋涡振荡2 min,离心10 min(12 000 r/min,4 ℃)后保留上清液,重复至无蛋白质层出现;然后取上清液加入1/3体积预冷的异丙醇,-20 ℃放置30 min后上下轻摇至出现白色絮状沉淀,离心10 min(12 000 r/min,4 ℃)后保留沉淀,自然风干,然后用75%的乙醇及5 M的醋酸钠清洗1次,75%乙醇及无水乙醇各清洗1次后自然干燥.
CTAB 法提取液:100 mM Tris-HCl(pH 8.0),1.4 M NaCl,10 mM EDTA(pH8.0),2% CTAB,2%β-巯基乙醇.
1.3 DNA质量检测方法
1.3.1紫外分光光度检测
取50μL用不同方法提取的DNA样品补加去离子水至4 mL,用紫外-可见分光光度计分别测量在230 nm、260 nm、280 nm的吸光度.
1.3.2琼脂糖凝胶电泳检测
样品DNA在0.8%琼脂糖凝胶、0.5×TBE缓冲液、电压4~5 V/cm,电泳30 min,0.5μg/mL EB溶液中染色20 min,凝胶成像系统观察并拍照,分析DNA质量.
1.3.3PCR扩增检测
为了进一步说明三种方法提取获得的高质量、高分子量的DNA,利用引物sll0853[1](上海生物工程技术服务有限公司产品)对三种样品DNA 模板进行PCR 扩增.PCR反应体系(25μL)为:12.5μL 2×Taq PCR MasterMix,正反引物上下游各1.0μL,DNA模板1.5μL,补ddH2O至25μL;PCR 反应条件为:94 ℃预变性2 min;94 ℃变性20 s;67.5 ℃复性25 s;72 ℃延伸30 s,20个循环;72 ℃延伸2 min,4 ℃保温.扩增产物用1.0%琼脂糖凝胶电泳进行检测,并用凝胶成像系统拍照.
2结果与讨论
2.1 紫外分光光度计检测结果
核酸的嘌呤、嘧啶碱基环上的许多原子中都含有共轭双键,在260 nm处对紫外光有较强的吸收[13].纯净DNA在260 nm处的吸收值与280 nm处吸收值的比值(OD 260/OD 280)约为1.80,小于1.80说明提取的DNA中有蛋白质污染,大于1.80则说明提取的DNA中可能还有RNA[14];并且OD 260与OD 230的比值应大于2.00,小于等于2.00说明可能残存核甘酸、氨基酸、酚类、有机溶剂、盐等小分子杂质[15].
提取的集胞藻基因组DNA在220~300 nm处的紫外吸收光谱表明:三种方法提取的集胞藻基因组DNA在260 nm附近均有吸收峰(如图1所示).与SDS法及PVP法相比,CTAB法提取的DNA 260 nm/280 nm的值为1.820,260 nm/230 nm的值为2.097(如表1所示),说明CTAB法提取的DNA纯度高,蛋白质、酚类及多糖类杂质除去更为完全,残留的氯仿、乙醇等小分子成分最少.SDS法提取的DNA 260 nm/280 nm、260 nm/230 nm的值分别为2.125和2.097,说明该方法提取的DNA还残存RNA,但小分子杂质去除的比较干净;而PVP法提取DNA 260 nm/280 nm、260 nm/230 nm的值分别为1.701及2.199,说明PVP法提取的DNA中不仅存在蛋白质污染,还残留了大量小分子的杂质.
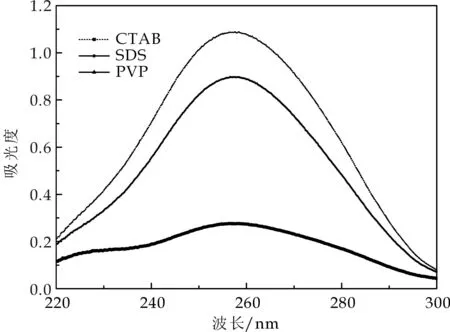
图1 三种方法提取的集胞藻6803基因组DNA在220~300 nm下的吸光度

样品颜色OD260/OD280OD260/OD230DNA样品浓度/(μg/mL)CTAB法a1.820±n2.097608PVP法b1.701±n1.075114SDS法b2.125±n2.199424
注:a为白色;b为淡黄色;n≤0.1
DNA 样品的浓度= OD260×DNA 稀释倍数×50(单位:μg /mL)[16].三种方法提取的DNA样品的OD260值表明,不同方法提取的DNA 产量不同.其中CTAB法提取的DNA浓度最高(608μg/mL),SDS法提取的DNA浓度较高(424μg/mL),PVP法提取的DNA浓度最小(114μg/mL).
2.2 琼脂糖凝胶电泳检测结果
将提取的基因组DNA进行琼脂糖凝胶电泳,以标准分子量DNA Hind Ⅲ为参照物进行对照,基因组DNA均大于23 kb,表明获得的是较大分子量的基因组DNA(如图2所示),完整性较好,无明显的拖尾现象.
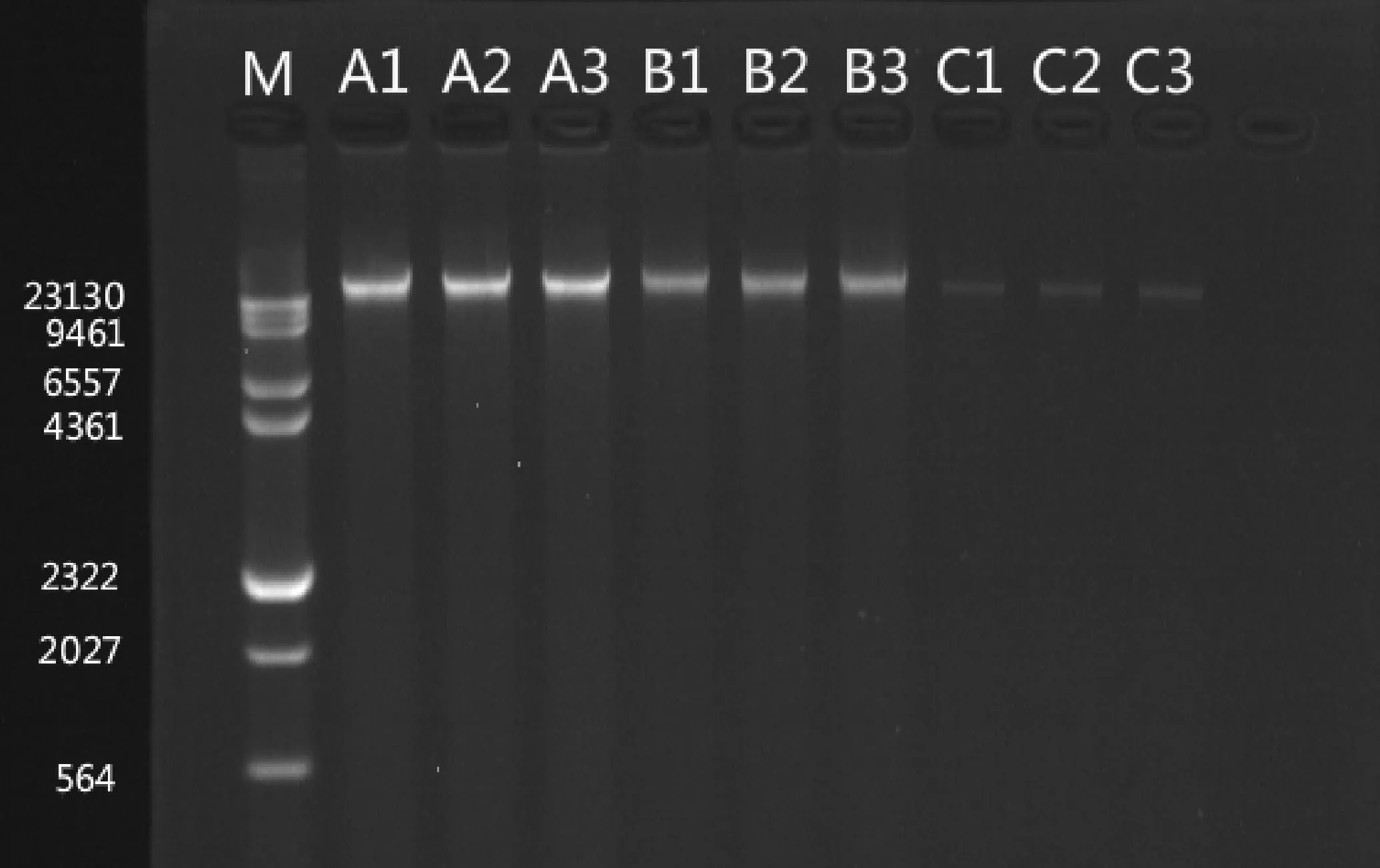
M为23 kb DNA Ladder;A为CTAB法;B为SDS法;C为PVP法.图2 三种方法提取的集胞藻6803基因组DNA琼脂糖凝胶电泳图谱
2.3 PCR检测结果
sll0853引物的PCR扩增结果如图3所示.从图3可以看出,用三种方法提取的基因组DNA在500~750 bp处均能扩增出与预期大小(561 bp)相符的条带.

M为Trans 2k DNA Maker;A为PVP法;B为CTAB法;C为SDS法.图3 sll0853引物的 PCR 扩增产物结果
3结论
本实验从实用的角度出发,选择了三种植物常用的基因组DNA提取方法(SDS法、CTAB法和PVP法)提取集胞藻基因组DNA,并对其质量进行了比较研究.综合DNA提取和PCR扩增结果,SDS法及CTAB法提取的DNA 质量好,产量高,该结果与张伟等人的研究结果相一致[17-19].其中CTAB法对集胞藻提取的基因组DNA 的质量和产量都略优于SDS法,这是由于CTAB提取缓冲液对细胞的裂解能力强于SDS提取缓冲液,并且CTAB与酚-氯仿结合使用效果较好,更适用于含细胞壁及含多糖、酚类等多种次生代谢产物的细胞[9,20].但SDS法较CTAB法操作步骤简单、温和,更适用于快速、大量的提取满足PCR实验要求的DNA样品,能够为后续进行高通量筛选提供可靠的技术支持.PVP法虽然可以简单快速的提取到基因组DNA,但其质量和产量均不太理想.由此可见,对于集胞藻,SDS法是这三种方法中最经济、高效、安全的基因组DNA 的提取方法.
参考文献
[1] 刘欣.集胞藻PCC6803基因sll0853的初步研究[D].武汉:中南民族大学,2012.
[2] 席超,王春梅,施定基.蓝藻基因工程应用研究进展[J].中国生物工程杂志,2010,35(3):105-111.
[3] 方萍.集胞藻PCC6803中sll1466的基因工程研究[D].武汉:华中科技大学,2009.
[4] Yukako Hihara,Ayako Kamei,Minoru Kanehisa,et al.DNA microarray analysis of cyanobacterial gene expression during acclimation to high light[J].The Plant Cell,2001,13(5):793-806.
[5] Takeshima Y,Takatsugu N,Sugiura M,et al.High-level express ion of human superoxide dismutase in the cyanobacterium anacystis nidulans 6301[J].Proceedings of the National Academy of Sciences,1994,91(21):9 685-9 689.
[6] 董学卫,李有志,何庆芳,等.聚球藻7002基因组DNA提取方法比较[J].北方园艺,2013(20):95-97.
[7] 邰丽华,于涛,张晓嵘,等.螺旋藻基因组DNA制备方法的摸索与比较[J].科学技术与工程,2007,7(22):5 755-5 758.
[8] 杨泽民,章群,谢数涛,等.金藻基因组DNA的提取与PCR扩增[J].生物学杂志,2008,26(1):60-63.
[9] 王永红.集胞藻6803封闭式光生物反应器混合营养培养及其生理学研究[D].上海:华东理工大学,2000.
[10] 易庆平,罗正荣,张青林,等.植物总基因组DNA提取纯化方法综述[J].安徽农业科学,2007,35(25):7 789-7 791.
[11] 王景雪,孙毅,高武,等.一种简便实用的植物总DNA提取方法[J].山西大学学报(自然科学版),2000,23(3):271-272.
[12] C.S.Kim,C.H.Lee,J.S.Shin,et al.A simple and rapid method for isolation of high quality genomic DNA from fruit trees and conifers using PVP[J].Nucleic Acids Research,1997,25(5):1 085-1 086.
[13] 彭秀玲,袁汉英,谢毅,等.基因工程实验技术[M].长沙:湖南科学技术出版社,1997.
[14] 曹凯鸣.核酸化学导论[M].上海:复旦大学出版社,1991.
[15] 姜泊.分子生物学常用实验方法[M].北京:人民军医出版社,1996.
[16] 李宪臻.生物化学[M].武汉:华中科技大学出版社,2008.
[17] 张伟,谢甫绨,曹萍,等.大豆叶片DNA提取方法的比较研究[J].大豆科学,2007,26(1):60-65.
[18] 尚啸,王健,龚胜,等.三色堇叶片DNA不同提取方法比较[J].湖北农业科学,2014,53(20):4 999-5 002.
[19] R.A.Ribeiro,M.B.Lovato.Comparative analysis of different DNA extraction protocols in fresh and herbariun specimens of the genus dalbergia[J].Genetics and Molecular Reseach,2007,6(1):173-187.
[20] 王珍,方宣钧.植物DNA分离[J].分子植物育种,2003,1(2):281-288.
【责任编辑:蒋亚儒】
Comparative analysis of different DNA extraction
protocols in Synechocystis 6803
CHEN Xue-feng, JIAO Wen-dong, LIU Ning, MA Wen-jin, SUN Yu-jiao
(School of Food and Biological Engineering, Shaanxi University of Science & Technology, Xi′an 710021, China)
Abstract:PVP method,CTAB method and SDS method were used in this paper to obtained good quality DNA extracted from Synechocystis sp.PCC6803.And the DNA quality was tested by the ultraviolet spectrophotometer,Agarose gel electrophoresis and PCR amplification.The results shows that genomic DNA conform to the requirements of molecular biology can be extracted by those three methods,and SDS method was better than the others in terms of the value and yields of genomic DNA extraction.The PCR amplification of the Synechocystis genomic DNA extracted in different ways were studied with primer sll0853.Both of CTAB and SDS method can amplificate more clear bands than the PVP method.
Key words:Synechocystis 6803; DNA extraction; PCR amplification
中图分类号:Q781
文献标志码:A
文章编号:1000-5811(2016)01-0123-05
作者简介:陈雪峰(1964-),男,陕西宝鸡人,教授,博士生导师,研究方向:食品功能成分及生物技术
基金项目:国家自然科学基金青年项目(31401633); 陕西科技大学国家基金后补助项目(2014XHBZ-10)
收稿日期:*2015-10-16

