刺参消化道中蛭弧菌类的生物多样性分析
韩民泳, 陈慧黠, 斯 晗, 刘 洋, 陈亚东
(大连海洋大学 农业部北方海水增养殖重点实验室,辽宁 大连 116023)
刺参消化道中蛭弧菌类的生物多样性分析
韩民泳, 陈慧黠*, 斯 晗, 刘 洋, 陈亚东
(大连海洋大学 农业部北方海水增养殖重点实验室,辽宁 大连 116023)
研究了刺参消化道中蛭弧菌类生物多样性。用刺参肠道内容物提取微生物总DNA,分别使用蛭弧菌类生物特异性引物Bd、Bac扩增获得16S rDNA目的片段,连接pMD19-T载体,转化到大肠埃希菌DH5α感受态细胞,经蓝白斑筛选及阳性克隆筛选,通过核糖体DNA扩增片段限制性内切酶分析(ARDAR)对阳性克隆进行分型,使用HaeⅢ和MspⅠ双酶切各60个阳性克隆,选取不同ARDAR型的阳性克隆测序,并进行生物学分析。结果显示:引物Bd、Bac的16S rDNA扩增产物分别分离获得3个和2个差异性序列,各属于一个类群,分属于蛭弧菌属(Bdellovibrio)和噬菌弧菌属(Bacteriovorax)。这些序列与数据库中相应菌株序列的最大相似性均为95.0%,这5个序列的菌株可能为潜在的新种。
蛭弧菌类生物;刺参;肠道;核糖体DNA扩增片段限制性内切酶分析(ARDRA);双酶切;多样性
蛭弧菌于1962年首次在土壤中被发现。Snyder等[1]使用“Bdellovibrio-and-like organisms(BALOs, 蛭弧菌及其类似生物)”一词描述这类细菌。近年来,国内外对蛭弧菌类生物的研究日益增多。研究表明BALOs对常见的水产养殖中主要病原菌具有较强的裂解作用[2-5],对真核细胞无不良影响,具有广阔的应用前景[6]。国内关于蛭弧菌多样性的研究较少,温崇庆等[7-8]对广东省湛江市海域多个集中对虾养殖区的BALOs多样性进行了系统的研究分析,薛明等[9]对对虾池中可分离蛭弧菌多样性进行了研究。而其他少量研究则是从动物消化道内分离蛭弧菌,用于防病研究[10-11]。在刺参养殖领域对BALOs多样性的研究鲜见报道,刺参肠道内蛭弧菌多样性的研究将为蛭弧菌的分离、培养、鉴定打下基础,有助于研究其对刺参疾病的防治作用。
1 材料与方法
1.1 材料
1.1.1 实验材料 刺参购自大连庄河宝发海珍品养殖厂(暂养于农业部北方海水增养殖重点实验室),平均体重10 g。随机选取10头活体刺参,解剖,每头参取等量肠道内容物混合,加入适量无菌生理盐水,-20 ℃保存。
1.1.2 引物 扩增16S rDNA目的条带使用引物分别为噬菌蛭弧菌特异性引物Bac(Bacteriovorax-specific):Bac676F(5′-ATTTCCGCATGTAGGGGTA-3′)和Bac1442R(5′-GCCACGGCTTCAGGTAAG-3′),蛭弧菌特异性引物Bd(Bdellovibrio-specific):Bd529F(5′-GGTAAGACGAGGGATCCT-3′)和Bd1007R(5′-TCTTCCAGTACATGTCAAG-3′)[12-13],阳性克隆筛选通用引物M13F(5′-CGCCAGGGTTTTCCCAGTCACGAC-3′)和M13R(5′-AGCGGATAACAATTTCACACA-3′)(生工生物工程(上海)股份有限公司)。
1.1.3 培养基 LB培养基;选择培养基:含100 μL氨苄青霉素(100 g/L)的LB培养基。
1.2 方法
1.2.1 总DNA提取及PCR扩增产物的回收 将刺参肠道内容物样品在4 ℃条件下,5 000 r/min离心10 min,取沉淀用DNA提取试剂盒提取微生物总DNA样品,1%的琼脂糖凝胶电泳进行检测,获得的DNA样品于-20 ℃贮存、备用。采用特异引物Bac和Bd分别对提取的总DNA进行扩增。PCR反应条件:94 ℃预变性5 min,94 ℃变性30 s,53 ℃退火30 s,72 ℃延伸30 s,72 ℃延伸7 min,4 ℃保存,40个循环。PCR产物使用DNA纯化回收试剂盒回收。
1.2.2 PCR产物的质粒载体连接和大肠埃希菌感受态细胞的转化 扩增得到的DNA片段克隆到pMD 19-T载体上,重组质粒转化到大肠埃希菌(E.coli)DH5α感受态细胞,经蓝白斑筛选,进行菌落通用引物PCR,筛选出阳性克隆。
1.2.3 阳性克隆的ARDRA双酶切分型 分别各选取60个阳性克隆菌株用限制性内切酶HaeⅢ和MspⅠ进行ARDRA分析。酶切反应体系如下:10 μL酶HaeⅢ反应体系包含酶HaeⅢ 0.5 μL,10×M buffer 1 μL,PCR产物8.5 μL;10 μL酶MspⅠ反应体系包含酶MspⅠ0.5 μL,10×T buffer 1 μL,BSA 1 μL,PCR产物7.5 μL。37 ℃水浴1 h,2%琼脂糖凝胶电泳进行检测,根据大小不同的酶切片段对其进行分型,将两种酶切带型一致的菌株定为同一种ARDRA型。分别在不同的ARDRA型中随机选取1~2个阳性克隆测序验证,由北京六合华大基因有限公司,通过M13引物双向测序。
1.2.4 序列分析 所得序列使用在线VecScreen(http://www.ncbi.nlm.nih.gov/tools/vecscreen/)工具去除载体片段,获得目的基因片段。应用BLASTN程序(http://blast.ncbi.nlm.nih.gov/Blast.cgi)搜索相似性序列,进行系统发育分析。利用Clusta1.81对序列进行多重比对,使用MEGA5软件中的Neighbor-Joining的建树方法构建系统发育树。
2 结果与分析
2.1 DNA的提取和特异性扩增
肠道中提取的微生物总DNA如图1所示,条带清晰,以总DNA为模板,进行特异性引物(Bac、Bd)PCR扩增,Bac引物扩增得到大小约为800 bp的特异性条带,引物Bd扩增得到大小约为500 bp的特异性条带(图2)。结果表明,在刺参消化道中存在数量众多的能被引物Bac、Bd特异扩增的BALOs。
2.2 阳性克隆检测
依据蓝白斑筛选,从两种引物阳性扩增转化平板上分别随机挑取单克隆60个,并编号Bac1-60和Bd1-60进行阳性验证。结果表明阳性结果清晰可见,仅少部分检测结果在目标区域没有条带出现,为阴性结果。分别保留阳性克隆菌株用作ARDRA分型。

图1 刺参消化道内容物微生物总DNAFig.1 Total DNA of microorganisms in alimentary canal of A. japonicas1:总DNA;M:Marker(DL2 000)1: Total DNA;M:Marker(DL2 000)

图2 16S rDNA PCR扩增产物Fig.2 PCR amplification of 16S rDNA1:Bac引物扩增产物;M:Marker(DL2 000);2:Bd引物扩增产物M:Marker(DL5 000)1: Amplification with primer Bac;M:Marker(DL2 000) 2: Amplification with primer Bd;M:Marker(DL5 000)
2.3 ARDRA分型
对于上述阳性克隆的ARDRA分型如图3、图4所示(为部分ARDRA分型图谱)。肉眼观测Bac扩增产物约有2种明显不同ARDRA型(类型A、B)及少量差异分型;Bd扩增产物约有4种明显不同ARDRA型(类型a~d)及少量差异分型。分别对引物Bac和Bd扩增产物对应的阳性克隆选取9和14个送样测序。
测序结果经过多重序列比对,共获得5个差异序列,引物Bac和Bd分别获得2和3个差异性16S rDNA序列。将该序列上传至GenBank核酸数据库中,Bd扩增序列分别获得接受号为GenBank KP303689~KP303691,Bac扩增序列分别获得接受号为GenBank KP303692、KP303693。如图5所示,Bac扩增产物阳性克隆中,Bac4(KP303693)代表菌株为优势种,ARDRA型(类型B)占总类型的56.0%;Bd扩增产物中Bd41(KP303689)代表菌株为优势种,有63.3%(类型a)。

图3 Bac扩增产物部分双酶切分型凝胶图Fig.3 Agarose gel electrophoresis of primer Bac amplified products digested by double enzymes上半部分为Bac扩增产物的Hae Ⅲ酶切;下半部分为其Msp Ⅰ酶切;M:Marker(DL2 000)The upper part of the map is the primer Bac amplified products digested by Hae Ⅲ ; the lower part is the products digested by Msp Ⅰ; M: Marker(DL2 000)

图4 Bd扩增产物部分双酶切分型凝胶图Fig.4 Agarose gel electrophoresis of primer Bd amplified products digested by double enzymes上半部分为Bd扩增产物的Hae Ⅲ酶切;下半部分为其Msp Ⅰ酶切;M:Marker(DL2 000)The upper part of the map is the primer Bd amplified products digested by Hae Ⅲ; the lower part is the products digested by Msp Ⅰ;M: Marker (DL2 000)
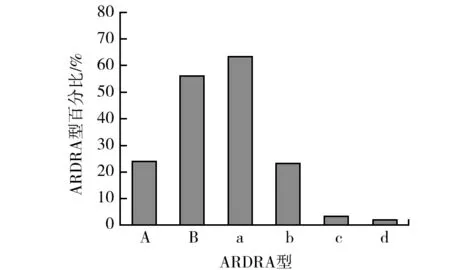
图5 ARDRA型在各60个蛭弧菌16S rDNA重组克隆子中的分布Fig.5 ARDRA distribution in 16S rDNA clones of BALOsA、B为Bac扩增产物双酶切分型类型;a~d为Bd扩增产物双酶切分型类型A、B are ARDRA patterns of primer Bac amplified products; a~d are ARDRA patterns of primer Bd amplified products
2.4 系统发育树分析
根据系统发育关系分析(图6),分离到的16S rDNA基因序列菌株分属于两个类群,其中Bac2(KP303692)和Bac4(KP303693)亲缘关系较近,分布于类群Ⅰ中,隶属于噬菌弧菌属(Bacteriovorax);Bd41(KP303689)、Bd58(KP303690)和Bd39(KP303691)亲缘关系较近,分布于类群Ⅳ中,隶属于蛭弧菌属(Bdellovibrio)。同时,上述菌株的序列与各自亲缘性最近的菌株的序列同源性最大值均为95.0%,可以认为这5个基因序列的菌株可能为潜在的新种。
3 讨 论
蛭弧菌类生物的生态学研究,以往依赖直接培养的方法,由于微生物分离培养技术的局限性,通常只能分离那些易于人工培养的微生物,因此限制了其中大多数不可培养的BALOs的研究。Zheng等[14]利用实时荧光定量PCR技术对一个海水样品中Bacteriovoraxspp.进行定量,其结果显示比经典双层平板法计数的结果高出两个数量级。本研究使用特异的16S rDNA克隆文库序列分析研究刺参消化道中的BALOs的系统多样性。该方法从类群的水平上分析了消化道BALOs的多样性及系统进化关系。

图6 基于16S rDNA基因序列的BALOs生物系统进化树Fig.6 Phylogenetic tree of intestine BALOs of A. japonicus based on the 16S rDNA gene sequences本研究中的序列用黑色实心圆圈标记;大于50%的置信值显示在系统进化树的各节点处;Cluster I:噬菌弧菌属;Cluster Ⅱ:吞菌弧菌属;Cluster III:其他;Cluster IV:蛭弧菌属The clones obtained in the present study are marked with black solid circle; Bootstrap values (>50%) are shown at nodes; Cluster I: Bacteriovorax; Cluster II: Peredibacter; Cluster III: other; Cluster IV: Bdellovibrio
蛭弧菌包括在δ-变形细菌纲(Deltaproteobacteria)蛭弧菌目(Bdellovibrionales),目前被分为蛭弧菌科(Bdellovibrionaceae)和噬菌弧菌科(Bacteriovoracaceae)两个科[15]。本研究从刺参消化道中分离的KP303689~KP303691和KP303692~KP303693分别分布在这两个科中。噬菌弧菌科(Bacteriovoracaceae)目前包含噬菌弧菌属(Bacteriovorax)和吞菌弧菌属(Peredibacter) ,特异性引物分别为Bac和Per。本研究对这两种引物同时进行扩增,然而只获得Bac引物的特异性扩增产物,肠道内容物中Per的特异性扩增产物并未得到,该产物在养殖水环境中扩增得到(另见报道)。而蛭弧菌科微生物由于耐盐性较差,目前未从大于5%盐水中分离到[16],本研究前期试验未在刺参养殖海水中检测到该类微生物,温崇庆等[7-8]也未在海水中检测到,而本研究从刺参消化道中检测到蛭弧菌科微生物。另外张志祥[10]、李楠[11]也从鳗鲡等动物肠道内容物中分离得到蛭弧菌菌株。说明蛭弧菌科菌株在海洋生物体内普遍存在,而在海洋环境中的分布量较噬菌弧菌科微生物少,而刺参肠道内噬菌弧菌科吞菌弧菌属的微生物相对较少。
本研究表明刺参消化道中存在着由Bacteriovorax和Bdellovibrio两大类别组成的多样性的BALOs。此次分离的5个16S rDNA序列的菌株为未报道的新菌株,与各自亲缘性最近的菌株序列同源性最大值均为95.0%,可以认为这5个基因序列的菌株可能为潜在的新种。由于环境条件的复杂性、研究方法的局限性,并且根据其较强的突变能力推断[17],刺参消化道中仍然存在未知的BALOs,其多样性的研究将在进一步工作中逐步完善。当前刺参养殖领域病害频发,将BALOs作为一种绿色环保的方式用于预防和清除养殖环境中的致病菌显得尤为重要,本研究工作致力于弄清刺参消化道中蛭弧菌的多样性及其优势种类,为BALOs作为疾病生物防治工具大规模应用打下基础。
[1] Snyder AR, Williams HN, Baer ML, et al. 16S rDNA sequence analysis of environmentalBdellovibrio-and-like organisms (BALO) reveals extensive diversity[J]. International Journal of Systematic Environmental Microbiology, 2002, 52(6): 2089-2094.
[2] 蔡姝文. 微生物制剂在水产养殖中的作用[J]. 现代农业科技, 2014, 18(1): 251-270.
[3] 王玉堂. 蛭弧菌及其在水产养殖动物疾病防治中的应用(连载一)[J]. 中国水产, 2014, (1): 50-51.
[4] 王玉堂. 蛭弧菌及其在水产养殖动物疾病防治中的应用(连载二)[J]. 中国水产, 2014, (2): 46-47.
[5] 王玉堂. 蛭弧菌及其在水产养殖动物疾病防治中的应用(连载三)[J]. 中国水产, 2014, (3): 52-54.
[6] Dwidar M, Monnappa AK, Mitchell RJ. The dual probiotic and antibiotic nature ofBdellovibriobacteriovorus[J]. Biochemistry and Molecular Biology Reports, 2012, 45(2): 71-78.
[7] 温崇庆. 海水养殖环境蛭弧菌类生物多样性及噬菌弧菌DA5对细菌的生物控制[D]. 广州: 中山大学, 2009.
[8] Wen CQ, Lai XT, Xue M, et al. Molecular typing and identification ofBdellovibrio-and-like organisms isolated from seawater shrimp ponds and adjacent coastal waters[J]. Journal of Applied Microbiology, 2009, 106(4): 1154-1162.
[9] 薛明,关敏丽,王飞燕.两种培养基对对虾苗池海洋蛭弧菌的分离及其多样性分析[J].微生物学通报,2014,41(9):1723-1732.
[10]张志祥. 鳗鲡肠道蛭弧菌的分离及其在预防水产养殖细菌性疾病的应用研究[D]. 厦门: 集美大学, 2009.
[11]李楠. 鸡源蛭弧菌的分离、生物学特性研究及对鸡白痢的治疗试验[D]. 雅安: 四川农业大学, 2005.
[12]Davidov Y, Friedjung A, Jurkevitch E. Structure analysis of a soil community of predatory bacteria using culture-dependent and culture-independent methods reveals a hitherto undetected diversity ofBdellovibrio-and-like organisms[J]. Environmental Microbiology, 2006, 8(9): 1667-1673.
[13]Davidov Y, Huchon D, Koval SF, et al. A new alpha-proteobacterial clade ofBdellovibrio-like predators: implications for the mitochondrial endosymbiotic theory[J]. Environmental Microbiology, 2006, 8(12): 2179-2188.
[14]Zheng G, Wang C, Williams HN, et al. Development and evaluation of a quantitative real-time PCR assay for the detection of saltwaterBacteriovorax[J]. Environmental Microbiology, 2008, l0(10): 2515-2526.
[15]Davidov Y, Jurkevitch E. Diversity and evolution ofBdellovibrio-and-like organisms (BALOs), reclassification ofBacteriovoraxstarriiasPeredibacterstarriigen. nov., comb. nov., and description of theBacteriovorax-Peredibacterclade asBacteriovoracaceaefam. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 2004, 54(5): 1439-1452.
[17]黄华伟, 邓时铭, 廖伏初. 蛭弧菌的生物学特性及其在刺参养殖中的应用前景[J]. 河北渔业, 2008, (5): 1-4.
Diversity Analysis ofBdellovibrio-Like Organisms in Spiny Sea Cucumber (Stichopusjaponicus) Intestine
HAN Min-yong, CHEN Hui-xia, SI Han, LIU Yang, CHEN Ya-dong
(KeyLab.ofMaricult. &StockEnhance’tinN.China,DalianOceanUni.,Minist.ofAgric.,Dalian116023)
The diversity ofBdellovibrio-like organisms inside the intestine of spiny sea cucumber (Apostichopusjaponicas) was studied. Total microbial DNA was extracted using intestinal content from the sea cucumber. The purpose fragments of 16S rDNA of BALOs were amplified using primers Bd and Bac respectively and connected to pMD 19-T vector, then transformed intoE.coliDH5 α. The 60 positive clones were selected respectively by blue white screening and verified by PCR detection. The patterns of different clones were determined using amplified ribosomal DNA restriction analysis (ARDRA) with double restriction enzymesHaeⅢ andMspI to cut 60 positive clones, respectively. Then, the positive clones sequence of different ARDAR patterns were selected and carried out biological analysis. The results showed that 3 and 2 different 16S rDNA sequences of primers Bd and Bac amplified products were obtained respectively. They belong to two different clusters ofBdellovibrioandBacteriovoraxrespectively. Comparison of these ssequences to those corresponding sequences in database all had a maximum similarity at 95%. These five sequences’ strains possibly are potential new species.
Bdellovibrio-like organisms; spiny sea cucumber (Apostichopusjaponicus); intestine; amplified ribosomal DNA restriction analysis (ARDRA); double enzyme digestion; diversity
辽宁省教育厅计划项目(L2012255)
韩民泳 男,硕士研究生。研究方向为海洋微生物研究。E-mail: minyhan@163.com
* 通讯作者。女,讲师,硕士生导师。研究方向为海洋生物研究。E-mail: chenhuixia@dlou.edu.cn
2015-06-02;
2015-07-08
Q78;Q93
A
1005-7021(2015)06-0044-05
10.3969/j.issn.1005-7021.2015.06.008

