合成Hg-DADPA柱分离含硒或硫化合物的研究
韩维娜,张 震,师 帅,柴 奇,刘学娜*
(1.哈尔滨医科大学药学院,黑龙江哈尔滨150081;2.哈尔滨医科大学大庆校区,黑龙江大庆163319)
研究植物及微生物体内生物大分子的结构修饰,对单一成分的纯化是一个重要且繁琐的过程[1-5]。到目前为止,在组成总核糖核酸(tRNA)中共存在107种不同结构修饰的核苷。越来越多的化学和生物学领域的科学家对硫或硒修饰的tRNA及其功能感兴趣[6],已发现近23种不同的硫和硒核苷修饰的tRNA[7]。到目前为止,研究微生物体 DNA与RNA的硒和硫结构修饰大多使用同位素示踪法,虽然灵敏度高,但是其放射性限制其在一般实验室的应用。因此,合成新的分离材料一直是一个热点。已有报道表明,用交联葡萄糖凝胶色谱从消化过的核苷酸混合物中选择性地分离出4-和2-硫代尿嘧啶核苷[8]。但是,目前还没有连接到对氯汞苯甲酸的报道。硫与硒在元素周期表中属于同一主族,因此其化学性质相似。如果某些载体能与硫结合,那么就能将含硒化合物分离。根据这个原则,笔者合成了一种新的汞柱,设计路线见图1。
合成的Hg-DADPA色谱纯化化合物是一个可逆过程。通过不同浓度的HCl洗脱,将不同位置硫或硒修饰的核苷酸选择性地分离。该研究方法简单,成本低,不破坏化合物的稳定性。Hg-DADPA色谱的高选择性和高回收率可推广至天然产物中含硫、含硒化合物及未知化合物的富集和分离,为下一步鉴定提供样品。
1 材料与方法
1.1 仪器和试剂 HR-MASS质谱仪(美国Waters公司);ZWY-200D恒温培养振荡器(上海智诚分析仪器制造有限公司)。碳酸氢钠(NaHCO3)pH 8.8,偶联缓冲液(0.1 mol/L MES,浓度 0.9%NaCl,pH 4.7),DADPA 琼脂糖购买于 Thermo公司;对氯汞苯甲酸、1-(3-二甲氨基丙基)-3-乙基碳二亚胺(EDC)和UTP购于Sigma公司;2-硫代尿嘧啶(2-SU)、2-硒代尿嘧啶(2-SeU)和4-硒代尿嘧啶核苷(4-SeUTP)由实验室提供。Baseline-ZEROTMDNase,RNase A,RiboShredderTMRNase Blend购买于Epicentre公司。
LB的组成:分别称取10 g胰蛋白胨、5 g酵母提取物和5 g氯化钠,加1 L双蒸水,定容,加热搅拌至溶解,灭菌后,在室温下放置,备用。
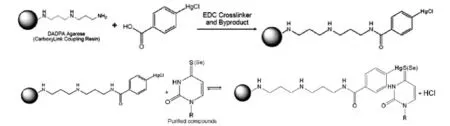
图1 设计路线
1.2 Hg-DADPA色谱的合成 取3 ml二氨基二异丙基琼脂糖 DADPA(16 ~20 μmol氨基/ml树脂),加入到0.5 cm ×8 cm柱中(最后柱床高度1 cm),自由滴下,使存储的缓冲液耗尽后,分3次加入偶合缓冲液(0.1 mol/L MES,浓度0.9%NaCl,pH 4.7),每次 3 ml,冲洗干净。在整个过程中,不要使树脂床干燥;平衡树脂,最后添加偶合缓冲液9 ml,转移DADPA琼脂糖至25 ml容量瓶中。取0.306 mmol对氯汞苯甲酸,溶解到6 ml二甲基甲酰胺中,混合几分钟,溶解后转移到 25 ml容量瓶中。另取0.774 mmol的 EDC 0.3 ml,溶解到偶合缓冲液中,然后加入装有树脂的圆底烧瓶中,在室温下搅拌,经过18 h反应,树脂转移到层析柱中,并且添加洗涤缓冲液(0.1 mol/L NaHCO3(pH=8.8)),洗涤至澄清,放至 4℃保存,备用。
1.3 Hg-DADPA色谱分离混合物 混合4-UTP、UTP、2-SU和2-SeU,使用Hg-DADPA色谱柱分离。步骤如下:使用移液器分别取2 μl的 10 mmol/L UTP(A)、2-SU(B)、2-SeU(C)和4-SeUTP(D),混合,然后加入1.8 ml双蒸水作为样品。使用Hg-DADPA色谱分离样品,洗脱剂分别是0.1 mol/L NaHCO3、0.002 mol/L HCl、0.005 mol/L HCl和0.5 mol/L HCl,其中0.6 ml/管,每份洗脱的馏分都要通过紫外全波长扫描检测,合并相同样品。其中,在13管、14管之间加双蒸水10 ml,洗脱残存的碳酸氢钠。
A=ECL计算样品回收率(n=3)
1.4 从大肠杆菌菌株K12(MG1665)提取tRNA 添加少量的大肠杆菌于LB中,4℃过夜,离心(6 000 r/min)8 min,弃上清液;留下约250 μl液体;加入含1 ml蛋白酶K组织和300 ml细胞裂解液,涡旋振荡5 min,65℃孵育120 min,再将样品置于冰上15 min,添加含175 ml的MPC蛋白沉淀试剂300 ml溶解样品,涡旋振荡1 min,4℃,离心(13 000 r/min)30 min;加入500 ml异丙醇,回收上清液;颠倒离心管30~40次,4℃离心20 min,小心倒出异丙醇;用浓度70%乙醇漂洗2次,不要接触沉淀;离心,用移液管取出所有残留的乙醇,得到总DNA和总RNA;加入6 ml不含RNA酶的游离水后,再加入 Baseline-ZEROTMDNase 200 μl(10 ×缓冲液,0.69 ml),37℃过夜;加入0.77 ml 3 mol/L NaCl,然后加入8 ml异丙醇,颠倒离心管30~40次;4℃,离心(3 000 r/min)30 min,得到tRNA。重复1次。
1.5 tRNA的消化和含硫核苷酸化合物的富集 将tRNA重新溶于3 ml双蒸水中,加入0.5 ml 5 μg/μl的 RNA 酶 A,混匀,并在37℃过夜孵育。然后,经Hg-DADPA色谱柱分离含硫或硒的核苷酸,洗脱剂分别是 0.1 mol/L NaHCO3、0.002 mol/L HCl、0.005 mol/L HCl和0.5 mol/L HCl,其中 0.6 ml/管,每份洗脱的馏分都要通过紫外全波长扫描检测,合并相同样品。
1.6 含硫核苷酸化合物的鉴定 水解tRNA后,经过Hg-DADPA色谱富集含硫、含硒核苷酸化合物,得到样品A和B,将样品经HPLC分析。HPLC条件如下:PRP-1反相柱,流速1 ml/min,柱温25℃,流动相A为10 mmol/L TEAAc(pH 7.1),流动相 B为含浓度60%CH3CN的10 mmol/L TEAAc(pH 7.1)。梯度洗脱,0~15 min 100%B,16~20 min 100%A到30%B,检测波长260、326 nm。对HPLC的每个组分进行ESI(-)-MS和HR-MS鉴定。
2 结果与分析
2.1 合成DADPA-Hg柱对混合物的分离和回收率测定 混合的UTP(A)、2-SU(B)、2-SeU(C)和4-SeUTP(D)有不同的最大紫外吸收峰,分别为260、300、307、365 nm。柱层析洗脱次序见图2。重复3次,使用计算公式:A=ECL,计算样品回收率,式中A代表吸光度;E代表吸光系数;C代表浓度;L代表比色皿厚度。UTP、2-SU、2-SeU、4-SeUTP的回收率分别约73%、87%、90%、85%(n=3)。Hg-DADPA色谱柱能够高效地通过不同浓度的HCl洗脱剂分离不同含硫或硒的化合物,且可重复使用。
2.2 样品稳定性 由表1可知,室温放置4 d后,2-硒代尿嘧啶核苷比4-硒代尿嘧啶核苷稳定,在酸性条件下4-硒代尿嘧啶核苷几乎100%降解;硒取代的化合物在中性和碱性条件下比酸性条件下要稳定,其中在中性条件下化合物几乎没有发生降解,pH越低,化合物的稳定性越差。2-硒代胸腺嘧啶核苷和4-硒代胸腺嘧啶核苷具有同样的试验结果,即2位比4位稳定,在酸性条件下化合物具有不稳定性。
2.3 未消化前提取大肠杆菌得到tRNA的总量和纯度 从1 L大肠杆菌中提取得到9 ml的RNA,取2 μl稀释到800 μl水中,得到紫外图谱,计算A260/A280=1.96,表明RNA纯度达到90%以上。
2.4 tRNA的消化和含硫、含硒核苷酸化合物的富集 0.1 mol/L盐酸洗脱液在326 nm处有吸收(11~13管,样品A),0.5 mol/L盐酸洗脱液在326和260 nm处均有吸收(样品B),通过紫外检测,大致能够判断水解程度,A260/A326为4~10(样品B),判断RNA没有彻底水解,每个修饰上面大概平均连接有4~6个未被修饰的碱基。由图3可知,所有馏分均显示在256 nm处有吸收。这可能由于是核苷酸降解后产生不完全水解。然而,11~13管均在326 nm处有强吸收,故此合并作为样品A,将进一步分析326 nm峰的组成。

表1 UTP、2-SU、2-SeU和4 SeUTP在不同溶剂、不同时间下的稳定性

图2 Hg-DADPA柱对硫硒修饰不同位置化合物的分离

图3 tRNA消化后,富集含硫及含硒核苷酸化合物

图4 加入不同浓度B2A6和不同pH样品A反应的紫外图谱
2.5 样品A的鉴定与分离
2.5.1 样品A的鉴定。首先,以B2H6为还原剂,加入后发现没有造成任何紫外吸收峰的改变(图4)。但是,样品A对pH敏感,尤其是在碱性条件下。样品吸收峰在酸性条件下转变非常缓慢,而在碱性条件下pH改变吸收峰也迅速发生改变。根据吸收峰的数值,这些变化可能是硫取代的核苷(硫尿嘧啶核苷)造成的。因此,据预测,样品A主要是寡核苷酸,且含有硫尿嘧啶的结构。
2.5.2 样品A的分离。样品A经过高效液相分析,收集到6个不同的组分,前5个无色,第6个黄色(图5)。
2.5.3 化合物鉴定。对HPLC的每个组分进行ESI(-)-MS and HR-MS鉴定。通过ESI(-)-MS(图6),分析所有的组分。684 m/z(M+-1)存在于后3个组分中,而668 m/z(M+-1)在6个组分中都存在,742 m/z(M+-1)存在于后3个组分中。水解RNA的酶核糖核酸酶A(RNA酶A)是一个内源性的残留3'端嘧啶,最终形成3'-嘧啶-3'-磷酸寡居核苷酸,因此根据紫外吸收峰和HR-MS,这2个峰分别对应到鸟嘌呤-2-硫代尿嘧啶核苷酸、腺嘌呤-3-硫代尿嘧啶核苷酸和鸟嘌呤-5-甲酰-2-尿嘧啶核苷酸(表2、图7)。

表2 大肠杆菌的tRNA消化后的核苷酸通过HR-MS的修饰和鉴定
3 讨论
使用二氨基二异丙基琼脂糖(DADPA)与对氯汞苯甲酸进行反应,得到Hg-DADPA色谱。用不同浓度的盐酸洗脱不同位置取代的硫或硒修饰化合物。这个反应是一个可逆反应。当增加HCl用量时,被Hg-DADPA色谱反应结合的产物会被游离出来,从而被洗脱,而当更换成其他酸时,不能达到洗脱和分离。Hg-DADPA色谱对酸是稳定的,因此Hg-DADPA色谱的再生可以使用0.5 mol/L HCl洗脱除杂,从而活化。Hg-DADPA还具有一些优点,如可重用性和高可靠性、高回收率。试验结果显示,最低的回收率达80%以上。该研究材料将帮助含硫和含硒修饰的内源性大分子的富集,从而有助于进一步确定生物大分子的修饰位置。
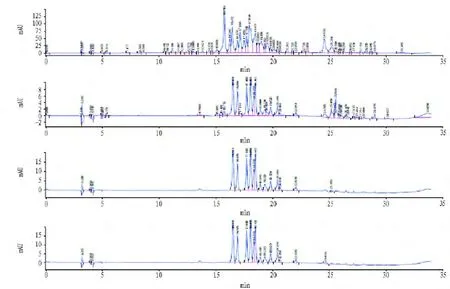
图5 HPLC分析和分离样品A
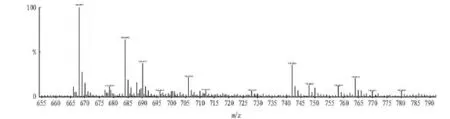
图6 大肠杆菌的tRNA消化后的核苷酸HR-MS

图7 化合物结构
使用合成的汞柱对来自大肠杆菌K12的tRNA中的含硫、含硒化合物分离,通过HR-MS鉴定得到鸟嘌呤-2-硫代尿嘧啶核苷酸、腺嘌呤-3-硫代尿嘧啶核苷酸和鸟嘌呤-5-甲酰-2-尿嘧啶核苷酸3个化合物。消化时使用RNA酶A,存在如下原因:核糖核酸酶A是一个内源性的核糖核酸酶,将最终水解得到3'-嘧啶-3'-磷酸根[9-10]。分离、富集后在326 nm处的吸收峰是类似合成的4-硫尿嘧啶核苷酸(峰值在330 nm处)。根据这一特点,进一步通过ESI-MS和HR-MS确定大肠杆菌核苷酸的RNA的G-4-硫代尿嘧啶核苷酸、A-3-硫代尿嘧啶核苷酸及G-5-甲酰-4-硫代尿嘧啶核苷酸。然而,没有发现硒修饰核苷酸。可能的原因是已知的自然发生的硫和硒核苷在所有类型的RNA修饰中非常低,因此下一步将扩大培养。
总之,已合成新的Hg-DADPA色谱,可用于分离硫和硒修饰的RNA。这是一个经济和安全的纯化方法,具有重复性和选择性。2005年,上海交通大学的科学家已发现一种新的DNA修饰,即DNA修饰中含有硫。这在核酸的研究上属于里程碑的科学文件[11-12]。这表明含有硒的DNA或RNA的修饰也可能存在于其他生物体中,或存在于更特殊的自然条件下生存的生物(如陨石坑的植物和微生物)。合成汞柱将有助于富集含硫含硒DNA或RNA修饰,使得检测简化,同时避免同位素对人体的伤害。今后,将使用这种新合成的Hg-DADPA色谱从天然植物中富集硫和硒衍生物。
[1]LIMBACH P A,CRAIN P F,MCCLOSKEY J A.Summary:The modified nucleosides of RNA[J].Nucleic Acids Res,1994,22(12):2183 -2196.
[2]CZERWONIEC A,DUNIN-HORKAWICZ S,PURTA E,et al.MODOMICS:A database of RNA modification pathways[J].Nucleic Acids Res,2013,41:262 -267.
[3]YU B,YANG Z,LI J,et al.Methylation as a crucial step in plant microRNA biogenesis[J].Science,2005,307:932 -935.
[4]MCCLOSKEY J A,ROZENSKI J.The small subunit rRNA modification database[J].Nucleic Acids Res,2005,33:135 -138.
[5]ROZENSKI J,CRAIN P F,MCCLOSKEY J A.The RNA modification database:1999 update[J].Nucleic Acids Res,1999,27:196 -197.
[6]DUNIN-HORKAWICZ S,CZERWONIEC A,GAJDA M J,et al.MODOMICS:A database of RNA modification pathways[J].Nucleic Acids Res,2006,34:145 -149.
[7]CANTARA W A,CRAIN P F,ROZENSKI J,et al.The RNA modification database,RNAMDB:2011 update[J].Nucleic Acids Research,2010,39:195-201.
[8]ONO M,KAWAKAMI M.Separation of newly-synthesized RNA by organomercurial agarose affinity chromatography[J].J Biochem,1977,81(5):1247-1252.
[9]VAN DEN WYMELENBERG A,GASKELL J,MOZUCH M,et al.Comparative transcriptome and secretome analysis of wood decay fungi postia placenta and phanerochaete chrysosporium[J].Appl Environ Microbiol,2010,76(11):3599 -3610.
[10]ZHAO K,LIU M,BURGESS R R.Promoter and regulon analysis of nitrogen assimilation factor,sigma54,reveal alternative strategy for E.coli MG1655 flagellar biosynthesis[J].Nucleic Acids Res,2010,38(4):1273-1283.
[11]ZHOU X,HE X,LIANG J,et al.A novel DNA modification by sulphur[J].Mol Microbiol,2005,57(5):1428 -1438.
[12]WANG L,CHEN S,XU T,et al.Phosphorothioation of DNA in bacteria by dnd genes[J].Nat Chem Biol,2007,3(11):709-710.

