在人转录调控网络中识别组织特异性功能模块
胡跃兵,李彭平(.南京大学医学院附属金陵医院神经外科,江苏南京 000;.南京大学生命科学院,江苏南京 003)
基因表达是调控子共同作用使特定的mRNA维持在稳定水平的过程。其中一类调控子是转录因子,它通过结合在其调控基因的启动子区,促进或抑制该基因的转录。它在调控细胞生长和凋亡中起到重要作用。很多转录因子还可作为疾病治疗的靶标[1]。而microRNA(miRNA)则是通过抑制mRNA表达或促进mRNA降解,在转录后水平调解基因的表达[2]。由此,转录因子和miRNA分别通过在转录水平和转录后水平调控基因表达,从而在影响基因功能过程中发挥重要作用。而转录因子本身的表达在其转录后水平也受到miRNA的调控,而miRNA自身的转录也要接受转录因子的调控。因此,细胞内基因的表达系统实际上是由转录因子、miRNA以及它们靶基因之间组成的一个复杂的调控网络。许多研究表明,特定生物学功能的实现是由一个包含多种相似功能分子的功能模块完成的。模块内部分子功能的相似度高于模块之间分子功能的相似度,并且分子在模块内的连接度远远高于模块间分子之间的连接度[3]。转录调控模块是由不同调控子调控特定靶基因集或同一类调控子调控不同靶基因集而形成的一个调控子网,是多个基因及其产物之间相互作用形成转录模块的结果[4]。研究转录调控网络模块结构有利于进一步了解细胞的转录调控、细胞分化及其功能。
组织特异性基因的表达在组织的发育、细胞类型、功能和转录调控中起到重要作用[5]。在已经识别的miRNA中,有很多表现出了组织特异性或发育阶段特异性的表达模式,并维持组织的特点和功能[6]。并且由转录因子形成的调控网络会控制组织特异性基因的表达。组织特异性的转录因子和miRNA对于组织特异性的调控和功能极其重要。基因转录表达过程实际上是调控子和靶基因之间形成功能模块作用的结果。由单个基因的组织特异性的表达调控的研究上升到模块的组织特异性调控模式的挖掘,可以进一步加深对基因组织特异性转录调控和组织功能的理解。为此,笔者从人体的7个组织调控网络出发,通过MCODE算法获得各个组织的调控模块,再通过计算模块间共有的节点数目获得组织间相似的模块或组织特异性模块,从而获得组织特异性的调控模式。
1 数据来源与方法
1.1 各组织表达谱数据来源 从UniGene[7]和 CGAP[8]获取人体的大脑、心脏、肾脏、肝脏、卵巢、脾和睾丸7个组织的蛋白质编码基因(包括TF和non-TF)的表达谱数据,而miRNA的表达谱数据则取自Landgraf等[6]的研究。
1.2 网络构建 通过以下方式构建包括人体大脑、心脏、肾脏、肝脏、卵巢、脾和睾丸7个组织的转录调控网络(TRN):首先在人基因组范围内预测TF、miRNA以及非蛋白质编码基因之间的调控关系,获得人的参考网络,然后通过对应组织的表达谱数据得到在7个组织中表达的基因,并从参考网络中获取这些基因之间的调控关系从而构建各个组织网络。在参考网络中,TF与靶基因之间的调控关系是先通过TRANSFAC数据库[9]找到靶基因上游1 kb的序列,之后在该段序列内寻找TF的靶点,该研究选取的是5个物种(人、狗、牛、小鼠和负鼠)保守的结果。而miRNA与靶基因之间的调控关系是通过取 Targetscan[10]、Pictar[11]和 Tarbase[12]3个预测结果的交集得到。
1.3 组织模块挖掘 生物网络可以直观地看成一个图形,图形节点和边分别表示生物分子和分子之间的关系。对于转录调控,则可以理解为一个有向的信号传递流,用有向图表示。一般从图中寻找子簇或局部紧密连接区域主要是依赖于网络流最小割理论或谱聚类。MCODE[13]是用节点聚类系数大小作为节点权重的衡量,从网络中获取分子复合体的模块挖掘方法。该算法主要分为节点权重计算、复合体预测、通过一定规则对复合体增加或减少节点3个阶段。MCODE在挖掘模块时会给每个模块计算一个得分并按照该得分对模块进行排序。模块得分越高,则排位越靠前,该模块内部连接也更加紧密。
1.4 模块相似性得分 用公式(1)计算模块相似性,式中N1、N2分别表示模块1和模块2的节点数,C表示2个模块共有节点个数。当2个模块节点大小和类型完全相同时,R等于1,表示2个模块完全相同。当2个模块之间的节点都不同时,共有节点数目C为0,此时R等于0。当R越接近1时,模块越相似。
得到模块相似得分后,以该得分作为模块间的距离,对模块进行系统聚类。聚类到一起的模块就是组织间相似的模块,而单个成簇的模块就是组织特异性模块。
2 结果与分析
2.1 组织模块 通过MCODE算法对人体7个组织的调控网络进行挖掘,得到大脑、心脏、肾脏、肝脏、卵巢、脾和睾丸的模块数目分别是 20、11、15、12、16、16 和 15(表 1)。得到模块后,统计模块中调控子的组成情况,发现所有组织大部分模块的调控子都包含转录因子和miRNA,只有少数模块里面的调控子只是转录因子或miRNA(表2),说明模块的作用模式主要是转录因子和miRNA协同调控,这与前人的研究结果“转录因子和miRNA在乳腺癌和前列腺癌中共同调控基因的表达”[14]是一致的。

表1 网络边的大小和模块数目
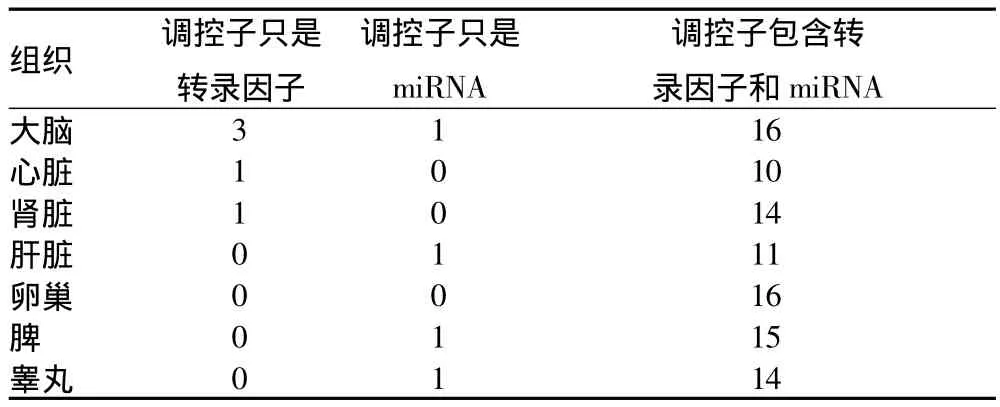
表2 模块的作用模式
计算每2个组织每2个模块之间的相似性得分。例如,计算大脑和心脏模块的相似性,则将大脑20个模块分别和心脏的11个模块一一进行计算。之后将组织相似性得分作为距离,对模块进行系统聚类。105个模块共聚为40类(图1)。其中,卵巢的第14个模块、肾脏的第14和第15个模块、心脏的第10个模块都各自聚类成一个簇,说明该4个模块和其他模块相似性很低,因此将其定义为组织特异性模块。
2.2 肾脏组织特异性模块 肾脏的第15个模块(图2)包含调控子 MTF1、hsa-miR-651、hsa-miR-708以及靶基因RAP1B和MTMR3。在该模块中,hsa-miR-651和 hsa-miR-708共同调控RAP1B和MTMR3的表达,同时hsa-miR-651又调控转录因子MTF1的表达。RAP1B是RAS-like小GTP结合蛋白超家族成员之一,其主要功能是调控细胞粘附、生长、分化、迁移增殖以及整合素介导的细胞信号转导[15]。有研究表明由RAP1B参与的cAMP/Rap1B/B-Raf通路可以调控结节性硬化症肾细胞癌p27的表达和p27-cyclin D1的细胞质的错误定位,从而影响肾细胞癌的发生和癌症的预后[16]。KEGG通路分析显示,RAP1家族与肾细胞癌通路密切相关[17]。RAP1家族主要通过细胞粘附、增殖和迁移而参与到肾细胞癌通路中(图3)。Hsa-miR-708被发现在肾细胞癌中能够诱导细胞凋亡并抑制肿瘤生长,是肾细胞癌中非常重要的肿瘤抑制子[18]。MTF1是一种核质穿梭作为转录因子,聚集在细胞核内并结合到含有金属反应元件(MRE)启动子上,主要是诱导金属硫蛋白及其他维持金属稳态相关蛋白的表达。而金属硫蛋白主要功能是抵抗重金属、清除自由基,许多研究表明其表达异常与肿瘤密切相关,已经作为结肠直肠癌和前列腺癌诊断的分子标记物[19]。综上所述,RAP1B和has-miR-708都与癌症密切相关,根据模块中分子功能相似性特点推测 hsa-miR-651可能是和has-miR-708协同调控RAP1B和MTMR3的表达。而has-miR-651又调控转录因子MTF1的表达,该转录因子又进一步作用于金属硫蛋白等相关蛋白,这些转录因子和miRNA形成的调控通路可能在肾细胞癌的形成发展中起重要作用。该模块中的节点及其调控关系需要通过试验进一步证实。
2.3 心脏组织特异性模块 心脏的第10个模块由GSK3B、CPEB3、hsa-miR-196b和转录因子HAND1组成(图4),在系统聚类中独自成簇,是心脏的一个组织特异性模块。HAND1是心脏特异性转录因子,主要功能是参与血管生成、发育以及肌肉组织形成。并且在心室隔和心脏形态学形成中也起着重要作用。HAND1和其另外一个家族成员以互补的方式工作,调节右心室和主动脉弓动脉的形成,提示HAND1和其家族成员可能会介导为先天性心脏病。Hatemi等研究表明HAND1序列的变异在心房异构的发生和发展中可能起重要作用[20]。而在先天性心脏病中也发现HAND1等基因的拷贝数变异,这些变异可能与心脏功能缺陷紧密相关[21]。
GSK3B是一种丝氨酸苏氨酸激酶,属于糖原合酶激酶亚家族,是为数不多的信号分子,调节关键的细胞内信号转导通路。在大鼠的胚胎发育过程中,GSK3B的缺失会导致胚胎干细胞中受损的心肌细胞的分化并促使心肌细胞显著增生,从而使心室被心肌细胞填充[22]。研究表明,GSK3B是调节心肌纤维化的重要调控子,心脏成纤维细胞中GSK-3β特异性缺失会导致心脏纤维化、左心室功能障碍和在缺血性心脏中产生过多的疤痕[23]。
Hsa-miR-196b主要功能是参与粒细胞生成、脂质代谢和造血功能[24]。而粒细胞升高会导致心内膜炎和心肌病。CPEB3、hsa-miR-196b可能也与心室隔形成、心肌细胞功能相关。具体作用机制可能是hsa-miR-196b作用于转录因子HAND1,HAND1再作用于GSK3B,该调控通路在心室隔的形成、维持心肌细胞正常功能中发挥重要作用,需要通过试验进一步证实。
3 结论
通过构建人体7个组织的调控网络,并对各调控网络进行模块挖掘与比较分析,得到了与肾癌密切相关的肾脏特异性功能模块,以及和室间隔的形成有关并参与心肌细胞功能的心脏特异性模块。这些模块中的基因以及基因之间的调控关系对于相应组织疾病的治疗可能有重要作用。
[1]WESTON V.Large scale screening for DNA damage-induced transcription factors as potential targets for treatment of CLL with p53 apoptotic defect[J].Blood,2009,114(22):1336.
[2]ERHARD F,HAAS J,LIEBER D,et al.Widespread context dependency of microRNA-mediated regulation[J].Genome Res,2014,24(6):906 -919.
[3]TORNOW S,MEWES H W.Functional modules by relating protein interaction networks and gene expression[J].Nucleic Acids Res,2003,31(21):6283-6289.
[4]SEGAL E,FRIEDMAN N,KOLLER D,et al.A module map showing conditional activity of expression modules in cancer[J].Nat Genet,2004,36(10):1090-1098.
[5]NIEHRS C,POLLET N.Synexpression groups in eukaryotes[J].Nature,1999,402(6761):483 -487.
[6]LANDGRAF P,RUSU M,SHERIDAN R,et al.A mammalian microRNA expression atlas based on small RNA library sequencing[J].Cell,2007,129(7):1401-1414.
[7]SAYERS E W,BARRETT T,BENSON D A,et al.Database resources of the national center for biotechnology information[J].Nucleic Acids Res,2012,36:13 -21.
[8]LASH A E,TOLSTOSHEV C M,WAGNER L,et al.SAGEmap:A public gene expression resource[J].Genome Res,2000,10(7):1051 -1060.
[9]WINGENDER E.The TRANSFAC project as an example of framework technology that supports the analysis of genomic regulation[J].Briefings in Bioinformatics,2008,9(4):326 -332.
[10]LEWIS B P,SHIH I H,JONES-RHOADES M,et al.Prediction of mammalian microRNA targets[J].Cell,2003,115(7):787 -798.
[11]KREK A,GRUN D,POY M N.Combinatorial microRNA target predictions[J].Nature Genetics,2005,37(5):495 -500.
[12]SETHUPATHY P,CORDA B,HATZIGEORGIOU A G.TarBase:A comprehensive database of experimentally supported animal microRNA targets[J].RNA,2006,12(2):192 -197.
[13]BADER G D,HOGUE C W.An automated method for finding molecular complexes in large protein interaction networks[J].BMC Bioinformatics,2003,4:2.
[14]WU J H,SUN Y J,HSIEH P H,et al.Inferring coregulation of transcription factors and microRNAs in breast cancer[J].Gene,2013,518(1):139-144.
[15]DUBE N,KOOISTRA M R,PANNEKOEK W J,et al.The RapGEF PDZGEF2 is required for maturation of cell-cell junctions[J].Cellular Signalling,2008,20(9):1608 -1615.
[16]COHEN J D,THAM K Y,MASTRANDREA N J,et al.cAMP-dependent cytosolic mislocalization of p27(kip)-cyclin D1 during quinol-thioetherinduced tuberous sclerosis renal ceu carcinoma[J].Toxicol Sci,2011,122(2):361-371.
[17]KANEHISA M,GOTO S,SATO Y,et al.Data,information,knowledge and principle:Back to metabolism in KEGG[J].Nucleic Acids Research,2014,42(D1):199 -205.
[18]BIRKHAUSER F D,KOYA R C,NEUFELD C,et al.Dendritic cell-based immunotherapy in prevention and treatment of renal cell carcinoma:Efficacy,safety,and activity of Ad-GM.CAIX in immunocompetent mouse models[J].J Immunother,2013,36(2):102 -111.
[19]ARRIAGA J M,BRAVO I A,BRUNO L,et al.Combined metallothioneins and p53 proteins expression as a prognostic marker in patients with Dukes stage B and C colorectal cancer[J].Human Pathology,2012,43(10):1695-1703.
[20]HATEMI A C,GULEC C C ,CINE N,et al.Sequence variations of NKX2-5 and HAND1 genes in patients with atrial isomerism[J].Anadolu Kardiyoloji Dergisi-the Anatolian Journal of Cardiology,2011,11(4):319-328.
[21]GOLDMUNTZ E,PALURU P,GLESSNER J,et al.Microdeletions and microduplications in patients with congenital heart disease and multiple congenital anomalies[J].Congenital Heart Disease,2011,6(6):592 -602.
[22]KERKELA R,KOCKERITZ L,MACAULAY K,et al.Deletion of GSK-3beta in mice leads to hypertrophic cardiomyopathy secondary to cardiomyoblast hyperproliferation[J].J Clin Invest,2008,118(11):3609 -3618.
[23]LAL H,AHMAD F,WOODGETT J,et al.The GSK-3 family as therapeutic target for myocardial diseases[J].Circulation Research,2015,116(1):138-149.
[24]LU M,ZHANG Q,DENG M,et al.An analysis of human microRNA and disease associations[J].PLoS One,2008,3(10):3420.

