清香型大曲细菌群落结构的比较分析
叶光斌 王彩虹 王 毅 甄 攀 王 勇 罗惠波
(1.四川理工学院生物工程学院,四川 自贡 643000;2.酿酒生物技术及应用四川省重点实验室,四川 自贡 643000;3.五粮液集团质量检测中心,四川 宜宾 644000;4.山西杏花村汾酒厂股份有限公司,山西 汾阳 032200)
清香型大曲是中国大曲清香型白酒的生产用曲,主要包括清茬曲、红心曲和后火曲3种大曲。这3种大曲在生产上使用的原材料相同,生产流程也基本相似,可简单概括为:卧曲→上霉期→晾霉期→潮火期→干火期→后火期→养曲→出房验收→贮曲。所不同的是在培曲过程中,各曲种对培曲温度的要求各不相同(见表1)。不同的制曲温度及生产工艺,使得不同大曲内的微生物生长和发酵特征各具特色,其产生的不同代谢产物对白酒风味形成也有所差异[1]。清香型白酒生产中将清茬曲、红心曲和后火曲按照4∶3∶3的比例混合后使用[2]。采用不同工艺生产的3种大曲共同发酵,相辅相成,便形成了清香型白酒(大曲清香)独特的风格。因此,认识清香型大曲内不同曲种间微生物群落结构与差异,对于研究清香型白酒发酵具有十分重要的意义。
原核细胞核糖体小亚基16SrRNA基因,长约1.5kb左右,大小适中,既含有高度保守的基因片段又具有种间变异的核酸片段,非常适合于进行序列测定和菌属间的分析比较,是理想的基因鉴定靶序列[3]。张守印等[4]对16SrDNA克隆文库用于细菌群落分析的可靠性进行了研究,结果表明,16SrRNA基因序列分析可以反映菌群中各种细菌的丰度,是一种较好的分析细菌菌群的方法。目前,16SrRNA基因克隆文库技术也已被广泛应用于发酵食品[5,6]、城市污水[7]、土壤[8]等各类复杂环境中细菌微生态的研究。
本研究拟以汾酒清香型大曲—清茬曲、后火曲、红心曲为研究对象,通过提取大曲微生物总DNA、PCR扩增及构建细菌16SrRNA基因文库,对其细菌群落组成进行了比较研究,确定了3种大曲的优势菌群,为进一步解析清香型大曲功能微生物、筛选优良酿造微生物奠定基础。
1 材料与方法
1.1 材料与仪器
大曲样品:采集自山西杏花村汾酒集团有限责任公司,全部为成品曲(见表1)。每个大曲样品取3个平行,按“四分法”[9]将不同类型大曲分别粉碎混合,用无菌封口袋分装后,置于-80℃保藏备用。
引物:由上海英俊生物技术有限公司合成;
Taq酶、pMD19-T载体和EscherichiacoliDH5α感受态细胞、限制性内切酶MspI和HhaI:大连宝生物有限公司;
PCR仪:BIO-RAD Thermal Cycle型,美国 BIO-RAD公司;
离心机:Eppendorf Centrifuge 5418型,美国Eppendorf公司。
表1 试验所用大曲样品信息表Table 1 Information of Daqu samples of the experiment

表1 试验所用大曲样品信息表Table 1 Information of Daqu samples of the experiment
来源山西杏花村。
名称 类型(制曲最高品温) 所产白酒香型汾酒清茬曲 中温曲(45~46℃)清香型清香型汾酒后火曲 中温曲(46~48℃) 清香型汾酒红心曲 中温曲(最高可达50℃)
1.2 试验方法
1.2.1 总DNA的提取及PCR扩增 DNA提取参照文献[10]。引物 Eub27f(5’-AGA GTT TGA TCC TGG CTC AG-3’)和 Eub1492r(5’-CGG TTA CCT TGT TAC GAC TT-3’)用于扩增细菌16SrDNA[11,12],为了防止 PCR 偏差,每个样品做3个重复。具体的PCR扩增体系及PCR扩增程序同叶光斌等的方法[10,12]。
1.2.2 细菌16SrRNA基因克隆文库的构建 将纯化后的PCR产物与pMD-19T载体16℃过夜连接,通过热激法(42℃热激90s)将连接产物转化到感受态细胞E.coliDH5α(感受态细胞的效价约108个/μg)中。
1.2.3 阳性克隆子的筛选及RFLP分析 菌落PCR筛选阳性克隆 子。引 物 RV-M(5'-GAG CGG ATA ACA ATT TCA CAC AGG-3')和 M13-47(5'-CGC CAG GGT TTT CCC AGT CAC GAC-3')用于菌落PCR扩增。PCR扩增体系及PCR扩增程序同叶光斌等的方法[10,12],利用MspI和HhaI双酶切阳性克隆子菌落PCR产物的酶切图谱,确定不同谱型,统计每一谱型出现的频率及数目,并将不同谱型的克隆子送到上海杰李生物技术有限公司进行DNA测序。
1.2.4 数据分析 将16SrDNA序列相似性≥97%的序列定义为同一OTU(operational taxonomic units,运算分类单位)[10,12]。根据测序结果和RFLP统计分析结果,统计各文库内OTU数目及每个OTU在个克隆文库的分布比例。将每个OTU的代表克隆子序列与NCBI数据库进行比对,找出与克隆子序列同源性最高的序列,用于构建系统发育树和确定其分类地位。Seaview4软件对齐序列,Mega 4软件构建系统发育树。文库的评估方法参考叶光斌等的方法[10,12]。共有32个克隆子的16SrRNA基因序列上传至NCBI Gen-Bank数据库,GenBank序列号为KF984353-KF984384。
2 结果与分析
2.1 细菌16SrRNA基因的PCR扩增
不同大曲细菌16SrRNA基因PCR扩增产物的琼脂糖电泳图谱见图1。由图1可知,3种大曲样品的细菌16SrDNA PCR扩增效果都很好,均得到了一条较亮的1 500bp左右的目的条带。

图1 细菌16SrRNA基因PCR产物的电泳图谱Figure 1 The electrophoresis diagram of PCR products of bacterial 16SrRNA gene
2.2 阳性克隆子检测及RFLP分析
部分菌落PCR产物电泳图谱见图2。菌落PCR产物为单条带,且片段长度在1 600bp左右的均可视为阳性克隆子。清茬曲、后火曲和红心曲细菌16SrRNA基因文库内分别有45,60和45个阳性克隆子用于后续的RFLP分析。将各文库的阳性克隆子菌落PCR产物通过MspI和HhaI双酶切后,进行RFLP分析,部分RFLP电泳图谱见图3。不同大曲样品根据RFLP分析结果,挑选不同谱型的32个代表克隆子进行测序,并与NCBI GenBank数据库比对(见表3)。

图2 部分克隆子菌落PCR产物电泳图谱Figure 2 The electrophoretogram of colony PCR products of partial colonies

图3 部分阳性克隆子菌落PCR产物双酶切产物电泳图谱(MspI和HhaI双酶切)Figure 3 The electrophoretogram of double enzyme digested products from PCR products of partial positive colonies(Digested with MspI and HhaI)
2.3 细菌16SrRNA基因文库的评估
3种大曲细菌16SrRNA基因文库的统计结果见表2。从文库覆盖率上看,在64%~72%,均未达到饱和;Shannon指数和Simpson指数显示3种清香型大曲内细菌群落多样性丰富;且从物种多样性上看,后火曲>清茬曲>红心曲。Buzas-Gibson均一度指数的分析结果表明,红心曲>后火曲>清茬曲。尽管以上3个文库均未达到饱和,但该文库能在一定程度上反映各样品内的主要细菌类群的多样性及丰度差异。
2.4 清香型大曲细菌群落结构
清茬曲、后火曲、红心曲的细菌16SrRNA基因文库内代表克隆子在文库中的分布及NCBI数据库比对信息见表3。由表3可知:克隆子与对应序列间的相似度均达到98%以上。代表克隆子参与构建的系统发育树见图4。由图4可知,所有清香型大曲内细菌群落主要分布于芽孢杆菌纲、放线菌纲及变形菌纲。芽孢杆菌纲内克隆子主要分布于乳杆菌目(Lactobacillales)和芽孢杆菌目(Bacillales)。其中乳杆菌目内克隆子种类最多,共有11个OTUs,主要分布于乳杆菌属(Lactobacillus)、魏斯氏菌属(Weissella)、明串株菌属(Leuconostoc)、乳球菌属(Lactococcus);芽孢杆菌目内克隆子包含有5个OTUs,分别为芽孢杆菌属(Bacillus)、葡萄球菌属(Staphylococcus)、高温放线菌属(Thermoactinomyces)。放线菌纲内克隆子主要分布于放线菌目(Actinomycetales)内的链霉菌属(Streptomyces)、短杆菌属(Brevibacterium)、考克氏菌属(Kocuria)及Brachybacterium属。变形杆菌纲内克隆子主要分布于不动杆菌属(Acinetobacter)和泛菌属(Pantoea)。
表2 不同清香型大曲细菌16SrRNA基因文库的统计分析Table 2 The statistical analysis of bacterial 16SrRNA gene libraries of different Fen-Daqus

表2 不同清香型大曲细菌16SrRNA基因文库的统计分析Table 2 The statistical analysis of bacterial 16SrRNA gene libraries of different Fen-Daqus
通常Shannon-Wiener指数数值越大,表明文库中克隆子多样性越丰富;Simpson指数越接近1,表明文库中克隆子多样性越丰富;Buza-Gibson均一度指数越接近1,表明文库中克隆子分布越均匀[10,12,13]。
文库来源 克隆子数目 OUT数目 文库覆盖率/%Buzas-Gibson均一度指数清茬曲Shannon-Wiener指数Simpson指数45 13 71.11 2.39 0.89 0.84 45 16 64.44 2.51 0.90 0.78后火曲 60 19 68.33 2.71 0.92 0.79红心曲
从克隆子分布表和系统发育树可以看出:乳杆菌属、葡萄球菌属、芽孢杆菌属、魏斯氏菌属、明串株菌属在3个大曲中同时存在;高温放线菌属只存在于制曲品温较高的后火曲和红心曲中;乳球菌属和短杆菌属只存在于清茬曲和后火曲中;泛菌属只存在于清茬曲和红心曲中;考克氏菌属和短杆菌属是清茬曲的特有菌群;链霉菌属和不动杆菌属是后火曲的特有菌群。其中,清茬曲的优势菌群为乳杆菌属(占全部克隆子的比例为37.78%,7OTUs)和葡萄球菌属(22.22%,1OTU);后火曲的优势菌群为乳杆菌属(占全部克隆子的 比 例 为 35%,8OTUs)、葡 萄 球 菌 属 (16.67%,2 OTUs)、芽孢杆菌属(16.67%,2OTUs)、魏斯氏菌属(13.33%,1OTU);红心曲的优势菌群为乳杆菌属(占全部克隆子的比例 为 26 .67%,6OTUs)、芽 孢 杆 菌 属 (28.89%,2 OTUs)、高温放线菌属(17.78%,1OTU)。总体而言,清香型3种曲的细菌群落结构存在一定的差异,后火曲分别与清茬曲和红心曲比较相似,而清茬曲和红心曲间群落相似性较低。
据周恒刚[14]、胡承等[15]报道,制曲品温在30℃左右较适合微生物的生长,而随着温度的上升微生物的活菌数也呈倍下降,大多数不耐高温的细菌逐渐死亡。汾酒3种大曲微生物群落的差异与大曲生产过程中温度的控制密切相关。
红心曲制曲温度最高,因此耐高温的芽孢杆菌和高温放线菌与不耐高温的细菌相比具有较大的竞争优势,在文库中占了较高的比例,但细菌群落多样性较其它两种曲低;后火曲的制曲温度较清茬曲的高,因此后火曲中芽孢杆菌所占的比例明显高于清茬曲,后火曲中也有较低含量的高温放线菌分布;而清茬曲制曲温度最低,未检测到高温放线菌的存在。不同的制曲温度使得微生物的生长和发酵特征也各具特色,其产生的不同代谢产物对白酒风味形成也有所差异,汾酒采用不同工艺生产的3种大曲共同发酵,三者相辅相成,便形成了汾酒独特的风格。
表3 不同清香型大曲细菌16SrRNA基因文库内代表克隆子的分布及比对信息Table 3 Blast and distribute information of represental clones from 16SrRNA gene libraries of different Fen-Daqus
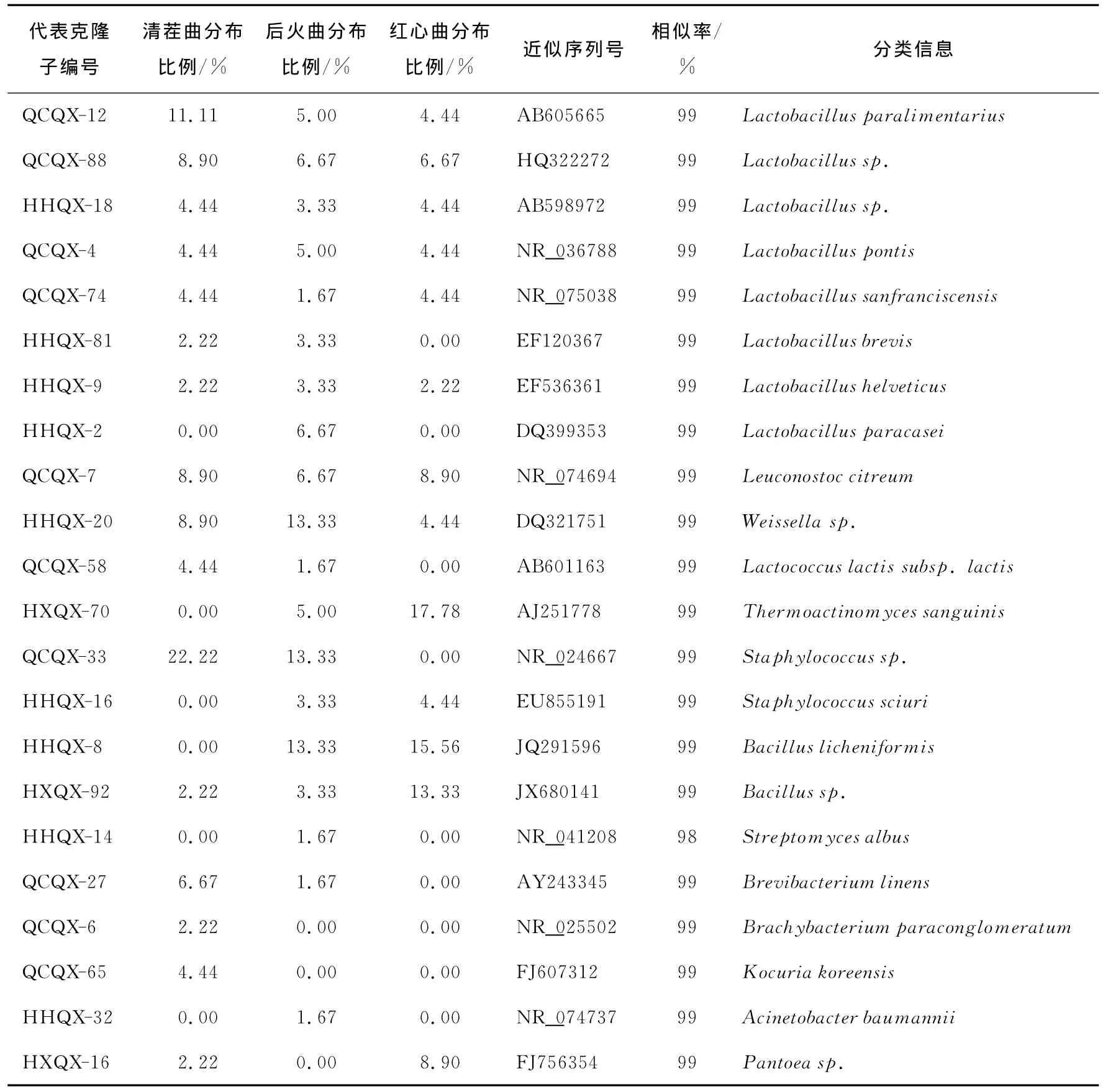
表3 不同清香型大曲细菌16SrRNA基因文库内代表克隆子的分布及比对信息Table 3 Blast and distribute information of represental clones from 16SrRNA gene libraries of different Fen-Daqus
“QCQX”、“HHQX”、“HXQX”分别代表清茬曲、后火曲、红心曲细菌16SrDNA文库克隆子。
代表克隆子编号清茬曲分布比例/%后火曲分布比例/%红心曲分布比例/% 近似序列号 相似率/%分类信息QCQX-12 11.11 5.00 4.44 AB605665 99 Lactobacillus paralimentarius QCQX-88 8.90 6.67 6.67 HQ322272 99 Lactobacillus sp.HHQX-18 4.44 3.33 4.44 AB598972 99 Lactobacillus sp.QCQX-4 4.44 5.00 4.44 NR_036788 99 Lactobacillus pontis QCQX-74 4.44 1.67 4.44 NR_075038 99 Lactobacillus sanfranciscensis HHQX-81 2.22 3.33 0.00 EF120367 99 Lactobacillus brevis HHQX-9 2.22 3.33 2.22 EF536361 99 Lactobacillushelveticus HHQX-2 0.00 6.67 0.00 DQ399353 99 Lactobacillusparacasei QCQX-7 8.90 6.67 8.90 NR_074694 99 Leuconostoc citreum HHQX-20 8.90 13.33 4.44 DQ321751 99 Weissella sp.QCQX-58 4.44 1.67 0.00 AB601163 99 Lactococcus lactis subsp.lactis HXQX-70 0.00 5.00 17.78 AJ251778 99 Thermoactinomyces sanguinis QCQX-33 22.22 13.33 0.00 NR_024667 99 Staphylococcus sp.HHQX-16 0.00 3.33 4.44 EU855191 99 Staphylococcus sciuri HHQX-8 0.00 13.33 15.56 JQ291596 99 Bacillus licheniformis HXQX-92 2.22 3.33 13.33 JX680141 99 Bacillus sp.HHQX-14 0.00 1.67 0.00 NR_041208 98 Streptomyces albus QCQX-27 6.67 1.67 0.00 AY243345 99 Brevibacterium linens QCQX-6 2.22 0.00 0.00 NR_025502 99 Brachybacterium paraconglomeratum QCQX-65 4.44 0.00 0.00 FJ607312 99 Kocuria koreensis HHQX-32 0.00 1.67 0.00 NR_074737 99 Acinetobacter baumannii HXQX-16 2.22 0.00 8.90 FJ756354 99 Pantoea sp.
潘勤春等[16]通过PCR—DGGE技术对汾酒大曲细菌群落结构进行了分析,结果表明制曲品温最低的清茬曲多样性最丰富,制曲品温最高的红心曲群落组成最简单,后火曲居中,且清茬曲和红心曲间群落组成差异较大,测序结果表明,清香型大曲内细菌主要为芽孢杆菌属、肠杆菌属、乳酸菌属、不动杆菌属、土壤球菌属,且芽孢杆菌属和肠杆菌属为优势类群。高亦豹[17]运用PCR—DGGE技术对不同工艺大曲(包括汾酒清茬曲、后火曲、红心曲)的微生物群落结构进行了研究,结果表明清香型大曲中乳酸菌多样性较丰富,包括Lactobacillu.helveticus、L.panis、L.pontis、L.fermentum、Weissellacibaria;地衣芽孢杆菌和枯草芽孢杆菌为大曲中的优势菌;而Thermoactinomycessanguinis仅存在于酱香型大曲中。Zhang等[18]通过454测序技术研究汾酒3种清香型大曲内细菌群落组成,结果表明3种大曲内细菌种群主要分布于厚壁菌门和放线菌门,厚壁菌门是优势种群,分别占清茬曲、后火曲和红心曲的72.2%,98.6%,81.4%;乳酸菌种类丰富,主要包括Weissellacibaria、Leuconostoccitreum、Lactobacillussakei、L.pontis、L.paralimentarius和L.mindensis;清茬曲内优势科为链霉菌科和明串珠菌科;后火曲内优势菌为芽孢杆菌;红心曲内优势菌为高温放线菌科和链霉菌科。但未见γ-变形杆菌的报道。本试验对汾酒大曲的研究中也发现乳酸菌的多样性非常丰富,芽孢杆菌是红心曲和后火曲的优势菌群,这一结论与潘勤春[16]、高亦豹[17]、Zhang[18]等的结论较一致;不同的是在具体的属种组成比例上存在差异,且本研究得到的细菌多样性更丰富。此外本研究在清香型大曲的后火曲和红心曲内均检测到了较高比例的高温放线菌(Thermoactinomycessanguinis)。导致以上差异的原因可能是由于试验方法或试验样品的不同导致的。

图4 清香型大曲细菌克隆子16SrRNA基因系统发育树Figure 4 Phylogenetic tree of bacterial 16SrRNA gene of Fen-Daqus
3 结论
通过细菌16SrRNA基因文库法和RFLP指纹技术系统研究了清香型大曲清茬曲、后火曲和红心曲内细菌群落结构差异。研究结果表明:制曲温度对于清香型3种大曲内细菌多样性具有显著影响,制曲温度较低的清茬曲和后火曲的细菌多样性要明显高于制曲温度较高的红心曲。3种大曲的细菌多样性丰富,均分布于芽孢杆菌纲、放线菌纲及变形菌纲,但在具体种属组成上存在明显差异;其中清茬曲的优势细菌群为乳杆菌属和葡萄球菌属;后火曲的优势细菌群为乳杆菌属、葡萄球菌属、芽孢杆菌属、魏斯氏菌属;红心曲的优势细菌群为乳杆菌属、芽孢杆菌属、高温放线菌属。
此外,本试验在进行测序序列与NCBI数据库比对过程中发现,部分克隆子的测序序列与NCBI数据库中多个同属不同种菌株的序列相似度都达到98%以上,如代表克隆子HHQX-20,与魏斯氏菌属的Weissellaconfusa、W.cibaria的相似度均为99%;HXQX-92与芽孢杆菌属的Bacillus aerophilus、B.stratosphericus、B.pumilus相似度均达到98%以上;QCQX-88与乳杆菌属的L.farciminis、L.futsaii、L.crustorum的相似度均达到98%以上。而在群落多样性分析时,由于只将序列相似度≥97%计为同一种OTU类型,因此可能会低估了清香型大曲的细菌多样性。后期的研究将结合高通量测序技术,以进一步确定以上大曲内的细菌群落组成信息,并有目的的针对清香型大曲的主要细菌类群进行分离、纯化和发酵机理的相关研究,以弄清其在清香型白酒固态发酵中的具体作用。
1 赵景龙.浅谈清香型酒三种大曲的差异性[J].酿酒,2000(5):43~44.
2 沈怡方.白酒生产技术全书[M].北京:中国轻工业出版社,2013.
3 都立辉,刘芳.16SrRNA基因在细菌菌种鉴定中的应用[J].乳业科学与技术,2006(5):207~209.
4 张守印,郭学青,李振军,等.16SrRNA基因克隆文库用于菌群分析的效能研究和评论[J].第三军医大学学报,2008,30(16):1 549~1 552.
5 胡佳,邓斌,张学文.浓香型白酒曲药中细菌组成及系统学分析[J].酿酒科技,2007(5):17~19.
6 陈希,沈锡权,翁佩芳,等.应用16SrDNA克隆文库法分析雪菜低盐腌制过程微生物群落的多样性[J].中国食品学报,2012,12(7):205~210.
7 窦娜莎,王琳.16SrDNA克隆文库法分析Biostyr曝气生物滤池处理城市污水的细菌多样性研究[J].环境科学学报,2011,31(10):2 117~2 124.
8 黄晟,陈旸,潘雪莲,等.甘肃西峰黄土中细菌16SrDNA文库的构建与组成初步分析[J].高校地质学报,2012,18(4):765~772.
9 吴谋成.食品分析与感官评定[M].北京:中国农业出版社,2002.
10 叶光斌,董瑞丽,王彩虹,等.基于免培养法的中高温大曲细菌群落结构研究[J].江苏农业科学,2013,41(8):333~336.
11 Delong E F.Archaea in coastal marine environments[J].Proc.Natl Acad Sci.USA,1992,89(12):5 685~5 689.
12 叶光斌,罗惠波,杨晓东,等.基于免培养法研究泸州地区浓香型白酒窖泥原核微生物群落结构[J].食品科学,2013,34(17):176~181.
13 叶光斌,王风平,肖湘.东太平洋中国多金属结核区锰结核样品中微生物群落结构的研究[J].台湾海峡,2010,29(2):218~227.
14 周恒刚.制曲热能[J].酿酒科技,2004(5):35~37.
15 胡承,邬捷锋,沈才洪,等.浓香型(泸型)大曲的研究及其应用[J].酿酒科技,2004(1):33~36.
16 潘勤春,孟镇,钟其顶,等.汾酒大曲细菌群落结构的PCR—DGGE分析[J].酿酒科技,2011(6):95~99.
17 高亦豹.PCR—DGGE研究中国白酒大曲中微生物群落结构[D].无锡:江南大学,2010.
18 Zhang X,Zhao J,Du X.Barcoded pyrosequencing analysis of the bacterial community of Daqu for light-flavour Chinese liquor[J].Letters in Applied Microbiology,2014,58(6):549~555.

