中国南海一株固氮类芽孢杆菌的筛选和分离鉴定
张丽红 , 刘艳芳 胡青平
(1.山西师范大学 生命学院, 山西 临汾 041004; 2.中国农业大学 农业生物技术国家重点实验室, 北京100083)
生物固氮是氮素循环的重要环节, 为整个生物圈提供了重要的氮素营养源。生物固氮反应是一种极其温和及零污染排放的生化反应, 它与人类发明的化学固氮相比有着无比优越性, 固氮微生物的深入研究、开发和利用, 有利于发展生态农业, 达到土地可持续利用的战略目标[1]。在生物固氮中, 大约40%的氮源由海洋固氮生物完成, 60%的氮源由陆生固氮生物完成。人们已对多个海域(阿拉伯海、红海、太平洋、中国南海、大西洋)进行了固氮微生物多样性的分析[2-4], 结果显示海洋中固氮微生物多集中于海洋沉积物中。目前文献报道的海洋固氮微生物包括三大类: 异养细菌类、光合细菌类以及蓝藻类[5-7]。2012年, Farnelid等[8]对海洋固氮微生物群落固氮酶基因库信息全面分析后提出: 海洋中占固氮微生物总数 42%的微生物群落属于非蓝藻细菌。这一结果表明在海洋生态系统中非蓝藻细菌发挥着不可低估的固氮作用。海洋中已报道的异养固氮细菌包括克氏杆菌属(Klebsiella)、固氮菌属(Azotobacter)、芽孢杆菌属(Bacillus)、梭菌属(Clostridium)、产气肠杆菌属(Enterobacter)、弧菌属(Vibrio)等属中的一些种[9]。
南海是仅次于阿拉伯海和珊瑚海的世界第三大陆缘海, 是海洋资源非常丰富的地区之一。迄今, 有关分离培养南海固氮菌的报道并不多见[10], 本研究采集南海底泥样品并采用煮沸淤泥样品结合无氮培养基富集培养的方法进行初筛, 进一步通过固氮酶分子标记及固氮酶活性检测进行复筛, 最终获得一株固氮菌。对所获菌株形态特征、生理生化指标及16S rDNA序列等结果进行分析确定其分类地位。这一研究为海洋自生固氮菌的多样性提供了新的证据。
1 材料和方法
1.1 固氮类芽孢杆菌的分离
1.1.1 海底淤泥样品的采集及处理
底 泥 样 品 采 自 南 海 东 经 110°51′227′′, 北 纬20°42′193′′; 水深 29 m 处(pH 8.30); 采集的样品封口于测序袋后并立即置于冰盒内(定期更换冰盒), 带回实验室后, 储存在4℃冰箱中保存备用。
1.1.2 培养基
无氮培养基: 用于分离、纯化固氮菌。2% (w/v)蔗糖; 1.2% (w/v) 磷酸氢二钾; 0.34% (w/v) 磷酸二氢钾; 0.05%(w/v) 硫酸镁; 0.001% (w/v) 氯化钠; 0.0015%(w/v) 氯化铁; 0.0005%(w/v) 钼酸钠; 1.2%~1.4%(w/v) 琼脂粉; pH 7.0。
限氮培养基: 用于测定固氮酶活性。1000 mL ddH2O 中溶解 26.3 g Na2HPO4•12H2O; 3.4 g KH2PO4;10 μg 生物素; 26 mg CaCl2•2H2O; 30 mg MgSO4;0.33 mg MnSO4•H2O; 36 mg 柠 檬 酸 铁 ; 7.6 mgNa2MoO4•2H2O; 10 μg 对氨基苯甲酸; 4 g 葡萄糖; 谷氨酸终浓度为2 mmol/L。
1.1.3 分离方法
称取适量淤泥样品以1/10比例加入液体无氮培养基中, 30℃富集培养1 h。系列梯度稀释后于沸水中煮沸 10 min, 以期获得能产芽孢的菌株(期间摇动3~5次), 每个稀释度各取 100μL涂布于无氮平板中,于30℃培养3 d。挑取单菌落多次划线纯化后保存备用。
1.2 菌株的鉴定
将菌株接种到LB培养基, 30℃培养过夜, 利用光学显微镜观察革兰氏染色结果; 利用电子显微观察细菌的大小、形态特征; 各项生理生化指标的测定参照《常见细菌系统鉴定手册》[11]和《微生物学实验》[12]进行。
1.3 nifH基因的扩增
采用菌落 PCR方法, 以固氮菌Paenibacillus sophoraeS27T(DSM23020T)为阳性对照(菌株由中国农业大学陈三凤教授提供), 检测固氮酶保守基因nifH。引物(PolF/PolR)参照参考文献[13]。具体序列如下: PolF 5′-3′TGCGAYCCSAARGCBGACTC; PolR 5′-3′ATSGCCATCATYTCRCCGGA, 扩增产物约 360 bp。扩增体系 20 μL, 组成如下: 10×PCR缓冲液 2 μL,dNTP 混合物(10 mmol/L)0.5 μL, 引物各 0.5 μL, Tap酶1~2 U, 菌落少许, 无菌水16.5μL。充分混匀。扩增程序: 95℃ 10 min; 95℃ 30 s; 55℃ 30 s; 72℃ 30 s。30个循环; 72℃ 10 min。用2%的琼脂糖凝胶电泳检测产物。将符合测序要求的PCR产物送北京博迈德公司测序, 序列相似性分析使用软件 DNAMAN完成; 基因比对通过NCBI数据库BLAST在线完成。
1.4 菌株16S rDNA的PCR扩增
引物: 采用 通 用引物 对 27F (5′-3′TACGGCTACCTTGTTACGACTT)以及 1492R(5′-3′AGA-GTT TGATCCTGGCTCAG)进行扩增, 扩增产物约1.5 kb。扩增体系为50μL, 反应条件如下: 95℃ 5 min, 94℃1 min, 50℃ 1 min, 72℃ 2 min, 30 个循环, 72℃ 10 min。PCR产物凝胶系统检测后送北京博迈德公司测序。序列相似性分析及基因比对方法同上。
1.5 固氮酶活性的检测
根据 Aranibar等[14]测定固氮菌酶活性的方法,将活化菌株接种到液体培养基LB中, 于30℃摇床振荡培养过夜, 用无菌去离子水清洗3遍后, 限氮培养基调整OD600=0.3左右, 分别取1 mL菌液加入灭菌的26 mL的厌氧管中, 反复抽充N2后, 用注射器注入厌氧管剩余体积的10%的乙炔, 30℃振荡培养, 每隔2 h, 从厌氧管中取100 μL气体, 采用气相色谱仪测定固氮酶活性(负对照为未接种的管, 固氮菌P.sophoraeS27T菌株为正对照)。固氮酶活[nmol/(mg·h)]的计算公式为:

1.6 序列比对及系统发育树的构建
nifH与 16S rDNA基因序到 GenBank数据库,BLASTN在线比对同源序列, 从 GenBank数据库下载与提交序列相似性较高的基因序列。使用DNAMAN、MEGA5软件中ClustalX功能比对序列,邻接法构建系统发育树, bootstrap值设为1000。
2 结果
2.1 类芽孢固氮菌的分离和筛选
2.1.1 固氮菌的分离
经过富集培养、从南海底泥中分离到30株能在无氮培养基上生长的菌株, 进一步分离纯化后, 保存备用。对30株菌株进行固氮菌保守基因nifH的扩增, 进一步进行复筛。被测试菌株菌落边缘整齐, 圆形扁平, 呈乳白色, 表面比较光滑、湿润。
2.1.2 固氮酶基因nifH的扩增及系统发育分析
nifH是固氮微生物的“分子标记”。目前, 固氮微生物分子水平的检验主要集中在nifH。用nifH兼并引物对挑选得到的30株菌进行了PCR扩增, 结果只从7株菌株中扩增到目的基因nifH, 7株菌株编号分别为: NH-1、NH-3、NH-9、NH-15、NH-21、NH-24及NH-30(图1)。将PCR产物进行回收测序, 对测序结果进行分析。通过DNAMAN软件分析, 7株菌株之间nifH同源性达到 99%以上, 因此, 后续试验选择其中一株NH-1作为代表菌株。根据其DNA序列推测的氨基酸序列, GenBank在线比对分析表明,NH-1的nifH与其他已知固氮菌的nifH具有较高的同源性(同源性 70%以上)。从 NCBI数据 GenBank中下载相关菌株的nifH序列构建系统发育树, 结果如图2所示。以上结果表明, 该菌基因组中确实存在固氮保守基因nifH。nifH基因序列已上传至 NCBI数据库中(GenBank登录号KJ627375)。
2.1.3 固氮酶活性测定
根据 Aranibar等[14]测定固氮菌酶活性的方法,对该菌株固氮酶活性进行了测定, 结果如下: 阳性菌株 S27T的固氮酶活为 283.3±33.1[nmol C2H4/(mg蛋白/h)], 该菌株固氮酶活为 897.4±11.8 [nmol C2H4/ (mg蛋白/h)], 最终确定该菌株具有固氮能力。
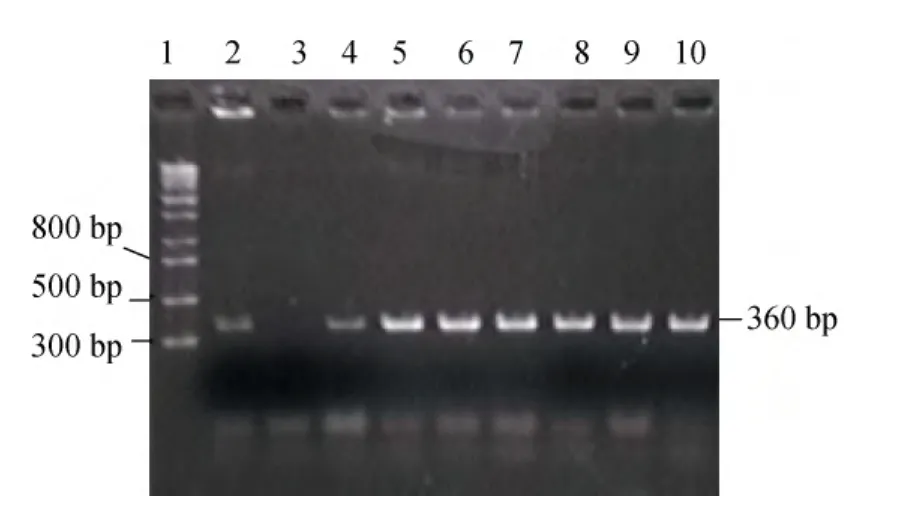
图1 nifH PCR扩增图Fig.1 Amplification of the nifH gene of the isolates
2.2 菌株NH-1的表型特征
菌株NH-1为杆状, 大小(0.5~0.7) μm×(1.5~2.5) μm,革兰氏染色为阳性, 能运动, 端生鞭毛(图3), 产芽孢(图4)。其生理生化特性见表1所示。
2.3 菌株NH-1 16S rDNA序列PCR扩增及系统发育分析
菌株NH-1 16S rDNA序列的测定结果已上传至NCBI数据库中(GenBank登录号KJ627376)。将序列导入 NCBI数据库, 利用 BLAST在线比对, 结果显示菌株 NH-1与固氮类芽孢杆菌属模式菌株Paenibacillus stellifer的 16S rDNA 同源关系最近(达到99%)。从GenBank中选出与NH-1同源性较高的10株菌株的16S rDNA序列构建系统发育进化树(图5)。结果显示, NH-1在进化树中与Paenibacillus stellifer的亲缘关系最近。根据固氮菌NH-1的形态特征、生理生化特性、固氮酶活性及16S rDNA序列分析, 将筛选到的产芽孢固氮菌定名为Paenibacillussp.NH-1。

图2 NH-1 nifH系统发育树Fig.2 Phylogenetic tree of nifH gene of NH-1 and other diazotrophic bacteria
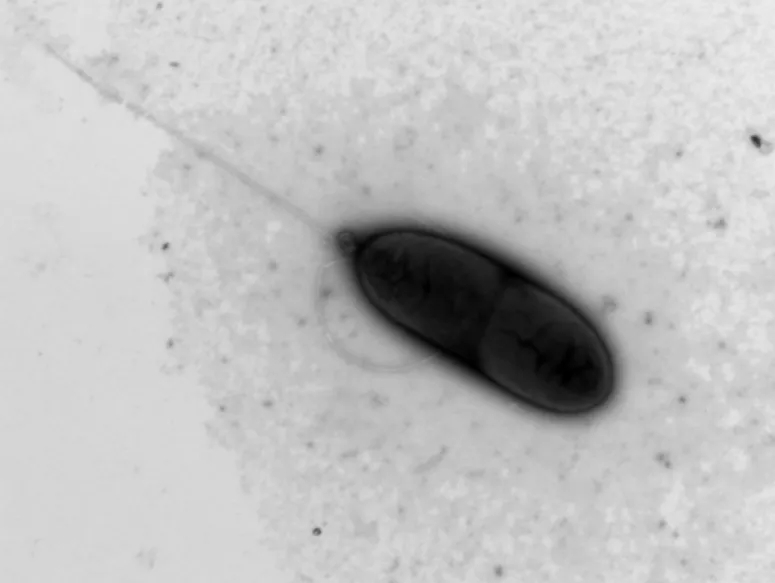
图3 NH-1菌株鞭毛透射电镜图Fig.3 Transmission electron microscopic image showing the flagellum of strain NH-1

图4 菌株NH-1芽孢扫描电镜图Fig.4 Scanning electron microphotograph of the isolate NH-1

表1 固氮类芽孢杆菌Paenibacillus sp.NH-1生理生化特征Tab.1 Morphological and biochemical characteristics of the isolate NH-1
3 讨论及结论
尽管人类已从多种环境中分离得到了固氮菌,但是到目前为止, 仍然有许多的固氮菌在实验条件下无法成功培养。所有固氮生物的铁蛋白均由nifH基因编码, 因此, 研究人员往往通过对nifH的分析来了解环境中固氮微生物的生物多样性及进化地位。目前, 海洋环境中异养固氮细菌的多样性研究大多集中在固氮酶保守基因nifH水平的检测上。1993年, 在对芽孢杆菌属51个种的16S rDNA序列比对后, 类芽孢杆菌(Paenibacillus)被单独分离出来形成一个新属[15]。该属细菌是一类 GC含量较低、能产芽孢, 兼性厌氧生长的革兰氏阳性细菌。到2013年统计为止, 该属已经包括100多个种和两个亚种, 其中19个种被确定为固氮菌[15-18]。固氮类芽孢菌纯培养物的报道多见于陆地土壤, 海洋中的报道并不多见。Steward等[19]利用固氮酶基因nifH检测技术对加利福尼亚高盐度莫诺湖水进行固氮菌多样性的分析研究, 结果显示该环境中存在有固氮类芽孢杆菌;Choi等[20]从韩国东海海底淤泥中分离到一株能够降解木聚糖的菌株Paenibacillus donghaensissp.Nov.,在DNA水平检测到固氮酶基因nifH后, 初步确定该菌株具有固氮作用。此外, Dang等[21]利用固氮酶基因nifH扩增技术对中国南海北部固氮细菌进行研究,结果表明该海洋同样分布有固氮类芽孢杆菌。由于这些固氮类芽孢菌的确认都是基于基因水平, 并未检测到它们的具体固氮酶活性, 因此, 无法比较这些菌株与本实验中分离得到的纯培养物 NH-1之间固氮酶活性的差异。本实验利用纯培养技术分离得到 NH-1, 通过固氮酶活性检测、形态观察、生理生化测定、系统发育分析等, 最终确定该菌株为固氮类芽孢菌。这一实验结果与Dang等[21]研究结果保持一致, 进一步丰富了海洋中固氮菌的多样性。
随着海洋固氮生物多样性的深入研究, 人们发现海洋自生固氮菌的多样性远远超出人类之前的认识。固氮菌的开发和研究对充分发挥生物固氮在农业中的作用具有十分重要的理论意义和实践价值[22-24]。自生固氮微生物因其独立生活条件下就能实现固氮而成为研究生物菌肥的热点。固氮类芽孢菌在微生物菌剂方面有着巨大的开发潜能和利用价值。目前已知的固氮类芽孢杆菌种类较少, 因此不同环境中固氮类芽孢杆菌的筛选, 对科学研究和农业生产有着十分重要的意义。
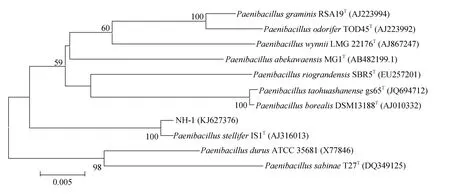
图5 NH-1 16S rRNA系统发育树Fig.5 Phylogenetic tree of 16S rRNA gene of NH-1 and those of other bacteria
致谢:感谢中国农业大学国家重点实验室李颖教授为本实验提供的南海海底淤泥样品; 感谢中国农业大学国家重点实验室陈三凤教授对本实验酶活性测定提供的帮助。
[1]周德庆.微生物学教程[M].北京: 高等教育出版社,2011.
[2]Church M J, Bjorkman K M, Karl D M, et al.Regional distributions of nitrogen-fixing bacteria in the Pacific Ocean[J].Limnology and Oceanography, 2008, 53(1) :63-77.
[3]Ren H, Sigman D M, Meckler A N, et al.Foraminiferal isotope evidence of reduced nitrogen fixation in the ice age Atlantic Ocean [J].Science, 2009, 323: 244-248.
[4]Karl D M, Church M J, Dore J E, et al.Predictable and efficient carbon sequestration in the North Pacific Ocean supported by symbiotic nitrogen fixation[J].Proceedings of the National Academy of Sciences,2012, 109(6): 1842-1849.
[5]Zehr J P.Nitrogen fixation by marine cyanobacteria[J].Trends in Microbiology, 2011, 19(4): 162-173.
[6]Berman-Frank I, Lundgren P, Falkowski P.Nitrogen fixation and photosynthetic oxygen evolution in cyanobacteria[J].Research in Microbiology, 2003,154(3): 157-164.
[7]Sohm J A, Webb E A, Capone D G.Emerging patterns of marine nitrogen fixation [J].Nature Reviews Microbiology, 2011, 9(7): 499-508.
[8]Farnelid H, Andersson A F, Bertilsson S, et al.Nitrogenase gene amplicons from global marine surface waters are dominated by genes of non-cyanobacteria[J].Plos One, 2011, 6(4): e19223.
[9]王晓姗.海洋固氮细菌的研究进展[J].科技资讯,2009, 6: 4-5.
[10]Moisander P H, Beinart R A, Voss M, et al.Diversity and abundance of diazotrophic microorganisms in the South China Sea during intermonsoon[J].The International Journal of Systematic and Evolutionary Microbiology, 2008, 2(9): 954-967.
[11]东秀珠, 蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社, 2001.
[12]沈萍, 范秀容, 李广武.微生物学实验(第三版)[M].吉林: 高等教育出版社, 1999.
[13]Poly F, Monrozier L J, Bally R.Improvement in the RFLP procedure for studying the diversity ofnifHgenes in communities of nitrogen fixers in soil [J].Research in Microbiology, 2001, 152(1): 95-103.
[14]Aranibar J, Anderson I, Ringrose S, et al.Importance of nitrogen fixation in soil crusts of southern African arid ecosystems: acetylene reduction and stable isotope studies[J].J Arid Environ, 2003, 54: 345-358.
[15]Heyndrickx M, Vandemeulebroecke K, Scheldeman P,et al.A polyphasic reassessment of the genusPaenibacillus, reclassification ofBacillus lautusasPaenibacillus lautuscomb.nov.and ofBacillus peoriaeasPaenibacillus peoriaecomb.nov., and emended descriptions ofP.lautusand ofP.peoriae[J].International Journal of Systematic Bacteriology, 1996,46(4): 988-1003.
[16]Jin H J, Lv J, Chen S F.Paenibacillus sophoraesp.nov., a nitrogen-fixing species isolated from the rhizosphere ofSophora japonica[J].International Journal of Systematic and Evolutionary Microbiology,2011, 61(4): 767-771.
[17]Jin H J, Zhou Y G, Liu H C, et al.Paenibacillus jilunliisp.nov., a nitrogen-fixing species isolated from the rhizosphere ofBegonia semperflorens[J].International Journal of Systematic and Evolutionary Microbiology,2011, 61(6): 1350-1355.
[18]Xie J B, Zhang L H, Zhou Y G, et al.Paenibacillus taohuashanensesp.nov., a nitrogen-fixing species isolated from rhizosphere soil of the root ofCaragana kansuensispojark [J].Antonie van Leeuwenhoek, 2012,102: 735-741.
[19]Steward G.Vertical distribution of nitrogen-fixing phylotypes in a meromictic, hypersaline lake[J].Microbial Ecology, 2004, 47(1): 30-40.
[20]Choi J H, Moon D S, Kim H J, et al.Paenibacillus donghaensissp.nov., a xylan-degrading and nitrogen-fixing bacterium isolated from East Sea sediment [J].Journal of Microbiology and Biotechnology, 2008, 18(2): 189-193.
[21]Dang H, Yang J, Li J, et al.Environment-dependent distribution of the sedimentnifH-harboring microbiota in the northern South China Sea [J].Applied and Environmental Microbiology, 2013, 79(1): 121-132.
[22]沈世华, 荆玉祥.中国生物固氮研究现状和展望[J].科学通报, 2003, 48(6): 535-540.
[23]张燕春.高效固氮芽孢杆菌的选育及其效应研究[D].福建: 福建农林大学, 2008.
[24]丁延芹, 王建平, 陈三凤, 等.几株固氮芽孢杆菌的分离与鉴定[J].农业生物技术学报, 2004, 12(6):690-697.
——固氮微生物

