FABP4基因在阿勒泰羊尾脂沉积与代谢模型中的表达变化规律
许瑞霞,高磊,赵伟利,张伟,宋广超,甘尚权,石国庆
基因在阿勒泰羊尾脂沉积与代谢模型中的表达变化规律
许瑞霞1,2,高磊2,赵伟利1,2,张伟1,2,宋广超1,2,甘尚权2,石国庆1,2
1. 石河子大学动物科技学院,石河子 832000;2. 新疆农垦科学院畜牧兽医研究所,石河子 832000
()参与细胞内脂肪酸的转运和脂肪酸代谢,是当前研究动物脂肪沉积与代谢的热门候选基因。为了研究基因在绵羊尾脂沉积与代谢中的作用,文章采用生物信息学方法分析了FABP4氨基酸序列在各物种中的保守性;利用半定量RT-PCR方法检测了该基因在阿勒泰羊主要组织中的表达;采用饥饿法成功建立了模拟阿勒泰羊尾脂沉积与代谢的动物模型,利用qPCR和iTRAQ(isobaric tags for relative and absolute quantitation)技术同时验证基因mRNA和蛋白在阿勒泰羊非饥饿组与饥饿组尾脂中的表达变化。序列分析结果表明,绵羊FABP4氨基酸序列在物种间高度保守,提示基因可能由于其重要的生物功能而在进化中表现出其物种的保守性。组织表达谱结果显示,mRNA在阿勒泰羊肠脂与尾脂中均高丰度表达,暗示基因可能在脂肪中行驶着重要的生理生化功能。qPCR与iTRAQ结果显示,mRNA与蛋白在两种极端条件下尾脂中的表达差异均不显著(>0.05),表明基因可能不是绵羊尾脂沉积与代谢两种极端差异表型的决定基因。以上研究结果为进一步研究基因在绵羊尾脂中的生物功能奠定了基础。
FABP4;阿勒泰羊;尾脂沉积与代谢;表达变化
阿勒泰羊臀尾合一,充斥着大量脂肪组织,其肥大的脂臀性状是在新疆生态恶劣的地理环境下长期进化与选择的结果。夏秋牧草丰茂时期,脂肪组织能够在阿勒泰羊尾臀部大量沉积,臀尾表现出极度充盈的表型。冬春牧草匮乏时,充盈的尾脂大量分解以维持机体新陈代谢能量之所需,其臀尾表现出“瘪瘦”表型。阿勒泰羊属于典型的脂肉型绵羊品种,其尾脂约占胴体重的1/4,这种高产脂能力不仅降低了其经济价值,其肉质也不利于健康饮食的需要。鉴于此,新疆农牧民大量引进外来低脂品种对其进行杂交改良,这种无组织性的杂交与串配使得阿勒泰羊基因库受到严峻挑战,其品种面临灭绝的险境。因此,发掘影响阿勒泰羊尾脂沉积与代谢的关键基因,继而培育出低脂阿勒泰羊新品系,无论对于阿勒泰羊保种还是提高其经济价值均具有重要的研究意义。
脂肪酸结合蛋白4(Fatty acid binding protein 4, FABP4)亦称aP2或A-FABP,属于脂肪酸结合蛋白超家族,编码132个氨基酸,相对分子质量约14.6 kDa。它在脂肪组织中高表达,是成熟脂肪细胞中主要的胞质蛋白,约占细胞总蛋白的6%[1~3]。研究表明FABP4能够可逆性地结合饱和及不饱和长链脂肪酸,促进脂肪酸的代谢和转运,调控脂类生成及降解,在介导胞内脂肪酸转运和能量代谢过程中起重要作用[4, 5]。猪和鸡的基因已被证实是肌内脂肪含量(IMF)的候选基因[6]。迄今为止,仍未见有关基因在绵羊尾脂沉积与代谢中的作用的文献报道。基于此,本文拟以阿勒泰羊为研究对象,采用饥饿法建立模拟阿勒泰羊在自然生理状态下尾脂沉积与代谢两种极端状态的动物模型,研究基因mRNA与蛋白在尾脂沉积与代谢两种状态中的表达变化规律,为进一步验证基因对阿勒泰羊尾脂沉积与代谢的影响积累基础数据。
1 材料和方法
1.1 材料
挑选年龄相近(约2~3岁)、体重接近、臀型一致、膘情良好的阿勒泰羊7只(购于阿勒泰羊种群核心区阿勒泰富蕴县)。隔离半月后饲养于新疆农垦科学院转基因羊场。非饥饿组:选取3只成年阿勒泰母羊,每天分早、中、晚3次饲喂草料,饮水自由,每日傍晚补饲玉米200 g,连续饲喂四周后手术取3只母羊左臀脂肪组织置于液氮中保存;饥饿组:上述3只阿勒泰羊尾脂组织样品采集完毕后缝合左侧创口,抗生素治疗1周,后连续恢复3周,期间自由采食与饮水。待其体况恢复至正常生理状态后,开始以周为单位进行限饲(每周饲喂量减半,自由饮水),3周后完全停止饲草料,停喂期间饮水照常,停喂4周后迅速采集阿勒泰羊右臀脂肪组织并置于液氮中保存备用。同时宰杀剩余4只处于正常饲喂条件下阿勒泰羊,迅速采集其心脏、肝脏、脾脏、肺脏、肾脏、肌肉、肠系膜脂肪和尾脂等组织样品,置于液氮中保存,用于基因组织表达谱研究试验。
1.2 方法
1.2.1 引物设计
以GenBank中公布的绵羊()基因(登录号:NM_001114667.1)与绵羊持家基因(登录号:NM_001190390)mRNA序列为参考序列,采用Oligo6.0软件设计相应的PCR扩增引物,引物由上海生工生物工程有限公司合成。引物序列及拟扩增片段大小见表1。
1.2.2 总RNA提取及cDNA第一链的合成
参照TRIzol®Reagent(Invitrogen)说明书提取阿勒泰羊心脏、肝脏、脾脏、肺脏、肾脏、肌肉、肠系膜脂肪、尾脂及饥饿与非饥饿试验组阿勒泰羊尾脂等组织总RNA。0.7%琼脂糖凝胶电泳检测其完整性,利用核酸蛋白分析仪测定RNA的纯度和浓度。cDNA第一链采用能够除去基因组DNA污染的PrimeScriptTMRT reagent Kit with gDNA Eraser (Perfect Real Time)(TaKaRa)反转录试剂盒进行合成。
1.2.3 序列生物信息学分析
在NCBI Nucleotide数据库中分别检索绵羊、牛、牦牛、小家鼠、猴、人等物种基因的核酸序列及蛋白序列,利用DNAMAN5.0软件进行物种间同源性分析;利用Clustalx和MEGA软件构建FABP4蛋白序列系统进化树。
1.2.4基因组织表达谱分析
为使RT-PCR结果可以反映基因在各组织中的真实表达量,本文首先确定了高丰度表达的内参基因的最佳扩增循环数。PCR反应体系为10×Reaction Buffer 2.5 µL,dNTP Mixture 2 µL,上下游引物(10 mmol/L)各0.5 µL,DNA 聚合酶0.5 µL,模板cDNA 1 µL,ddH2O 18 µL。分别设定循环数20、22、24、26、27、28、29和30,根据PCR扩增琼脂糖检测结果选择合适的循环数。
以正常饲喂条件下阿勒泰羊的心脏、肝脏、脾脏、肺脏、肾脏、肌肉、肠系膜脂肪和尾脂cDNA为扩增模板进行PCR扩增,反应体系同上。PCR程序如下:94℃预变性 1 min;94℃变性 30 s;60℃复性30 s;72℃延伸30 s,共29个循环;72℃延伸10 min。半定量RT-PCR产物经1.5%琼脂糖凝胶电泳,用溴化乙锭染色后,在紫外光下用GelDoc XR凝胶成像系统(Bio-Rad,美国)成像。持家基因的RT-PCR步骤与程序同基因。
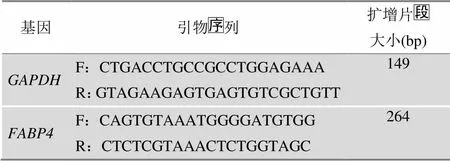
表1 引物序列和PCR产物长度
1.2.5 荧光实时定量PCR
标准曲线的制备:以阿勒泰羊饥饿与非饥饿试验组阿勒泰羊尾脂cDNA为模板进行PCR扩增,扩增产物经琼脂糖凝胶回收试剂盒纯化回收后,与pMD18-T载体连接、转化大肠杆菌感受态细胞(DH5α),提取经PCR和测序鉴定的阳性克隆质粒,将质粒稀释成20 ng/µL,并以此浓度为始浓度依次稀释10倍,共稀释9个梯度,以最后6个梯度绘制定量标准曲线。
荧光实时定量PCR检测:利用荧光定量PCR仪Lightcycler480(Roche),采用SYBR GreenⅠ染料法分别检测和基因在饥饿和非饥饿阿勒泰羊尾脂中的mRNA表达水平,饥饿组与非饥饿组各3个生物学重复,并设置相应的阴性对照(扩增模板为双蒸水)。荧光定量PCR为25 µL反应体系:2×QuantiFast SYBR Green PCR Master Mix(QIAGEN) 12.5 µL,上下游引物各0.5 µL,Rnase-free water 10.5 µL,cDNA模板1 µL。PCR反应程序:95℃热激活 5 min,95℃变性 1 s,60℃复性/延伸 30 s,45个循环。同时记录PCR产物的熔解曲线,检验产物的特异性。
1.2.6 饥饿与非饥饿组尾脂蛋白组绝对定量分析
iTRAQ标记:非饥饿组和饥饿组尾脂蛋白样品各3份,然后取等量各样品肽段进行混合并以此作为内参对照。每份样品各取约60 μg,通过iTRAQ试剂标记后混合进行定量测试。本研究共采用7种同量异位素编码的标签对以上样本进行iTRAQ标记,标记后进行串联质谱分析,同时比较7种不同样品中蛋白质的绝对含量。在质谱图中,任何一种iTRAQ标签标记的不同样本中的同一蛋白质表现为相同的质荷比。而在串联质谱中,信号离子表现为不同质荷比的峰,根据波峰的高度和面积,可以得到较为准确的蛋白质定量信息。
毛细管高效液相色谱分离与质谱分析:每份样品采用纳升流速HPLC液相系统Easy nLC进行分离。样品经SCX分级并脱盐后用Q-Exactive质谱仪(Thermo Finnigan)进行质谱分析。分析时长:120 min,检测方式:正离子,母离子扫描范围:300~1800 m/z,一级质谱分辨率:70 000 at m/z 200,AGC target:3e6,一级Maximum IT:10 ms,Number of scan ranges:1,Dynamic exclusion:40.0 s。多肽和多肽碎片的质量电荷比按照下列方法采集:每次全扫描后采集10个碎片图谱(MS2 scan),MS2 Activation Type: HCD,Isolation window:2 m/z,二级质谱分辨率:17 500 at m/z200,Microscans:1,二级Maximum IT:60 ms,Normalized collision energy:30eV,Underfill ratio:0.1 %。
1.2.7 数据分析
荧光定量数据分析:以持家基因为参照,将阿勒泰羊基因在非饥饿试验尾脂中的表达量定义为 1,荧光实时定量PCR结果利用2-ΔΔCt法[7]计算基因在饥饿与非饥饿试验阿勒泰羊尾脂中的相对表达量,并利用SPSS(18.0)数据分析软件对定量数据进行单因素方差分析。阿勒泰羊组织表达谱相对灰度值定量:采用quantity one软件分别检测凝胶上与带型的灰度值,以灰度值/灰度值为相对灰度值,将所有组织相对灰度值除以心脏组织的相对灰度值所得到的数据为相对灰度值定量数据,定量数据以心脏组织的相对灰度值基调(心脏relative gray value=1)进行比较。iTRAQ数据分析:采用Mascot2.2软件进行查库鉴定分析蛋白质谱;采用Proteome Discoverer1.4(thermo)软件对肽段报告离子峰强度值进行定量分析。定量时以各样品等量肽段混合物Mix标记标签的为内参,根据不同iTRAQ标签的丰度对来源于不同组的各肽段进行定量,表现为各组蛋白质与内参的比值。
2 结果与分析
2.1 总RNA提取及基因的克隆测序
0.7%的琼脂糖凝胶电泳检测结果显示,所提取不同组织完整性(28S、18S和5S)较好,无DNA和蛋白质污染,所有样品经核酸蛋白分析仪检测260/280均在1.8~2.0之间,说明提取的总RNA纯度较高,可以用于后续试验。
以阿勒泰羊尾脂组织cDNA为模板,利用和基因特异性引物进行PCR扩增,获得了一条与预期大小相符的片段,经回收测序,并与GenBank中公布的绵羊基因序列进行比对,确定为绵羊基因。
2.2 绵羊FABP4同源性及系统发育分析
利用DNAMAN软件对绵羊、山羊、牛、牦牛、小家鼠、猕猴、原鸡、人、野猪、马鹿等物种的核苷酸序列及蛋白质序列进行同源性比较。结果表明,绵羊的核苷酸序列与山羊、牛、牦牛、小家鼠、原鸡、猕猴、人、野猪、马鹿的同源性分别为:99.25%、95.99%、96.24%、82.71%、72.93%、88.47%、89.47%、90.23%、96.49%。蛋白质序列的同源性与mRNA序列同源性大致相同,分别为:99.24%、93.18%、95.45%、86.36%、72.73%、86.36%、84.09%、96.21%。运用Mega5.0软件,利用N-J法建立绵羊与其他物种FABP4蛋白系统进化树,结果显示,绵羊FABP4与山羊、牛等反刍动物FABP4的遗传距离最近,与禽类(原鸡)的遗传距离最远(图1)。

图1 各物种FABP4蛋白序列系统进化树
2.3 FABP4基因组织表达谱研究
RT-PCR结果显示,mRNA在肠系膜脂肪和尾部脂肪组织中的表达丰度最高,两者表达均高于持家基因的表达,且尾部脂肪组织中的表达量高于肠系膜脂肪(图2)。此外,除了肠系膜脂肪与尾脂,肺脏和肌肉中也有微量mRNA表达(图3),这可能是由于肺脏与肌肉组织含有少量脂肪所致,但其表达量相对于肠系膜脂肪与尾脂可以忽略不计。

图2 FABP4在阿勒泰羊组织中的表达谱
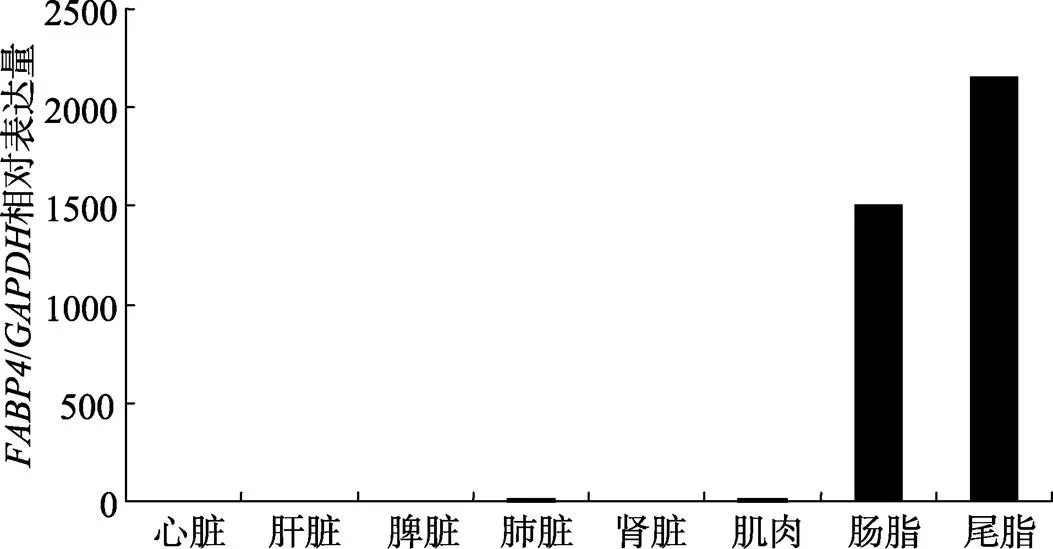
图3 阿勒泰羊组织表达谱相对灰度值定量
2.4 FABP4基因的荧光实时定量检测
阿勒泰羊基因在饥饿与非饥饿试验尾脂中的表达规律显示,3个非饥饿组生物学重复尾脂基因值均略低于饥饿组。以为持家基因,以2-ΔΔCt法计算基因相对表达量,从图4可以看出,阿勒泰羊基因在非饥饿组尾脂中的表达量略高于饥饿组尾脂,但二者差异不显著(>0.05)。
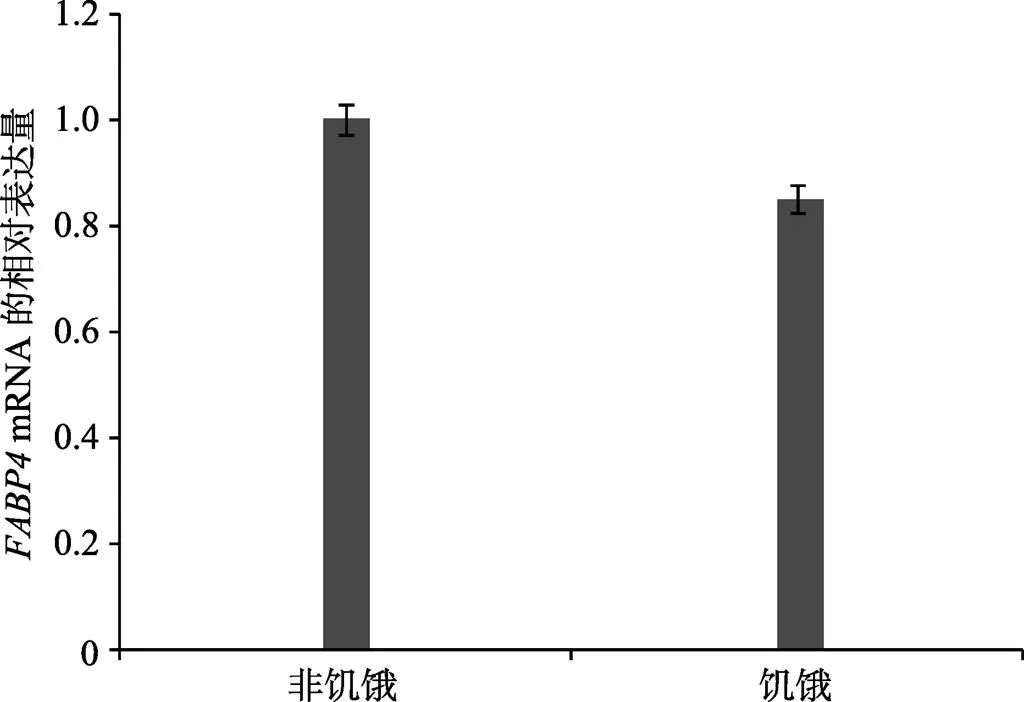
图4 FABP4基因在阿勒泰羊饥饿组与非饥饿组尾脂中的表达水平比较
2.5 iTRAQ质谱数据分析
iTRAQ技术结合液相色谱质谱技术构建阿勒泰羊饥饿组与非饥饿组尾脂中的蛋白差异表达谱系。经质谱分析及Mascot查库,结果合并以后以肽段FDR≤0.01筛选过滤,最终共鉴定到2094个唯一肽段和1100个蛋白质,图5A为质谱鉴定总离子流图。将其中一高丰度表达的肽段进行二级质谱鉴定,二级质谱图如图5B所示,经比对蛋白质二级质谱数据库,鉴定该高丰度表达的蛋白肽段为绵羊FABP4蛋白。
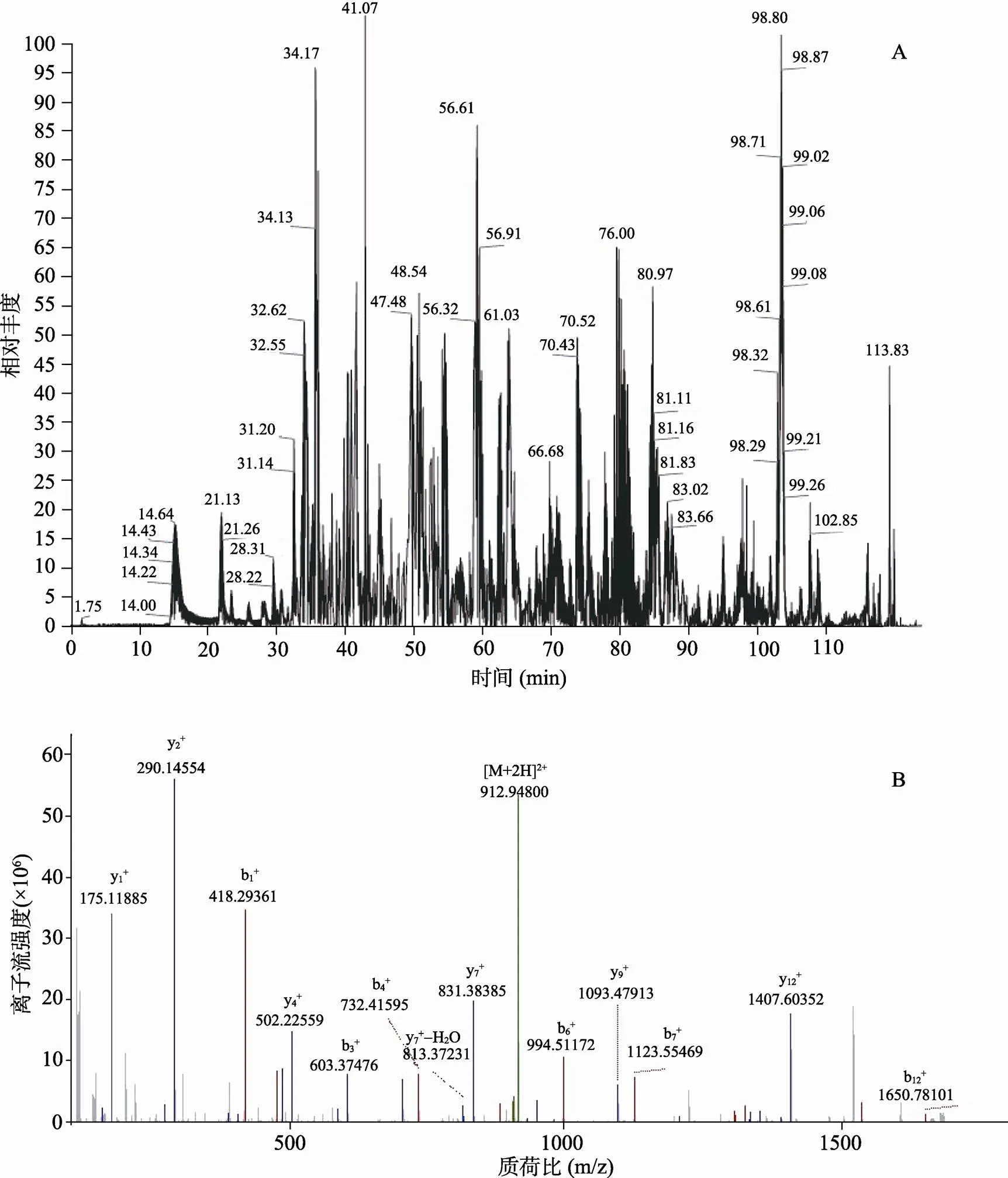
图5 质谱鉴定总离子流图(A)与FABP4的二级质谱图(B)
对FABP4蛋白肽段标记数据进行显著性分析并计算其显著性指数(-Value),结果显示FABP4蛋白在阿勒泰羊非饥饿组尾脂中的表达量略高于饥饿组,但二者差异不显著(>0.05)此结果与mRNA定量结果类似,说明基因mRNA与蛋白质表达趋势相统一(图6)。

图6 阿勒泰羊非饥饿组与饥饿组尾脂中的FABP4蛋白水平比较
3 讨 论
FABP4是低分子量、可溶性胞内脂质转运载体,在脂肪细胞中高度表达,可受PPARγ激动剂(如罗格列酮)、胰岛素和脂肪酸调节[5, 8~10]。研究表明在脂肪细胞分化过程中,FABP4的表达及活性显著增强[11];在甘油三酯形成及脂解过程中,FABP4能够储存或释放大量脂肪酸,参与调控甘油三酯的形成和脂解的生化循环[12]。因此,已成为调控动物脂肪代谢的重要候选基因。
本文采用饥饿法模拟了阿勒泰羊在自然生理状态下尾脂沉积与代谢两种极端状态,成功建立了阿勒泰羊饥饿与非饥饿状态下尾脂沉积与代谢的动物模型,并在此基础上分析了基因在阿勒泰羊饥饿组与非饥饿组尾脂中的表达变化。mRNA与蛋白质在非饥饿组尾脂中的表达量虽略高于饥饿组,但二组间差异并不显著(>0.05),提示基因可能不是造成阿勒泰羊尾脂沉积与代谢两种尾脂表型的关键基因。我们推测,基因参与脂肪沉积与代谢过程,但不是绵羊尾脂沉积与代谢过程中脂肪代谢的关键限速酶。Hotamisligil等[13]、Coe等[14]和Shaughnessy等[15]发现基因敲除小鼠正是由于受基因表达的补偿效应,脂肪发育和新陈代谢才得以正常。Hertzel等[12]发现过表达转基因小鼠基因表达上升10倍,其体内基因表达却下降了2倍,转基因小鼠脂解相关蛋白的表达(和)及脂解水平均呈显著的上升趋势,但其体重、血清FFA和脂肪垫质量与野生型相比均无显著性差异。这一研究结果暗示基因可能在脂解发挥作用,但并不是脂解所必需的蛋白因子。与Hertzel等[12]的发现相反,Coe等[14]、Uysal等[16]与Scheja等[17]等研究发现基因敲除小鼠的基础脂解和异丙肾上腺素诱导的脂解作用均显著降低。基因缺失小鼠的脂解作用显著降低,从侧面提示基因可能参与脂解这一生化过程。从以上数据我们分析,基因的缺失或表达下降可造成脂解水平的上升或下降,关键看其他的家族成员(如)是否能够补偿这一由于缺失或表达下降带来的影响。
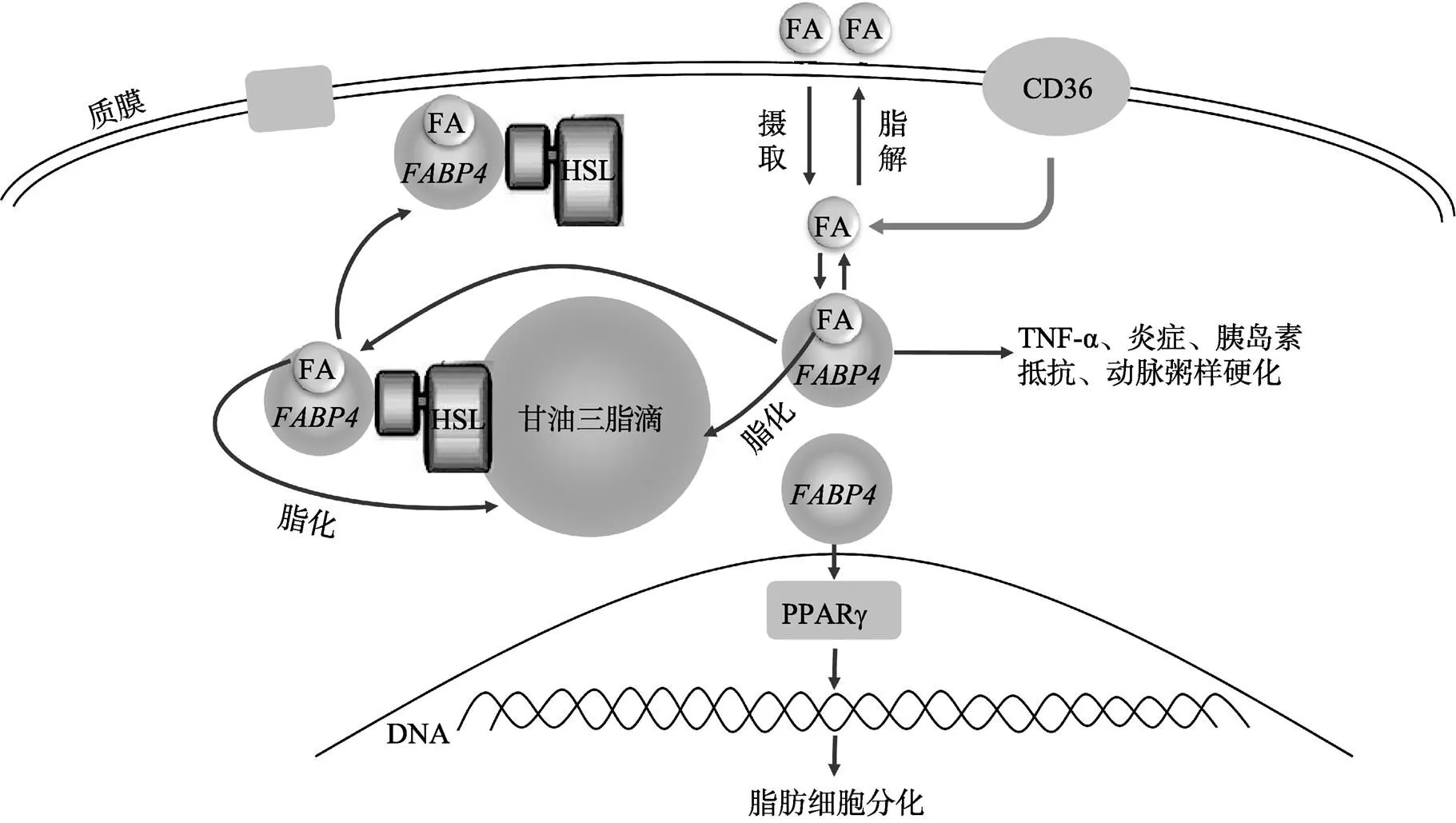
图7 FABP4基因在脂质代谢中的调控图
Baar等[18]发现基因缺失还可显著降低脂肪组织中非酯化脂肪酸的释放。与此相反,Smith等[19]发现基因敲除小鼠脂肪组织中游离的非酯化脂肪酸增多,这主要是由于FABP4结合一个脂肪酸后又与脂滴表面磷酸化活化的激素敏感脂肪酶(Hormone sensitive lipase, HSL)相互作用并形成复合物,最后共同将脂肪酸从脂滴转运到质膜,进而有利于脂肪酸的释放,但同时释放的脂肪酸又反馈抑制脂肪组织脂解水平。这一发现与Baar等研究结果有效统一,提示基因在酯化中扮演重要作用。本研究发现mRNA与蛋白在非饥饿组阿勒泰羊尾脂中表达均要高于饥饿组,与FABP4在酯化过程中的作用的推理相契合。
除了在脂解与酯化等方面影响脂质代谢作用外,基因还可以通过PPARγ途径促进脂肪细胞的分化[20]。Fu等[21]还发现基因的启动子区存在(PPAR应答元件)PPREs,PPARγ激动剂在激活PPARγ的同时,还可以促进基因的表达[22],形成FABP4与PPARγ之间的正反馈环[23]。CCAAT增强子结合蛋白也能够与基因的启动子相互作用并提高基因的表达[24]。
此外,基因还是全身性胰岛素敏感和糖代谢的重要调控因子[1]。当将膳食肥胖[13]和遗传性肥胖[16]鼠基因敲除,肥胖鼠不表现高胰岛素血症、高血糖和胰岛素抵抗。大量研究已证实血清FABP4水平与肥胖症、高胰岛素血症、胰岛素抵抗和代谢综合征呈正相关[2, 25, 26],Tuncman等[27]发现人类基因的启动子处一处突变会导致脂肪组织基因mRNA表达减少,从而降低患Ⅱ型糖尿病和心血管疾病的风险。在巨噬细胞[28, 29]和内皮细胞[30]中也有一定的生理功能,巨噬细胞中的可以被氧化性低密度脂蛋白[31]和toll样受体激动剂[32]所诱导,也能被降胆固醇抑制素药物抑制[33],从而调节炎症细胞因子的产生和胆固醇酯的积累[34]。
基于基因以上4个方面的主要功能,我们绘制一幅较为全面的基因生理生化作用的调控图(图7),从这张图中我们可以看出在哺乳动物的脂肪细胞分化、脂质代谢、葡萄糖稳态、胰岛素敏感以及炎症反应等诸多生理过程中发挥着重要作用。目前,基因在阿勒泰羊尾脂沉积与代谢中的作用机制仍不明晰,本研究成功建立了阿勒泰羊饥饿与非饥饿状态下尾脂沉积与代谢的模型,分析了基因mRNA和蛋白在阿勒泰羊饥饿与非饥饿组尾脂中的表达变化,研究结果为进一步研究基因在绵羊尾脂沉积与代谢中的作用机制提供依据。
[1] Makowski L, Hotamisligil GS. Fatty acid binding proteins-the evolutionary crossroads of inflammatory and metabolic responses., 2004, 134(9): 2464S–2468S.
[2] Xu AM, Wang Y, Xu JY, Stejskal D, Tam S, Zhang JL, Wat NMS, Wong WK, Lam KSL. Adipocyte fatty acid-binding protein is a plasma biomarker closely associated with obesity and metabolic syndrome., 2006, 52(3): 405–413.
[3] Baxa CA, Sha RS, Buelt MK, Smith AJ, Matarese V, Chinander LL, Boundy KL, Bernlohr DA. Human adipocyte lipid-binding protein: purification of the protein and cloning of its complementary DNA., 1989, 28(22): 8683–8690.
[4] Coe NR, Bernlohr DA. Physiological properties and functions of intracellular fatty acid-binding proteins., 1998, 1391(3): 287–306.
[5] Hertzel AV, Bernlohr DA. The mammalian fatty acid-binding protein multigene family: molecular and genetic insights into function., 2000, 11(5): 175–180.
[6] 张军, 董飚, 张红, 储冬生, 徐国庆, 龚道清, 段修军, 赵旭庭, 顾志良. 鸭A-FABP基因的克隆及其组织表达. 畜牧兽医学报, 2008, 39(6): 819–822.
[7] Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Δ ΔCT method., 2001, 25(4): 402–408.
[8] Hunt CR, Ro JH, Dobson DE, Min HY, Spiegelman BM. Adipocyte P2 gene: developmental expression and homology of 5'-flanking sequences among fat cell-specific genes., 1986, 83(11): 3786–3790.
[9] Melki SA, Abumrad NA. Expression of the adipocyte fatty acid-binding protein in streptozotocin-diabetes: effects of insulin deficiency and supplementation., 1993, 34(9): 1527–1534.
[10] Distel RJ, Robinson GS, Spiegelman BM. Fatty acid regulation of gene expression. Transcriptional and post- transcriptional mechanisms., 1992, 267(9): 5937–5941.
[11] Bernlohr DA, Bolanowski MA, Kelly TJ Jr, Lane MD. Evidence for an increase in transcription of specific mRNAs during differentiation of 3T3-L1 preadipocytes., 1985, 260(9): 5563–5567.
[12] Hertzel AV, Bennaars-Eiden A, Bernlohr DA. Increased lipolysis in transgenic animals overexpressing the epithelial fatty acid binding protein in adipose cells., 2002, 43(12): 2105–2111.
[13] Hotamisligil GS, Johnson RS, Distel RJ, Ellis R, Papaioannou VE, Spiegelman BM. Uncoupling of obesity from insulin resistance through a targeted mutation in, the adipocyte fatty acid binding protein., 1996, 274(5291): 1377–1379.
[14] Coe NR, Simpson MA, Bernlohr DA. Targeted disruption of the adipocyte lipid-binding protein (aP2 protein) gene impairs fat cell lipolysis and increases cellular fatty acid levels., 1999, 40(5): 967–972.
[15] Shaughnessy S, Smith ER, Kodukula S, Storch J, Fried SK. Adipocyte metabolism in adipocyte fatty acid binding protein knockout mice (aP2-/-) after short-term high-fat feeding: functional compensation by the keratinocyte [correction of keritinocyte] fatty acid binding protein., 2000, 49(6): 904–911.
[16] Uysal KT, Scheja L, Wiesbrock SM, Bonner-Weir S, Hotamisligil GS. Improved glucose and lipid metabolism in genetically obese mice lacking aP2., 2000, 141(9): 3388–3396.
[17] Scheja L, Makowski L, Uysal KT, Wiesbrock SM, Shimshek DR, Meyers DS, Morgan M, Parker RA, Hotamisligil GS. Altered insulin secretion associated with reduced lipolytic efficiency in aP2-/- mice., 1999, 48(10): 1987–1994.
[18] Baar RA, Dingfelder CS, Smith LA, Bernlohr DA, Wu C, Lange AJ, Parks EJ. Investigation of in vivo fatty acid metabolism in AFABP/aP2(-/-) mice., 2005, 288(1): E187–E193.
[19] Smith AJ, Thompson BR, Sanders MA, Bernlohr DA. Interaction of the adipocyte fatty acid-binding protein with the hormone-sensitive lipase: regulation by fatty acids and phosphorylation., 2007, 282(44): 32424–32432.
[20] Kralisch S, Fasshauer M. Adipocyte fatty acid binding protein: a novel adipokine involved in the pathogenesis of metabolic and vascular disease?, 2013, 56(1): 10–21.
[21] Fu YC, Luo LH, Luo NL, Garvey WT. Lipid metabolism mediated by adipocyte lipid binding protein (ALBP/aP2) gene expression in human THP-1 macrophages., 2006, 188(1): 102–111.
[22] Pearson SL, Cawthorne MA, Clapham JC, Dunmore SJ, Holmes SD, Moore GB, Smith SA, Tadayyon M. The thiazolidinedione insulin sensitiser, BRL 49653, increases the expression of PPAR-gamma and aP2 in adipose tissue of high-fat-fed rats., 1996, 229(3): 752–757.
[23] 闫桂蕊, 张小东, 蔡海燕, 朱维良, 王贺瑶. 糖尿病和动脉粥样硬化的潜在靶标——FABP4(aP2). 生命科学, 2009, 21(2): 270–275.
[24] Christy RJ, Yang VW, Ntambi JM, Geiman DE, Landschulz WH, Friedman AD, Nakabeppu Y, Kelly TJ, Lane MD. Differentiation-induced gene expression in 3T3-L1 preadipocytes: CCAAT/enhancer binding protein interacts with and activates the promoters of two adipocyte-specific genes., 1989, 3(9): 1323–1335.
[25] Yeung DC, Xu AM, Cheung CW, Wat NM, Yau MH, Fong CH, Chau MT, Lam KS. Serum adipocyte fatty acid-binding protein levels were independently associated with carotid atherosclerosis., 2007, 27(8): 1796–1802.
[26] Xu AM, Tso AWK, Cheung BMY, Wang Y, Wat NMS, Fong CHY, Yeung DCY, Janus ED, Sham PC, Lam KSL. Circulating adipocyte-fatty acid binding protein levels predict the development of the metabolic syndrome: a 5-year prospective study., 2007, 115(12): 1537–1543.
[27] Tuncman G, Erbay E, Hom X, De Vivo I, Campos H, Rimm EB, Hotamisligil GS. A genetic variant at the fatty acid-binding protein aP2 locus reduces the risk for hypertriglyceridemia, type 2 diabetes, and cardiovascular disease., 2006, 103(18): 6970–6975.
[28] Boord JB, Fazio S, Linton MF. Cytoplasmic fatty acid-binding proteins: emerging roles in metabolism and atherosclerosis., 2002, 13(2): 141–147.
[29] Pelton PD, Zhou LB, Demarest KT, Burris TP. PPARγ activation induces the expression of the adipocyte fatty acid binding protein gene in human monocytes., 1999, 261(2): 456–458.
[30] Elmasri H, Karaaslan C, Teper Y, Ghelfi E, Weng MQ, Ince TA, Kozakewich H, Bischoff J, Cataltepe S. Fatty acid binding protein 4 is a target of VEGF and a regulator of cell proliferation in endothelial cells., 2009, 23(11): 3865–3873.
[31] Fu YC, Luo NL, Lopes-Virella MF. Oxidized LDL induces the expression of ALBP/aP2 mRNA and protein in human THP-1 macrophages., 2000, 41(12): 2017–2023.
[32] Kazemi MR, McDonald CM, Shigenaga JK, Grunfeld C, Feingold KR. Adipocyte fatty acid-binding protein expression and lipid accumulation are increased during activation of murine macrophages by toll-like receptor agonists., 2005, 25(6): 1220–1224.
[33] Llaverias G, Noé V, Peñuelas S, Vázquez-Carrera M, Sánchez RM, Laguna JC, Ciudad CJ, Alegret M. Atorvastatin reduces CD68, FABP4, and HBP expression in oxLDL-treated human macrophages., 2004, 318(1): 265–274.
[34] Makowski L, Brittingham KC, Reynolds JM, Suttles J, Hotamisligil GS. The fatty acid-binding protein, aP2, coordinates macrophage cholesterol trafficking and inflammatory activity. Macrophage expression of aP2 impacts peroxisome proliferator-activated receptor γ and IκB kinase activities., 2005, 280(13): 12888–12895.
(责任编委: 赵要风)
Analysis ofexpression pattern in rump fat deposition and metabolism of Altay sheep
Ruixia Xu1,2, Lei Gao2, Weili Zhao1,2, Wei Zhang1,2, Guangchao Song1,2, Shangquan Gan2, Guoqing Shi1,2
() is a hot candidate gene in fat deposition and lipid metabolism and participates in the transport and metabolism of intracellular free fatty acids. We aim to study the role ofin fat deposition and metabolism of the rump fat in Altay sheep. In this study, bioinformatics method was used to analyze the protein sequence homology among 10 species, and RT-PCR was employed to detecttissue profiling of Altay sheep. An animal model simulating the rump fat deposition and metabolism of Altay sheep was established by continuous starvation, and qPCR and iTRAQ (isobaric tags for relative and absolute quantitation) were used to detectemRNA and protein expression changes in the control and continuous starvation groups, respectively. Sequence analysis showed that FABP4 protein sequence is highly conserved among species, suggesting an important biological function during evolution for. The RT-PCR result confirmed thatmRNA was highly expressed in intestinal and rump fat, suggesting thatplays an important physiological role in fat tissues. We did not find significant differences inmRNA and protein between control and continuous starvation groups (>0.05), which indicates thatmay not be the key gene in fat deposition and metabolism in Altay sheep.The results above lay a foundation for further studies ofin rump or tail fat.
FABP4; Altay sheep; rump(tail) fat deposition and metabolism; expression change
2014-10-28;
2014-11-24
国家自然科学基金新疆联合基金重点项目(编号:U1130302)和“973”前期专项项目(编号:2011CB111501)资助
许瑞霞,硕士研究生,专业方向:绵羊分子育种设计。Tel: 0993-6683739;E-mail: ruixiaxu2013@163.com
甘尚权,副研究员,研究方向:分子遗传学。E-mail: shangquangan@163.com石国庆,研究员,研究方向:动物繁殖与绵羊育种。E-mail: nkkxyxms@163.com
10.16288/j.yczz.14-369
网络出版时间: 2014-12-23 10:11:59
URL: http://www.cnki.net/kcms/detail/11.1913.R.20141223.1011.001.html

