珠美海棠Mz2NHX1基因的克隆和序列分析
张永利等


摘要:以珠美海棠幼苗根系的cDNA为模板,根据珠美海棠NHX1(GQ503257)的保守序列设计引物,通过 RT-PCR 扩增得到目的基因全长1 865 bp,包含1个1 635 bp的开放阅读框,编码544个氨基酸,其核苷酸序列与苹果(GU338395)、玫瑰(KC188664)、杨树(ACU01853)的核苷酸序列同源性分别是95%、93%、91%。其对应的氨基酸序列与拟南芥(AAF21755)、玫瑰(BAD93487.1)和大叶补血草(BAB11940)液泡型Na+/H+逆向转运蛋白的氨基酸序列的同源性分别是79%、87%、79%,说明该蛋白是一种定位于液泡膜的Na+/H+逆向转运蛋白,该基因属于液泡膜Na+/H+逆向转运蛋白基因。它编码的蛋白质分子量为60.5 ku,理论等电点(pI)为8.85,二级结构主要由α-螺旋、β-折叠和不规则卷曲构成,其N-末端具有12个跨膜疏水片段,C-末端具有亲水的长链尾巴,在跨膜片段内含有氨基酸保守序列85-LFFIYLLPPI-94,是Na+/H+逆向转运蛋白抑制剂氨氯吡嗪咪的结合位点。
关键词:珠美海棠;Mz2NHX1基因;克隆;生物信息学;耐盐机理
中图分类号: Q785;S685.120.1文献标志码: A文章编号:1002-1302(2015)09-0020-05
盐害是影响植物生长发育和农作物产量的主要限制因子之一,它对植物的伤害表现为离子不平衡、水分亏缺和离子毒害[1]。土壤中过多的Na+导致植物细胞的膜功能失调、代谢活动衰减及其他次生影响,使植物生长受到抑制,直至细胞死亡[2-3]。为避免盐的毒害作用,植物形成了特殊的耐盐机制,即限制Na+吸收、增加Na+外排及Na+区隔化[4]。这3种途径主要与细胞质膜上和液泡膜上的Na+/H+逆向转运蛋白有关。编码质膜Na+/H+逆向转运蛋白的基因首先从酵母中克隆得到[5],随后从拟南芥中克隆到SOS1[6],SOS1基因与真菌中编码质膜Na+/H+逆向转运蛋白的基因有很高的同源性,并且SOS1基因的突变体对盐胁迫非常敏感,盐胁迫可以增SOS1基因的表达,说明SOS1在拟南芥的耐盐机制中起重要作用。由于Na+/H+逆向转运蛋白可以将Na+外排或区隔化到液泡中,因此它们在维持细胞渗透平衡中具有重要作用[7]。在植物中,液泡膜Na+/H+逆向转运蛋白(NHX1)的活性最初在甜菜碱储藏组织的液泡形成体的小泡囊中观察到,后来从拟南芥、水稻和冰叶日中花中分别克隆了液泡膜Na+/H+逆向转运蛋白基因AtNHX1[8]、OsNHX1[9]和McNHX1[10],并且发现盐胁迫可以增加它们的表达,说明NHX1基因在植物的耐盐机制中起关键作用。因此通过研究Na+/H+逆向转运蛋白基因的耐盐机理,可以为培育优良的耐盐种质资源提供理论依据。
珠美海棠为蔷薇科(Rosacaeae)苹果属(Malus)多年生木本植物,有较高的应用价值。该树种耐盐碱、抗寒能力强,在盐碱地较重的地区可作砧木嫁接苹果,成活率较高。它在含盐量0.6%、pH值9.2的土壤中也能正常生长[11]。推测这种高度的耐盐能力与植物的耐盐基因有关。
关于植物的Na+/H+逆向转运蛋白基因的研究已有很多报道,但是对珠美海棠的相关研究报道很少。笔者在前期研究(GQ503257)的基础上,以珠美海棠组培生根苗为试材,克隆了珠美海棠NHX1的同源基因Mz2NHX1,并对Mz2NHX1基因进行了序列分析和生物信息学分析,旨在为研究该基因的功能、探讨珠美海棠的耐盐机制奠定基础,同时也为耐盐基因的利用提供理论依据。
1材料与方法
1.1材料与试剂
珠美海棠[Malus zumi (Matsum.) Reder]组培生根苗,由天津农学院果树学重点实验室提供。Recombinant DNase Ⅰ(RNase-free)、Ribonuclease Inhibitor、Oligo d(T)18 Primer、Reverse Transcriptase M-MLV(RNase H-)、TaKaRa Ex Taq、TaKaRa MiniBEST Agarose Gel DNA Extraction Kit Ver.4.0购自TaKaRa公司;RNase/DNase Free枪头、RNase/DNase Free离心管、Ampicillin、X-gal、IPTG、SanPrep柱式质粒DNA小量抽提试剂盒购自生工生物工程(上海)股份有限公司;Marker购自天根生化科技(北京)有限公司;克隆载体试剂盒 pEASY-T1 Cloning Kit 和大肠杆菌感受态Trans1-T1 Phage Resistant Chemically Competent Cell购自北京全式金生物技术有限公司;引物由生工生物工程(上海)股份有限公司合成,DNA测序由华大基因有限公司完成。其他药品均为分析纯,购自生工生物工程(上海)股份有限公司。
1.2方法
1.2.1珠美海棠总RNA提取和反转录取0.1 g珠美海棠幼苗根系,用改良的CTAB法提取RNA,并用DNaseⅠ处理去除DNA污染,电泳检测其质量;利用微量紫外分光光度计(NANODROP 2000)测定RNA浓度,根据宝生物反转录酶说明书取适量M-MLV酶和RNA进行反转录,合成cDNA第一链,-80 ℃保存备用。
1.2.2引物设计与目的基因的扩增根据已克隆的珠美海棠NHX1基因的保守序列,设计1对特异引物P1:5′-AGGAGGCGGATACAATGGCT-3′和P2:5′-CAACCGATGTGCTTGGGAC-3′,利用RT-PCR反应扩增目的基因的全长序列。根据全长序列设计特异引物P3:5′-CGGGGTACCCCGATGGCTGTTCCACATTTG-3′和P4:5′-CCGGAATTCCGGTTGCCACTGAACGTTGTTG-3′,扩增目的基因的开放阅读框序列。扩增体系(20 μL):10×Ex PCR buffer 2.0 μL,dNTP Mix 1.6 μL,引物P1和引物P2各0.8 μL,cDNA模板1.5 μL,Ex Taq 0.1 μL,DEPC-d2H2O 13.2 μL。反应条件:94 ℃预变性4 min;94 ℃变性30 s,55.3 ℃退火30 s,72 ℃延伸 2 min,后延伸10 min,30个循环,4 ℃短暂保存。endprint
1.2.3目的片段的检测与鉴定使用TaKaRa MiniBEST Agarose Gel DNA Extraction Kit回收目的片段,将目的片段与pEASY-T1载体连接15 min,然后转化大肠杆菌感受态细胞,整个过程要轻柔。在不含Amp的LB液体培养基中37 ℃条件下、200 r/min摇菌1 h,然后在含有IPTG和X-gal的 LB(+Amp) 固体培养基上37 ℃恒温培养过夜。挑取白色单菌落至LB(+Amp)液体培养基中,在37 ℃、200 r/min下摇菌培养8 h,以菌液为模板做PCR,鉴定阳性重组子。提取质粒,通过双酶切法(EcoRⅠ,KpnⅠ)鉴定目的片段是否成功连入载体,用鉴定正确的质粒送华大基因公司测序。
1.2.4目的基因的生物信息学分析利用NCBI的BLASTn和BLASTp进行核苷酸及氨基酸序列相似性分析;采用DNAMAN软件进行多序列比对,并预测蛋白质的疏水性和亲水性,采用MEGA 5.0软件进行系统进化树分析;利用TMpred在线准确预测跨膜蛋白的跨膜片段,利用TMHMM在线对目的基因进行跨膜结构域的预测;采用ProtParam软件分析蛋白质的理化性质。
2结果与分析
2.1珠美海棠Mz2NHX1基因的克隆和测序分析
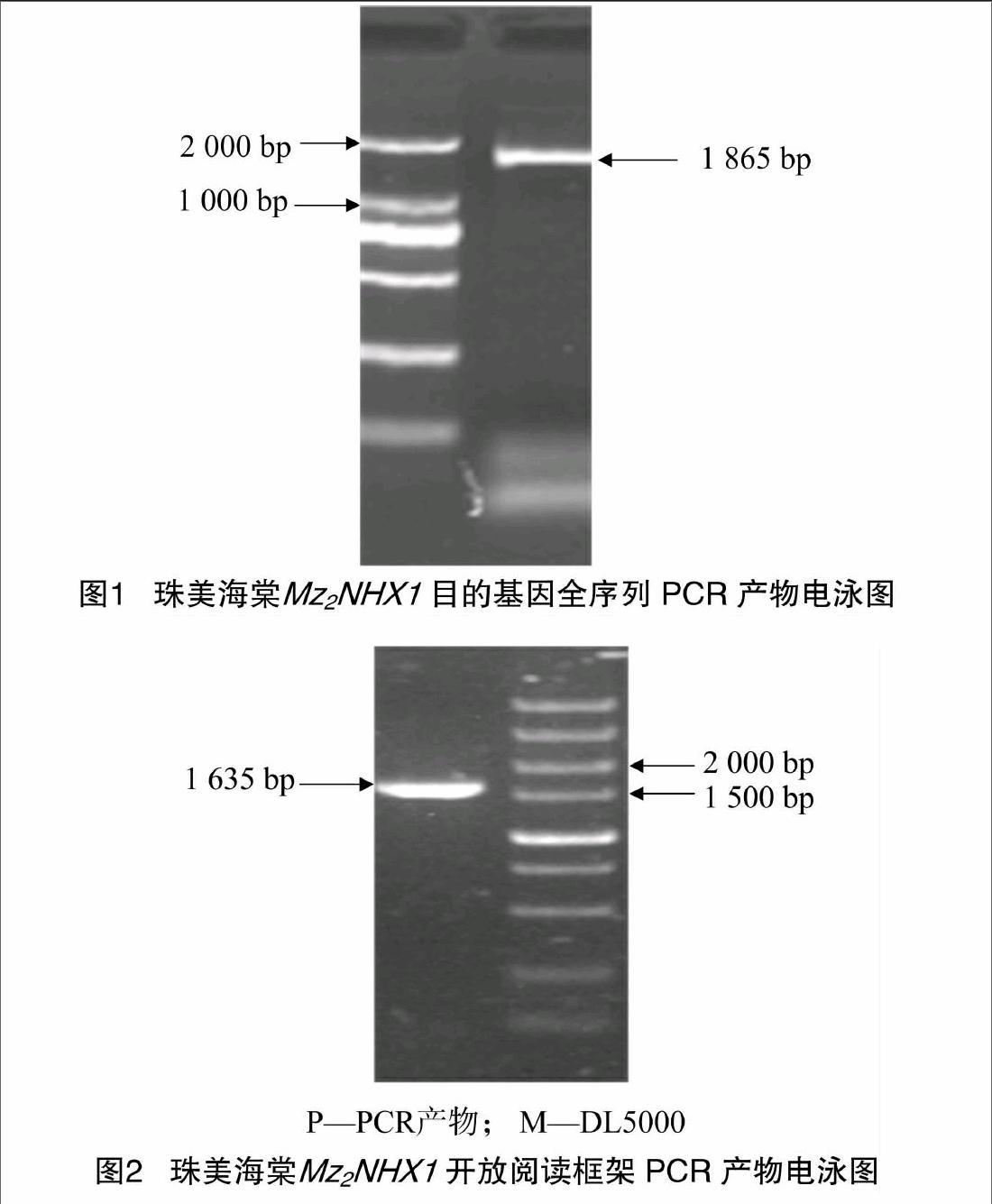
用质量分数1%的琼脂糖凝胶检测从珠美海棠中提取的RNA质量,总RNA基本无降解,无蛋白质污染,28 S、18 S和5 S条带完整。RNA反转录为cDNA第1条链后,以cDNA为模板,用引物P1和P2进行PCR扩增得到1条1865 bp的目的条带(图1),与苹果(GU338395)、玫瑰(KC188664)、杨树(XM_002307158)的核苷酸序列的同源性分别为95%、93%、91%。通过NCBI的ORF Finder在线分析,预测开放阅读框长1 635 bp。根据该核苷酸序列设计引物P3、P4,PCR扩增出其开放阅读框序列(图2),经与pEASY-T1连接、转化、蓝白斑筛选,菌液PCR和酶切鉴定后测序,得到1 635 bp的核苷酸序列,测序结果与预测序列吻合,我们将该基因命名为Mz2NHX1。
2.2珠美海棠Mz2NHX1基因的生物信息学分析
2.2.1Mz2NHX1氨基酸序列分析经 NCBI 的 ORF Finder在线软件分析,Mz2NHX1基因开放阅读框长 1 635 bp,编码1
个由 544 个氨基酸组成的蛋白 Mz2NHX1(图3)。
通过Protparam在线分析可知,Mz2NHX1蛋白的分子质量为60.5 ku,等电点(pI)为8.85,分子式为C2802H4377N699O749S20。用DNAman软件分析发现,珠美海棠与拟南芥(AAF21755)、大叶补血草(BAB11940)和玫瑰(BAD93487.1)等液泡膜 Na+/H+ 逆向转运蛋白的氨基酸序列有较高的同源性,分别为79%、79%和87%,并且含有保守序列85-LFFIYLLPPI-94,是Na+/H+逆向转运蛋白抑制剂氨氯吡嗪咪的结合位点;以及钙调素类蛋白(AtCaM15)的结合位点的氨基酸保守序列515-RKFDNAFMRPVFGGRG-536(图4)。
用MEGA 5.0软件对珠美海棠Mz2NHX1编码的氨基酸与拟南芥、水稻及牵牛花、番杏、玫瑰、杨树的Na+/H+逆向转运蛋白的氨基酸序列进行了进化树分析。结果(图5)发现,Mz2NHX1与苹果(ADB92598)和玫瑰(BAD93487)的 Na+/H+ 逆向转运蛋白的氨基酸序列同源性达100%,而与牵牛花同源性达95%,与番杏和杨树的同源性为 90%,与水稻的同源性为86%,亲缘关系比较近。以上植物的NHX基因编码蛋白均定位在液泡膜上,推测Mz2NHX1也是定位于液泡膜上。其主要功能是将过量的Na+区隔入液泡,避免Na+积累对细胞造成离子毒害,降低细胞渗透势提高植物吸收水分的能力,抵御盐胁迫对植物的渗透胁迫。
2.2.2Mz2NHX1基因编码蛋白的理化性质对Mz2NHX1编码的蛋白进行分析发现,该蛋白编码544个氨基酸,包含参与生物组成的所有氨基酸。用DNAman软件分析,Mz2NHX1蛋白分子量为60.5 ku,等电点(PI)为8.85。由疏水性分析(图6)可知,Mz2NHX1蛋白N-末端有12个跨膜疏水区域和1个较长的C-末端亲水性尾巴。用TMHMM Server v.2.0在线软件对其跨膜区域(图7)进一步预测表明,Mz2NHX1的
N-端面向细胞质,推测N-端的疏水区域可能含有与Na+结合的重要氨基酸残基,并且可以形成跨膜螺旋,几乎全部存在于液泡内的C-端亲水尾巴可能在对Na+和K+的选择性吸收过程中发挥调节作用。研究发现,将拟南芥AtNHX1的亲水C-端去掉后,则可导致Na+/H+运输的相对比率增长[12]。因此,NHX1的跨膜结构和亲水尾巴可能在Na+的转运过程中起着重要作用。
2.2.3Mz2NHX1蛋白结构分析利用PORTER在线预测Mz2NHX1编码蛋白的二级结构,结果表明,Mz2NHX1蛋白由47.43%的α-螺旋、17.16%的β-折叠片、3.68%的β-转角和31.80%的无规则卷曲组成(图8)。
3讨论与小结
植物Na+/H+逆向转运蛋白是一种电中性转运蛋白,不同植物中的该蛋白的N-末端具有高度同源性,该端是负责Na+转运的区域,对Na+的竞争性抑制剂氨氯吡嗪脒及其衍生物敏感,C-末端是亲水性长链尾巴,位于细胞质或液泡囊腔内,此结构域内含有多个蛋白激酶作用位点,能与钙调素结合,参与启动多种信号的反应,是调节活性的区域 [13]。
有研究将AtNHX1基因在拟南芥上进行过量表达,结果发现转拟南芥植株在200 mmol/L NaCl溶液浇灌下能正常生长发育[8]。将AtNHX1基因转入番茄中,过量表达AtNHX1基因的转基因番茄在200 mmol/L NaCl浓度中能够正常开花和结实,但是叶片中Na+浓度高于果实中Na+浓度 [14]。这些结果表明,转单一的Na+/H+逆向转运蛋白基因能够明显提高植物耐盐性,其原因可能是Na+/H+逆向转运蛋白基因导入植物细胞后,激活了一系列与耐盐相关的基因,从而明显提高植物的耐盐性,说明Na+/H+逆向转运蛋白基因属于耐盐关键基因。因此,在利用转基因技术培育耐盐品种中有很高的应用价值。endprint
本研究通过将Mz2NHX1编码的Mz2NHX1与已知Na+/H+逆向转运蛋白氨基酸序列的同源性对比及系统发育分析,认为Mz2NHX1是一种定位于液泡膜的Na+/H+逆向转运蛋白,与拟南芥(AAF21755)、大叶补血草(BAB11940)和玫瑰(BAD93487.1)等液泡膜Na+/H+逆向转运蛋白的氨基酸序列有较高的同源性,分别为79%、79%和87%,因此,
Mz2NHX1基因属于液泡膜Na+/H+逆向转运蛋白基因。对Mz2NHX1核酸序列所翻译的蛋白序列预测发现N-末端含有12个跨膜疏水区域和1个较长的C-末端亲水性尾巴。对该蛋白质二级结构预测分析可知,Mz2NHX1由47.43%的α-螺旋、17.16%的β-折叠片、3.68%的β-转角和3180%的无规则卷曲组成。
参考文献:
[1]Hamada A,Shono M,Xia T,et al. Isolation and characterization of Na+/H+antiporter gene from the halophyte Atriplex gmelini [J]. Plant Molecular Biology,2001,46:35-42
[2]Yeo A R. Molecular biology of salt tolerance in the context of whole-plant physiology[J]. J Exp Bot,1998,49:915-929
[3]Glenn E P,Brown J J,Blumwald E. Salt tolerance and crop potential of halophytes[J]. Crit Rev Plant Sci,1999,18:227-255
[4]Padan E,Venturi M,Gerchan Y,et al. Na+/H+antiporter[J]. Biochim Biophys Acta,2001,1 505(1):144-157
[5]Jia Z P,McCullough N,Martel R,et al. Gene amplification at a locus encoding a putative Na+/H+antiporter confers sodium/lithium tolerance in fission yeast[J]. EMBO J,1992,11:1631-1640
[6]Shi H,Ishitani M,Kim C,et al. The Arabidopsis thaliana salt tolerance gene SOS1 encodes a putative Na+/H+antiporter[J]. Proc Natl Acad Sci USA,2000,97:6896-6901
[7]Rausch T,Kirsch M,Low R,et al. Salt stress responses of higher plants:the role of proton pumps and Na+/H+-antiporters[J]. Plant Physiol,1996,148:425-433
[8]Apse M P,Aharon G S,Snedden W A,et al. Salt tolerance conferred by overexpression of a vacuolar Na+/H+antiport in Arabidopsis[J]. Science,1999,285(5431):1256-1258.
[9]Fukuda A,Nakamura A,Tanaka Y. Molecular cloning and expression of the Na+/H+exchanger gene in Oryza sativa[J]. Biochimica et Biophysica Acta,1999,1446(1/2):149-155.
[10]Barkla B J,Zinggarelli L,Blumwald E,et al. Topolast Na+/H+antiport activity and its energization by the vacuolar H+-ATPase in the halophytic plant Mesembryanthemum crystallinum L.[J]. Plant Physiol,1995,109:549-556
[11]顾逎良,赵惠祥,马继龙,等. 珠美海棠对盐碱地适应范围及应用[J]. 天津农学院学报,1996,3(3):48-52.
[12]Yamaguchi T,Apse M P,Shi H,et al. Topological analysis of a plant vacuolar Na+/H+antiporter reveals a luminal C terminus that regulates antiporter cation selectivity[J]. Proceedings of the National Academy of Sciences of the United States of America,2003,100(21):12510-12515.
[13]石乐义,李美茹,李洪清,等. 植物Na+/H+逆向转运蛋白功能及调控的研究进展[J]. 广西植物,2006,26(6):602-607.
[14]Zhang H X,Blumwald E. Transgenic salt-tolerance tomato plants accumulate salt in foliage but not in fruit[J]. Nat Biotechnol,2001,19:765-768endprint
[15]Apse M P,Blumwald E. Na+transport in plants[J]. FEBS Letters,2007,581(12):2247-2254.
[16]Blumwald E,Aharon G S,Apse M P. Sodium transport in plant cells[J]. Biochimica et Biophysica Acta ,2000,1465(1/2):140-151.
[17]Ohta M,Hayashi Y,Nakashima A,et al. Introduction of a Na+/H+antiporter gene from Atriplex gmelini confers salt tolerance to rice[J]. FEBS Letters,2002,532(3):279-282.
[18]Shi H Z,Lee B H,Wu S,et al. Overexpression of a plasmamembrane Na+/H+antiporter gene improves salt tolerance in Arabidopsis thaliana[J]. Nat Biotechnol;2003,21:81-85
[19]Xue Z Y,Zhi D,Xue G P,et al. Enhanced salt tolerance of transgenic wheat(Tritivum aestivum L.) expressing avacuolar Na+/H+antiporter gene with improved grain yields in saline soils in the field and a reduced level of leaf Na+[J]. Plant Sci,2004,167:849-859.
[20]Fukuda A,Nakamura A,Tagiri A,et al. Function,intracellular localization and the importance in salt tolerance of a vacuolar Na+/H+ antiporter from rice[J]. Plant & Cell Physiology,2004,45(2):146-159.
[21]Zhao F,Wang Z,Zhang Q,et al. Analysis of the physiological mechanism of salt-tolerant transgenic rice carrying a vacuolar Na+/H+antiporter gene from Suaeda salsa[J]. Journal of Plant Research,2006,119(2):95-104.
[22]李金耀,张富春,马纪,等. 植物分子水平的耐盐机制[J]. 植物生理学通讯,2003,39(6):715-719.
[23]吕慧颖,李银心,孔凡江,等. 植物Na+/H+逆向转运蛋白研究进展[J]. 植物学通报,2003,20(3):363-369.
[24]吕慧颖,李银心,陈华,等. 番杏Na+/H+逆向转运蛋白基因的克隆及特性分析[J]. 高技术通讯,2004,14(11):26-31.
[25]郭会敏,顾春笋,刘兆磊,等. 荷花液泡膜型Na+/H+逆向转运蛋白基因NnNHX1的克隆与特性分析[J]. 植物生理学通讯,2010,46(10):1025-1032.
[26]严一诺,孙淑斌,徐国华,等. 菊芋Na+/H+逆向转运蛋白基因的克隆与表达分析[J]. 西北植物学报,2007,27(7):1291-1298.endprint

