老年患者医院感染的肠外致病性大肠埃希菌毒力因子检测及SNP分析
曹阳,刘双庆,魏殿军,陈薇
临床研究
老年患者医院感染的肠外致病性大肠埃希菌毒力因子检测及SNP分析
曹阳,刘双庆,魏殿军△,陈薇
目的了解老年患者医院感染肠外致病性大肠埃希菌(ExPEC)30种毒力因子的检出情况及其分子流行病学特点。方法收集天津地区引起老年患者医院感染的非重复肠外致病性大肠埃希菌140株,采用多重PCR方法检测其毒力因子基因的存在情况,比较不同标本来源ExPEC毒力因子基因的检出率。从检出fimH基因的菌株中选取50株,进行fimH基因的扩增与测序,将全部测序结果与大肠埃希菌对照株CFT037、UTI89和参考株K-12 Gen⁃Bank数据库中的fimH基因序列均输入软件DNAMAN 6.0.3.93中寻找单核苷酸多态性(SNPs),后将53个fimH基因序列输入软件MEGA4,进行基因分型和树状图的构建。分型结果结合病例回顾,综合分析医院内大肠埃希菌克隆流行情况。结果ExPEC中毒力因子基因fimH、traT、fyuA、iutA和kpsMT II的检出率较高,非尿标本来源ExPEC的毒力因子基因kpsM II、K5、papC、papEF、papG allele II(Internal)、papA、cnf1(CNF)、sfa/focDE和rfc的检出率均比尿标本来源ExPEC的高。53株菌fimH基因SNPs分析显示,在57个多态位点中发现有60种SNPs。53株菌被分为25个基因型,其中有11组菌株的基因指纹图谱分别完全一致。未发现不同医院的菌株具有相同的基因型。结论老年患者医院感染的肠外致病性大肠埃希菌携带多种毒力因子基因;非尿标本来源的大肠埃希菌可能比尿液标本来源的菌株致病性更强;fimH基因SNPs分型适用于医院大肠埃希菌克隆流行的调查,值得在今后的临床工作中推广。
大肠杆菌;交叉感染;毒力因子类;多态性,单核苷酸;fimH基因
引起肠道外感染的大肠埃希菌统称肠外致病性大肠埃希菌(extraintestinal pathogenic E.coli,ExPEC),是导致尿路感染、医院获得性肺炎、败血症及褥疮伤口感染的主要致病菌之一[1]。而以上多种感染多为医院感染,其患者以老年人为主。ExPEC中存在多种毒力因子,如黏附素、保护素、毒素等[2],在医院感染的致病机制中发挥着重要作用。ExPEC也是院内暴发克隆流行的常见菌株之一,而对老年患者医院感染的不同来源标本中ExPEC毒力因子的检出情况,国内相关报道较少,本研究采用一种新型的大肠埃希菌基因分型方法fimH单核苷酸多态性(SNPs)分型对ExPEC进行流行病学调查,以期为临床预防和控制老年患者的ExPEC感染提供依据。
1 材料与方法
1.1菌株2010年6月—2012年6月收集天津地区3所三级甲等综合医院的老年医院感染患者140例,女85例,男55例,患者年龄65~97岁,感染种类包括医院感染性肺炎、尿路感染、褥疮感染和败血症等。收集引起其感染的非重复肠外致病性大肠埃希菌140株,其中尿标本来源76株(尿标本组),其他来源标本(包括痰、分泌物和血液等)64株(非尿标本组)。菌株采用常规生化鉴定。医院感染诊断标准参照卫生部2001年《医院感染管理规范》。
1.2方法
1.2.1毒力因子基因的扩增煮沸法提取细菌DNA。采用六重三步PCR方法检测140株大肠埃希菌中的30种毒力因子。PCR体系:Premix 10.5 μL,上、下游引物各1 μL,DNA模板2.5 μL,水10 μL,共25 μL。反应条件为95℃12 min;94℃30 s,63℃30 s,68℃3 min,共25个循环;72℃10 min。30种大肠埃希菌毒力因子基因的名称、引物序列、产物长度及具体检测方法见文献[3-4]。
1.2.2fimH基因的测序与SNPs分型分析从检出fimH基因的菌株中选取50株,以普通PCR方法扩增fimH基因,反应体系及条件与多重PCR相同,其中27株来自于尿标本,23株来自其他来源标本。将其fimH基因PCR扩增产物送至北京Invitrogen生物公司进行测序。测序结果经软件Se⁃quence Scanner version 1.0拼接后,选取其中测序结果最可靠的674 bp序列(位于大肠埃希菌参考株K-12 fimH基因的119 bp~792 bp)用于SNPs分析。将50株菌的fimH基因674 bp序列与大肠埃希菌对照株CFT037、UTI89和参考株K-12 GenBank数据库中fimH基因674 bp序列均输入到软件DNAMAN 6.0.3.93中,以参考株K-12的fimH基因序列为参比序列,找出SNPs。将全部53个fimH基因的674 bp序列输入软件MEGA4,进行基因分型和树状图的构建[5]。
1.3统计学方法采用SPSS15.0软件包进行统计分析,计数资料以例(%)表示,组间比较采用χ2检验与Fisher确切概率法,P<0.05为差异有统计学意义。
2 结果
2.1大肠埃希菌毒力因子基因检测30个毒力因子基因中,检测到26个毒力因子基因,未检测到papG internal I、gafD、cdtB、bmaE。经检测的140株ExPEC中,毒力因子基因fimH、traT、fyuA、iutA和kpsMT II的检出率较高,均超过50%,PAI、K5、papC、papEF、papG allele II和papA的检出率在20%~50%,其他毒力因子基因的检出率较低,均<20%。非尿标本组的大肠埃希菌毒力因子基因kpsM II、K5、papC、papEF、papG allele II(Internal)、papA、cnf1(CNF)、sfa/focDE和rfc的检出率均比尿标本组高,见表1。

Tab.1 Detection rate of virulence factor from 140 E.coli strains表1 140株大肠埃希菌毒力因子基因检出情况例 (%)
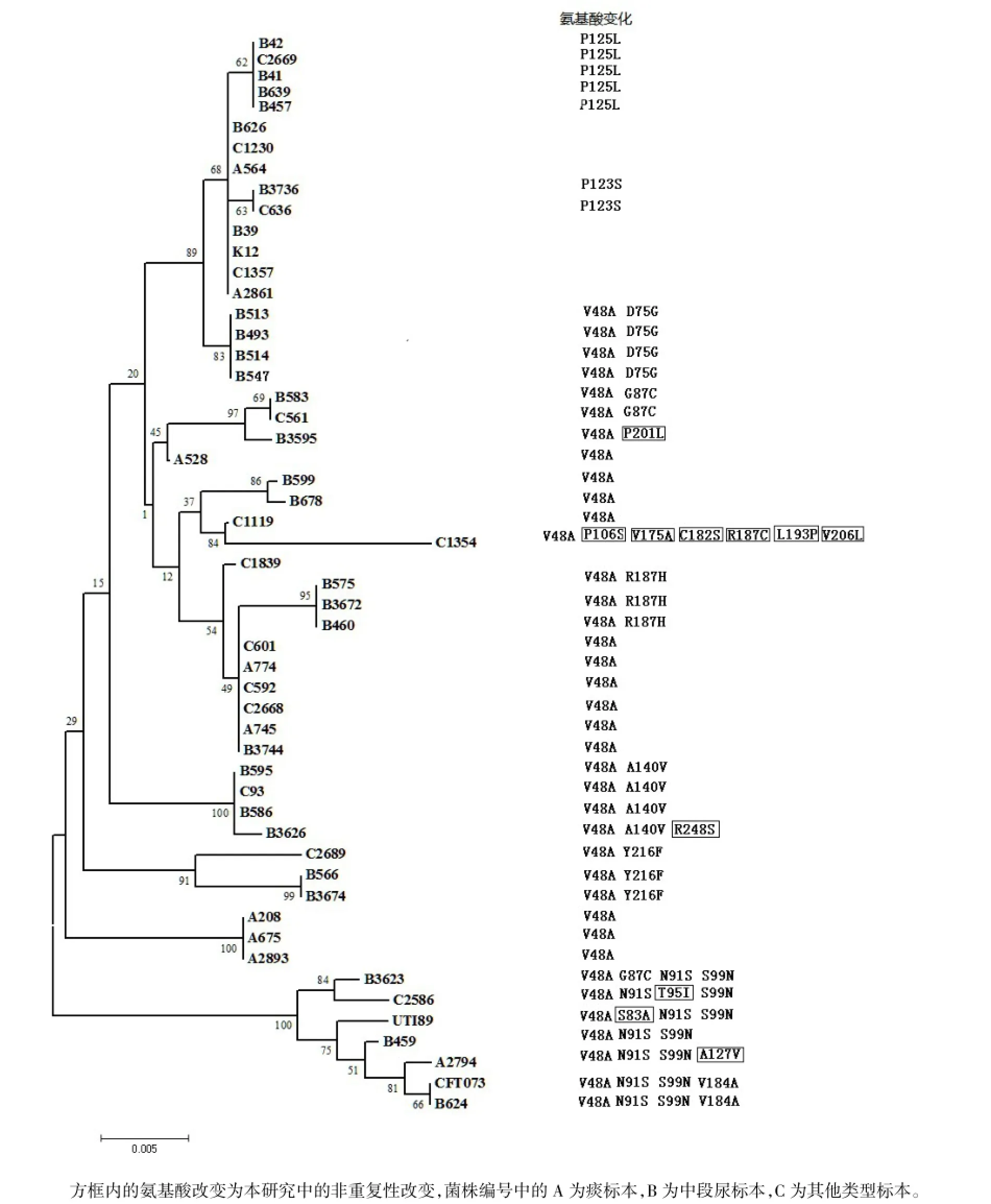
Fig.1 Dendrogram of fimH SNP from 50 clinical isolates and the control isolates CFT073 and UT189 as well as the reference isolate K-12图1 50株临床菌株与对照株CFT073、UTI89和参考株K-12的FimH SNPs分型树状图
2.2fimH基因SNPs分型结果分析见图1。将50株临床菌株,对照株CFT037、UTI89和参考株K-12 的fimH基因674 bp序列输入DNAMAN软件后,同参考株K-12进行对比后发现,在57个多态位点中有60种SNPs。其中,28.33%(17/60)的SNPs为颠换,71.67%(43/60)的SNPs为转换;23种SNPs导致22种氨基酸的替换,37种 SNPs为静默替换。71.70%(38/53)的序列含有V48A的氨基酸替换。将53个fimH序列输入到软件MEGA4进行同源性分析,53株菌被分为25个基因型。其中B42、C2669、B41、B639和B457;B626、C1230、A564、B39、K12、C1357和 A2861;B3736和C636;B513、B493、B514和B547;B583和C561;B575、B3672和B460;C601、A774、C592、C2668、A745和B3744;B595、C93 和 B586;B566和 B3674;A208、A675和 A2893;CFT073和B624的基因指纹图谱分别完全一致。经病例回顾性分析,未发现不同医院的菌株具有相同的基因型。B42、B41、B639、B457;B513、B493、B514、B547分别来自同一年度、同一科室的不同患者的中段尿标本。C601、A774、C592、C2668、A745、B3744来自同一年度、同一科室患者的各类型标本,其中A745、B3744来自同一患者间隔4个月的痰标本和中段尿标本,且药敏结果完全一致。B583、C561为同一患者间隔3 d的中段尿标本和血液标本,且药敏结果完全一致。其他克隆流行组内具有相同基因型的菌株分别属于各自医院的不同科室。
3 讨论
ExPEC毒力因子的存在和毒力的大小决定了所致疾病的种类和严重程度。研究其毒力因子基因有助于了解ExPEC的致病机制。本研究发现老年患者医院感染的ExPEC中携带多种毒力因子基因,其中检出率比较高的毒力因子fimH、traT、fyuA、iutA和kpsMT II的检出率与国外相关报道结果相符[6]。这几种基因编码的产物分别为黏附素、保护素和铁摄取系统,其中黏附素是菌株黏附宿主细胞不被清除的最主要结构,保护素和铁摄取系统是菌株能够在宿主内生存的关键因素。这些毒力因子中某些是在大肠埃希菌进化的早期已经存在,某些是在大肠埃希菌受到了比较强的环境压力下通过较高频率的水平转移而获得。毒力因子的以上特点为制备相应的疫苗预防ExPEC感染指明了方向[7]。
本研究中P菌毛相关毒力因子papG allele II (Internal)、papC、papA、papEF的总体检出率相对较高,在20%~50%,但其在痰液、血液等非尿标本中的检出率均比尿液来源的标本检出率高。原因可能是:(1)P菌毛的受体主要分布在近端肾小管,与尿路致病性大肠埃希菌引起的肾盂肾炎的关系更密切[2]。而本研究中收集的尿标本多来自于老年性的下尿路感染,大肠埃希菌上行侵犯到肾盂者少见,主要黏附在膀胱上皮细胞,与1型菌毛的作用相关,P菌毛对其作用有限[8],因此这类尿标本中的大肠埃希菌P菌毛检出率较低。(2)非尿标本大肠埃希菌可来源于肾盂。本研究中部分大肠埃希菌败血症患者前期有肾盂肾炎,推测其血液中的大肠埃希菌是由于感染未得到控制,进入血流所致,实为肾盂来源,而其他部位感染的大肠埃希菌其来源尚待证实。(3)在其他组织器官上也可能存在P菌毛的高亲和力受体,除P菌毛相关毒力因子外,kpsM II、K5、cnf1 (CNF)、sfa/focDE和rfc在非尿标本中的检出率也比尿标本来源的高,推测由于非尿标本来源的大肠埃希菌携带有更多的毒力因子,其可能比尿液标本来源的菌株致病性更强,相关机制需要进一步研究。
在过去的20多年里,多种基因分型方法应用到大肠埃希菌的流行病学调查。其中多位点序列分型(MLST)和脉冲场凝胶电泳(PFGE)的稳定性和重复性都要强于其他分型方法,但操作步骤繁琐,成本高昂限制其临床应用。fimH基因SNPs分型是近年来出现的一种新型的大肠埃希菌分型方法,研究证明该方法对尿路致病性大肠埃希菌的分型能力与MLST相仿[9]。fimH基因是大肠埃希菌十分重要的黏附基因,是ExPEC在感染部位趋化运动的关键因素[10];绝大多数的ExPEC都具有fimH基因,本研究结果也证实该种论点。而且有报道称fimH基因中存在着大量的有义突变[11]。由于毒力因子基因相对于管家基因更容易受到宿主免疫应答所造成的压力,因此其在短期内就更容易发生变异,所以与MLST相比,fimH基因SNPs分型更适合医院内临床菌株克隆流行的调查。
本研究中,53株菌被分为25个基因型,其中有11组克隆流行株,且不同医院的菌株没有发生交叉流行,多组菌株都分别来自于同一年度,同一科室患者的标本,表明当时这些科室中都存在着大肠埃希菌的克隆流行;有的患者间隔时间4个月后的不同标本都培养出大肠埃希菌,且药敏结果一致,SNPs分型为同一基因型,表明该株大肠埃希菌在患者体内长期定植,一旦患者免疫力低下,就会造成感染。有的患者间隔3 d的中段尿标本和血液标本都培养出大肠埃希菌,且药敏结果一致,SNPs分型也为同一基因型,提示该患者的血液感染来源于尿路感染。由于fimH基因SNPs分型的应用,使临床医生能早期发现医院内大肠埃希菌的克隆流行,能提供患者(尤其是长期感染的患者)感染来源的线索。且fimH基因SNPs分型与MLST一样也是基于基因序列分析,所以试验结果易于标准化,便于实验室之间的交流与沟通,可用于大范围的菌株流行病学调查,甚至进行国际间的基因分型比较。这种方法仅需要扩增1个基因,明显少于MLST的7个基因,无论从操作步骤,还是试验成本上都大大降低。且其分型能力与MLST相仿,适用于院内大样本大肠埃希菌克隆分型的初筛,值得在今后的临床工作中推广。
[1]Jakobsen L,Gameau P,Kurbasic A,et al.Mieroarray-based detec⁃tion of extended virulence and antimierobial resistance gene pro⁃files in phylogroup B2 Escherichia coli of human,meat and animal origin[J].J Med Microbiol,2011,60(Pt 10):1502-1511.
[2]Gao XL,Chen JY,Hou M,et al.Experimental study on distribu⁃tion of virulence factors genes in uropathogenic Escherichia coli [J].Chin J Prev Contr Chron Dis,2009,17(6):551-553.[高新蕾,陈锦英,侯敏,等.致肾盂肾炎大肠杆菌毒力因子的基因分布[J].中国慢性病预防与控制,2009,17(6):551-553].
[3]Kodinba T,Kong F,Johnson JR,et al.Multiplex PCR-based re⁃verse line blot Assay for simultaneous detection of 22 virulence genes in uropathogenic Eseherichia coli[J].Appl Environ Microbi⁃ol,2012,78(4):1198-1202.
[4]Johnson JR,Stell AL.Extended virulence genotypes of Escherich⁃ia coli strains from patients with urosepsis in relation to phylogeny and host compromise[J].J Infect Dis,2000,181:261-272.
[5]Tamura K,Dudley J,Nei M,et al.MEGA4:Molecular Evolution⁃ary Genetics Analysis(MEGA)software version 4.0[J].Mol Biol Evol,2007,24:1596-1599.
[6]Kaczmarek A,Budzynska A,Gospodarek E.Prevalence of genes en⁃coding virulence factors among Escherichia coli with K1 antigen and non-K1 E.coli strains[J].J Med Microbiol,2012,61(Pt 10): 1360-1365.
[7]Scharenberg M,Schwardt O,Rabbani S,et al.Target Selectivity of FimH Antagonists[J].J Med Chem,2012,55(22):9810-9816.
[8]Zhou G,Mo WJ,Sebbel P,et al.Uroplakin Ia is the urothelial recep⁃tor for uropathogenic Escherichia coli:evidence fromin vitro FimH binding[J].J Cell Sci,2001,114(Pt 22):4095-4103.
[9]Tartof SY,Solberg OD,Riley LW.Genotypic analyses of uropatho⁃genic Escherichia coli based on fimH single nucleotide polymor⁃phisms(SNPs)[J].J Med Microbiol,2007,56(Pt 10):1363-1369.
[10]Hung CS,Bouckaert J,Hung D,et al.Structural basis of tropism of Escherichia coli to the bladder during urinary tract infection[J].Mol Microbiol,2002,44(4):903-915.
[11]Sokurenko EV,Feldgarden M,Trintchina E,et al.Selection footprint in the FimH adhesin shows pathoadaptive niche differentiation in Escherichia coli[J].Mol Biol Evol,2004,21(7):1373-1383.
(2014-09-19收稿2014-10-20修回)
(本文编辑闫娟)
Virulence factors detection and single nucleotide polymorphism assay of extraintestinal pathogenic E.coli in elderly nosocomial infection
CAO Yang,LIU Shuangqing,WEI Dianjun△,CHEN Wei
Department of Clinical Laboratory,Second Hospital of Tianjin Medical University,Tianjin 300211,China
△Corresponding Author E-mail:weidianjun01@163.com
ObjectiveTo examine the detection rate of 30 known virulence factors(VFs)of extraintestinal pathogenic Escherichia coli(ExPEC),and to investigates the epidemiology of ExPEC in elderly nosocomial infection.MethodsA to⁃tal of 140 ExPEC clinical isolates from elderly nosocomial patients in hospitals in Tianjin were investigated.Multiplex PCR was performed to detect the 30 virulence factors among the E.coli strains and the detection rate of virulence factors for Ex⁃PEC were compared between isolates from different sites of infection.Fifty E.coli strains were shown to carry fimH gene that was amplified and sequenced.These sequences were used besides3 references strains(CFT037、UTI89 and K-12)to detect SNPs of fimH gene using DNAMAN Version 6.0.3.93 these 53 fimH sequences were used for genotyping and building dendrogram by MEGA4 software.ResultsIn ExPEC,the following virulence factor genes,fimH,traT,fyuA,iutA and kpsMT II,had a higher detection rate than those of the rest.The following virulence factor genes,kpsMT II,K5,papC,papEF,papG allele II(Internal),papA,cnf1(CNF),sfa/focDE and rfc had a a higher detectionrate from non-urine origin sam⁃ples than those from urine origin samples.fimH SNPs analysis of the 50 clinical isolated samples and 3 references samples showed 60 SNPs at 57 polymorphic sites.The fimH SNPs analysis classified the 53 strains into 25 genotype.The genetic fin⁃gerprintings of 11 isolates were exactly the same.ConclusionMany kinds of virulence factors can be found in ExPEC of el⁃derly nosocomial infection.The ExPEC strain isolated from non-urine origin had a stronger pathogenicity than those from urine-origin specimens.fimH SNPs analysis is suitable for molecular epidemiological investigation of ExPEC in hospital.
Escherichia coli;cross infection;virulence factors;polymorphism,single nucleotide;fimH gene
R37
ADOI:10.11958/j.issn.0253-9896.2015.02.014
天津市卫生局科技基金(2013KZ099),天津医科大学第二医院科研基金(Y1202)
天津医科大学第二医院检验科(邮编300211)
曹阳(1979),男,主管技师,硕士,主要从事医院感染的分子流行病学研究
△E-mail:weidianjun01@163.com

