理性设计提高β-葡萄糖醛酸苷酶的热稳定性
汤恒,黄申,冯旭东,李春,
(1 石河子大学化学化工学院,新疆 石河子 832003;2 北京理工大学生命学院,北京 100081)
引 言
甘草酸(glycyrrhizin,GL)是一种五环三萜皂苷类化合物,同时也是甘草的主要有效成分之一,常作为食品和药物添加剂,并有抗炎、抗过敏、抗肝毒、抗肿瘤、抗病毒、抗高血压、抗高血脂及促进肾上腺皮激素分泌等作用;可用于人体抗衰老、抗炎症、降压、增强机体免疫力、提高生理机能、改善高血脂症、维持水盐代谢平衡等[1-2]。另外,其在化妆品和食品添加剂领域也有广泛的应用,是一种重要的精细化工产品[3]。但是作为药物,甘草酸极性较强,无法有效透过脂溶性的细胞膜,限制了其药效的发挥。将甘草酸进行水解后得到的单葡萄糖醛酸基甘草次酸 (glycyrrhetic acid monoglucuronide, GAMG)和甘草次酸(glycyrrhetinic acid,GA)更易被人体吸收,更好地发挥其药理作用,两者的生物利用度都明显优于甘草酸[4-5]。特别是GAMG 更具有甜度高(甘草酸的5 倍,蔗糖的1000 倍)、溶解性好、便于体内运输以及低毒性等特性[6-7],使得其应用前景更加广阔。
β-葡萄糖醛酸苷酶(β-glucuronidase,EC:3.2.1.31)是糖苷类水解酶的一种,能催化各种类型的β-葡萄糖醛酸酸苷键水解,释放β-葡萄糖醛酸和相应的配基。该酶一般属于GH1 和GH2 家族,在GH79 家族中也有分布[8-9]。本课题组前期从新疆甘草种植区筛选到的真菌Penicillium purpurogenum Li-3,能在以甘草酸为唯一碳源的培养基上生长,特异性催化GL 外侧的糖苷键水解,将GL 转化为GAMG。但因野生菌的诱导产酶量较低,本课题组前期构建了重组β-葡萄糖醛酸苷酶的大肠杆菌[E.coli BL21(DE3)]表达体系,简称PGUS-E。PGUS-E的酶学性质较原始酶发生了改变,除了可以将GL催化生成GAMG,还可以进一步催化GAMG 生成GA,如图1 所示。
研究过程中发现,重组酶的热稳定性较差,不能满足工业生产的需要;另一方面,在较高温度下进行反应具有反应黏度低、微生物污染较少且反应速度快等优点[10]。因此提高β-葡萄糖醛酸苷酶的热稳定性具有一定现实意义,不仅可以改进β-葡萄糖醛酸苷酶的工业应用效果,而且可以此为基础研究β-葡萄糖醛酸苷酶结构与功能之间的关系。蛋白质分子理性设计是在对蛋白质结构与功能的认识的基础上,以定点突变技术和定点饱和突变技术为主的,对蛋白质进行改造的方法[11-13]。随着X 射线晶体衍射技术的进步,越来越多的蛋白质结构被解析出来。在研究某个目标蛋白时,即使没有其立体结构的信息,也可以同源结构为模板,借助分子模拟技术预测其结构[14]。
随着研究的深入,相继发现具有不同热稳定性的蛋白质,其维持热稳定性的机制不断被了解,使得采用理性设计对蛋白质进行改造以提高其热稳定性变得越来越具有理论意义。采用理性设计策略,一般可在短时间内将设计完成,并获得性质改善的突变体。而通过理性设计得到的结果,也将加深对蛋白质结构与功能关系的理解。
近年来国内外学者大量运用理性设计对多种工业用酶的性能进行了改造,取得较好的效果。例如,D 型氨基酸在高温环境下对保持蛋白质结构具有很强的作用。Andersen 课题组[15]将色氨酸笼(Trp-cage)迷你蛋白TC10b 的10 号位氨基酸突变为D-丙氨酸,在α螺旋内部转角处有效地加强了刚性,该蛋白的溶解温度(Tm)也由原来的 56℃提升为72℃。对于β-葡萄糖醛酸苷酶的研究,目前国内外研究较少,尤其是在β-葡萄糖醛酸苷酶的热稳定性理性设计研究方面,尚未见相关报道。

图1 重组β-葡萄糖醛酸苷酶转化甘草酸示意图Fig.1 Biotransformation of glycyrrhizin by recombinant β-glucuronidase (PGUS-E)
本研究基于分子模拟的PGUS-E 结构,将通过与其他耐热β-葡萄糖醛酸苷酶的同源比对,以及对酶结构的分析,利用定点突变技术对重组β-葡萄糖醛酸苷酶进行理性设计分子改造,以得到热稳定性明显提高的突变体,并分析热稳定性提高的机理。
1 实验材料和方法
1.1 材料
1.1.1 菌种 E.coli DH5α和E.coli BL21(DE3)感受态细胞购自北京博迈德科技发展公司。重组大肠杆菌(E.coli BL21(DE3)/pET-28a(+)-pgus)由实验室前期构建[16]。β-葡萄糖醛酸苷酶基因 gus(GenBank 收录号为:EU095019.1)由课题组构建的原核表达质粒pET-28a(+)-pgus 中克隆获得。原核表达载体pET-28a(+)购自Novagen 公司。
1.1.2 主要试剂 TransStart FastPfu DNA polymerase 购自北京金式金生物技术有限公司;PCR Master Mix,多功能DNA 纯化回收试剂盒,高纯质粒小量快速提取试剂盒购自北京博迈德生物技术公司;FastDigest Dpn I 购自Thermo Fisher Scientific;Protein Marker, 14.4×103~116.0×103,购自北京庄盟国际生物基因科技公司;2000 DNA Marker,1 kb DNA Ladder 购自北京特可美生物技术有限公司;Nucleic Acid Stain 核酸染料购自北京鼎国昌盛生物技术有限公司;琼脂购自Amresco 公司;酵母提取物,蛋白胨购自Oxford 公司;IPTG,卡那霉素购自北京赛百盛基因有限公司;其余试剂均为国产分析纯。Ni-NTA FF 亲和层析填料购自德国Qiagen 公司。本实验用到的引物由金唯智生物科技有限公司合成,测序工作也由金唯智公司完成。
1.1.3 培养基 LB 培养基 (g·L-1):胰蛋白胨10,酵母提取物5,氯化钠10,琼脂粉15 (固体培养基),固体和液体培养基在需要时加入卡那霉素至50 μg·ml-1。
1.2 方法
1.2.1 酶结构同源模拟与突变位点的选择 在SWISS-MODEL 数据库[17-20](http://swissmodel.expasy.org/workspace) 中输入β-葡萄糖醛酸苷酶氨基酸序列并选择β-葡萄糖醛酸苷酶3k46 为模板,生成同源模拟结构。将该结构与GH2 家族中其他耐热β-葡萄糖醛酸苷酶进行同源比对,获得关键的突变位点。借助PYMOL 蛋白质三维结构分析软件并根据预测的结构信息,采用脯氨酸策略对酶进行理性改性。
1.2.2 定点突变 采用全质粒PCR 构建目标突变,将突变设计在引物上。PCR 体系如表1 所示。

表1 质粒克隆体系Table 1 Plasmid-cloning system
1.2.3 模板消除 用FastDigest Dpn1 酶,在37℃下酶解PCR 产物,以去除模板链,酶切时间为1 h。酶切体系如表2 所示。

表2 模板消除体系Table 2 System for eliminating template
将酶切产物直接用于转化感受态细胞E.coli DH5α,涂布卡那霉素 (50 mg·L-1) 抗性平板筛选阳性克隆。测序正确后将质粒再次转化感受态细胞E.coli BL21(DE3)进行表达。
1.2.4 目标蛋白的纯化 纯化过程全部在4℃下进行,离心收集重组菌,充分倒尽培养基。将菌体重悬于 10 ml 高渗缓冲液[500 g·L-1蔗糖, 50 mmol·L-1Tris-HCl (pH 9.0), 2.5 mmol·L-1EDTA],冰浴中放置30 min。4℃ 8000 r·min-1离心10 min,倒尽上清。加入10 ml 低温冻存的冰水,涡旋振荡使细胞悬浮后持续振荡5 min。4℃ 12000 r·min-1离心20 min,分别收集上清即为粗酶液。为方便蛋白纯化,在构建pET-28a(+)-pgus 载体时,在β-葡萄糖醛酸苷酶的N 端引入6×His-tag。采用AKTA Purifier 10 (GE Healthcare)层析系统和 HisTrap affinity column 亲和层析获得目标蛋白,纯酶液稀释至同一浓度后用于后续实验。
1.2.5 酶活力的测定 取200 μl 2 g·L-1pH 5.0 甘草酸溶液,加入50 μl 一定浓度的纯酶液,37℃水浴中反应30 min,立即取100 μl 反应液,加入至装有900 μl 甲醇的1.5 ml 离心管,样液经孔径为0.45 μm 的有机滤膜过滤至样品池中。用高效液相色谱检测GL、GAMG 和GA 的含量。检测仪器参数设置以及GL、GAMG 和GA 的标准曲线参考文献[21]。在此条件下,底物GL、产物GAMG 和GA的保留时间分别为7.5、15.9 和22.7 min。β-葡萄糖醛酸苷酶活力定义为:在上述反应条件下,每分钟转化1 μmol 甘草酸所需要的β-葡萄糖醛酸苷酶量即为一个活力单位。
1.2.6 野生酶和热稳定性提高突变体的热稳定性测定 纯化得到的酶液分别在60℃的水浴中热变性30、60、90、120、150min,再立即放入冰浴20 min,测定酶活力。将酶溶液不经热变性处理时的酶活力作为100%,以不同温度对相应的残余酶活力百分比作图。定义β-葡萄糖醛酸苷酶60℃下酶活丢失一半的时间为其半衰期,记为T1/2。
1.2.7 野生酶和热稳定性提高突变体的Michaelis-Menten 动力学分析 用50 mmol·L-1pH 4.2 乙酸-乙酸钠缓冲液配制0.2、0.4、0.6、0.8、1.0、1.2 g·L-1的甘草酸溶液,在37、60、70℃下反应30 min 后,检测酶活,采用Lineweaver-Burk作图法计算酶动力学参数。
2 实验结果与讨论
2.1 酶结构的同源模拟及对比分析
将PGUS-E 的氨基酸序列(Genebank 登录号为EU095019)提交SWISS-MODEL,以大肠杆菌K-12 MG1605 的β-葡萄糖醛酸苷酶3k46 (0.25 nm)为PDB 模板,同源性达到55.97%,得到该酶的三维模拟结构,如图2 所示。同源建模的结果由PROCHECK 验证。
糖苷酶家族的活性中心大多在(α/β)8TIM-barrel 的中心[22-23],糖苷酶催化水解糖苷键过程中一般通过两个催化氨基酸(一般为谷氨酸或天冬氨酸)进行催化:一个是质子供体(又称亲核试剂),另一个是酸性/碱性基团。Henrissat 等[24]已通过定点突变的方法证实β-葡萄糖醛酸苷酶3k46 的活性位点为451 位的谷氨酸和540 位的谷氨酸。

图2 PGUS-E 的三维模拟结构Fig.2 3D structure of PGUS-E based on homology modeling
借助PYMOL 蛋白质三维结构分析软件,通过与已经得到晶体结构的β-葡萄糖醛酸苷酶3k46 进 行同源比对,发现PGUS-E 的414 位的谷氨酸和505位的谷氨酸与β-葡萄糖醛酸苷酶3k46 的451 位的谷氨酸和540 位的谷氨酸重叠,推测PGUS-E 活性中心区是以414 位的谷氨酸和505 位的谷氨酸为核心的(α/β)8TIM-barrel 结构域,见图3。
2.2 突变位点的选择
有研究表明,引入脯氨酸可以降低蛋白质去折叠时的骨架熵,提高蛋白质的刚性。脯氨酸包含一个亚氨基,一个羧基及一个吡咯烷环侧链。脯氨酸由于自身吡咯烷环的束缚,因而具有更小的构象自由度,并且还可以将与其相连的其他氨基酸固定在较小的构象空间内,说明脯氨酸的引入可以降低蛋白质去折叠时的骨架熵,即增加蛋白质的刚性。因此有研究表明,在蛋白质构象中柔性较大的区段引入脯氨酸,可以提高蛋白质热稳定性[25-27]。并非所有的脯氨酸引入都会改善热稳定性,选择正确的突变位点是理性设计的关键。酶的核心在于活性中心,如果选择的突变位点距离活性中心过远,则热稳定性可能提高的并不显著;如果选择过近,则可能影响到酶的活性。本实验选择了(α/β)8TIM-barrel 结构域桶型结构底部的Gly280 进行脯氨酸置换,该位点处于距离活性中心区距离适中(3.0~4.0 nm)的不稳定的区段β-转角处(图3)。

图3 PGUS-E 的催化活性中心和突变位点Fig.3 Active sites and mutation sites in catalytic domain of PGUS-E
同时,应用同源比对策略,将PGUS-E 的蛋白序列与糖苷酶家族2 的其他嗜热蛋白进行同源比对,如图4 所示。首先进行位置筛选,挑选距离活性中心区适中(3.0~4.0 nm)的位点,再分析这些位点的突变效应,最终选择了将Ile130 置换为Val(图3)。其原因可能是替换后的缬氨酸增加蛋白质内部活性中心附近的疏水包装密度。由于疏水侧链避开水的需要而相互接近,蛋白质的折叠总是倾向于把疏水残基包裹在内部,但是分子量越大的蛋白质,其疏水包装密度就越难达到100%,因为总会有疏水的氨基酸暴露在蛋白质表面[28]。所以在蛋白质内部更换疏水氨基酸就有可能通过调节内部空穴的体积来容纳更多的疏水氨基酸,并提高整体的疏水包装密度,有效增加蛋白质的稳定性。
最后,将两个突变位点进行组合,构建了I130V+G280P 双点突变体。
2.3 野生酶和热稳定性提高的突变酶的热稳定性测定
纯化得到的酶液在60℃的水浴中分别保温30~150min,立即冰浴20 min,按照1.2.5 节描述方法测定酶活力。记酶溶液不经过热处理时的酶活力为100%,以不同温度对相应的残余酶活力百分比作图。β-葡萄糖醛酸苷酶在60℃下酶活丢失一半的时间为其半衰期T1/2(60℃)。实验结果表明 (图5),突变酶I130V,G280P 和I130V+G280P 在60℃下的半衰期T1/2分别比野生酶23 min 提高3.5 倍、5 倍和5.5 倍,达到82 min、117 min 和128 min。该结果要优于文献中先前的报道,例如,Liu 等[29]对碱性α-淀粉酶进行理性设计,获得了有组合效应的3 组双点突变体,这些突变体在60℃下的半衰期T1/2分别比野生酶3 min 提高了1.5 倍、2.5 倍和3倍,达到5 min、7 min 和11 min。
2.4 野生酶和热稳定性提高突变体的Michaelis- Menten 动力学分析
通过理性设计,酶的热稳定性和催化活性同时得到提高[30],但以活性的损失来换取热稳定性的提高在定向进化中非常普遍。Fang 等[31]对细菌漆酶Lac15 进行理性设计,突变酶在45℃下的半衰期T1/2比野生酶72 min 提高2 倍,达到150 min,但突变酶的Kcat/Km较野生酶下降了10%。因为酶的活性是依靠其活性中心的柔性,而酶的热稳定性需要活性中心外围区域具有刚性,以抵御高温作用的破坏[32]。所以理性设计的选点要求,在尽量小地影响酶的活性中心的同时,提高维持活性中心构象的外围区域的刚性。

图4 PGUS-E 与糖苷酶家族2 的其他嗜热蛋白氨基酸序列比对Fig.4 Sequence alignment of PGUS-E and other thermophilic proteins in GH2
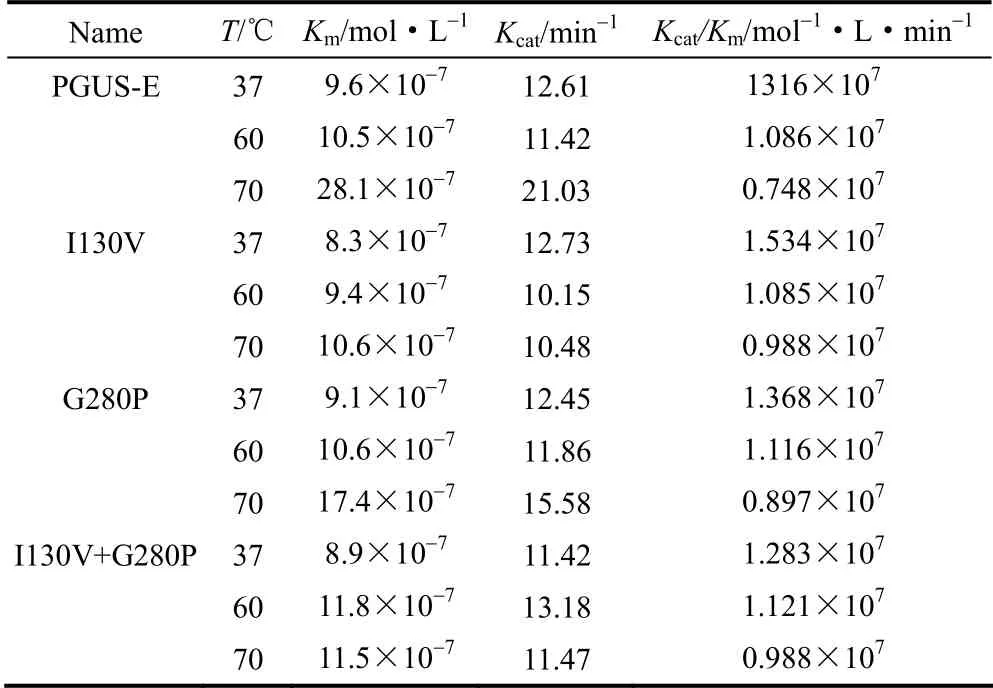
表3 PGUS-E 及其突变体动力学参数Table 3 Kinetic parameters of PGUS-E and its mutants
用50 mmol·L-1pH 4.2 乙酸-乙酸钠缓冲液配制0.2、0.4、0.6、0.8、1.0、1.2 g·L-1的甘草酸溶液,在37、60、70℃下反应30 min 后,检测酶活,采用Lineweaver-Burk 作图法计算酶动力学参数,结果如表3 所示。突变体I130V 和G280P 距离活性中心均超过3.0 nm,将对活性中心的影响降到了最低,在37℃下,突变体对底物的亲和力和催化效率与PGUS-E 基本相同很好地证明了这一点。随着温度的提高,酶与底物的亲和力有所提高,催化效率逐渐下降,但突变体的催化效率下降的幅度较小,这也从另一方面证明了突变体的热稳定性得到了提高。
3 结 论
通过同源比对替换策略和脯氨酸替换策略,得到3 株β-葡萄糖醛酸苷酶突变体I130V、G280P 和I130V+G280P,其热稳定性较野生型分别提高了3.5倍、5 倍和5.5 倍。酶学性质及动力学性质分析表明,突变体在热稳定性提高的同时,对底物的亲和力基本不变,稳定β-葡萄糖醛酸苷酶的工业应用效果,进一步适应了工业生产的要求。同时,成功将同源比对替换策略和脯氨酸替换策略应用于理性设计β-葡萄糖醛酸苷酶,一方面,为研究β-葡聚糖酶结构与功能之间的关系提供了基础,另一方面,也为同源比对替换和脯氨酸替换理性设计策略的应用提供了参考。
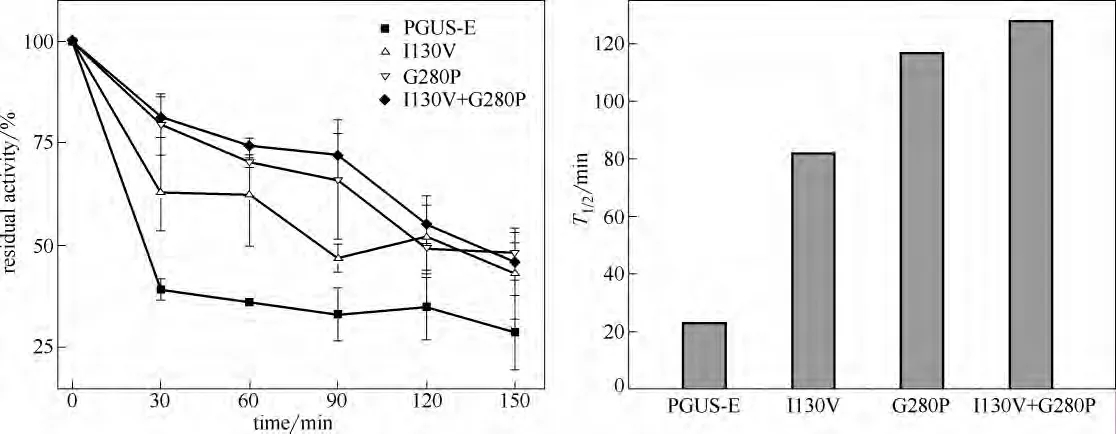
图5 PGUS-E 及其突变体在60℃下的半衰期以及热稳定性 Fig.5 Thermostability and T1/2 of PGUS-E and its mutants at 60℃
[1]Zhou Jiaju(周家驹), Xie Guirong(谢桂荣), Yan Xinjian (严新建).Traditional Chinese Medicines Molecular Structures(中药原植物化学成分手册)[M].Beijing: Chemical Industry Press, 2004.
[2]Cinatl J, Morgenstern B, Bauer G, Chandra P, Rabenau H, Doerr H.Glycyrrhizin, an active component of liquorice roots, and replication of SARS-associated coronavirus [J].The Lancet, 2003, 361(9374): 2045-2046.
[3]Zhou Xueliang(周学良).Handbook of Fine Chemical Products—Medicine(精细化工产品手册:药物)[M].Beijing: Chemical Industry Press, 2003.
[4]Chubachi A, Wakui H, Asakura K I, Nishimura S, Nakamoto Y, Miura A B.Acute renal failure following hypokalemic rhabdomyolysis due to chronic glycyrrhizic acid administration [J].Internal Medicine (Tokyo, Japan), 1992, 31(5): 708-711.
[5]Krausse R, Bielenberg J, Blaschek W, Ullmann U.In vitro anti-Helicobacter pylori activity of Extractum liquiritiae, glycyrrhizin and its metabolites [J].Journal of Antimicrobial Chemotherapy, 2004, 54(1): 243-246.
[6]Matsui S, Matsumoto H, Sonoda Y, Ando K, Aizu-Yokota E, Sato T, Kasahara T.Glycyrrhizin and related compounds down-regulate production of inflammatory chemokines IL-8 and eotaxin 1 in a human lung fibroblast cell line [J].International Immunopharmacology, 2004, 4(13): 1633-1644.
[7]Feng S, Li C, Xu X, Wang X.Screening strains for directedbiosynthesis of β-d-mono-glucuronide-glycyrrhizin and kinetics of enzyme production [J].Journal of Molecular Catalysis B: Enzymatic, 2006, 43(1-4): 63-67.
[8]Michikawa M, Ichinose H, Momma M, Biely P, Jongkees S, Yoshida M, Kotake T, Tsumuraya Y, Withers S G, Fujimoto Z.Structural and biochemical characterization of glycoside hydrolase family 79 β-glucuronidase from Acidobacterium capsulatum [J].Journal of Biological Chemistry, 2012, 287(17): 14069-14077.
[9]Cantarel B L, Coutinho P M, Rancurel C, Bernard T, Lombard V, Henrissat B.The carbohydrate-active enzymes database (CAZy): an expert resource for glycogenomics [J].Nucleic Acids Research, 2009, 37(suppl.1): D233-D238.
[10]Sierks M R, Svensson B.Protein engineering of the relative specificity of glucoamylase from Aspergillus awamori based on sequence similarities between starch-degrading enzymes [J].Protein Engineering, 1994, 7(12): 1479-1484.
[11]Siegel J B, Zanghellini A, Lovick H M, Kiss G, Lambert A R, Clair J L S, Gallaher J L, Hilvert D, Gelb M H, Stoddard B L.Computational design of an enzyme catalyst for a stereoselective bimolecular Diels-Alder reaction [J].Science, 2010, 329(5989): 309-313.
[12]Röthlisberger D, Khersonsky O, Wollacott A M, Jiang L, DeChancie J, Betker J, Gallaher J L, Althoff E A, Zanghellini A, Dym O.Kemp elimination catalysts by computational enzyme design [J].Nature, 2008, 453(7192): 190-195.
[13]Jiang L, Althoff E A, Clemente F R, Doyle L, Röthlisberger D, Zanghellini A, Gallaher J L, Betker J L, Tanaka F, Barbas C F.De novo computational design of retro-aldol enzymes [J].Science, 2008, 319(5868): 1387-1391.
[14]Gribenko A V, Patel M M, Liu J, McCallum S A, Wang C, Makhatadze G I.Rational stabilization of enzymes by computational redesign of surface charge-charge interactions [J].Proceedings of the National Academy of Sciences, 2009, 106(8): 2601-2606.
[15]Williams D V, Barua B, Andersen N H.Hyperstable miniproteins: additive effects of D- and L-Ala mutations [J].Organic & Biomolecular Chemistry, 2008, 6(23): 4287-4289.
[16]Song Zhanke(宋占科),Wang Xiaoyan(王小艳),Chen Guoqiang(陈国强),Li Chun(李春).Cloning and prokaryotic expression of β-glucuronidase from Penicillium purpurogenum Li-3 [J].Journal of Chemical Industry and Engineering(China)(化工学报), 2008, 59(12): 3101-3106.
[17]Arnold K, Bordoli L, Kopp J, Schwede T.The SWISS-MODEL workspace: a web-based environment for protein structure homology modelling [J].Bioinformatics, 2006, 22(2): 195-201.
[18]Biasini M, Bienert S, Waterhouse A, Arnold K, Studer G, Schmidt T, Kiefer F, Cassarino T G, Bertoni M, Bordoli L, et al.SWISS-MODEL: modelling protein tertiary and quaternary structure using evolutionary information [J].Nucleic Acids Research, 2014, 42(Web Server issue): W252-258.
[19]Guex N, Peitsch M C, Schwede T.Automated comparative protein structure modeling with SWISS-MODEL and Swiss-PdbViewer: a historical perspective [J].Electrophoresis, 2009, 30 (suppl.1): S162-173.
[20]Kiefer F, Arnold K, Kunzli M, Bordoli L, Schwede T.The SWISS-MODEL repository and associated resources [J].Nucleic Acids Research, 2009, 37(Database issue): D387-392.
[21]Song Zhanke( 宋占科).Overexpression, purification and characterization of recombinant β-glucuronidase from Penicillium purpurogenum Li-3 [D].Beijing: Beijing Institute of Technology, 2008.
[22]Dias F M, Vincent F, Pell G, Prates J A, Centeno M S, Tailford L E, Ferreira L M, Fontes C M, Davies G J, Gilbert H J.Insights into the molecular determinants of substrate specificity in glycoside hydrolase family 5 revealed by the crystal structure and kinetics of Cellvibrio mixtus mannosidase 5A [J].Journal of Biological Chemistry, 2004, 279(24): 25517-25526.
[23]Islam M R, Tomatsu S, Shah G N, Grubb J H, Jain S, Sly W S.Active site residues of human beta-glucuronidase.Evidence for Glu(540) as the nucleophile and Glu(451) as the acid-base residue [J].Journal of Biological Chemistry, 1999, 274(33): 23451-23455.
[24]Henrissat B, Callebaut I, Fabrega S, Lehn P, Mornon J P, Davies G.Conserved catalytic machinery and the prediction of a common fold for several families of glycosyl hydrolases [J].Proceedings of the National Academy of Sciences, 1995, 92(15): 7090-7094.
[25]Goihberg E, Dym O, Tel-Or S, Levin I, Peretz M, Burstein Y.A single proline substitution is critical for the thermostabilization of Clostridium beijerinckii alcohol dehydrogenase [J].Proteins: Structure, Function, and Bioinformatics, 2007, 66(1): 196-204.
[26]Takano K, Higashi R, Okada J, Mukaiyama A, Tadokoro T, Koga Y, Kanaya S.Proline effect on the thermostability and slow unfolding of a hyperthermophilic protein [J].Journal of Biochemistry, 2009, 145(1): 79-85.
[27]Prajapati R S, Das M, Sreeramulu S, Sirajuddin M, Srinivasan S, Krishnamurthy V, Ranjani R, Ramakrishnan C, Varadarajan R.Thermodynamic effects of proline introduction on protein stability [J].Proteins: Structure, Function, and Bioinformatics, 2007, 66(2): 480-491.
[28]Pace C N, Fu H, Fryar K L, Landua J, Trevino S R, Shirley B A, Hendricks M M, Iimura S, Gajiwala K, Scholtz J M.Contribution of hydrophobic interactions to protein stability [J].Journal of Molecular Biology, 2011, 408(3): 514-528.
[29]Liu L, Deng Z, Yang H, Li J, Shin H D, Chen R R, Du G, Chen J.In silico rational design and systems engineering of disulfide bridges in the catalytic domain of an alkaline α-amylase from Alkalimonas amylolytica to improve thermostability [J].Applied and Environmental Microbiology, 2014, 80(3): 798-807.
[30]Kim M S, Lei X G.Enhancing thermostability of Escherichia coli phytase AppA2 by error-prone PCR [J].Applied Microbiology and Biotechnology, 2008, 79(1): 69-75.
[31]Fang Z, Zhou P, Chang F, Yin Q, Fang W, Yuan J, Zhang X, Xiao Y.Structure-based rational design to enhance the solubility and thermostability of a bacterial laccase Lac15 [J].PloS One, 2014, 9(7): e102423.
[32]Nakazawa H, Okada K, Onodera T, Ogasawara W, Okada H, Morikawa Y.Directed evolution of endoglucanase III (Cel12A) from Trichoderma reesei [J].Applied Microbiology and Biotechnology, 2009, 83(4): 649-657.

