分子模拟指导化学修饰对有机相中脂肪酶的性能强化
郭丽,徐刚,吴坚平,杨立荣
(浙江大学化学工程与生物工程学系生物工程研究所,浙江 杭州 310027)
脂肪酶对底物具有良好的立体选择性,并且可以适应包括疏水性有机溶剂在内的非天然反应环 境[1-4],因此成为应用最为广泛的生物催化剂之一。长期以来,提高脂肪酶在有机溶剂中的催化性能是酶学研究的热点。Kazlauskas 等[5]提出了脂肪酶对手性底物立体识别的分子机理,认为脂肪酶的催化中心附近区域是一个容纳底物的空腔结构,这一空腔可以看作是由大小不同的两个子空腔组成,即大、小口袋。当手性底物靠近酶的催化中心时,一种构型的手性中心上较大的基团位于“大口袋”中、较小基团位于“小口袋”中,则该构型的底物受到的空间位阻较小,因而容易与催化中心结合而被催化;相反,如果较大基团位于小口袋中,受到的空间位阻较大,因此与催化中心结合较为困难而不易被催化。脂肪酶正是依靠这种催化中心空腔大小口袋与底物手性基团的性状匹配来识别底物的手性构型的[6-7]。
根据上述机理,很多研究者成功地通过基因修饰[8-12]或者化学修饰[13-16]等方法缩小脂肪酶的“小口袋”,使其不能容纳原本勉强能够容纳的手性基团,从而进一步提高脂肪酶的立体选择性。如 Chen 等[17]通过基因突变在产碱假单胞菌脂肪酶(Pseudomonas alcaligenes lipase,PaL)活性中心附近引入一个 Cys272,并使用 5,5-二硫代双(2-硝基苯甲酸) 对半胱氨酸进行特异性修饰,将其活性中心的小口袋减小,从而将该酶催化水解 L-薄荷醇丙酸酯的选择性提高了3 倍。Tuomi 等[18]使用四甲基甲烷硝基化 PcL 的酪氨酸,将其活性中心的小口袋减小,从而将该酶催化水解 2-苯氧基-1-丙醇乙酸酯的选择性由 E = 17 提高到了 36。
然而,上述修饰改造在提高选择性的同时,却严重降低了脂肪酶的催化活力(如 Chen 等的修饰脂肪酶残余酶活仅有原始酶的 1/35)。此外,尽管脂肪酶能够很好的适应疏水有机溶剂,现有关于脂肪酶的选择性强化,却主要是针对水溶液中的反应体系。对有机溶剂中脂肪酶立体选择性的计算模拟,以及在此指导下的理性改造鲜有报道。本文基于分子动力学(molecular dynamics,MD)技术,开发了模拟有机溶剂中脂肪酶的立体识别的新方法。并运用该方法,成功将 Kazlauskas 规则推广到疏水有机溶剂体系中。以PcL 在正己烷中催化多种手性仲醇的选择性转酯化为模型,在模拟计算指导下对PcL 的催化空腔进行特异性化学修饰,在不损失酶活的条件下,从而成功提高了其对多种手性仲醇的选择性,最好E 值提高到了 48.1。
1 实验部分
1.1 实验材料
洋葱假单胞菌脂肪酶(Pseudomonas cepacia lipase,缩写为PcL,商品名Lipase PS)购自Amano Enzyme Inc.。胰蛋白酶(trypsin)购自北京 Promega 公司。手性仲醇,如仲丁醇、仲己醇、仲辛醇及其光学纯对映体标准品购自 Tokyo Chemical Industry Co. Ltd.。酰基供体如乙酸乙烯酯和化学修饰剂 N-乙酰咪唑购于 Sigma-Aldrich Inc.。其余试剂均为分析纯。
1.2 分子模拟
首先在 Discovery Studio 3.0 软件中处理PcL蛋白结构(PDB:2NW6)[19]。使用Calculate Protein Ionization and Residue pK 工具设定蛋白表面可电离氨基酸的质子化状态,并补全晶体结构中缺失的氢原子,删除晶体结构中原有的配体小分子(POT612),保留全部结晶水(HOH615~893)和离子(CA613,NA614),再将催化中心三联体 Ser87-His286-Asp264中组氨酸的质子化状态设为 HIP,从而得到受体蛋白结构。使用 Discovery Studio 的 Sketching 工具生成底物(手性仲醇和酰基供体)同时与催化中心结合时的四面体过渡态结构,并使用 LigandFit 工具[20]将其对接到PcL 活性中心,再以共价单键连接底物羰基碳原子与PcL的催化中心 Ser87-Oγ。
将得到的酶-底物过渡态复合物导入到 AMBER11 软件[21]中,给复合物周围加上六面体形的TIP3P 水溶剂盒子[22],溶剂边界到蛋白表面最远距离 10Å(1Å=0.1nm),并以Na+和Cl-平衡蛋白所带电荷,进行2.5ns 恒温恒压MD 以获得接近真实情况的酶-底物复合物构象。接下来进行5ns 正己烷中的MD 模拟。从水溶液MD 结果中剔除后来加入的TIP3P 溶剂水,将提取出的带有结晶水、离子的复合物导入Packmol 软件,在其周围加上六面体形的正己烷溶剂盒子,溶剂边界到蛋白表面最远距离10Å,再导入到AMBER11 软件中进行5ns 有机相MD 模拟。模拟结束后使用AMBER PTRAJ 工具分析结果。
1.3 手性仲醇的动力学拆分
手性仲醇(100μmol)、酰基供体(100μmol)、正己烷(1mL)与PcL 酶粉(10mg,约含 0.2mg 蛋白)充分混合,200 r/min 搅拌下 25℃温浴一段时间后离心取上清液。反应式如图式1 所示。反应液用气相色谱分析[Hewlett-Packard 1890 型气相色谱仪,Varian CP-Cyclodextrin-β-2,3,6-M-19 毛细管手性柱(50m × 0.25mm × 0.25μm)]。参照文献[23],计算反应的对映体选择性:E = ln[1 - c(1 + eep)]/ln[1 - c(1 - eep)],其中eep为产物对映体过剩值,c 为底物转化率,eep= {[(R)-酯] - [(S)-酯] } / { [(R)-酯] + [(S)-酯] },方括号表示浓度。
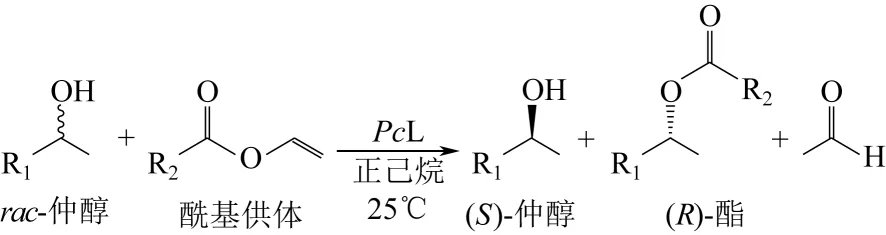
图1 PcL 催化拆分仲醇
1.4 PcL 的化学修饰
N-乙酰咪唑(N-acetylimidazole,NAI) 修饰酪氨酸残基(Tyr)的方法参照文献[24],10mg 脂肪酶PcL溶于1mL 磷酸缓冲液中(50mmol/L,pH = 7.5),于2mL 平底离心管中配成酶液,加入5mg NAI,并加入200μL 界面激活剂正己烷,形成两相界面,激活脂肪酶打开“盖子”,从而利于口袋内部氨基酸修饰[25]。冰浴下200r/min 搅拌反应3h,使用脱盐柱(HiTrap Desalting,GE Healthcare Corp.)将修饰酶液中的目标蛋白与各种盐离子和反应后的修饰剂 NAI 进行分离,最终溶解在去离子水中,立即置于 -80℃冰箱预冻 6~8h,然后真空冷冻干燥。冷冻干燥方法参照文献[26],冻干完成后将样品取出,储存于 -20℃保存待用。PcL 的剩余水解活力对修饰时间作图得到修饰失活动力学曲线,水解酶活测定采用 p-NPP 法[27]。
1.5 修饰PcL 蛋白质谱分析
化学修饰的PcL 蛋白通过十二烷基磺酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)胶内酶切方法获取肽段[28],采用基质辅助激光解析电离飞行时间质谱(MALDI-TOF-MS)检测方法,在 UltraFleXtreme 质谱仪(Bruker Corp.)上进行肽段质谱分析,从而获得修饰位点的相关信息。本实验采取基质 α-氰-4-羟基肉桂酸(HCCA)对肽段进行质谱分析。PcL蛋白经胰蛋白酶(trypsin)酶解的肽谱由 ExPASy PeptideMass(http://web.expasy.org/peptide_mass/)在线工具检索产生。搜索时漏切数选择1 并显示质荷比 Z/A 大于 500 Da 的肽段序列。
2 结果与讨论
2.1 PcL 在正己烷中对手性仲醇的立体识别的模拟计算
分别对PcL 在正己烷中催化(R)-及(S)-2-丁醇、2-己醇、2-辛醇与乙酸乙烯酯的转酯化反应进行了MD 模拟。以(R)-2-丁醇与乙酸乙烯酯的转酯化为例,图2显示了2-丁醇与乙酸乙烯酯的乙酰基在PcL催化中心结合形成的过渡态构象。图中青色的部分为底物分子,其醇部较大的手性基团(乙基)朝向催化空腔开口一侧,其附近空间开阔,即为PcL 的“大口袋”,较小的手性基团(甲基)朝向催化空腔内侧,附近被构成空腔壁的氨基酸残基所包围,较为拥挤,即为“小口袋”。Tyr29残基位于底物的大小手性基团之间,是大小口袋的分界点。Tyr29的酚羟基朝向催化空腔内侧(即“小口袋”),在这一位置通过化学修饰连接合适的基团,会使“小口袋”更为拥挤,根据Kaslauskaz 规则,这将提高PcL 对底物的选择性。表1 列出了对Tyr29酚羟基进行乙酰化修饰的PcL 以及原始的PcL 催化一系列手性仲醇的酶-底物过渡态结合能的计算数据。对于一对手性底物,酶与两者的结合能差的绝对值越大,选择性越好。表1 数据显示,Tyr29-酚羟基乙酰化的PcL结合自由能差的绝对值明显大于原始PcL,表明这一修饰能够显著提高选择性。
2.2 对PcL 的 Tyr29 的化学修饰
NAI 是一种修饰酪氨酸酚羟基的特异性化学试剂。在水-疏水界面上用NAI 可以修饰PcL 靠近活性中心的Tyr 残基。如图3 所示,NAI 修饰PcL,修饰反应持续6hPcL 的本征活力(p-NPP 水解活力) 已降至最低,表明NAI 修饰影响到了PcL 的活性中心附近区域。将原始PcL 酶液和脱盐后的NAI 修饰PcL 酶液,进行SDS-PAGE 分析,如图4 所示。2 号泳道(原始PcL)和3 号泳道(修饰PcL)都仅在33 kDa 左右出现的单一条带,与文献报道的PcL 分子量一致[19]。这一电泳结果结合图3 的修饰-失活过程,表明PcL 分子上仅有少数Tyr 被修饰。
用特异性切开 N-端为 Arg/Lys 残基的胰蛋白酶水解PcL,可以得到特异性的定酶切肽指纹图谱,谱中的肽段分子量可以通过软件计算,通过MALDI-TOF-MS 蛋白质谱表征,也可以精确测定这些肽段的分子量。通过对比计算预测的肽谱和测得的肽谱中特定肽段的质量迁移,即可定位发生修饰反应的氨基酸残基。表2 给出了预测和测定的肽指纹图谱数据,可以看到10 号肽段的分子量相比理论值(9 号肽段质量)高出 42 Da,即乙酰基的分子量,而 Tyr29刚好位于这一肽段中,从而证明修饰特异性发生在 Tyr29位点。图4 是原始PcL 的 MALDI-TOF-MS 原始图谱,可以看到其中9 号肽段的分子量与表2 中的预测值一致,而图5 中9 号肽段的响应值几乎消失,10 号肽段则与9 号肽段相差乙酰基的分子量。对比图4 和图5,也证明了修饰发生在位于10 号肽段上的 Tyr29残基。

图2 2-丁醇与乙酸乙烯酯的乙酰基在PcL 催化中心结合形成的过渡态构象
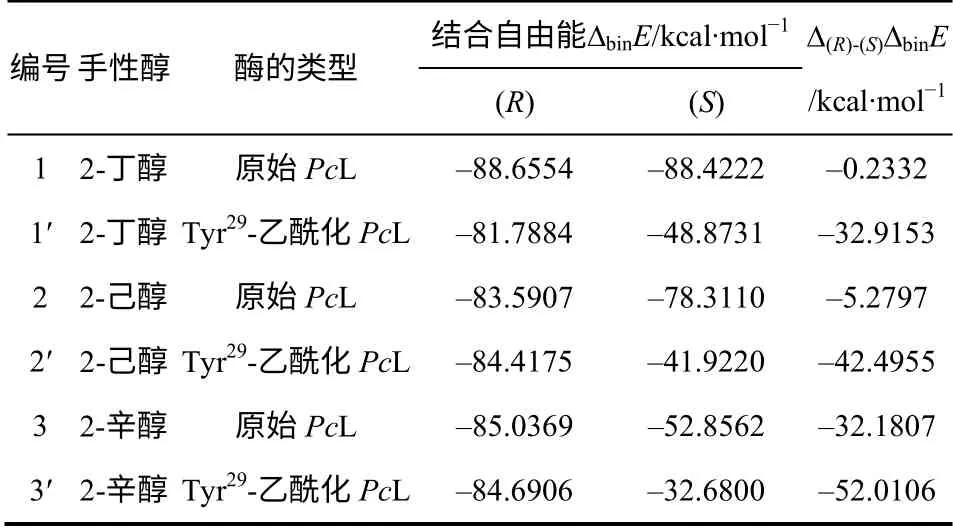
表1 分子动力学模拟计算的PcL 与不同手性醇过渡态复合物的结合能
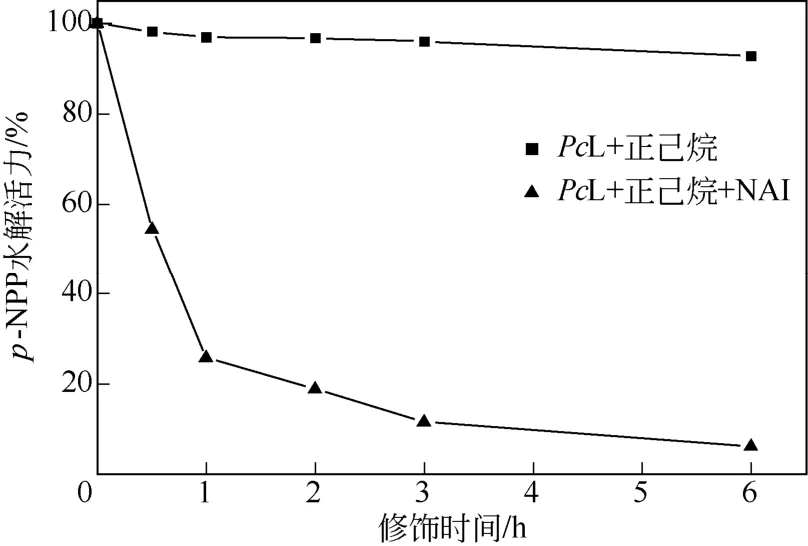
图3 NAI 修饰PcL 失活动力学

图4 脂肪酶PcL 蛋白的 SDS-PAGE 结果
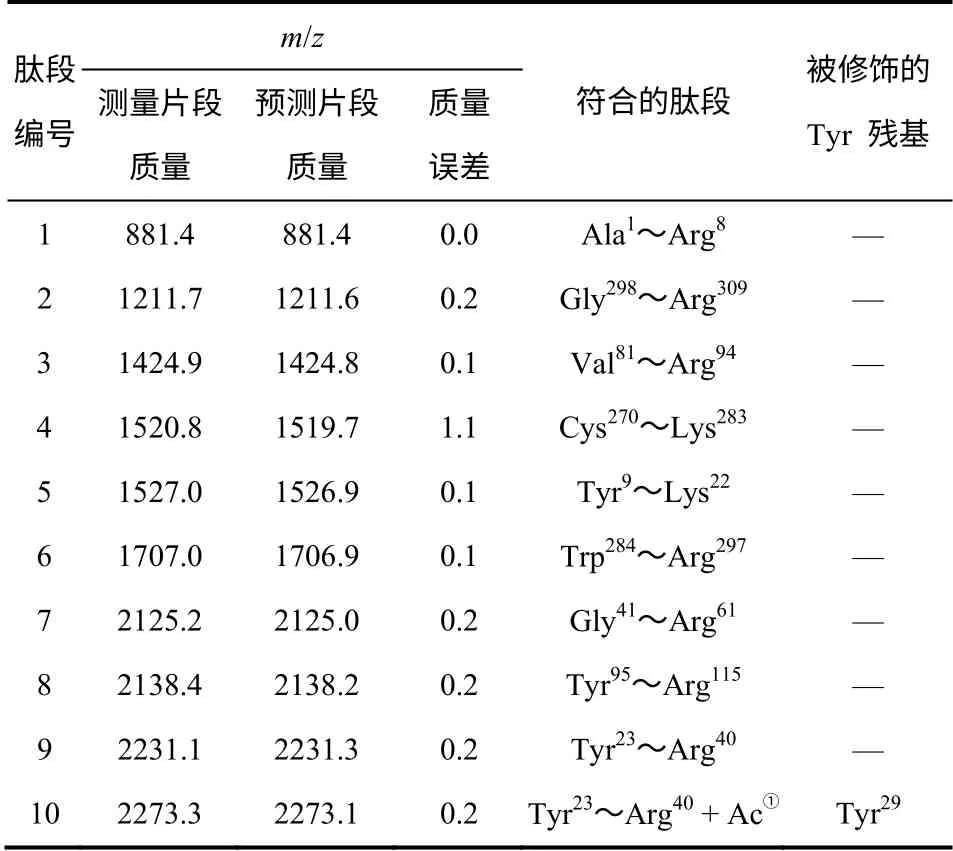
表2 NAI-修饰的PcL 的MALDI-TOF 质谱表征

图5 原始脂肪酶PcL 的胰蛋白酶酶切蛋白样品指纹图谱
2.3 修饰 Tyr29 对选择性的影响
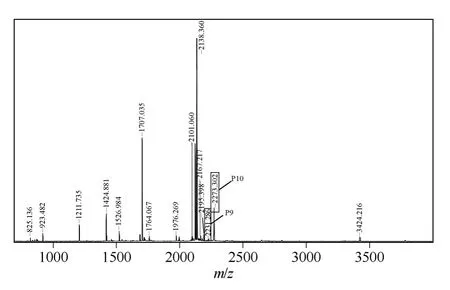
图6 NAI 修饰脂肪酶的胰蛋白酶酶切蛋白样品指纹图谱
前文表1 已经对PcL 在正己烷中识别手性底物进行了模拟计算,考察了PcL 催化3 种不同结构仲 醇的不对称转酯化,Tyr29-酚羟基乙酰化的PcL 结合自由能差的绝对值明显大于原始PcL,表明这一修饰能够显著提高拆分仲醇的选择性,见图6。进而对PcL 进行了 NAI 特异性修饰实验研究,对表3 中考察的反应而言,Tyr29-酚羟基乙酰化PcL 的选择性较原始PcL 均有明显提高,其中修饰PcL 催化 2-己醇与乙酸乙烯酯的转酯化反应的选择性提高最多,由原来的 E = 12.6 提高到 48.1,这与表1 的模拟计算是一致的。此外,在选择性提高的同时,修饰PcL 催化这些仲醇底物的活力还有小幅提高。
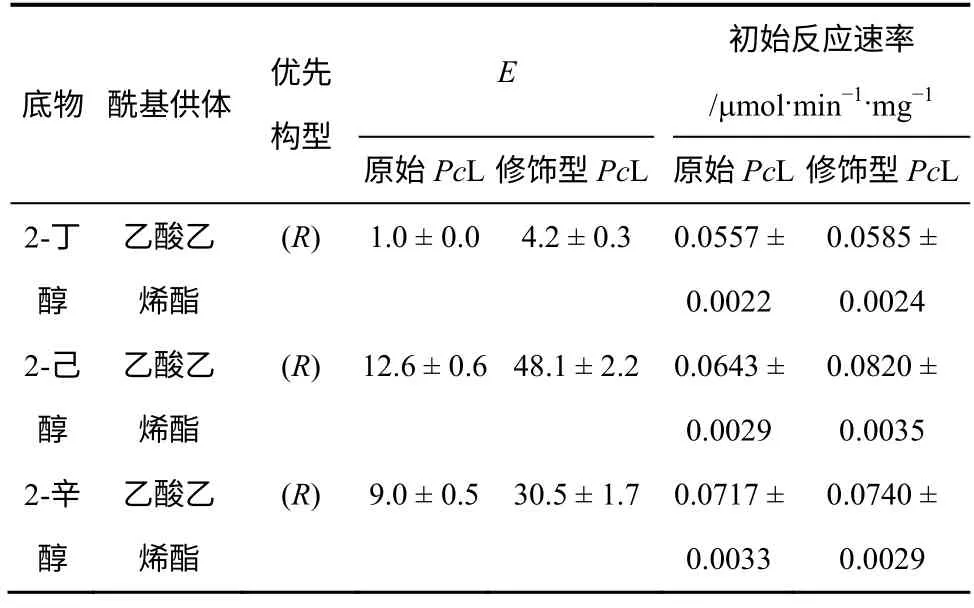
表3 对PcL 氨基酸残基侧链的特异性化学修饰
3 结 论
根据 Kazlauskas 规则,通过有机相中MD 模拟的新方法,提出了对PcL 催化空腔中的关键残基进行特异性修饰来提高酶的立体选择性的研究策略。并通过MALDI-TOF-MS 表征和PcL 催化3 种不对称转酯化反应的实验数据证明这一修饰的有效性。这一方法成功地在不损失酶的催化活力的情况下提高了其立体选择性,并且其研究方法借助独立于分子生物学之外的化学修饰方法,结合蛋白质谱解析和分子动力学模拟等现代技术手段,开创了一条崭新的灵活、高效、可靠的酶构效关系研究。
[1] 崔丽娟,徐刚,孟枭,等. 多元电解质对脂肪酶有机相拆分炔戊醇的激活[J]. 化工进展,2014,33(8):2150-2154.
[2] 戴晓庭,孟枭,徐刚,等. 酰基供体对动态动力学拆分1-四氢萘胺的影响[J]. 化工进展,2014,33(9):2421-2424.
[3] Bajaj A,Lohan P,Jha P N,et al. Biodiesel production through lipase catalyzed transesterification:An overview[J]. J. Mol. Catal. B:Enzym.,2010,62(1):9-14.
[4] Fan Y X,Qian J Q. Lipase catalysis in ionic liquids/supercritical carbon dioxide and its applications[J]. J. Mol. Catal. B:Enzym.,2010,66(1):1-7.
[5] Laane C,Boeren S,Vos K,et al. Rules for optimization of biocatalysis in organic solvents[J]. Biotechnol. Bioeng.,1987,30(1):81-87.
[6] Yadav G D,Lathi P S. Kinetics and mechanism of synthesis of butyl isobutyrate over immobilized lipases[J]. Biochem. Eng. J.,2003,16(3):245-252.
[7] Lemke K,Lemke M,Theil F. A three-dimension predictive active site model for lipase from Pseudomonas cepacia[J]. J. Org. Chem.,1997,62(18):6268-6273.
[8] Fernandez L R. Lipase from Thermomyces lanuginosus:Uses and prospects as an industrial biocatalyst[J]. J. Mol. Catal. B:Enzym.,2010,62(3):197-212.
[9] Contesini F J,Lopes D B,Macedo G A,et al. Aspergillus sp. lipase:Potential biocatalyst for industrial use[J]. J. Mol. Catal. B:Enzym.,2010,67(3):163-171.
[10] Li N,Zong M H. Lipases from the genus Penicillium:Production,purification,characterization and applications[J]. J. Mol. Catal. B:Enzym.,2010,66(1):43-54.
[11] Rodrigues R C,Fernandez L R. Lipase from Rhizomucor miehei as a biocatalyst in fats and oils modification[J]. J. Mol. Catal. B:Enzym.,2010,66(1):15-32.
[12] Rodrigues R C,Fernandez L R. Lipase from Rhizomucor miehei as an industrial biocatalyst in chemical process[J]. J. Mol. Catal. B:Enzym.,2010,64(1):1-22.
[13] Cabrera Z,Fernandez L G,Fernandez L R,et al. Enhancement of Novozym-435 catalytic properties by physical or chemical modification[J]. Proc. Biochem.,2009,44:226-231.
[14] Palomo J M,Fernandez L G,Guisan J M,et al. Modulation of immobilized lipase enantioselectivity via chemical amination[J]. Adv. Synth. Catal.,2007,349(7):1119-1127.
[15] Rodrigues R C,Berenguer M A,Fernandez L R. Coupling chemical modification and immobilization to improve the catalytic performance of enzymes[J]. Adv. Synth. Catal.,2011,353(13):2216-2238.
[16] Mateo C,Palomo J M,Fernandez L G,et al. Improvement of enzyme activity,stability and selectivity via immobilization techniques[J]. Enzym Microb. Technol.,2007,40(6):1451-1463.
[17] Chen H,Wu J P,Yang L R,et al. A combination of site-directed mutagenesis and chemical modification to improve diastereopreference of Pseudomonas alcaligenes lipase[J]. Biochim. Biophys Acta,2013,1834(12):2494-2501.
[18] Tuomi W V,Kazlauskas R J. Molecular basis for enantioselectivity of lipase from Pseudomonas cepacia toward primary alcohols. Modeling,kinetics,and chemical modification of Tyr29 to increase or decrease enantioselectivity[J]. J. Org. Chem.,1999,64(8):2638-2647.
[19] Kim K K,Song H K,Shin D H,et al. The crystal structure of a triacylglycerol lipase from Pseudomonas cepacia reveals a highly open conformation in the absence of a bound inhibitor[J]. Sturcture,1997,5(2):173-185.
[20] Peretz A,Pell L,Gofman Y,et al. Targeting the voltage sensor of Kv7.2 voltage-gated K+channels with a new gating-modifier[J]. PANS,2010,107(35): 15637-15642.
[21] Case D A,Darden T A,Cheatham T E ,et al. AMBER 11[C]. San Francisco:University of California,2011,
[22] Jorgensen W L,Chandrasekhar J,Madura J D,et al. Comparison of simple potential functions for simulating liquid water[J]. J. Chem. Phys.,1983,79(2):926-935.
[23] Chen C S,Fujimoto Y,Girdaukas G,et al. Quantitative analyses of biochemical kinetic resolutions of enantiomers[J]. J. Am. Chem. Soc.,1982,104(25):7294-7299.
[24] Furth A J,Hope D B. Studies on the chemical modification of the tyrosine residue in bovine neurophysin-II[J]. Biochem. J.,1970,116:545-553.
[25] Norin M,Olsen O,Svendsen A,et al. Theoretical studies of Rhizomucor miehei lipase activation[J]. Protein Eng.,1993,6(8):855-863.
[26] Ru M T,Dordick J S,Reimer J A,et al. Optimizing the salt-induced activation of enzymes in organic solvents:Effects of lyophilization time and water content[J]. Biotechnol. Bioeng.,1999,63(2):233-241.
[27] Gupta N,Rathi P,Gupta R. Simplified para-nitrophenyl palmitate assay for lipases and esterases[J]. J. Med. Chem.,2003,46(4):499-511.
[28] Zehl M,Lescic L,Abramic M,et al. Characterization of covalently inhibited extracellular lipase from Streptomyces rimosus by matrix-assisted laser desorption/ionization time-of-flight and matrix-assisted laser desorption/ionization quadrupole ion trap reflectron time-of-flight mass spectrometry:Localization of the active site serine[J]. J. Mass Spectrom.,2004,39(12):1474-1483.

