GITRL基因SNP-rs2236876分型方法的比较
吴晶晶,仝佳,陈娟,袁靖,田洁,汤新逸,王胜军
(江苏大学医学院检验医学研究所,江苏镇江212013)
GITRL基因SNP-rs2236876分型方法的比较
吴晶晶,仝佳,陈娟,袁靖,田洁,汤新逸,王胜军
(江苏大学医学院检验医学研究所,江苏镇江212013)
目的:对3种单核苷酸多态(single nucleotide polymorphism,SNP)分型方法进行比较,选择准确有效的SNP分型方法对GITRL SNP-rs2236876A/G进行基因分型。方法:采用Taqman-MGB探针法,限制性片段长度多态性聚合酶链式反应(cleaved amplification polymorphism sequence-tagged sites,PCR-RFLP)法和直接测序法对GITRL SNP-rs2236876A/G进行分型并比较。结果:PCR-RLFP法分型结果直接通过电泳图可见,适合少量的实验对象;Taqman-MGB探针法通过不同的荧光区分不同的基因型,适合大量的实验对象;直接测序法交由测序公司测定,样本数量浮动范围较大,费用较高。结论:Taqman-MGB探针法适用于GITRL基因rs2236876A/G基因分型,PCR-RFLP法适用于基因分型结果的验证,直接测序法适用于鉴定有争议的PCR-RFLP法的验证结果。
Taqman-MGB探针;单核苷酸多态分型;PCR-RFLP;GITRL
快速经济地对糖皮质激素诱导的肿瘤坏死因子受体配体(GITRL)基因SNP-rs2236876进行分型,对于实验室研究SNP而言非常重要。目前,SNP分型的方法有多种,如探针法,限制性片段长度多态性聚合酶链式反应(cleaved amplification polymorphism sequence-tagged sites,PCR-RFLP)等,为了配合研究SNP的大量样本分析,我们需要一种经济、精确、有效的SNP分型方法。
1 材料和方法
1.1 材料和仪器
1.1.1 材料 常规PCR试剂(TaKaRa公司),PCRRFLP所用TaiⅠ限制性内切酶(TaKaRa公司),Taqman-MGB探针所用的组分(上海闪晶)。
1.1.2 仪器 定量PCR扩增仪CFX 96 Real Time system(Bio-Rad公司),凝胶成像及分析系统(北京赛智公司),电泳仪(Bio-Rad公司),低温台式高速离心机(Eppendorf公司)。
1.2 方法
1.2.1 确立用于比较实验方法的标准品 提取10例不同个体的外周血基因组DNA,提取方法参考文献[1]。对GITRL基因SNP-rs2236876A/G进行引物设计,设计的原则是将rs2236876A/G突变的位点包含在PCR产物内,并保证与引物上下端有一定的距离。PCR-RFLP所设计的引物序列见表1。用引物对所提取的10例基因组DNA进行常规PCR,产物交由上海英潍捷基公司测序,分析PCR结果,选择rs2236876A/G 3种不同基因型(AA,AG,GG)的样本作为标准品进行下一步分析。
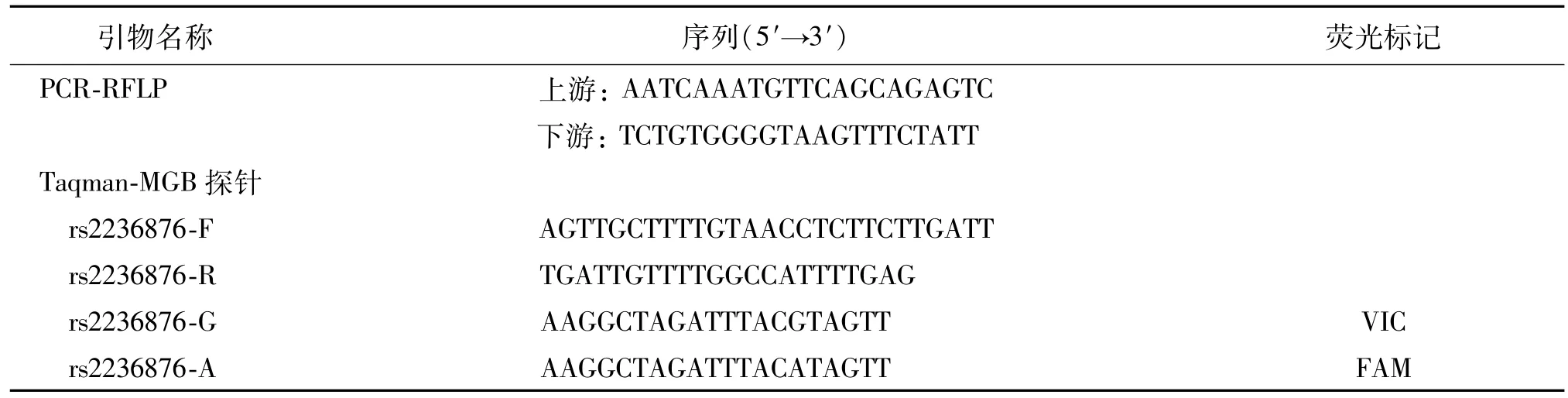
表1 PCR-RFLP与Taqman-MGB探针的引物序列
1.2.2 基因分型方法
1.2.2.1 PCR-RFLP法[2]将选出的3例不同基因型的标准品进行常规PCR,针对rs2236876A/G选择能识别于该位点的限制性内切酶TaiⅠ,然后对PCR产物进行限制性内切酶反应,酶切结果行琼脂糖凝胶电泳,然后EB染色,最后在紫外凝胶成像仪中观察酶切结果,根据酶切结果的不同进行基因分型。具体反应体系如下:10×PCR反应缓冲液2 μL、dNTP 1.6μL、上游引物0.4μL、下游引物0.4 μL、DNA聚合酶0.2μL。
1.2.2.2 Taqman-MGB探针法[3]设计Taqman-MGB探针法所需的引物及探针,引物和探针序列见表1。在荧光定量PCR仪上选择两种荧光(FAM,VIC)进行基因分型。具体反应体系如下:双蒸水8 μL、TaqMan®SNP探针0.5μL、TaqMan®SNP引物0.5μL、TaqMan®Genotyping Master Mix 10μL、模板1.0μL。
1.2.2.3 直接测序法 将PCR产物直接送至上海英潍捷基公司进行直接测序,获得结果后,通过Chromas软件进行测序结果的分析。将分析结果在NCBI数据库中进行比对,确认基因型。
2 结果
2.1 PCR-RFLP法进行基因分型的结果
由图1可见3种不同的酶切结果,rs2236876GG样本被完全酶切,rs2236876AG样本部分被酶切,而rs2236876AA样本完全未被酶切,与预期结果相符。

图1 PCR-RFLP法对GITRL基因rs2236876进行基因分型的结果
2.2 Taqman-MGB探针法进行基因分型的分型图谱
根据FAM和VIC两种不同的荧光与不同基因型的结合分析,方块点所代表的基因型是rs2236876-GG,三角形点所代表的基因型是rs2236876-AG,圆形点所代表的基因型是rs2236876-AA,与预期结果相符合。见图2。

图2 Taqman-MGB探针法对GITRL基因rs2236876进行基因分型的荧光分型图
2.3 直接测序法进行基因分型的测序结果
从图3中序列的峰图谱可看出,在rs2236876A/ G位点处可见3种不同的峰图谱,rs2236876AG所对应的峰图谱中可见两种不同颜色的混合峰,而rs2236876GG和rs2236876AA分别对应的是不同颜色的单一峰,与预期结果相符合。

图3 直接测序法进行基因分型的测序结果分析图谱
2.4 3种基因分型方法的费用和耗时比较
预估计实验所要检测约400个样本的SNP,因此需要考虑费用和时间各方面因素,故需选择一种经济、快捷、准确性较高的基因分型方法。综合考虑,对3种不同的基因分型方法从费用和耗时的角度进行了比较,见表2。由表可知,PCR-RFLP法进行基因分型费用低,但耗时较长;TaqMan-MGB法费用较高,但耗时很短;直接测序法费用较高,耗时较长。
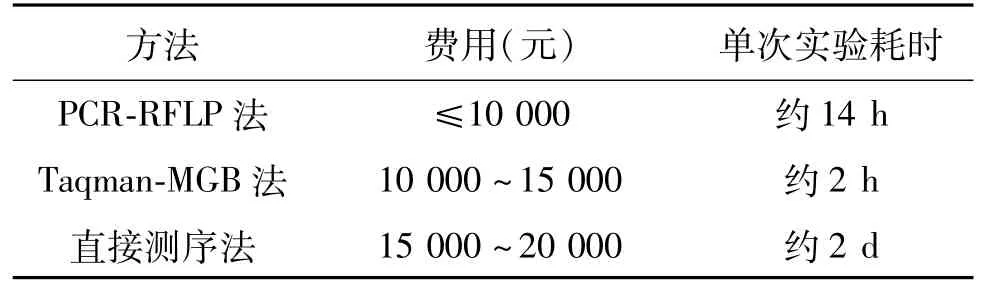
表2 不同基因分型方法的费用和时间比较
3 讨论
目前对基因多态性的研究越来越多,临床上多种疾病都与SNP有关。多数实验室都配有基因直接测序的仪器,而对无直接测序仪器的实验室而言,若要研究SNP,则需要找到一种高效、经济的基因分型方法。
作为第3代遗传标记的SNPs是基因组变异最常见的一种形式,大规模SNPs的鉴定和SNPs公共数据库的建立大大促进了复杂性疾病相关基因研究的发展[4]。研究证实,SNPs与消化道肿瘤有密切的关系[5]。此外,对于GITRL与肿瘤关系的研究亦是热点,考虑到GITRL与疾病有密切的关系[6],本实验室拟从GITRL基因组学的角度分析其与疾病间的关系,从而为疾病的鉴定及治疗提供可能的依据[7]。
近几年来,GITR及其配体GITRL在抗肿瘤免疫中的作用引起了学者的广泛关注,尤其是通过调控GITR/GITRL系统作为一种治疗肿瘤的新途径的理念更是成为关注焦点。研究报道,GITRL基因SNP-rs2236876与过敏性哮喘有关[8]。本实验室针对GITRL基因rs2236876A/G进行基因分型方法的比较,根据实验室的情况,选择了Taqman-MGB探针法、PCR-RFLP法、直接测序法进行基因分型比较。我们发现采用PCR-RFLP法进行基因分型费用低,但耗时较长,且步骤较为烦琐;此外,虽然基因型能直接在琼脂糖凝胶电泳中鉴别出来,但易受客观因素(如PCR、电泳等)影响;Taqman-MGB法费用较高,但耗时很短,可在1 d之内检测出500例样本的SNP;直接测序法需要交由专业的公司测定,费用较高,耗时较长,得到结果后仍要做峰状图谱的分析,在本实验中不适用。
因此,我们选择了Taqman-MGB探针法作为GITRL基因rs2236876A/G基因分型的方法,将PCR-RFLP作为基因分型结果的验证方法,另外将直接测序法作为有争议的PCR-RFLP结果的确定方法。
[1] 赵嘉慧,张华屏.外周血DNA提取方法的比较及改良[J].山西医科大学学报,2006,37(1):12-13.
[2] Miao YZ,Xu H,Fei BJ,et al.PCR-RFLP analysis of the diversity of phytate-degrading bacteria in the Tibetan Plateau[J].Can JMicrobiol,2013,59(4):245-251.
[3] Huang J,Gao C,Ding X,et al.MGB-based one-step multiplex real-time PCR method for rapid detection of HPV[J].Cancer Biomark,2012,12(3):107-113.
[4] Baeten D,Van Damme N,Van den Bosch F,etal.Impaired Th1 cytokine production in spondyloarthropathy is restored by anti-TNFα[J].Ann Rheum Dis,2001,60(8):750-755.
[5] Demontis D,Pertoldi C,Loeschcke V,et al.Efficiency of selection,asmeasured by single nucleotide polymorphism variation,is dependent on inbreeding rate in Drosophila melanogaster[J].Mol Ecol,2009,18(22):4551-4563.
[6] Mosbruger TL,Duggal P,Goedert JJ,etal.Large-scale candidate gene analysis of spontaneous clearance of hepatitis C virus[J].J Infect Dis,2010,201(9):1371-1380.
[7] Chattopadhyay K,Ramagopal UA,Mukhopadhaya A,et al.Assembly and structural properties of glucocorticoidinduced TNF receptor ligand:implications for function[J].Proc Natl Acad Sci U S A,2007,104(49):19452-19457.
[8] Bottema RW,Postma DS,Reijmerink NE,et al.Interaction of T-cell and antigen presenting cell co-stimulatory genes in childhood IgE[J].Eur Respir J,2010,35(1):54-63.
Com parison ofmethod for GITRL SNP-rs2236876 genotyping
WU Jing-jing,TONG Jia,CHEN Juan,YUAN Jing,TIAN Jie,TANG Xin-yi,WANG Sheng-jun
(Institute of Laboratory Medicine,School of Medicine,Jiangsu University,Zhenjiang Jiangsu 212013,China)
Objective:Comparing three single nucleotide polymorphism(SNP)genotypingmethods,to choose an accurate and effective SNP genotypingmethod for rs2236876A/G of GITRL gene.M ethodsSNP genotyping for rs2236876A/G of GITRL were performed and compared by Taqman-MGB probe,the PCRRLFP and direct sequencing.Results:PCR-RLFP showed the genotyping result by electrophoresis,which was suitable for a small number of experimental subjects.Taqman-MGB probe showed the genotyping result by fluorescence,which was suitable for a large number of experimental subjects.Direct sequencingmethod was operated by the sequencing company which was expensive.Conclusion:For rs2236876A/G of GITRL gene,Taqman-MGB probemethod could be chosen for genotyping,PCR-RFLPmethod confirmed the genotyping results and,at the same time,the direct sequencing finally validated the result of PCR-RFLP.
Taqman-MGB probe;SNP genotyping;PCR-RFLP;GITRL
R393
A
1671-7783(2015)04-0308-03
10.13312/j.issn.1671-7783.y130120
国家自然科学基金资助项目(81072453,31170849,30972748);江苏省自然科学基金资助项目(BK2011472);江苏省普通高校研究生科研创新计划项目(CXLX11_0608);江苏省卫生厅医学科研基金资助项目(Z201313);江苏省“青蓝工程”项目;江苏大学“拔尖人才工程”和高级人才基金项目
吴晶晶(1988—),女,硕士研究生;王胜军(通讯作者),教授,博士生导师,E-mail:sjwjs@mail.ujs.edu.cn
2013-06-04 [编辑] 刘星星

