橡胶树Lhcb2基因的克隆与表达特性分析
邹 智 王丹华 戴雪梅 朱家立 谢贵水
(中国热带农业科学院橡胶研究所,农业部儋州热带作物科学观测试验站,海南儋州571737)
橡胶树Lhcb2基因的克隆与表达特性分析
邹 智 王丹华 戴雪梅 朱家立 谢贵水
(中国热带农业科学院橡胶研究所,农业部儋州热带作物科学观测试验站,海南儋州571737)
基于从基因组中获得的基因序列,应用RT-PCR技术从橡胶树叶片中分离得到1条849 bp的cDNA;该cDNA包含1个798 bp的ORF,编码265个氨基酸,理论分子量为28.66 kD,等电点为5.42,属于叶绿体定位的类囊体膜蛋白;蛋白含有3个跨膜螺旋,2个C端螺旋,1个保守的捕光叶绿素a/b结合蛋白结构域,1个三聚化基序,以及多个类胡萝卜素和叶绿素结合位点,根据进化关系可归为LHCB2亚家族;蛋白与蓖麻、野草莓、可可、葡萄和棉花中同源蛋白的一致性均接近95%,在进化上显示出高度的保守性,将基因命名为HbLhcb2.1。表达分析显示,HbLhcb2.1倾向于在叶片中表达,且其转录水平随着叶片的成熟而逐渐增加,但随着叶片的衰老而明显下调。
橡胶树;叶绿素a/b结合蛋白;Lhcb2
LHCB是植物光系统II(PSII)中一类丰度极高的膜蛋白,通过保守的叶绿素a/b结合蛋白结构域与色素分子结合形成捕光色素蛋白复合体,广泛参与光能的捕获与传递、叶片发育、类囊体膜结构的维持以及各种胁迫应答[1-3]。基因组范围内的分析显示,LHCB可分为LHCB1-LHCB8等8个亚家族[4-5]。在前期工作中,研究组从橡胶树(Hevea brasiliensis)中克隆了1个与叶片发育相关的Lhcb1类基因和1个Lhcb2类基因[3,6]。此外,研究组还采用生物信息学手段从近期释放的橡胶树基因组[7]中鉴定出15个Lhcb类基因,其中,有2个可归为Lhcb2亚家族,由于转录组和试验数据的缺乏,当初仅预测出HbLhcb2.1 1 367的基因编码区[5]。随着第2代测序技术的应用与推广,橡胶树的胶乳、树皮、叶片和芽等不同组织的转录组数据也相继释放[8-14],这为基因结构的鉴定提供了条件。本文重点报道HbLhcb2.1基因的序列特征和表达特性。
1 材料与方法
1.1 试验材料
橡胶树的EST、转录组和基因组数据下载于NCBI(http://www.ncbi.nlm.nih.gov/)。试验材料详见文献[3,6,15]。生化试剂和试验耗材详见文献[16]。
1.2 试验方法
1.2.1 基因克隆 根据前期从橡胶树基因组中鉴定出的基因序列[5],用Primer 5引物设计软件设计1对引物HbLhcb2.1(F:5′-TCA CAA CGC AAT TTA GCC ATG G-3′、R:5′-AGG ACT GAG TAA TGT TGT AACCTG-3′)。然后进行叶片总RNA的提取、cDNA第一链的合成,并以成熟期叶片反转录的cDNA作为模板进行PCR扩增、凝胶电泳、挖胶回收、连载体、转化大肠杆菌感受态细胞、蓝白斑筛选、PCR验证及基因测序[16]。
1.2.2 序列的生物信息学分析 蛋白序列的理化特性、保守结构域、跨膜螺旋、翻译后修饰、亚细胞定位预测,以及同源比对与进化分析参照文献[6,15]。1.2.3 基因的表达分析 基因在叶片(NCBISRA登录号SRX278515)、胶乳(SRX278514)和树皮(SRX278513)中的表达谱分析参照文献[17],以RPKM值表示基因的相对转录水平。采用半定量RT-PCR分析基因在不同发育时期叶片中的表达模式,具体为:根据测序结果,设计引物HbLhcb2.1Fq(5′-CCT CAA CCT CCT CAG CCA AA-3′)和HbLhcb2.1Rq(5′-TGA ACC AAT CTT GCG GAC G-3′),以18 S rRNA作为内参基因(引物序列详见文献[6]),目的基因和内参的扩增循环数分别设为25个和18个,72℃延伸时间设为20 s,每个样品至少重复3次。
2 结果与分析
2.1 基因克隆
研究通过将NCBI数据库中橡胶树胶乳、树皮、叶片和芽等不同组织的转录组数据比对到基因组上,发现HbLhcb2.1的cDNA全长为2 006 bp,其中包括:798 bp的ORF、234 bp的5'UTR和974 bp的3'UTR;其基因序列中另外含有1个569 bp的内含子。
为克隆HbLhcb2.1,本研究以成熟叶片反转录的cDNA作为模板,应用引物对HbLhcb2.1F和HbLhcb2.1R(目标片段为849 bp,其中包括ORF,以及18 bp的5'UTR和33 bp的3'UTR)进行PCR扩增,结果获得1条800余bp的清亮条带。测序结果(3个单克隆)显示,除存在2个碱基的差异(均未影响蛋白序列)外,PCR扩增得到的序列与基因组的对应区域一致(图1)。
2.2 序列的生物信息学分析
2.2.1 HbLhcb2.1基因及其编码蛋白的一级结构分析 HbLhcb2.1预测编码265个氨基酸(aa),理论分子量为28.66 kD,等电点为5.42;在构成蛋白的20种aa中,甘氨酸含量最高(12.5%),丙氨酸次之(10.6%),半胱氨酸含量最低(0.8%);蛋白含有22个强碱性aa,27个强酸性aa,103个疏水aa和53个极性aa,脂肪族指数为81.06,不稳定系数为26.93;蛋白的N-端为主要的亲水区,而C-端含有3个明显的疏水区,疏水性最高的为167位的亮氨酸,其分值为2.378,亲水性最高的为53位的天冬氨酸,其分值为-2.111;蛋白的总平均疏水指数为-0.047,整体表现为亲水性。
2.2.2 蛋白的高级结构分析 SMART分析显示,HbLhcb2.1含有1个保守的捕光叶绿素a/b结合蛋白结构域(PF00504,65~232位,E值为6×10-51,图2)。MotifScan分析显示,蛋白含有4个潜在的N-豆蔻酰化位点(即28~33、108~113、122~127和178~183位)、3个潜在的蛋白激酶C磷酸化位点(即36~38、40~42和234~236位)、1个潜在的酪氨酸激酶磷酸化位点(即236~243位)。WoLF PSORT分析显示,HbLhcb2.1是1个叶绿体定位的类囊体膜蛋白,且蛋白的前37位被ChloroP预测为叶绿体导肽序列;TMpred分析显示,蛋白的97~ 119、156~174和219~235位为潜在的跨膜螺旋区。


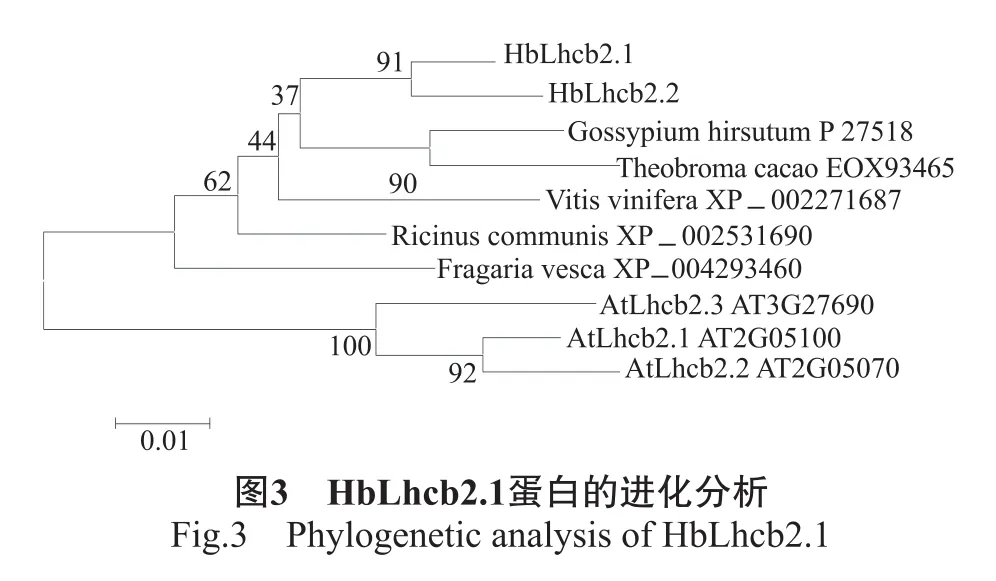
2.2.3 同源比对与进化分析 同源比对显示,HbLhcb2.1与蓖麻(Ricinus communis,XP_002531690)、棉花(Gossypium hirsutum,P27518)、葡萄(Vitis vinifera,XP_002271687)、野草莓(Fragaria vesca,XP_004293460)和可可(Theobroma cacao,EOX93465)中同源蛋白的一致性依次为95.5%、95.1%、95.1%、94.7%和94.7%,且与拟南芥(Arabidopsis thaliana)中3个同源蛋白(即AtLhcb2.1、AtLhcb2.2和AtLhcb2.3)的一致性也在88%以上(图3)。通过与已结晶的菠菜(Spinacia oleracea)SoLhcb1[18](一致性为76.2%)和豌豆(Pisum sativum)PsLhcb1[19](一致性为75.1%)进行比对,发现HbLhcb2.1含有1个三聚化基序、3个跨膜α-螺旋、2个C端螺旋、8个叶绿素a结合位点、6个叶绿素b结合位点和4类胡萝卜素结合位点(图2)。
2.3 基因的表达分析
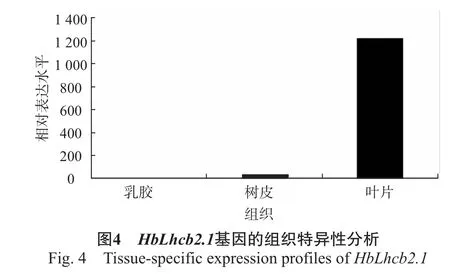
及至当前,NCBI中已释放的橡胶树转录组数据已涵盖胶乳、树皮、叶片、芽、根和体细胞胚[8-14]。同源比对显示,HbLhcb2.1目前还没有对应的EST,但在叶片和树皮的转录组文库中找到同源读段(read)。基于RPKM值的相对定量显示,基因在叶片中的表达丰度比树皮中高1 000倍以上(图4)。
半定量RT-PCR分析显示,HbLhcb2.1在古铜期叶片中的转录水平相对较低,后面随着叶片的成熟其表达水平逐渐增加,淡绿期达到最大值,后期随着叶片的衰老而明显降低(图5)。

3 讨 论
采用RT-PCR技术分离并报道了橡胶树HbLhcb2.1的cDNA序列。基因与先前报道的HbLhcb2.2[3]在核酸和蛋白水平上的一致性分别为98.0%和98.9%,而与LHCB1亚家族的HbCAB1[6]在核酸和蛋白水平上的一致性分别为72.7%和78.4%。与HbLhcb2.2相同,HbLhcb2.1预测编码265个氨基酸,其N端含有一段类囊体膜定位的导肽序列,总平均疏水指数<0,不稳定系数<40,表明HbLhcb2.1是一个稳定的、叶绿体定位的亲水性类囊体膜蛋白。序列分析显示,HbLhcb2.1含有3个跨膜螺旋、2个C端螺旋、1个三聚化基序、1个保守的捕光叶绿素a/b结合蛋白结构域、1个N端(T40)磷酸化位点,以及多个叶绿素和类胡萝卜素结合位点。其中,叶绿素和类胡萝卜素结合位点在不同LHC蛋白中均非常保守[4-5];三聚化基序为LHCB1-3和LHCB5亚家族所特有,其对于同源或异源三聚体的形成是必须的[20-21];T40磷酸化位点在LHCB1-4亚家族中均非常保守,其对于植物应答环境胁迫的可逆磷酸化所必须[22-23]。序列比对和进化分析表明,HbLhcb2.1与蓖麻、棉花、葡萄、野草莓、可可和拟南芥中同源蛋白的一致性均在88%以上,在进化上显示出高度的保守性。就表达模式而言,在本研究所分析的各种组织中,基因仅在叶片和树皮中被检测到,这表明HbLhcb2.1不在胶乳、芽、根和体细胞胚等组织中表达,或者基因在这些组织中的表达极低以至于在当前的测序深度下还不能被检测到,当然,也不能排除基因在这些组织中的表达需要某种特定的刺激。在叶片中,HbLhcb2.1的表达与叶绿素含量密切相关,其转录水平随着叶片的成熟而逐渐增加,而随着叶片的衰老逐渐下调。因此,HbLhcb2.1可作为叶片发育的标记基因,尤其是用于评估叶片的衰老状况。
[1] Barros T,Kühlbrandt W.Crystallisation,structure andfunction of plant light-harvesting Complex II[J]Biochim Biophys Acta,2009,1787(6):753-772.
[2] Xia Y,Ning Z,Bai G,et al.Allelic variations of a light harvesting chlorophyll a/b-binding protein gene(Lhcb1)associated with agronomic traits in barley[J].PLoS One,2012,7(5):e37573.
[3] 刘建汀,谢贵水,莫业勇,等.橡胶树捕光叶绿素结合蛋白基因Lhcb2.2的克隆与分析[J].西南农业学报,2014,27(6):2267-2271.
[4] Jansson S.A guide to the Lhc genes and their relatives in Arabidopsis[J].Trends Plant Sci,1999,4(6):236-240.
[5] 邹智,安峰,杨礼富,等.大戟科Lhcb基因家族的全基因组鉴定、分类与进化分析[J].中南林业科技大学学报,2013,33(12):40-45.
[6] 邹智,杨礼富,安峰,等.橡胶树AtCAB1同源基因的克隆及其在稳定与衰老期叶片中的差异分析[J].热带农业科学,2013,33(4):30-35.
[7] Rahman A Y,Usharraj A O,Misra B B,et al.Draft genome sequence of the rubber tree Hevea brasiliensis[J].BMC Genomics,2013,14:75.
[8] Xia Z,Xu H,Zhai J,etal.RNA-Seq analysis and de novo transcriptome assembly of Hevea brasiliensis[J].Plant Mol Biol,2011,77(3):299-308.
[9] Triwitayakorn K,Chatkulkawin P,Kanjanawattanawong S,et al.Transcriptome sequencing of Hevea brasiliensis for development ofmicrosatellitemarkers and construction of a genetic linkage map[J].DNA Res,2011,18(6):471-482.
[10] Chow K S,Mat-Isa M N,Bahari A,et al.Metabolic routes affecting rubber biosynthesis in Hevea brasiliensis latex[J].JExp Bot,2011,63(5):1863-1871.
[11] Chow K S,Ghazali A K,Hoh C C,et al.RNA sequencing read depth requirement for optimal transcriptome coverage in Hevea brasiliensis[J].BMCRes Notes,2014,7:69.
[12] Duan C,Argout X,Gébelin V,et al.Identification of the Hevea brasiliensis AP2/ERF superfamily by RNA sequencing[J].BMCGenomics,2013,14:30.
[13] Mantello CC,Cardoso-Silva CB,da Silva CC,etal.De novo assembly and transcriptome analysis of the rubber tree(Hevea brasiliensis)and SNPmarkers development for rubber biosynthesis pathways[J].PLoSOne,2014,9(7):e102665.
[14] Salgado L R,Koop D M,Pinheiro D G,et al.De novo transcriptome analysis of Hevea brasiliensis tissues by RNA-seq and screening for molecular markers[J].BMC Genomics,2014,15:236.
[15] 邹智,杨礼富.橡胶树半胱氨酸蛋白酶基因HbCP3的克隆与表达特性分析[J].湖南农业大学学报(自然科学版),2015,41(1):53-57.
[16] 邹智,杨礼富.巴西橡胶树铁硫簇支架蛋白cDNA的克隆与分析[J].热带作物学报,2010,31(10):1752-1756.
[17] Zou Z,Huang Q X,An F.Genome-wide identification,classification and expression analysis of Lhc supergene family in castor bean(Ricinus communis L.)[J].Agr Biotechnol,2013,2(6):44-48,51.
[18] Liu Z,Yan H,Wang K,et al.Crystal structure of spinach major light-harvesting complex at2.72 A°resolution[J].Nature,2004,428(6980):287-292.
[19] Standfuss J,Terwisscha van Scheltinga A C,Lamborghini M,et al.Mechanisms of photoprotection and nonphotochemical quenching in pea light-harvesting complex at2.5 A resolution[J].EMBO J,2005,24(5):919-928.
[20] Hobe S,Förster R,Klingler J,et al.N-proximal sequencemotif in light-harvesting chlorophyll a/b-binding protein is essential for the trimerization of light-harvesting chlorophyll a/b complex[J].Biochemistry,1995,34(32):10224-10228.
[21] Standfuss J,Kühlbrandt W.The three isoforms of the light-harvesting complex II:spectroscopic features,trimer formation,and functional roles[J].JBiol Chem,2004,279(35):36884-36891.
[22] Andersson J,Wentworth M,Walters R G,et al.Absence of the Lhcb1 and Lhcb2 proteins of the light-harvesting complex of photosystem II-effects on photosynthesis,grana stacking and fitness[J].Plant J,2003,35(3):350-361.
[23] Bellafiore S,Barneche F,Peltier G,et al.State transitions and light adaptation require chloroplast thylakoid protein kinase STN7[J].Nature,2005,433(7028):892-895.
(责任编辑 赵粉侠)
Molecular Cloning and Expression Analysis of an Lhcb2 Gene from Hevea brasiliensis
Zou Zhi,Wang Danhua,Dai Xuemei,Zhu Jiali,Xie Guishui
(Rubber Research Institute,Chinese Academy of Tropical Agricultural Sciences,Danzhou Investigation&Experiment Station of Tropical Crops,Ministry of Agriculture,Danzhou Hainan 571737,China)
Based on the gene sequence identified from the rubber tree(Hevea brasiliensis)genome,an 849 bp cDNA containing an ORF of 798 bp was isolated from the leaf tissue by using the RT-PCR technique.The gene denoted HbLhcb2.1 was predicted to encode 265 amino acids with a theoreticalmolecular weight of 28.66 kD and isolectric point of 5.42;the protein was promised to be a thylakoid protein of chloroplast;the protein contains one conserved light-harvesting chlorophyll a/b binding protein domain,three transmembrane helices and two C-terminal helices,a trimerization motif,several chlorophyll and carotenoid binding sites,which could be grouped into the LHCB2 subfamily based on the evolutionary relationship.Homologous analysis showed that HbLhcb2.1 shared an identity of about 95%with the homologs in Ricinus communis,Gossypium hirsutum,Vitis vinifera,Fragaria vesca or Theobroma cacao,suggesting a highly conserved evolution.Expression analysis indicated that HbLhcb2.1 wasmainly expressed in the leaves,with a gradual increase as along with the leafmaturity,but a sharp decrease in senescing leaves.
rubber tree(Hevea brasiliensis);chlorophyll a/b binding protein;Lhcb2
S794.1
:A
:2095-1914(2015)06-0014-05
10.11929/j.issn.2095-1914.2015.06.003
2015-03-17
国家自然科学基金项目(31371556)资助;海南省自然科学基金项目(312026)资助;现代农业产业技术体系建设专项资金项目(CARS-34-GW5)资助;中国热带农业科学院橡胶研究所基本科研业务费专项(1630022015004)资助。
第1作者:邹智(1982—),男,助理研究员。研究方向:分子生物学。Email:zouzhi2008@126.com。

