芒果转录组中SSR位点信息分析与引物筛选
罗纯 武红霞 姚全胜 王松标 许文天 马小卫

摘 要 利用MicroSAtellite软件分析筛选芒果转录组中的SSR位点。结果表明,在54 207条Unigene中共搜索得到4 103个SSR位点,出现频率为7.57%。其中,三核苷酸重复为主导重复类型,占SSR总数的38.19%,其次是单核苷酸重复(26.91%)。AG/CT和AAG/CTT分别是二核苷酸和三核苷酸重复的优势基元。根据SSR侧翼序列共设计6 915对SSR引物,随机挑选了230对进行PCR检测,获得93对多态性引物。由此可见,芒果转录组数据可以作为大量开发SSR标记的资源,这些SSR标记也将有助于芒果遗传多样性和种质资源鉴定的研究。
关键词 芒果;转录组;SSR信息;多态性
中图分类号 S667.7 文献标识码 A
Abstract A MicroSAtellite software was used to find the SSR loci in the mango transcriptome. As a result, a total of 4 103 SSRs were found from 54 207 unigenes, with a frequency of 7.57%. The trinucleotide repeats were the most frequent SSRs among them(38.19%). AG/CT and AAG/CTT were the most common motifs in the dinucleotide and trinucleotide repeats, respectively. Based on the flanking sequences of these SSR loci, 6 915 SSR primer pairs were designed. A total of 230 primer pairs were randomly selected to be verified by PCR, and 93 pairs(40.43%)of them exhibited to be polymorphism. The results showed that transcriptome data would be an effective source for the exploration of SSR markers for mango and these SSR markers would contribute greatly to the research into genetic diversity and germplasm characterization in mango.
Key words Mango; Transcriptome; SSR information; Polymorphism
doi 10.3969/j.issn.1000-2561.2015.07.013
Simple sequence repeat(SSR,简单重复序列)是指以1~6个核苷酸为单位多次串联重复的DNA序列,在人类和其他动植物基因组中普遍存在且随机分布[1]。SSR重复单位的数目高度变异,但是某一特定SSR序列的侧翼序列通常为高度保守的单一序列,根据SSR侧翼序列设计引物通过PCR技术即可分析SSR的多态性。与其它分子标记技术相比,SSR标记具有多态性高、共显性、易用PCR检测、重复性高、数量丰富和对基因组有很好的覆盖性等特点[2]。基于以上这些特点,SSR已成为在生物上应用广泛和重要的DNA分子标记。在植物遗传育种中,SSR标记已经在植物遗传多样性研究、品种鉴定、分子标记辅助选择育种(MAS)、遗传连锁图谱的构建和外源染色体片段或目的基因鉴定和定位等方面得到应用。根据SSR的来源可将其分为基因组SSR和EST-SSR。传统的基因组SSR标记引物设计过程较慢,需要构建基因组DNA文库,探针杂交,重复序列克隆的识别和筛选,测序等许多繁琐的过程,不但耗时费力,而且开发成本也比较高,效率较低[3]。另外,利用该方法所获得的SSR还会受开发过程所采用探针的SSR重复单元种类限制[4]。而EST-SSR分子标记与基因组SSR标记相比,在不同的物种之间具有较好通用性,其开发方法较简单而且成本又较低。
芒果(Mangifera indica L.)是世界五大水果之一,且因其风味独特、营养丰富,经济效益高等优点,种植面积不断扩大,产量也逐年上升。中国是世界芒果主要生产国之一,芒果种植在国民经济上具有重要作用。目前,国内外学者利用多种DNA分子标记技术如RAPD[5-7]、ISSR[8]、AFLP[9-10]等对芒果进行了品种或真实杂种鉴定,并且对野生、砧木或栽培品种等资源进行了亲缘关系及遗传多样性分析。芒果SSR标记也有开发[11-13],但是目前基于EST数据开发的SSR标记非常少,限制了芒果分子水平的研究发展。因此,本研究对芒果转录组测序产生的海量EST序列进行全转录组SSR鉴定及引物设计,揭示芒果转录组SSR的分布规律和特性,为芒果遗传图谱构建、系谱分析和MAS育种提供高效的分子标记资源。
1 材料与方法
1.1 材料
以‘Zill4个发育时期的果皮和果肉混合样品为材料进行转录组测序,共获得54 207条Unigene[14],总长度为45 425 223 bp。
1.2 方法
1.2.1 芒果转录组SSR的筛选 利用SSR软件 MicroSAtellite(MISA,http://pgrc.ipk -gatersleben.de/misa/)对‘Zill转录组Unigene序列进行SSR查找,具体筛选标准为:SSR重复基元的长度为1~6 bp,单核苷酸重复次数≥12,二核苷酸重复次数≥6,三、四核苷酸重复次数≥5,五、六核苷酸重复次数≥4。只保留SSR重复单元在Unigene上前后序列均不小于150 bp的SSR,使用Primer3软件设计引物。
1.2.2 芒果SSR-PCR扩增及产物检测 PCR反应体系20 μL,其中包括:1×buffer,40 ng DNA模板,1.8 mmol/L MgCl2,0.4 mmol/L dNTPs,0.2 μmol/L引物,0.75 U Taq酶。扩增反应是在TaKaRa PCR Thermal Cycler Dice仪器上进行,扩增程序如下:94 ℃预变性2.5 min;94 ℃变性30 s,58 ℃复性45 s,72 ℃延伸1 min,35个循环;72 ℃延伸5 min。扩增产物在6%变性聚丙烯酰胺凝胶上电泳分离。
2 结果与分析
2.1 芒果转录组中SSR位点的数量与分布
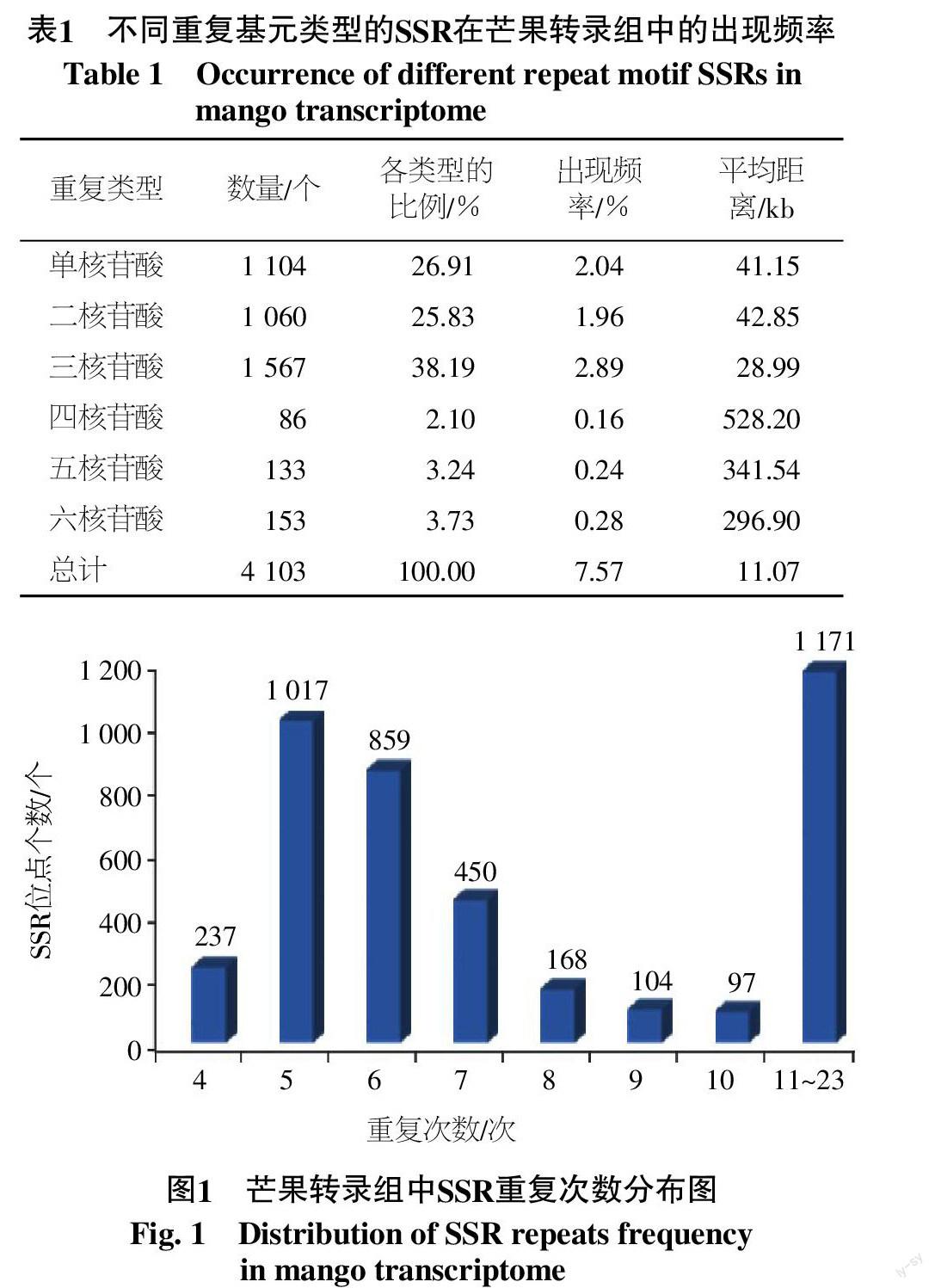
利用软件MISA对芒果转录组54 207条Unigene进行分析查找,共3 683条Unigene含有SSR,发生频率(含有SSR的Unigene数目/总Unigene数目)为6.79%。其中,3 301条Unigene含有单个SSR位点,占含有SSR序列条数的89.63%,其余382条Unigene含有多个SSR位点,占含有SSR序列条数的10.37%。芒果转录组中共查找到SSR位点4 103个,出现频率为(检出的SSR个数/总Unigene数目)7.57%,平均距离(芒果转录组Unigene总长度/SSR数目)为11.07 kb,即芒果转录组中平均每11.07 kb出现1个SSR(表1)。
芒果转录组SSR数量较为丰富,单核苷酸重复至六核苷酸重复均有分布,但是类型SSR出现频率存在差异(表1),主要是单、二和三核苷酸重复类型。三核苷酸重复类型数量最多,占全部SSR的38.19%;其次是单核苷酸和二核苷酸重复类型,分别占26.91%和25.83%,四核苷酸重复类型数量最少,占2.10%。
芒果转录组中SSR位点重复次数以5次(1 017)最多,占总SSR的24.79%;其次为重复次数6、7、4、8、9和10次,SSR位点个数在97~859。统计4~10次重复的SSR位点共有2 932个,占71.46%;11~23次重复的SSR位点共有1 171个,占总SSR数量的28.54%(图1)。
2.2 芒果转录组中SSR特性
芒果转录组中共查找到153种重复基元,其中以单核苷酸重复基元A/T最多,占总SSR位点的26.81%,其次是二核苷酸重复基元AG/CT和三核苷酸重复基元AAG/CTT,分别占总SSR数目的19.79%和13.21%。具体来说,二核苷酸重复基元共查找到3种,AG/CT占主导地位,占该重复类型的76.60%,其它2种AC/GT和AT/AT数量相当;三核苷酸重复基元共10种,出现次数最多的是AAG/CTT,占该重复类型的34.59%,其次是ATC/ATG和ACC/GGT,分别占16.46%和13.08%,出现次数最少的是ACT/AGT,仅占该重复类型的1.21%;四、五和六核苷酸重复基元最多的分别是AAAG/CTTT、AAGAG/CTCTT和ACCCTG/AGGGTC,其它重复基元出现频率较低(表2)。
2.3 芒果转录组SSR的可用性评价
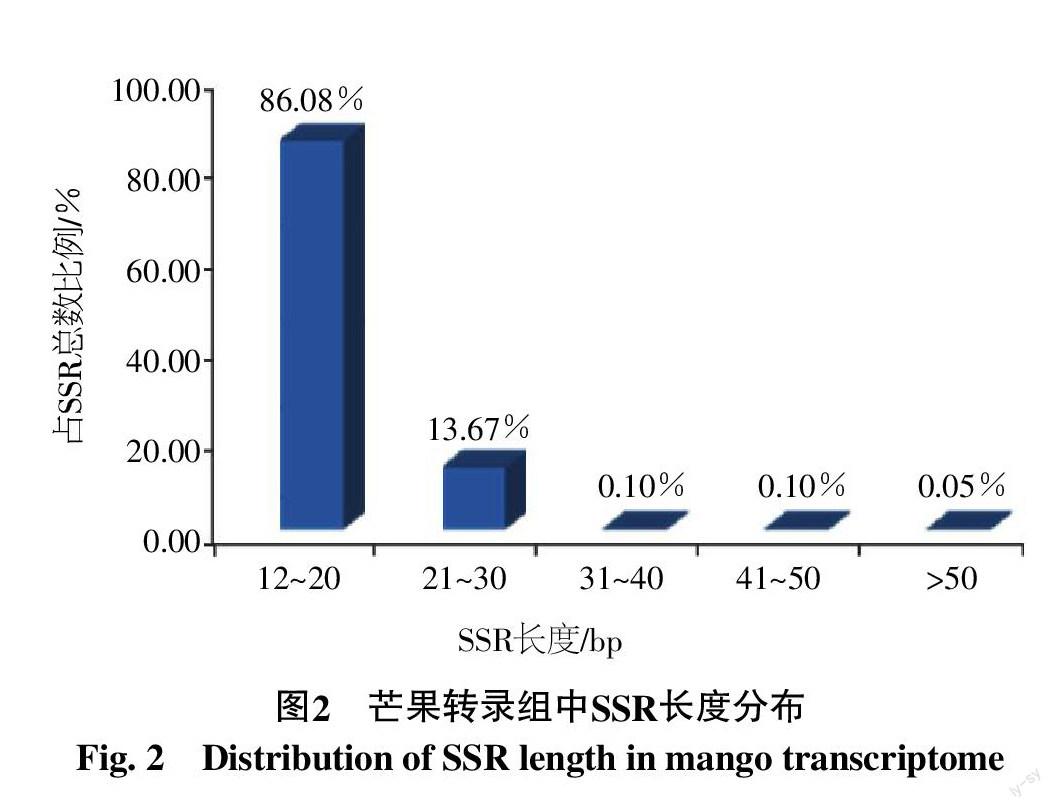
SSR多态性的高低是评价其有用性的主要标准,而SSR的长度影响其多态性。当SSR长度在20 bp以上时,在不同品种间显示出较高的多态性,长度在12~20 bp的SSR多态性低些,而长度在12 bp以下的SSR多态性很低[15],因此本研究在查找SSR时将长度小于12 bp的已经除去。芒果转录组SSR的长度为12~60 bp,长度在12~20 bp的SSR数量最多,共3 532个,占全部SSR的86.08%。其次是21~30 bp的SSR,共561个(13.67%)(图2)。依据高级基元SSR多态性普遍比低级基元的低[16],而在长度大于20 bp的芒果SSR中,主要是低级基元SSR,包含单、二和三核苷酸SSR,共378个,可以预计这部分多态性潜能高的SSR在芒果上应具有较高的利用价值。
2.4 芒果转录组SSR引物设计与筛选
使用Primer 3软件对上下游序列均不小于150 bp的SSR设计引物,每条序列产生5对引物,共有1 383条芒果SSR序列获得6 915对引物。为了验证这些引物的有效性和多态性,随机挑选230对引物对7个不同的芒果品种进行PCR扩增(图3)。结果表明除12对引物未获得目的产物外,其余218对引物均获得稳定和可重复的目的产物,表现多态性的引物共93对,多态性比例为40.43%(表3)。
3 讨论与结论
本研究在芒果转录组54 207条Unigene序列中搜索到SSR位点4 103个,分布于3 683条Unigene上,出现频率为7.57%,平均距离为11.07 kb。与其他植物相比,芒果转录组SSR出现频率低于枇杷(22.02%)、油棕(22.6%)和橡胶树(35.58%)[17-19],但高于大麦(2.8%)、棉花(5.29%)和小麦(7.4%)[20-22],与莲花(7.6%)和木豆(7.6%)相当[23-24]。SSR的出现频率在不同物种、不同研究结果中存在差异主要是搜索重复类型和长度等标准不同以及分析数据量的大小不同所造成的[25]。
已报道的大多数植物中SSR以二核苷酸和三核苷酸重复类型最为丰富,与芝麻、猕猴桃、咖啡、橡胶树等二核苷酸SSR为主导的植物不同[26-29],芒果转录组中三核苷酸SSR数量最多,占全部SSR的38.19%(表1),与甘蔗、小麦、红薯和柑橘等植物类似[30-33]。芒果中二核苷酸优势重复基元为AG/CT,与多数植物中报道的情况相同[26,29]。前人研究发现双子叶植物中三核苷酸重复以AAG/CTT重复为主,CCG/CGG出现频率很低[34-35],芒果三核苷酸SSR中AAG/CTT数量最多,占总SSR的13.21%,而CCG/CGG仅占0.95%,验证了前人研究。
在芒果上,国内外已报道的SSR标记数量并不多,主要是通过构建基因组文库,利用探针杂交富集筛选得到,如Duval等[36]、Viruel等[37]、Honsho等[38]和Chiang等[39]通过该方法分别开发了28、16、6和20个SSR标记。国内黄启星等[40]利用公共数据库中1 665条芒果EST序列分析查找到EST-SSR位点32个,EST-SSR平均距离为20.2 kb,大于本研究中利用转录组数据查找的SSR的平均距离,可能是由于转录组测序数据较丰富,从而含有SSR位点也更丰富。同时黄启星等[40]还发现芒果EST-SSR以三核苷酸重复类型数量最多,其中又以AAG/CTT重复为主,与本研究得到的结果一致。
总的来说,芒果转录组中SSR的出现频率虽然不高,但是类型比较丰富,长度大于20 bp的主要是低基元重复,多态性潜能高,设计的引物经实验验证其有效性也高。本研究结果为进一步构建芒果遗传图谱、系谱分析和MAS育种提供了高效的分子标记资源。
参考文献
[1] Tautz D, Renz M. Simple sequence repeats are ubiquitous repetitive components of eukaryotic genomes[J]. Nucl Acids Res, 1984, 12(10): 4 127-4 138.
[2] Powell W, Machray G C, Provan J. Polymorphism revealed by simple sequence repeat[J]. Trends Plant Sci, 1996, 1(7): 215-222.
[3] Roder M S, Korzun V, Wendehake K, et al. A microsatellite map of wheat[J]. Genetics, 1998, 149(4): 2 007-2 023.
[4] Chen C, Zhou P, Choi Y A, et al. Mining and characterizing microsatellites from citrus ESTs[J]. Theor Appl Genet, 2006, 112(7): 1 248-1 257.
[5] Karihaloo J L, Dwivedi Y K, Sunil A, et al. Analysis of genetic diversity of Indian mango cultivars using RAPD markers[J]. J Hortic Sci Biotech, 2003, 78: 285-289.
[6] Shukla M, Babu R, Mathur V K, et al. Diverse genetic bases of Indian polyembryonic and monoembryonic mango(Mangifera indica L)cultivars[J]. Curr Sci, 2004, 87: 870-871.
[7] Rahman M L, Rabbani M G, Siddique M N A, et al. Molecular characterization of 28 mango germplasm using RAPD[J]. Plant Tiss Culture Biotech, 2007, 17(1): 71-77.
[8] Rocha A, Carlos L, Salomao C, et al. Genetic diversity of ‘Ubá mango tree using ISSR markers[J]. Mol Biotechnol, 2012, 50(2): 108-113.
[9] 房经贵, 章 镇, 马正强, 等. AFLP标记在两个芒果品种间杂交F1代的多态性及分离方式[J]. 中国农业科学, 2000, 33(3): 19-24.
[10] Kashkush K, Fang J G, Tomer E, et al. Cultivar identification and genetic map of mango(Mangifera indica)[J]. Euphytica, 2001, 122: 129-136.
[11] Hirano R, Ishii H, Oo T H, et al. Propagation management methods have altered the genetic variability of two traditional mango varieties in Myanmar, as revealed by SSR[J]. Plant Genet Resour, 2011, 9(3): 404-410.
[12] Tsai C C, Chen Y H, Chen C H, et al. Cultivar identification and genetic relationship of mango(Mangifera indica)in Taiwan using 37 SSR markers[J]. Sci Hortic, 2013, 164: 196-201.
[13] Surapaneni M, Vemireddy L R, Begum H, et al. Population structure and genetic analysis of different utility types of mango(Mangifera indica L.)germplasm of Andhra Pradesh state of India using microsatellite markers[J]. Plant Syst Evol, 2013, 299(7): 1 215-1 229.
[14] Wu H X, Jia H M, Ma X W, et al. Transcriptome and proteomic analysis of mango(Mangifera indica Linn)fruits[J]. J Proteomics, 2014, 105: 19-30.
[15] Temnykh S, De Clerck G, Lukashova L, et al. Computational andexperimental analysis of microsatellites in rice(Oryzasativa L.)[J]. Genome Res, 2001, 11: 1 441-1 452.
[16] Dreisigacker S, Zhang P, Warburton M L, et al. SSR and pedigree analyses of genetic diversity among CIMMYT wheat lines targeted to different mega environments[J]. Crop Sci, 2004, 44: 381-388.
[17] Li X Y, Xu H X, Feng J J, et al. Development and application of genic simple sequence repeat markers from the transcriptome of loquat[J]. J Amer Soc Hort Sci, 2014, 139(5): 507-517.
[18] 周丽霞, 肖 勇, 杨耀东. 油棕转录组SSR标记开发研究[J]. 广东农业科学, 2014(14): 136-138.
[19] Mantello C C, Cardoso-Silva C B, da Silva C C, et al. De Novo assembly and transcriptome analysis of the rubber tree (Hevea brasiliensis)and SNP markers development for rubber biosynthesis pathways[J]. PLoS ONE, 2014, 9: e102 665.
[20] Varshney R, Grosse I, Hahnel U, et al. Genetic mapping and BAC assignment of EST-derived SSR markers shows non-uniform distribution of genes in the barley genome[J]. Theor Appl Genet, 2006, 113(2): 239-250.
[21] Zhang X W, Ye Z W a, Wang T K, et al. Characterization of the global transcriptome for cotton(Gossypium hirsutum L.)anther and development of SSR marker[J]. Gene, 2014, 551(2): 206-213.
[22] Peng J, Lapitan N L V. Characterization of EST-derived microsatellites in the wheat genome and development of eSSR markers[J]. Funct Integr Genomic, 2005, 5(2): 80-96.
[23] Zhang W, Tian D, Huang X, et al. Characterization of flower-bud transcriptome and development of genic SSR markers in asian lotus(Nelumbo nucifera Gaertn.)[J]. PLoS ONE, 2014, 9(11): e112 223.
[24] Dutta S, Kumawat G, Singh B P, et al. Development of genic-SSR markers by deep transcriptome sequencing in pigeonpea [Cajanus cajan(L.)Millspaugh][J]. BMC Plant Biol, 2011, 11: 17.
[25] Varshney R K, Graner A, Sorrells M E. Genic microsatellite markers in plants: features and applications[J]. Trends Biotechnol, 2005, 23(1): 48-55.
[26] Wei W, Qi X, Wang L, et al. Characterization of the sesame (Sesamum indicum L.)global transcriptome using Illumina paired-end sequencing and development of EST-SSR markers[J]. BMC Genomics, 2011, 12: 451.
[27] Fraser L G, Harvey C F, Crowhurst R N, et al. EST-derived microsatellites from Actinidia species and their potential for mapping[J]. Theor Appl Genet, 2004, 108(6): 1 010-1 016.
[28] Aggarwal R K, Hendre P S, Varshney R K, et al. Identification, characterization and utilization of EST-derived genic microsatellite markers for genome analyses of coffee and related species[J]. Theor Appl Genet, 2007, 114(2): 359-372.
[29] Li D, Deng Z, Qin B, et al. De novo assembly and characterization of bark transcriptome using Illumina sequencing and development of EST-SSR markers in rubber tree(Hevea brasiliensis Muell. Arg.)[J]. BMC Genomics, 2012, 13: 192.
[30] Marconi T G, Costa E, Miranda H R, et al. Functional markers for gene mapping and genetic diversity studies in sugarcane[J]. BMC Res Notes, 2011, 4: 264.
[31] La Rota M, Kantety R V, Yu J K, et al. Nonrandom distribution and frequencies of genomic and EST-derived microsatellite markers in rice, wheat, and barley[J]. BMC Genomics, 2005, 6: 23.
[32] Wang Z, Fang B, Chen J, et al. De novo assembly and characterization of root transcriptome using Illumina paired-end sequencing and development of cSSR markers in sweet potato(Ipomoea batatas)[J]. BMC Genomics, 2010, 11: 726.
[33] Chen C, Zhou P, Choi Y, et al. Mining and characterizing microsatellites from citrus ESTs[J]. Theor Appl Genet, 2006, 112(7): 1 248-1 257.
[34] Kumpatla S, Mukhopadhyay S. Mining and survey of simple sequence repeats in expressed sequence tags of dicotyledonous species[J]. Genome, 2005, 48(6): 985-998.
[35] Liang X, Chen X, Hong Y, et al. Utility of EST-derived SSR in cultivated peanut(Arachis hypogaea L.)and Arachis wild species[J]. BMC Plant Biol, 2009, 9: 35.
[36] Duval M F, Bunel J, Sitbon C, et al. Development of microsatellite markers for mango(Mangifera indica L.)[J]. Mol Ecol Notes, 2005, 5(4): 824-826.
[37] Viruel M A, Escribano P, Barbieri M, et al. Fingerprinting, embryo type and geographic differentiation in mango(Mangifera indica L., Anacardiaceae)with microsatellites[J]. Mol Breeding, 2005, 15(4): 383-393.
[38] Honsho C, Nishiyama K, Eiadthong W, et al. Isolation and characterization of new microsatellite markers in mango(Mangifera indica)[J]. Mol Ecol Notes, 2005, 5(1): 152-154.
[39] Chiang Y C, Tsai C M, Chen Y K H, et al. Development and characterization of 20 new polymorphic microsatellite markers from Mangifera indica(Anacardiaceae)[J]. Am J Bot, 2012, 99(3): e117-e119.
[40] 黄启星, 左 娇, 孔 华, 等. 11种热带植物EST-SSR标记的开发和多样性分析[J]. 热带作物学报, 2012, 33(7): 1 208-1 214.

