野生杏遗传多样性研究
努尔买买提,满 莉,温亚梅,肖洪兴
(1.伊犁师范学院新疆维吾尔自治区教育厅普通高等学校天然产物化学与应用重点实验室,新疆 伊宁 835000;2.东北师范大学生命科学学院,吉林 长春 130024;3.四平市环境保护监测站,吉林 四平 136000)
野生杏遗传多样性研究
努尔买买提1,满 莉2,温亚梅3,肖洪兴2
(1.伊犁师范学院新疆维吾尔自治区教育厅普通高等学校天然产物化学与应用重点实验室,新疆 伊宁 835000;2.东北师范大学生命科学学院,吉林 长春 130024;3.四平市环境保护监测站,吉林 四平 136000)
采用DNA测序技术对原产于我国新疆伊犁河谷地区的杏(ArmenicavulgarisLamarck)进行了群体样本测序.对所获得的野生杏单拷贝核基因Adh1序列进行了分析.结果表明:不同野杏居群的核苷酸多态性和单倍型多态性没有显著差异.Gapdh基因序列的分析显示,各野生杏居群的核苷酸多态性和单倍型多态性相对较低,特别是巩留县核桃沟的野生杏居群,其核苷酸多态性和单倍型多态性均为0.此外,AMOVA分析结果表明,遗传变异主要发生在居群内,而居群之间没有明显的遗传分化.研究结果不仅揭示了野生杏居群的遗传多样性,而且为杏的遗传资源研究保护提供了科学依据.
野生杏;遗传多样性;SNP
杏(ArmenicavulgarisLamarck),蔷薇科(Rosaceae)李亚科(Prunoideae)杏属(Armeniaca)落叶乔木,我国各地均产,多数为栽培;华北、西北和华东地区种植较多,少数地区逸为野生,新疆各地均有栽培.杏原产中国新疆,在新疆伊犁一带野生成为纯林或与新疆野苹果林混生.野生杏主要分布于在伊犁的北天山等区域,西起于霍城县的大西沟、小西沟、果子沟,伊宁县的匹里青沟和吉里格朗沟,并向东延伸至新源县的铁木尔勒克.[1]上述区域的南向山沟和山脉的西坡、东坡,由于阳光充足、雨水充沛,非常有利于野杏的生长.从垂直分布来看,新疆野杏主要分布在阔叶林带的下半部,海拔950~1 400 m的低山带山坡和河谷中.
野生杏具有抗寒、抗旱、耐贫瘠、适应性强、栽培容易等特点,是绿化荒山、保护农田、实行果粮间作的优良树种.其果实的形状、颜色、品质及抗旱特性,都比栽培杏具有更为丰富的遗传多样性.因此,加强对野生杏资源的搜集、保存与利用,对于扩大栽培杏的遗传多样性具有重要意义.
近年来,国内外关于杏种质资源的研究越来越多,于希志等[2]对新疆杏资源进行了系统考察,揭示了新疆杏的资源特点;王磊等[1]从分布区域、形态生理、生长特性等方面描述了新疆杏的多样性;刘黎明等[3]对新疆野生杏生殖生态进行了初步的研究;吕英民等[4]应用同工酶进行了杏属植物演化关系和分类的研究.同时,也有部分学者采用分子标记技术对新疆野生杏的遗传多样性进行了探讨,如何天明等[5]利用简单重复序列(simple sequence repeat,SSR)对伊犁河谷野生杏种群遗传结构进行了分析.然而,以往对新疆野生杏遗传多样性的研究大多采用简单重复序列(SSR)[6]、随机扩增DNA多态性(randomly amplified polymorphic DNA,RAPD)[7]、 扩增片段长度多态性 (amplified restriction fragment polymorphism,AFLP)等分子标记,这些分子标记由于所携带的遗传信息较少,有的稳定性不强,近年来逐渐开始被淘汰.
随着基因组测序研究的发展,单核苷酸多态性(single nucleotide polymorphism,SNP)已成为生命科学中的研究热点.它是继RAPD和SSR等第一、二代分子标记技术之后的最新的一代分子标记,因其在基因组中分布广泛、位点丰富、遗传稳定、易实现等优点而被广泛应用于遗传多样性、分子标记辅助育种、品种鉴定和种质资源管理的研究中.目前,在玉米(ZeamayL.)、大豆(Glycinemax)、小麦(TriticumaestivumL.)及其他植物物种中已经发现了大量的SNP.[8]然而,利用SNP分子标记对新疆野生杏的遗传多样性进行研究的报道较少.因此,本研究采用以PCR和DNA测序为基础的SNP分子标记技术,对野生杏的遗传多样性和遗传结构进行了初步研究,以期为野生杏资源的有效保护和科学利用提供一些理论支持,同时也为进一步开展新疆野杏的遗传多样性研究奠定基础.
1 材料与方法
1.1 材料的收集与基因组DNA提取
研究材料采集于新疆伊犁河谷地区伊宁县的阿吾赞、巩留县的核桃沟和霍城县的大西沟(见图1和表1).采用快捷型植物基因组提取试剂盒(康为世纪生物科技有限公司,北京)提取新疆野生杏的基因组DNA,具体步骤参考产品说明书.
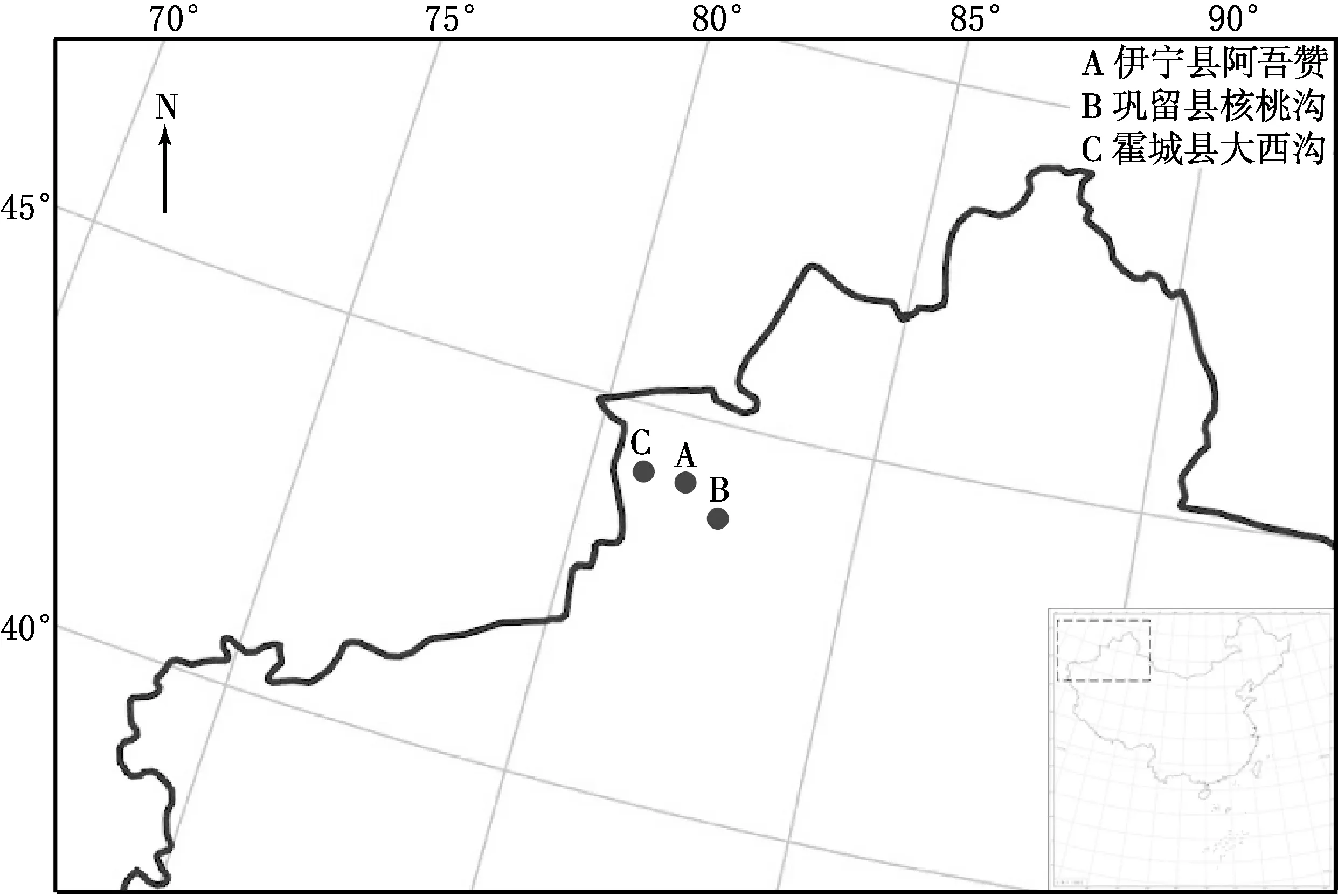
图1 样品采集分布图
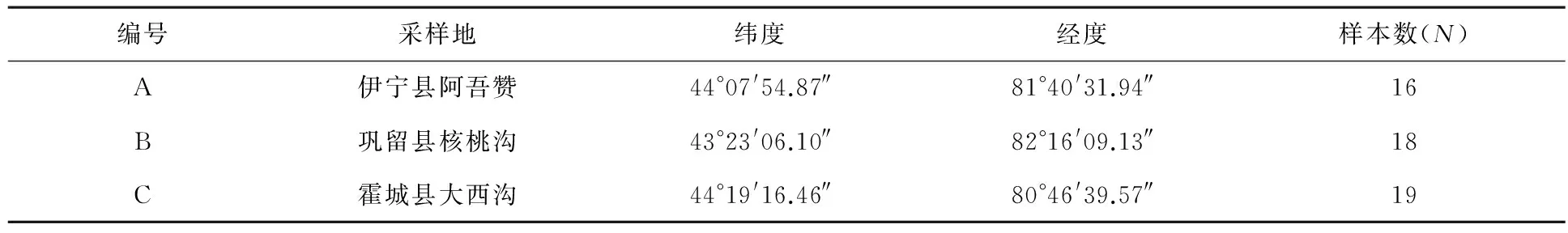
表1 新疆野生杏居群的采样地点
1.2 实验方法
1.2.1 引物设计和PCR扩增
根据NCBI(National Center for Biotechnology Information)中公布的拟南芥的乙醇脱氢酶(alcohol dehydrogenase,Adh1)和甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,Gapdh)的基因序列与杏属的EST文库进行比对,获得杏属的Adh1和Gapdh基因的EST序列.基于所获得序列(Adh1基因序列号为CV046978,Gapdh基因序列号为CV044191)利用Primer Premier 5.0软件设计引物(见表2).
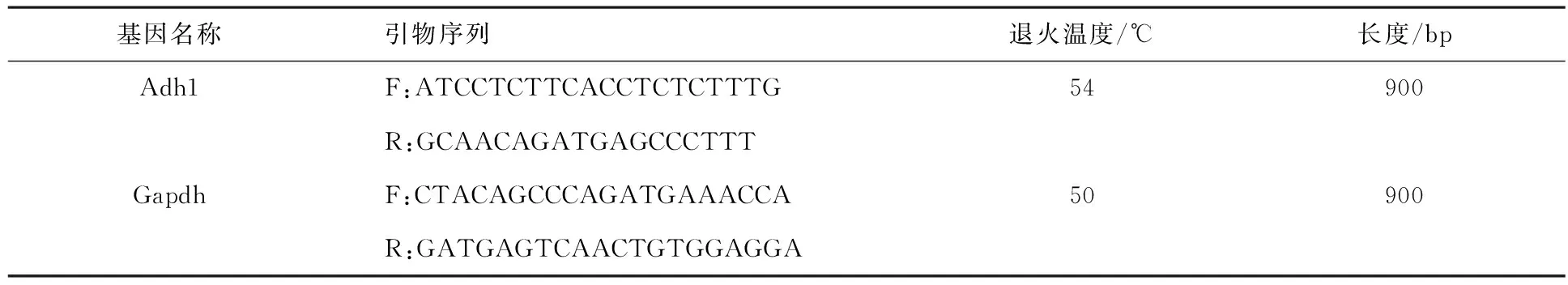
表2 引物信息
实验所用的PCR反应体系包括:10×Buffer(Mg2+plus)2.0 μL,dNTPs (10 mmol/L) 0.4 μL,正反引物(10 pmol/μL)均为0.5 μL,rTaq酶(1 U/μL) 0.2 μL,模板基因组DNA(50 ng/μL)2 μL,ddH2O 14.4 μL.扩增程序为:95℃预变性5 min,94℃变性30 s,54℃退火30 s,72℃延伸60 s,35个循环;72℃延伸10 min.4℃保存.扩增产物用1.5%琼脂糖凝胶电泳检测.
1.2.2 PCR产物的连接、转化与测序
将得到的PCR产物进行切胶回收后连接到pMD18-T载体(Takara)上,再将连接了目的片段的载体转入大肠杆菌感受态JM109(Takara)内,置于无氨苄的LB液体培养基中摇床培养2 min;将菌液均匀涂在含有氨苄(AMP)的LB固体培养基中,37℃条件下培养12 min.挑取透明的单一菌落进行菌液PCR检测.反应体系15 μL,反应程序为:95℃预变性5 min,94℃变性30 s,50℃退火30 s,72℃延伸60 s,35个循环;最后72℃延伸10 min.4℃保存.选择检测为阳性菌落的菌液送到上海立菲生物技术有限公司测序.
1.3 数据分析
根据测序所获得的数据,采用序列比对软件Clustalx1.81[9]对Gapdh和Adh1基因进行序列的比对,然后利用Bioedit[10]编辑软件进行人工调整和编辑.
遗传多样性分析主要采用DnaSP5.0[11]软件进行统计,具体参数包括核苷酸多态性(Pi)、单倍型多态性(Hd)、多态位点数(s)和单倍型数目(h).同时,采用DnaSP5.0软件对Gapdh和Adh1基因进行Tajima’sD分析.此外,采用Arlequin 3.1[12]软件中的AMOVA分析遗传变异在野杏居群中的分布情况,并根据Fst来探讨群体间的遗传分化程度.
2 结果与分析
2.1 遗传多样性检测
我们对野生杏3个居群总共53个个体的Gapdh和Adh1基因进行了测序,一共获得53条Gapdh基因序列;因为有些个体在Adh1基因序列上有不同拷贝,所以共有68条Adh1基因序列(见表3).其中,Adh1基因片段的长度大约在900 bp,与实验中根据杏属序列设计的引物所对应的片段长度相符.基于Adh1基因序列分析,野生杏居群的核苷酸多态性(Pi)较高,其范围在0.024~0.025之间.此外,不同野生杏居群间的核苷酸多态性没有明显的差异.而基于Gapdh基因序列分析结果,野生杏居群的核苷酸多态性(Pi)较低,其范围在0.000~0.003之间,其中巩留居群序列核苷酸多态性为0.
Adh1基因序列的单倍型多态性(Hd)分析显示,霍城县大西沟野生杏居群的单倍型多态性最高(0.995),其次是伊宁县阿吾赞野生杏居群(0.989),最低的是巩留县核桃沟野生杏居群(0.988).该结果与各居群的核苷酸多态性一致.Gapdh基因序列分析显示,伊宁县阿吾赞野生杏居群的单倍型多态性最高(0.988),霍城县大西沟野生杏居群次之,Hd值为0.702;巩留县核桃沟野生杏居群最小,单倍型多态性是0.000.结合核苷酸多态性和单倍型多态性可以看出,伊宁县阿吾赞野生杏居群和霍城县大西沟野生杏居群的遗传多样性较高,巩留县核桃沟野生杏居群的遗传多样性低于这两个居群.此外,通过对比2个基因的结果,发现Adh1的核苷酸多态性和单倍型多态性都要明显高于Gapdh基因(见表3).
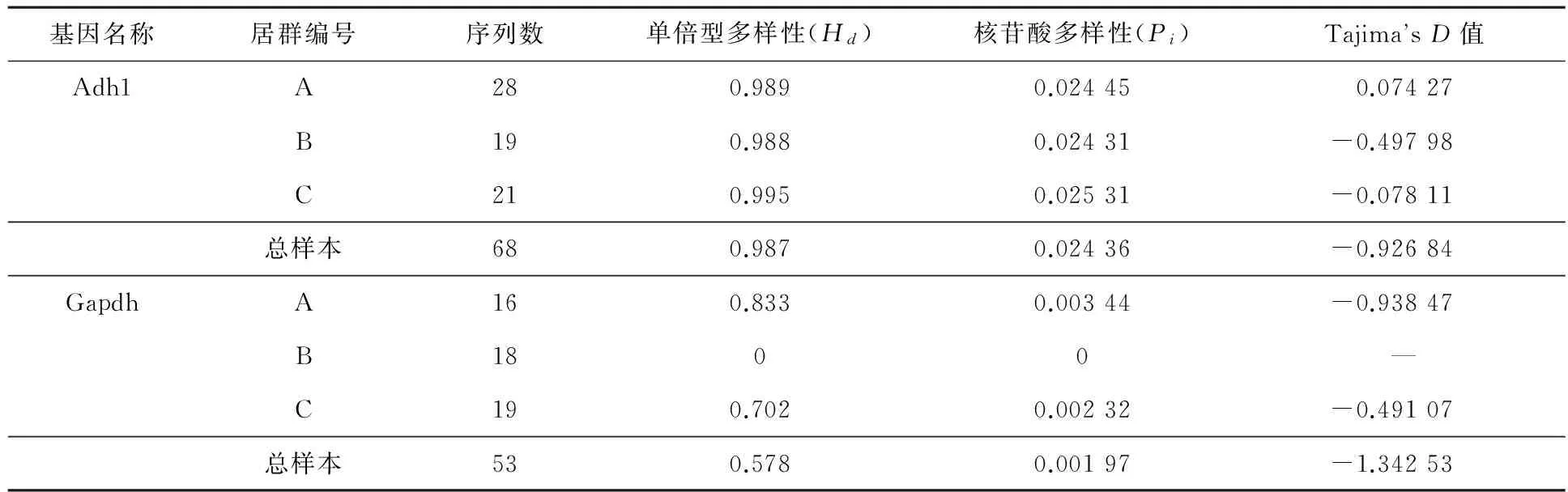
表3 野生杏居群的遗传多样性
2.2 中性检验与AMOVA分析
Tajima’sD是用于检验分子进化过程中中性学说的一个统计参数.从物种水平上对野生杏居群的Adh1基因和Gapdh基因序列进行中性检验,结果(见表3)显示,Adh1基因序列的Tajima’s D值不显著(P>0.10),说明该基因并未受到自然选择.与此类似,Gapdh基因序列的分析结果显示,除了巩留县核桃沟的野生杏居群外,另外2个居群的Tajima’sD为负值.负的D值表明含有比理论上更多的低频率的突变位点[13].
此外,通过对这2个基因进行的AMOVA分子方差分析表明,遗传变异主要发生在居群内,居群间变异很小(Adh1基因FST=-0.031 42,Gapdh基因FST=0.070 38)(见表4),这说明这3个群体的遗传分化较低.
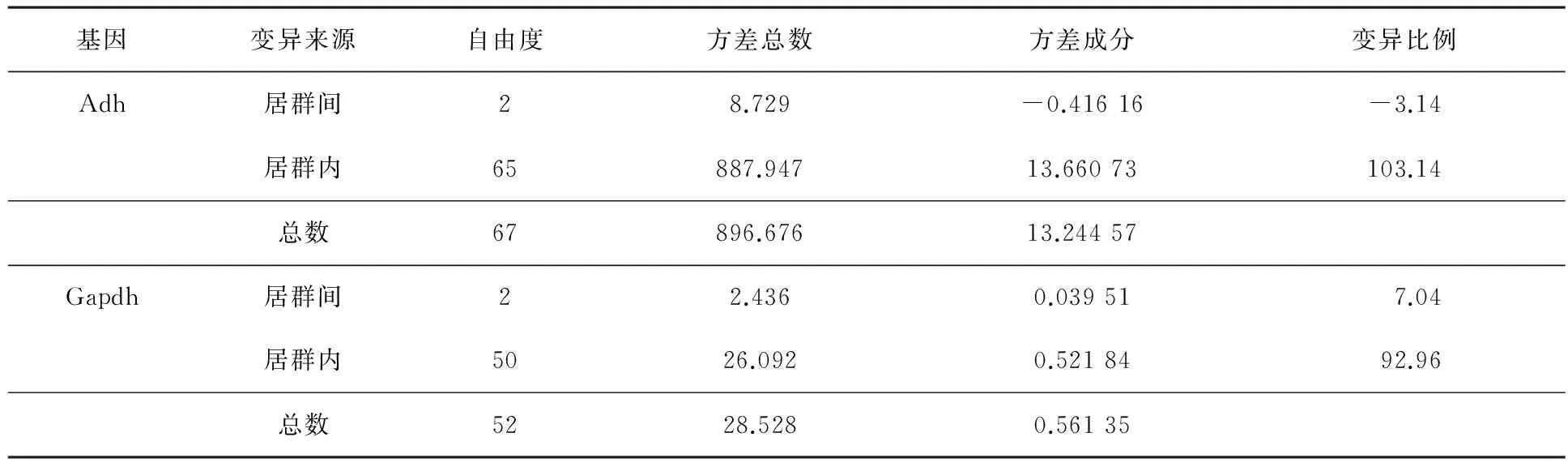
表4 基于Adh1和Gapdh基因序列的分子变异分析
3 讨论
3.1 野生杏群体的遗传多样性
原产新疆的杏是我国重要的野生果树之一,因此已有众多的学者对其遗传多样性进行了探讨.何天明等[5]利用SSR分子标记技术对伊犁河谷野生杏种群的遗传结构和多样性进行了研究,发现野生杏种群保持较高的遗传多样性(多态性带百分率P=82.4%,Shannon多样性指数为0.428);此外,苑兆和等[14]利用荧光AFLP标记技术对普通杏4个生态群(伊犁生态群、中亚生态群、欧洲生态群、华北生态群)的群体遗传结构进行了研究,结果表明伊犁生态群(野生杏)具有较高的遗传多样性(P=43.59%,H=0.131,I=0.202).本研究基于SNP分子标记技术对野生杏的遗传多样性进行了分析,结果表明这3个野生杏居群的Adh1基因具有较高的单倍型多态性和核苷酸多态性.相对而言,Gapdh基因的分析结果却显示,这3个野生杏居群的单倍型多态性和核苷酸多态性较低.
3.2 野生杏群体的遗传变异
根据AMOVA分子方差分析,野生杏群体的遗传变异主要发生在居群内,居群间的遗传变异很小.导致该现象的原因可能有2个:首先,各个居群间可能存在较大的基因流,从而阻止了居群之间的分化.已有的对野生杏的研究结果表明,野生杏居群间的遗传距离与居群间的地理分布间隔具有相关性,这说明了地理距离相近的群体之间存在基因流.[5]其次,这些居群可能是最近才分化的.图1显示,霍城县、巩留县分别位于新疆伊犁自治州的西北部和东南部,阿吾赞位于伊宁县境内,处于中部,这种地理分布对野生杏的遗传分化有着显著的影响.
3.3 野生杏遗传多样性资源保护
高水平的遗传多样性是种群稳定的基础.本研究中的野生杏居群保持了较高的遗传多样性,因此,对这些自然群体的保护是对野生杏种质资源保护的首要任务.此外,还可以考虑建立保护基地,建立核心种质基因库,通过人工授粉等措施促进基因交流,最大限度地保持野生杏种群的遗传多样性.在进行遗传多样性保护和种质资源保存的同时,应充分重视居群内不同类型个体的保存,同时也要在分布区不同区域内保存不同的群体.[15]
[1] 王磊.新疆野苹果和新疆野杏[J].新疆农业科学,1989(6):33-34.
[2] 于希志,廖新宇,张加延.新疆杏的资源特点与开发利用前景[J].落叶果树,1998(增刊):60-62.
[3] 刘黎明.新疆野杏生殖生态初步研究[D].乌鲁木齐:新疆农业大学,2010.
[4] 吕英民,吕增仁,高锁柱.应用同工酶进行杏属植物演化关系和分类的研究[J].华北农学报,1994,9(4):69-74.
[5] 何天明,陈学森,徐正,等.利用SSR标记对伊犁河谷野杏种群遗传结构的分析[C].中国园艺学会第十届会员代表大会暨学术讨论会论文集,2005.
[6] HORMAZA J I. Molecular characterization and similarity relationships among apricot (PrunusarmeniacaL.)genotypes using simple sequence repeats[J].Theor Appl Genet,2002,104:321-328.
[7] MARILYN L WARBURTON,FREDRICK A BLISS.Genetic diversity in peach (PrunuspersicaL.Batch) revealed by randomly amplified polymorphic DNA (RAPD) markers and compared to inbreeding coefficients[J].J AMER SOC HORT SCI,1996,121(6):1012-1019.
[8] 邹洪锋.野生、半野生和栽培大豆的单核苷酸多态性[D].南昌:南昌大学,2005.
[9] THOMPSON J D,GIBSON T J,PLEWNIAK F,et al.The CLUSTAL_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997,25:4876-4882.
[10] AHMED MANSOUR ALZOHAIRY.Bioedit:an important software for molecular biology[J].GERF Bulletin of Biosciences,2011(2):60-61.
[11] LIBRADO P,ROZAS J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009,25(11):1451-1452.
[12] EXCOFFIER L,LAVAL G,SCHNEIDER S.Arlequin3.0:an integrated software package for population genetics data analysis[D].Switzerland:Institute of Zoology,University of Berne,2006.
[13] ADA CHING,KATHERINE S CALDWELL,MARK JUNG,et al. SNP frequency,haplotype structure and linkage disequilibrium in elite maize inbred lines[J].BMC Genetics,2002(3):1-14.
[14] 苑兆和,陈学森,张春雨,等.普通杏群体遗传结构的荧光AFLP分析[J].园艺学报,2008(3):319-328.
[15] 李飞飞,崔大方,廖文波,等.中国新疆野苹果(Malussieversii(Ldb.) Roem.)种群地理分布格局及其遗传关系研究[J].干旱区地理,2011,34(6):926-932.
(责任编辑:方 林)
Genetic diversity of wild apricot (PrunusarmeniacaL.) in Xinjiang
NU ER Mai-mai-ti1,MAN Li2,WEN Ya-mei3,XIAO Hong-xing2
(1.Key Laboratory at Universities of Education Department of Xinjiang Uygur Autonomous Region,Yili Normal College,Yining 835000,China;2.School of Life Sciences,Northeast Normal University,Changchun 130024,China;3.Environmental Monitoring Station of Siping City,Siping,136000,China)
This study employed DNA sequencing technique to investigate the genetic diversity of three natural populations (AWZ,GONGLIU and HUOCHENG) of wild apricots (PrunusarmeniacaL.) distributing in Yili Valley,Xinjiang.According to the resulted sequences of Adh1 gene,the genetic population analyses showed that three natural populations of wild apricot possessed high levels of nucleotide and haplotype diversity.In contrast,the results of Gapdh revealed that relatively low nucleotide and haplotype diversity were found in the three populations.Specifically,no SNPs were identified in the population in GONGLIU.In addition,the analysis of AMOVA demonstrated that no significantly genetic differentiations were observed between the three populations.The findings provided framework for future studies of genetic conservation of wild apricots.
wild apricot;genetic diversity;SNP
1000-1832(2015)04-0111-05
10.16163/j.cnki.22-1123/n.2015.04.024
2014-04-02
自治区科技支疆项目(2014915);新疆教育厅普通高等学校重点实验室项目(2013YSHXYB08).
努尔买买提(1971—),男,博士研究生,讲师;通讯作者:肖洪兴(1963—),男,博士,教授,博士研究生导师,主要从事植物系统与进化研究.
Q 941+.2 [学科代码] 180·5155
A

