结核分枝杆菌Rv3134c蛋白抗原表位的预测
崔瑞娜,白雪娟,阳幼荣,梁 艳,张俊仙,赵卫国,吴雪琼
结核分枝杆菌Rv3134c蛋白抗原表位的预测
崔瑞娜1,2,白雪娟1,阳幼荣1,梁 艳1,张俊仙1,赵卫国2,吴雪琼1
目的 预测结核分枝杆菌Rv3134c的抗原表位,了解其免疫学特性。方法 利用DNAStar软件包中Protean软件对Rv3134c氨基酸序列进行分析,采用包括二级结构、亲水性、抗原性、表面可能性、柔韧性等多参数预测其二级结构及T细胞和B细胞抗原表位,最后BLAST分析其与人类抗原表位的同源性。结果 Rv3134c蛋白具有丰富的二级结构和多处抗原指数较高的区段,含有较多潜在的B细胞抗原表位,可能位于1-7、30-37、65-81、89-100、111-120、138-148、184-195、209-216、233-238位氨基酸残基或其附近,这些区域基本上含有β转角结构,亲水性、表面可能性和柔韧性指数都较高。该蛋白潜在的T细胞抗原表位较少,可能位于62-76、89-97、185-195、203-213、219-231、248-255位氨基酸残基或其附近。BLAST分析结果显示,其与人类抗原表位的同源性很低。结论 结核分枝杆菌Rv3134c是一个即含有较多B细胞抗原表位又含有较多T细胞抗原表位的蛋白抗原,实验结果为该蛋白抗原表位的进一步研究与合成肽疫苗奠定了基础。
结核分枝杆菌;Rv3134c蛋白;抗原表位;二级结构;预测
目前,结核病仍是全球最主要的健康问题之一,而结核潜伏感染(Latent tuberculosis infection,LTBI)者约占全球人口的三分之一,而大多数结核病患者来源于内源性复燃。研究结核分枝杆菌(Mycobacterium tuberculosis,MTB)潜伏感染相关蛋白抗原的免疫学特性,阐明MTB的休眠机制,对于结核病疫情的控制具有重要意义。免疫细胞识别抗原分子引起机体免疫应答,而抗原分子表面的抗原表位(Epitope)是免疫细胞受体识别的位点,因此,对MTB蛋白抗原表位的免疫学特性研究及优势表位的筛选和鉴定具有重要意义,并能进一步揭示免疫反应和结核病致病机理,为研发新疫苗和抗结核潜伏感染药物提供理论依据。
MTB Rv3134c编码基因全长807 bp,编码268个氨基酸,蛋白理论分子量为28.008 kDa,等电点为7.965。在低氧、一氧化氮和一氧化碳等刺激下,其表达上调[1],编码一种富含丙氨酸-缬氨酸的胁迫蛋白( universal stress protein)[2-3],对MTB抵抗胁迫环境、适应能量缺乏等起重要作用。Rv3134c基因与双组份系统DevR-DevS共转录,共同参与MTB 应对外界环境刺激所发生的代谢改变。Rv3134c基因启动子区域包含两个DevR绑定位点,与hspX、narK2和 Rv1738等多个DevR基因调节子共同调节MTB向非复制的休眠表型转变,在MTB向潜伏感染转变过程中起重要的调控作用[4-5]。由此可见,该蛋白可能成为清除潜伏感染的结核分枝杆菌、抑制其复燃的潜在的药物靶点。因此,本研究运用DNAStar生物信息学软件对MTB Rv3134c蛋白的二级结构、T细胞和B细胞抗原表位进行初步的预测,为进一步地筛选、鉴定Rv3134c蛋白抗原的优势表位,并进行功能分析及免疫学特性研究奠定理论基础。
1 材料与方法
1.1 MTB Rv3134c蛋白氨基酸序列 从NCBI网站获取其氨基酸全长序列(268个氨基酸)如下:

1.2 方法
1.2.1 抗原表位分析软件 DNAStar软件包是DNASTAR有限公司开发的生物软件,可在计算机上进行DNA和蛋白质分析。用DNAStar软件包中Protean软件对蛋白的二级结构和抗原表位进行分析和预测[6-7]。
1.2.2 BLAST分析 采用EXPASY在线软件(NCBI BLAST2 service)对Rv3134c蛋白氨基酸序列与人类蛋白质的同源性进行分析,具体方法:利用Internet 网络进入EXPASY主页(http://web.expasy.org/blast/),选择蛋白数据库[blastp - query against the UniProt Knowledgebase (Swiss-Prot +4 TrEMBL)],选择database (Homo sapiens),选定Run BLAST,输入Rv3134c蛋白的氨基酸序列后进行比对。
1.2.3 Rv3134c蛋白二级结构的预测 采用Gamier-Robson方法、Chou—Fasman方法和Coiled Coil方法预测Rv3431c蛋白二级结构。
1.2.4 Rv3134c蛋白抗原表位的预测 用Kyte-Doolittle方法、Hopp-Woods方法和Eisenberg方法分析Rv3134c蛋白的疏水性和亲水性;用Emini方法预测蛋白表面可及性;用Karplus-Schulz方法预测蛋白质骨架区的柔韧性;以Jameson-wolf方法预测蛋白潜在的抗原表位,以AMPHI方法预测蛋白免疫优势辅助性T淋巴细胞抗原位点,以Rothbard-Taylor方法预测含有特定基序(motif)的潜在T淋巴细胞抗原表位,以Sette MHC Motifs预测短肽上与老鼠 MHC II d 型蛋白质相互作用的抗原位点,最后综合评价Rv3134c蛋白抗原表位。
2 结 果
2.1 Rv3134c蛋白二级结构和抗原表位预测
2.1.1 Rv3134c蛋白二级结构的预测 综合应用Gamier-Robson、Chou-Fasman和Coiled Coil 3种方法预测Rv3134c蛋白二级结构,结果见(图1):α螺旋较多,分布较均匀,位于17-35、49-52、58-75、79-86、88-103、120-124、126-129、143-212、214-221、226-234、242-252、265-268位氨基酸残基;不存在跨膜区的α螺旋结构;β折叠也较多,位于7-15、36-52、80-86、104-115、120-125、130-137、143-147、149-154、160-165、171-174、214-220、238-242、258-264位氨基酸残基;Chou-Fasman法显示存在较多长度相仿的转角,且分布较均匀;在2-5、55-57、76-79、138-142、211-213位氨基酸残基存在无规则卷曲。
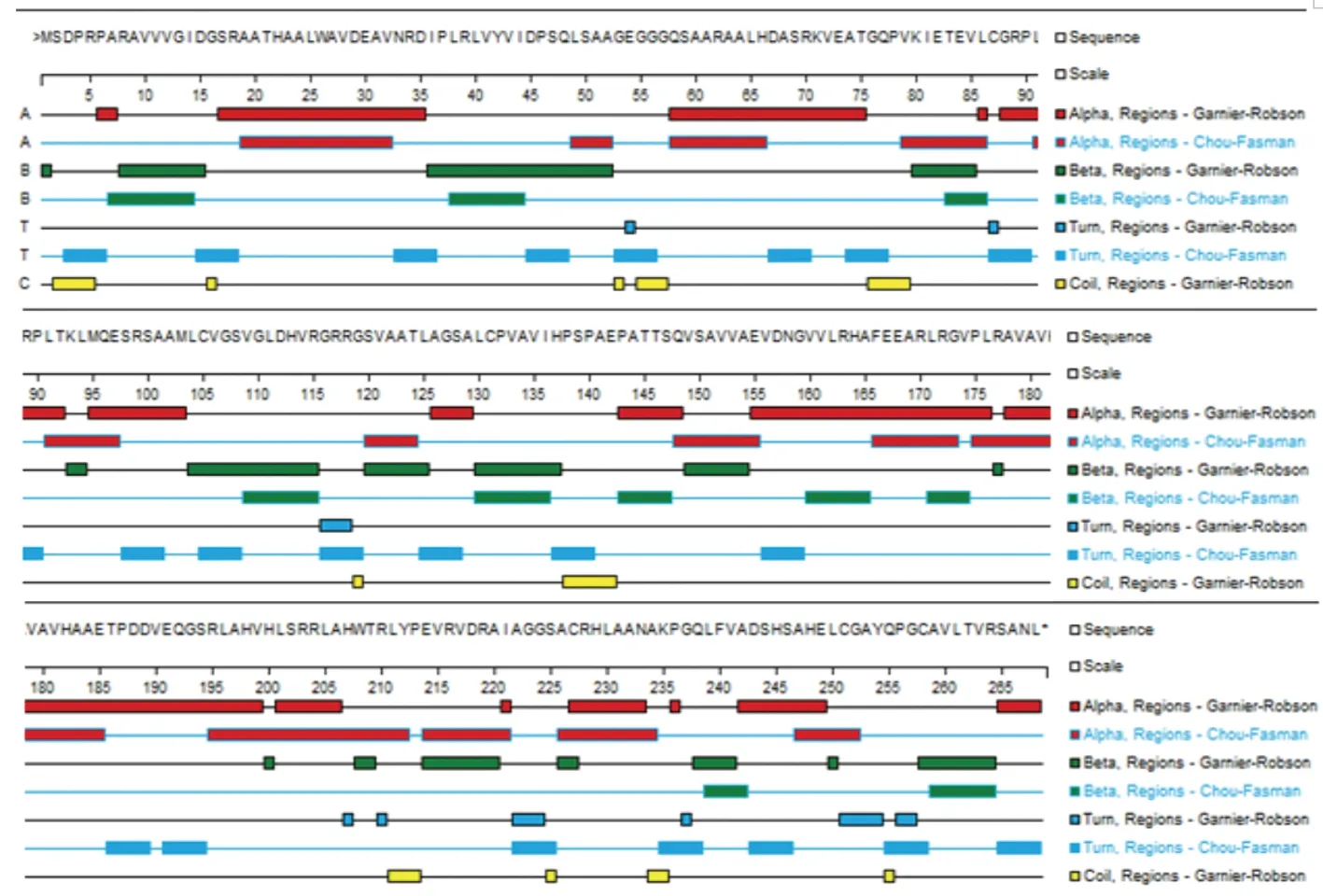
图1 结核分枝杆菌Rv3134c蛋白二级结构预测
2.1.2 Rv3134c蛋白的亲水性分析 用Kyte-Doolittle方法分析Rv3134c蛋白的疏水性和亲水性,结果(图2)显示该蛋白疏水性区域位于8-16、21-29、38-48、82-85、87-91、101-110、121-138、148-160、162-164、175-183、221-226、254-267位氨基酸残基,以8-16、101-110、121-138、148-160、175-183、254-267位氨基酸残基疏水性指数较高;亲水性区域1-7、17-20、30-37、49-52、54-60、65-81、92-100、111-120、139-147、165-174、184-196、199-217、231-237、244-249、251-253,以1-7、65-81、92-100、112-120、184-196、199-217位氨基酸残基等处的亲水性指数较高。
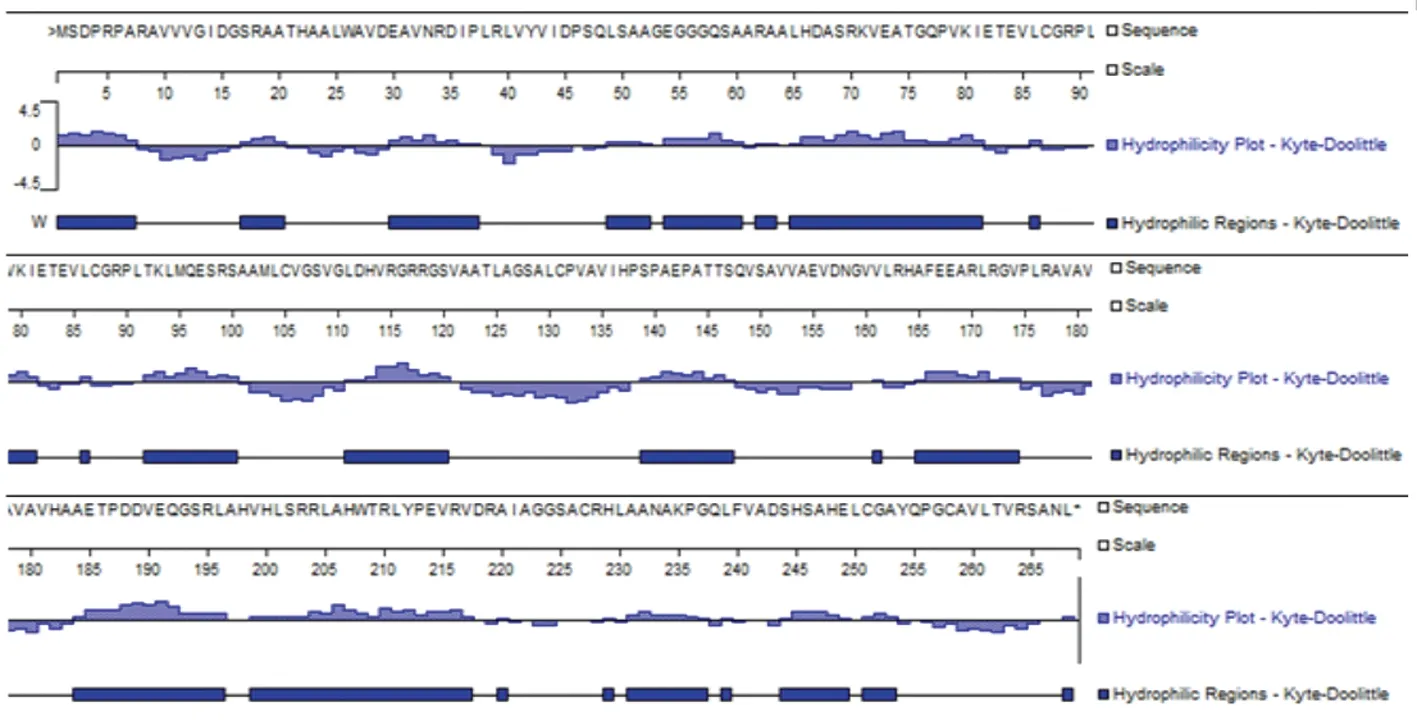
图2 结核分枝杆菌Rv3134c蛋白的亲水性和疏水性分析
2.1.3 Rv3134c蛋白的表面可能性分析 Rv3134c蛋白呈现在表面可能性较大的区域主要是在1-7、67-72、94-99、114-117、139-145、184-193、209-216、233-236位氨基酸残基,其他部位展示的可能性较小或表现为负值,见图3。
2.1.4 Rv3134c蛋白的柔韧性分析 Rv3134c蛋白含有较多的柔韧性区域位于5-7、14-19、33-35、45-48、53-59、68-83、89-93、96-100、115-120、138-148、156-159、185-195、223-225、234-238位氨基酸残基,见(图4)。

图3 结核分枝杆菌Rv3134c蛋白表面可能性区域

图4 结核分枝杆菌Rv3134c蛋白柔韧性区域
2.1.5 Rv3134c蛋白B细胞抗原表位的预测分析 Rv3134c蛋白含有许多抗原指数较高的区域,见(图5)。提示这些区段含有潜在优势抗原表位,如1-9、15-22、30-39、45-49、51-62、64-102、112-122、137-146、154-159、165-176、185-196、201-206、212-229、234-239、243-248、256-258、265-268位氨基酸残基区域。再结合蛋白质二级结构、亲水性、表面可能性和柔韧性,综合分析 Rv3134c蛋白潜在的蛋白抗原决定簇,在1-7、30-37、65-81、89-100、111-120、138-148、184-195、209-216、233-238位氨基酸残基区域β转角和无规则卷曲结构较多、亲水性和表面可能性指数较高、柔韧性较大,暴露于表面的几率较大,且易发生扭曲、折叠,提示作为B细胞抗原表位的可能性最大。
2.1.6 Rv3134c蛋白T细胞抗原表位的预测分析 用Rothbard-Taylor方法预测含有特定基序(motif)的潜在T淋巴细胞抗原表位,结果(图6)显示,18-22、30-33、62-66、73-76、93-96、159-167、182-185、195-198、204-213、219-223、229-233、252-255位氨基酸残基处是潜在的T细胞抗原表位,且分布较均匀。以AMPHI方法预测48-53、55-59、65-75、89-97、123-125、142-147、149-156、181-183、185-195、203-212、224-231、248-254位氨基酸残基可能是免疫优势的辅助性T淋巴细胞抗原位点,7-12、14-19、38-43、46-51、113-119、121-126、144-154、160-165、176-186、195-206、215-220、260-266位氨基酸残基可能是与小鼠 MHC IId型蛋白质相互作用的抗原位点。综合分析,62-76、89-97、185-195、203-213、219-231、248-255位氨基酸残基处作为T细胞抗原表位的可能性较大。

图5 结核分枝杆菌Rv3134c蛋白抗原指数分析
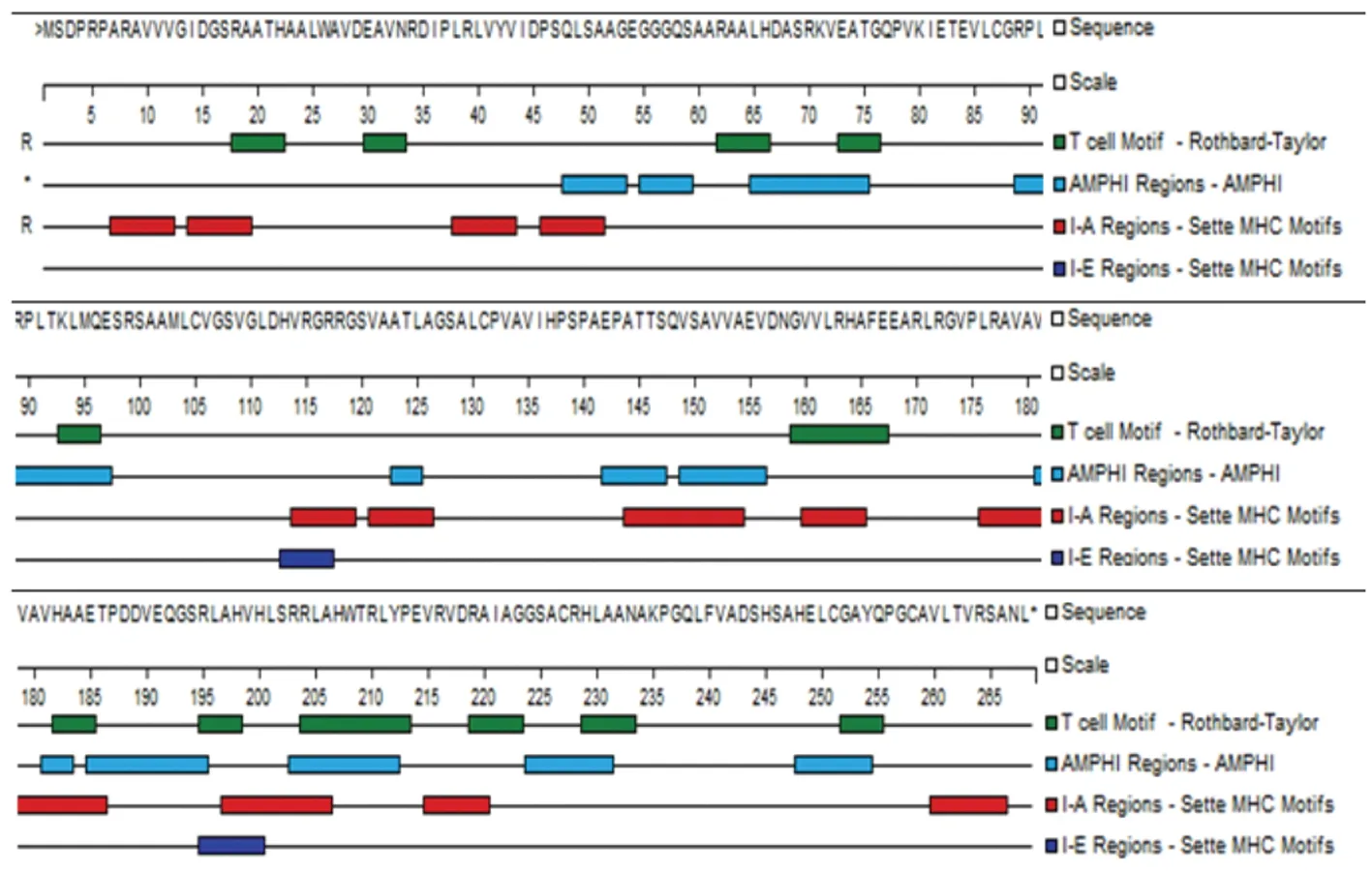
图6 结核分枝杆菌Rv3134c蛋白T细胞抗原表位的预测
2.2 MTB Rv3134c蛋白与人类蛋白质氨基酸序列BLAST分析 通过BLAST分析,发现Rv3134c蛋白的氨基酸序列与人半椎蛋白-2前体(Hemicentin-2 precursor)只有12%(32/268个氨基酸)的同源性,同源性主要位于第76~160位氨基酸残基,这段同源性达到30%。
3 讨 论
由于MTB生长缓慢,天然蛋白抗原获取困难,常常成为研究MTB致病机理的障碍。通过基因工程技术获得重组蛋白抗原是一个简便、有效的途径[8-9]。而生物信息学分析和预测蛋白优势抗原表位为重组蛋白抗原奠定了前期基础,增强了实验的目的性,减少了不必要的人力、物力和时间的浪费,目前已被越来越多的研究者采用[10-11]。
MTB的双组分调节系统DevS、DevT和DevR分别由Rv3132c、Rv2027c和Rv3133c基因编码,其中Rv3133c、Rv3132c和 Rv3134c基因构成操纵子,Rv3134c/Rv3133c/Rv3132c基因组在缺氧情况下能被快速的诱导[2,5]。Sherman[12]研究发现,敲除Rv3134c 编码序列后,下游的基因表达受影响,Rv3134c蛋白可能参与了双组分系统的磷酸化过程。Saini[13]等研究发现MTB强毒株H37Rv在低氧条件下DevR蛋白增加的水平与Rv3134c-devR-devS操纵子诱导的RNA水平上调是相关的。Kaur[14]等最近也发现,DevRS A-ext和DevRS D模拟肽抑制Rv3134c启动子活性的同时,DevR调节子其他成员的表达也受损。这些研究结果均表明Rv3134c蛋白在MTB适应缺氧中起重要作用。
本文通过生物学软件对Rv3134c蛋白的抗原表位首次进行初步的预测,发现1-7、67-72、96-100、115-119、139-145、185-194、234-236位氨基酸残基是潜在的B细胞抗原表位;73-75、93-97、204-212、229-231、252-254位氨基酸残基是潜在的T细胞抗原表位。另外,通过BLAST分析,发现Rv3134c蛋白的氨基酸序列与人半椎蛋白-2前体的同源性很低,少数同源性主要位于第76~160位氨基酸残基。因此,在抗原表位设计、合成时避开这些同源性区域就可获得交叉反应性低、特异性高的抗原表位。这些预测数据为进一步研究其在潜伏感染中的作用奠定了理论基础。但由于预测软件的局限性和机体免疫反应的复杂性,预测结果只能作为参考,应综合应用多个软件进行预测并做体外试验验证。
总之,MTB Rv3134c蛋白既含有潜在的B细胞抗原表位,又含有潜在的T细胞抗原表位,本研究的抗原表位预测结果为该蛋白免疫学特性的深入研究,揭示MTB的潜伏感染机制奠定了理论基础。
[1]Hingley-Wilson SM, Lougheed KEA, Ferguson K, et al. IndividualMycobacteriumtuberculosisuniversal stress protein homologues are dispensableinvitro[J]. Tuberculosis, 2010, 90(4): 236-244. DOI: 10.1016/j.Tube.2010.03.013
[2]Bagchi G, Chauhan S, Sharma D, et al. Transcription and autoregulation of the Rv3134c-devR-devS operon ofMycobacteriumtuberculosis[J]. Microbiology, 2005, 151: 4045-4053. DOI: 10.1099/mic.0.28333-0
[3]Fallow A, Domenech P, Reed MB. Strains of the East Asian (W/Beijing) lineage of Mycobacterium tuberculosis are DosS/DosT-DosR two-component regulatory system natural mutants[J]. J Bacteriol, 2010, 192(8): 2228-2238. DOI: 10.1128/JB. 01597-09
[4]Mayuri, Bagchi G, Das TK. Molecular analysis of the dormancy response inMycobacteriumsmegmatis: expression analysis of genes encoding the DevR-DevS two-component system, Rv3134c and chaperone K-crystallin homologues[J]. FEMS Microbiol Lett, 2002: 231-237.DOI: 10.1111/j.1574-6968.2002.tb11230.x
[5]Bandyopadhyay A, Biswas S, Maity AK, et al. Analysis of DevR regulated genes inMycobacteriumtuberculosis[J]. Systems Synthetic Biol, 2014, 8(1): 3-20. DOI: 10.1007/s11693-014-9133-y
[6]Zhu YJ, Liu B, Zheng WW, et al. Gene sequence of the protein structural analysis software Protean use of technology[J]. Fujian Agr Biotechnol Communications, 2004, 6(4): 8-12. (in Chinese) 朱育菁,刘波,郑伟文,等.基因序列蛋白质结构分析软件Protean使用技术[J].福建农业生物技术通讯,2004, 6(4):8-12.
[7]Wang GX, Shang YJ, Lü ZL, et al. Prediction of secondary structures and cell epitopes of F1L protein of ORFV[J]. Chin J Zoonoses, 2012, 28(12): 1185-1190. DOI: 10.3969/cjz.j.issn.1002-2694.2012.12.006 (in Chinese) 王光祥,尚佑军,吕占禄,等.羊口疮病毒F1L蛋白二级结构分析与表位测[J].中国人兽共患病学报, 2012, 28(12):1185-90.
[8]Yang YR, Wu XQ, Zhao WG, et al. Preparation and preliminary application of fusion protein Rv0057 Rv1352 ofMycobacteriumtuberculosis[J]. Chin J Biologicals, 2013, 26(6): 851-856. (in Chinese) 阳幼荣,吴雪琼,赵卫国.结核分枝杆菌Rv0057-Rv1352融合蛋白的制备及其初步应用[J].中国生物制品学杂志,2013, 26(6): 851-856.
[9]Lu JB, Chen BW, Wang GZ, et al. Recombinant tuberculosis vaccine AEC/BC02 induces antigen-specific cellular responses in mice and protects guinea pigs in a model of latent infection[J]. J Microbiol Immunol Infect, 2014, pii: S1684-1182(14)00060-7. DOI: 10.1016/j.jmii.2014.03.005
[10]Greory AP, lnna GO, Kenneth LJ, et al. The role of mass spectrometry in vaccine development[J]. Vaccine, 2001(17-19): 2692-2700. DOI: 10.1016/S0264-410X(00)00505-3
[11]Zhu DD, Duan YN, Chen YL. Prediction on the antigenic epitopse of the kexin protein fromPneumocystiscarinii[J]. Mod Prevent Med, 2011, 38(6): 1077-1079. (in Chinese) 朱丹丹,段义农,陈金铃.卡氏肺孢子虫 kexin 蛋白抗原表位预测[J].现代预防医学,2011,38(6):1077-1079.
[12]Sherman DR, Voskuil M, Schnappinger D, et al. Regulation of the Mycobacterium tuberculosis hypoxic response gene encoding α-crystallin[J]. Proc Natl Acad Sci U S A, 2001, 98(13): 7534-7539. DOI: 10.1073/pnas.121172498
[13]Saini DK, Malhotra V, Dey D, et al. DevR-DevS is a bona fide two-component system of Mycobacterium tuberculosis that is hypoxia-responsive in the absence of the DNA-binding domain of DevR[J]. Microbiology, 2004, 150: 865-875.DOI: 10. 1099/mic.0.26218-0
[14]Kaur,Taneja NK, Dhingra S, et al. DevR (DosR) mimetic peptides impair transcriptional regulation and survival ofMycobacteriumtuberculosisunder hypoxia by inhibiting the autokinase activity of DevS sensor kinase[J]. BMC Microbiol, 2014, 14: 195. DOI: 10.1186/1471-2180-14-195
s: Wu Xue-qiong, Email: wu-xueqiong@263.net; Zhao Wei-guo, Email: zhaowg309@163.com
Prediction of epitopes ofMycobacteriumtuberculosisRv3134c protein
CUI Rui-na1,2,BAI Xue-juan1,YANG You-rong1,LIANG Yan1, ZHANG Jun-xian1,ZHAO Wei-guo2,WU Xue-qiong1
(1.ArmyTuberculosisControlandPreventionKeyLaboratory/BeijingKeyLaboratoryofTuberculosisNewDiagnosisandTreatmentTechnology/InstituteofTuberculosisResearch,Beijing100091,China; 2.DepartmentofRespiratoryMedicine,the309thHospitalofChinesePLA,Beijing100091,China)
In this study, we predicted the epitopes ofMycobacteriumtuberculosisRv3134c protein and investigated its immunological characteristics. The amino acid sequence of Rv3134c protein was input and predicted the epitopes of B cell and T cell using multi-parameters including the secondary structure, hydrophilicity, antigenicity, accessibility, flexibility, charge distribution by Protean software of DNAStar software package. Results showed that the Rv3134c protein had rich secondary structure and multiple sections with higher antigenicity. There were a few potential B cell epitopes at 1-7, 30-37, 65-81, 89-100, 111-120, 138-148, 184-195, 209-216, and 233-238 amino acid residues or nearby, and theses epitopes had better antigenicity, contained beta angle and irregular coil structure, presented in the surface probability and had larger flexibility. There also were more potential T cell epitopes of the protein containing at 62-76, 89-97, 185-195, 203-213, 219-231, 248-255, amino acid residues or nearby. There was low homology between Rv3134c protein and human protein by BLAST analysis. In conclusion,MycobacteriumtuberculosisRv3134c protein had more B-cell epitopes and T-cell epitopes, which will lay the foundation for its further study and the development of vaccine.
Mycobacteriumtuberculosis; Rv3134c protein; epitope; secondary structure; prediction
国家重大传染病防治科技重大专项基金资助项目(No.2012ZX10003008002),军队医学科技“十二五”重点项目(No.BWS11J050),北京市科技创新基地培育与发展工程专项项目(No.Z141107004414021)
吴雪琼,Email: wu-xueqiong@263.net; 赵卫国,Email: zhaowg309@163.com
1.解放军第309医院全军结核病研究所,全军结核病防治重点实验室,结核病诊疗新技术北京市重点实验室;北京 100091; 2.解放军第309医院呼吸科,北京 100091
Supported by grants from the National Science and Technology Major Infectious Diseases of Major Funded Project (No. 2012ZX10003008002), the Military Medical Science "Twelve Five" Key Project (No. BWS11J050), the Beijing Technology Innovation Base Nurturing and Development Special Projects (No. Z141107004414021)
10.3969/cjz.j.issn.1002-2694.2015.06.010
R378.91
A
1002-2694(2015)06-0541-06
2014-12-05;
2015-03-30

