组蛋白甲基化转移酶G9a的最新研究进展
薛伟丽,孙瑜,郑凌
(武汉大学生命科学学院,武汉430072)
引言
组蛋白修饰指发生在组蛋白氨基酸残基上的一系列化学修饰,包括乙酰化、甲基化、磷酸化、泛素化和ADP核糖基化等[1,2]。组蛋白修饰是表观遗传学的一种主要机制,在多种生命过程中扮演着重要的角色[1,2]。其中,组蛋白甲基化因其参与了异染色质形成、基因印记、X染色体失活和基因的转录调控等过程,而日益受到大家重视[3]。
组蛋白甲基化转移酶(Histone methyltransfereases,HMTs)是一类催化1~3个甲基基团转移到组蛋白赖氨酸或精氨酸上的酶[4],分成组蛋白-赖氨酸N-甲基转移酶(Histone lysine methyltransfereses, HKMTs)和组蛋白-精氨酸N-甲基转移酶(Histone arginine methyltransferases , HRMTs)。其中,HKMTs可细分为含SET结构域和不含SET结构域的两个亚类[5]。SET结构域由最早发现含有该结构域的3个基因,Su(var)3-9, Enhancer of zeste和Trithorax来命名。该结构域含约110个氨基酸,序列高度保守,具有独特的折叠结构[6,7]。以酵母组蛋白甲基化转移酶Clr4为例,其HMT催化区域含Pre-SET 序列、SET结构域和Post-SET序列(图1)。研究发现,含有SET结构域的蛋白质多具有HMT活性。但并非所有含有SET结构域的蛋白质都能甲基化组蛋白,如甲基转移酶Rubisco LSMT(Rubisco large subunit methyltransferase),虽然含有SET结构域,但不能甲基化组蛋白, 只能对一些非组蛋白进行甲基化修饰[8]。同样也存在一些不含SET结构域的HMTs,如Dot1是首个被发现的无SET结构域的HKMT,可以催化H3K79甲基化[9,10],在一定程度上扩展了HMT的研究领域。此外,不少HMTs除了能甲基化组蛋白,还能甲基化非组蛋白,并通过这种修饰来调控这些蛋白的功能[11,12]。蓝色、红色、灰色和绿色分别表示N端、Pre-SET 序列、SET结构域和Post-SET序列;圆柱形表示α螺旋、箭头表示β折叠、线段表示无规则卷曲结构;N、C分别表示氨基酸序列的氨基端和羧基端。
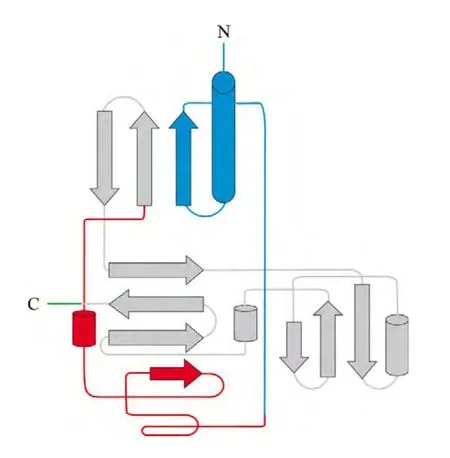
图1酵母Clr4中HMT催化结构域的模式结构图(根据[13]进行一定修改)Fig. 1The structure of HMTase domain of histone methyltransferase Clr4 from Schizosaccharomyces pombe
G9a属于含SET结构域的Suv39h蛋白家族的成员之一,是一种重要的常染色质HMT[14]。由于各种敲除或过表达G9a细胞系的获得,以及全身性和多种组织特异性G9a敲除小鼠的研发,使得近年对G9a的研究进展十分迅速。本文对G9a结构和功能上的最新研究进展做一综述,并就其在多种疾病的预防与治疗中的作用进行相关展望。
2 G9a的发现与结构特点
G9a,又称常染色质组蛋白赖氨酸N-甲基转移酶2 (euchromatic histone-lysine N-methyltransferase 2,EHMT2),在Homo sapiens中由EHMT2基因编码[15]。研究发现其C端含有SET结构域,并且与两个富含半胱氨酸的区域相邻,具有HMT活性[16]。
G9a在哺乳动物中序列十分保守[17],人源、牛源、大鼠与小鼠的G9a同源性高达95.0%。小鼠G9a的N端含富含谷氨酸和半胱氨酸区域,可能参与G9a自身的甲基化[18],并含有核定位信号(NLS);中间区域是G9a的功能核心区域,由具有锚定功能的六组Ankyrin(ANK)重复序列组成,形成α螺旋-β转角-α螺旋-β转角的空间结构,用于对底物的甲基化位点识别[19];C端区域主要是SET结构域,行使蛋白甲基化功能[20]。在SET结构域的前后分别有Pre-SET和Post-SET序列,分别起着稳定G9a蛋白结构和促进甲基化酶活性中心形成的作用[21](图2)。一般说来,G9a有两种不同长短的异构体。长异构体(L-G9a)表达全长的G9a氨基酸序列,而短异构体(S-G9a)在N端存在一段氨基酸的缺失[22](图2)。但这两种异构体在功能上的差异还有待进一步研究。
G9a在多种组织中广泛表达。我们检测了正常小鼠不同组织中G9a的表达情况(图3A)。结果表明,G9a在肌肉、心脏、脑等组织中高表达,而在白色脂肪、脾脏及肺等组织中表达较低。即使在同一种组织的不同类型细胞中,G9a的表达量也有较大差异。我们发现在体外培养的大鼠视网膜不同细胞系中,G9a在神经节细胞系RGC-5中的表达量明显高于其在神经胶质细胞系rMC-1中的表达量(图3B)。我们进一步发现G9a在人神经母细胞瘤细胞系SH-SY5Y中表达量也较高(图3B)。
3 G9a调控基因转录的功能
3.1 G9a通过甲基化组蛋白来抑制基因转录
G9a是继Suv39h1后发现的第二个哺乳动物中甲基化赖氨酸的HMT,主要催化组蛋白H3K9和K27位点。G9a 将甲基从SAM(S-adenosyl-l-methionine)上转移到目的赖氨酸残基的ε—氨基上[16]。Suv39h1/h2和G9a都可以催化H3K9位点的单、双以及三甲基化反应[23,24],其中Suv39h1/h2催化组蛋白H3K9异染色质区域的甲基化,而G9a则催化其在常染色质区域的甲基化[14, 25, 26]。
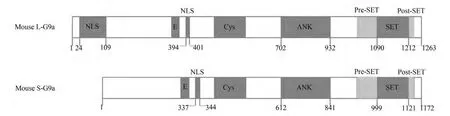
图2鼠源G9a的两种异构体结构示意图[22]Fig. 2Schematic information about different domains of mouse G9aNLS:核定位信号;E:谷氨酸富集区;Cys:半胱氨酸富集区;ANK:ANK重复序列;Pre-SET:Pre-SET序列;SET:SET结构域;Post-SET:Post-SET序列;数字表示氨基酸残基
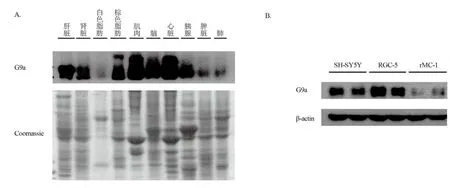
图3不同组织和细胞中G9a的表达情况Fig. 3The expression pattern of G9a in different mouse tissues and cultured cell linesA. 雄性C57BL/6小鼠(3个月大)不同组织中G9a的表达情况(上图)和相应样本中考马斯亮蓝的染色结果(下图)。B. 不同类型的细胞中G9a的表达情况(上图)和相应样本中β-actin的表达情况(下图)。
G9a与GLP(G9a-like protein)关系密切,人源G9a与GLP的相似度达到45%,主要在N端存在较大差异[27],两者结构也极其相似[28]。体外实验表明,两者都有H3K9甲基化酶活性,可以单独行使HMT功能[16,23,29]。但体内研究却发现,两者通常通过SET结构域形成异二聚体,共同行使HMT功能。在细胞内单独敲除任何一个,都会造成H3K9me1和 H3K9me2水平显著性下降;但两者的双敲除不会进一步降低H3K9me1和H3K9me2水平[20],表明两者不能补偿对方的甲基化酶功能的缺失。
化合物BIX01294和UNC0638特异性抑制负责甲基化H3K9位点的HMTs活性,构象研究发现它们通过占据HMTs中与底物结合的部位,从而抑制甲基化反应[30],用它们处理细胞都可以显著性降低H3K9me2的水平[24,31]。 BIX01294被认为是G9a的特异性抑制剂,UNC0638是G9a及GLP的甲基化酶活性抑制剂,比BIX01294具有更强的抑制作用[32]。
由于H3K9位点的单/双甲基化(H3K9me1/2)和H3K27位点甲基化(H3K27me)会募集抑制子,如HP1家族成员,导致DNA和组蛋白结合更加紧密,从而抑制基因转录[33]。因此,G9a可以通过其HMT活性对一些基因的启动子区域上的组蛋白进行H3K9和H3K27位点的甲基化修饰,从而调控基因的转录沉默[34]。同时,生物芯片结果显示,胚胎干细胞中G9a的缺失可以激活一系列启动子区域富含H3K9me2的基因的表达[35],表明G9a抑制基因转录是广泛存在的。
3.2 G9a通过维持启动子区域DNA甲基化来抑制基因转录
G9a不仅能调控组蛋白的甲基化,而且也能调控DNA甲基化程度。与组蛋白修饰相同,DNA甲基化也是一种表观遗传修饰,是指在DNA甲基转移酶(DNA methylation transferase,DNMT)的作用下,DNA链上CG中的胞嘧啶接受一个甲基形成5'-mCG-3'[36]。因此,DNA甲基化多发生在5'-CG-3'富集区域。DNMTs主要分为两大类,DNA甲基化维持酶DNMT1和DNA从头甲基化酶DNMT3a、DNMT3b及DNMT3L等[37]。DNA甲基化通常与基因转录抑制相关,在哺乳动物的胚胎发育和细胞分化过程中起着不可或缺的作用,其异常也与多种疾病如肿瘤的发生发展密切相关[38]。
研究表明,在胚胎干细胞中敲低G9a会引起下游靶基因Mage-a2启动子和Wfdc15a的5'UTR区域甲基化程度下调[39]。对该种细胞整个基因组的DNA甲基化水平进行研究发现,G9a缺失会造成大部分CpG岛中CG位点的低甲基化状态,特别是逆转录转座子和卫星DNA区域的甲基化显著减少[40,41]。
G9a一方面可通过其甲基化酶活性来调控DNA的甲基化。研究发现在粗糙脉孢菌和植物中,H3K9的甲基化可以调控DNA甲基化水平[42,43]。在小鼠胚胎干细胞DNA复制过程中,半甲基化DNA和复制叉处的H3K9me2/3可先招募UHRF1(Ubiquitin-like, containing PHD and RING finger domains,1),定位于复制叉上的UHRF1进而招募DNMT1完成DNA全甲基化[44],从而实现DNA甲基化在细胞增殖过程中的延续。另一方面,G9a还能直接通过DNMTs来调控DNA甲基化水平。多种细胞实验表明,在DNA复制过程中,DNMT1和G9a相互结合来维持DNA复制过程中甲基化信息的传递[45]。除了DNMT1之外,G9a还通过ANK结构域募集DNMT3a和DNMT3b,来维持基因启动子上DNA甲基化,从而调控基因沉默[46]。这种G9a调控DNA甲基化的方式并不依赖其甲基化酶活性,因为在G9a敲除的胚胎干细胞中过表达SET结构域缺失的G9a,仍然可以改善由于G9a敲除引发的逆转录转座子低甲基化状态[40]。此外,G9a 的SET结构域点突变也不影响G9a和DNMT3a/3b的相互作用[46]。因此,在G9a通过DNMTs来调控DNA甲基化的过程中,G9a一般扮演着脚手架蛋白的作用,与DNMTs共同调控基因沉默。
3.3 G9a作为脚手架蛋白募集转录激活子从而激活基因转录
Bittencourt等在对敲低G9a的人肺癌A549细胞进行基因表达谱分析中,发现在200个差异表达的基因中,有约25%的基因表达显著降低[47],这与G9a的转录抑制作用不符。不仅在肺癌细胞中,在乳腺癌细胞中,G9a的敲低可以正调控一部分雌激素诱导的靶基因表达,同时抑制另一部分靶基因表达[48]。同样的,在神经母细胞瘤细胞和骨肉瘤细胞中,过表达G9a可以促进细胞周期相关基因的表达。这些数据都表明在某些特定条件下,G9a对基因转录有激活作用。
进一步研究表明,G9a可与多种转录激活子相互结合,来协同激活基因转录。例如,在类固醇激素受体调控其靶基因转录的过程中,类固醇激素的刺激可以诱导 G9a募集该受体,并通过G9a结构域进一步募集,如谷氨酸受体相互作用蛋白(Glutamate receptor interacting protein1, GRIP1)、精氨酸甲基化酶(Coactivator-associated arginine methylthrasferase,CARM1)和组蛋白乙酰化酶p300(E1A binding protein p300,p300)等转录激活子,形成巨大的转录复合体,协同促进下游靶基因的转录[49]。在糖皮质激素受体诱导下游基因表达的过程中,也存在着相似的G9a激活基因转录的作用。在人肺癌细胞中,G9a在糖皮质激素刺激下,能够募集CARM1和p300到糖皮质激素受体调控的靶基因ENaCα和CDH16的启动子上,促进这些基因的转录[47]。在这些过程中,G9a与激活子协同激活基因转录的作用并不依赖其甲基化酶活性,而是通过其相关结构域来募集其它转录激活子[47,49]。
4 G9a参与多种生物反应过程
由于其在基因转录上的重要调控作用,所以G9a对机体的生长和发育是至关重要的。最直接的证据是全身性G9a敲除小鼠胚胎致死[14,20]。通过对G9a全身性和条件性敲除小鼠的研究,发现该蛋白与胚胎发育、生殖细胞的发育及减数分裂、肿瘤细胞的生长及转移、认知及适应性行为和脂肪细胞分化等生物过程都有着密切联系。
4.1 G9a 与胚胎发育
G9a全身敲除小鼠由于严重的发育缺陷,在胚胎9.5天(ED9.5)左右死亡[14,20]。由于G9a SET结构域突变的小鼠也像全身敲除小鼠一样,具有胚胎致死的表型[14,20],因此胚胎的正常发育需要G9a的HMT活性。缺乏G9a或其HMT活性使得小鼠胚胎中H3K9me2水平显著下降,同时H3K4甲基化水平显著升高[14,20],因此推测这两种组蛋白甲基化修饰的改变,共同抑制了发育过程中某些关键基因的表达,从而引发胚胎致死。进一步研究表明,G9a全身敲除小鼠的胚胎中,细胞凋亡的数目明显增多[50],这和细胞水平上G9a敲除能引发自噬,从而引起细胞死亡是相符的[51,52],但遗憾的是,论文作者并没有对这一方面进行深入研究,因此,G9a调控胚胎发育的具体机制仍需进一步研究。
在生殖细胞中特异性敲除G9a,导致生殖细胞无法进行减数分裂,从而引发生殖细胞发育缺陷[50]。在该品系小鼠的生殖细胞中,H3K9me1/2水平下降,但H3K9me3的水平不变。由于H3K9me1/2在常染色质结合相关蛋白的过程中充当脚手架作用,而这些结合的蛋白在同源染色体联会阶段帮助同源染色体正确配对[53],因此,G9a的敲除通过降低H3K9甲基化修饰影响常染色质相关结合蛋白的募集,使得减数第一次分裂时同源染色体不能完成联会,从而无法进行正常减数分裂[50]。
4.2 G9a与肿瘤
在多种癌症患者,如肝癌、膀胱癌、骨髓癌、结肠癌和前列腺癌等癌变组织中都发现G9a高表达[54]。与此同时,在各种人癌细胞系中,例如乳腺癌、结肠癌、膀胱癌和肺癌等细胞系中,G9a也出现高表达[54,55]。肺癌患者尸检结果显示肺组织中G9a的表达水平与病情进展以及愈后性呈正相关[56],但具体机制并不清楚。在一些肿瘤细胞系中,G9a异常表达可以显著地促进细胞增殖,并促进与细胞周期相关的基因如Cyclin A2和Cyclin B1的表达,也可以显著地增强肿瘤细胞的非贴壁性生长[52]。相反的,在体外敲低G9a可以显著抑制癌细胞的生长[57]。裸鼠致瘤实验也表明,敲低G9a可以显著抑制癌细胞的侵袭转移[56]。这些都表明G9a与癌症的发生和发展有着密切关系,抑制G9a也许是治疗癌症的有效措施。
那么G9a对癌细胞生长的调控是否需要其HMT活性呢?现有的研究并没有得出一致的结论。Ding研究组表明G9a可以通过单甲基化PHGDH、SHMT2等基因的H3K9位点,从而激活丝氨酸-甘氨酸合成途径,而该途径是氨基酸、核苷酸和脂类等大分子生物合成的前体以及辅酶的重要来源,在促进癌细胞增殖和存活的过程中发挥关键作用[52]。Huang等人也证实,在乳腺癌细胞中,G9a可以双甲基化p53,一种非常重要的抑癌基因,从而造成p53抑癌功能的失活;而敲低G9a后,p53恢复活性,造成凋亡细胞的数目明显增多[54]。Chen等人在研究中虽然发现G9a可以沉默上皮细胞粘附分子Ep-CAM,从而促进肺癌细胞的入侵和转移,但在肺癌细胞中H3K9me2的水平与G9a的表达水平并不对应,因此,G9a对该基因表达的抑制很可能不依赖其甲基化酶活性,而是通过调控DNA甲基化水平实现的[56]。
近年来,多个实验室在多种不同组织来源的癌细胞系以及一些癌症患者的原代细胞中发现,利用siRNA技术或者特异小分子抑制剂来降低G9a水平或其HMT活性,可以造成细胞自噬的过度激活,引起关键细胞组分的过度消耗,进而造成细胞自噬性死亡[51,52]。这种细胞死亡是caspase非依赖性的,有别于经典的细胞凋亡[51]。抑制G9a引发细胞自噬性死亡的分子机制现有两种解释:一种是抑制G9a可以引起细胞内活性氧基团(ROS)的积累[51],持续的 ROS可以引发自噬,从而引起癌细胞的自噬性死亡。然而,G9a的下调是如何造成细胞内ROS的积累还需要进一步阐明。另一种是抑制G9a可以显著抑制丝氨酸-甘氨酸合成途径,造成细胞内能量缺失,继而引发癌细胞的自噬性死亡[52]。虽然抑制G9a引发细胞自噬性死亡的分子机制并没有完全研究清楚,但是两个研究共同表明G9a的抑制剂可以作为癌细胞自噬的诱导剂,为癌症治疗提供新思路。
4.3 G9a 与神经系统疾病
对前脑神经元特异性G9a敲除小鼠的研究发现,该品系小鼠虽然没有明显的神经元发育缺陷,但由于神经细胞内H3K9me2水平下降,造成大量基因表达上调,特别是重新激活了Dach2,一个只在胚胎期表达的转录因子[58]。人类的DACH2基因位于X染色体中与多种智力发育迟滞有关的区域上[59]。因此,该品系小鼠成年后表现出认知水平和适应环境的能力下降,这种表型类似于人类第9号染色体缺陷引发的智力发育迟滞综合征[58]。这种G9a在神经元中的作用在进化上是保守的,因为G9a缺陷的果蝇在学习和记忆方面也出现明显缺陷[60]。
除了对学习和记忆有影响外,G9a在可卡因成瘾方面也起着关键的作用。反复的可卡因刺激会改变小鼠伏隔核神经元中部分基因的表达,进而改变神经元的形态,表现出明显的可卡因成瘾。进一步检测发现,G9a 和 GLP 在这种小鼠的伏隔核神经元中表达水平明显下降,相应的,H3K9me2水平也显著性下降[61]。利用伏隔核神经元特异性的G9a转基因和敲除小鼠进一步研究,证明G9a及其介导的H3K9me2在可卡因诱导的神经反应中起着关键作用[61]。因此,调控G9a水平或其HMT活性有可能成为治疗可卡因成瘾的新靶标。但该文章并没有进一步研究GLP,是否调控GLP水平也能达到调控H3K9me2水平,从而影响小鼠的可卡因成瘾性呢?还需要设计相关实验来进一步证明G9a对可卡因成瘾调控的特异性。
4.4 G9a与脂肪形成
在脂肪细胞分化过程中有两类重要的转录因子,PPARγ(Peroxisome proliferator-activated receptors γ,过氧化物酶体增殖激活受体γ)会促进脂肪分化,而Wnts会抑制脂肪分化[62~64]。研究发现,在3T3-L1前脂肪细胞中PPAR γ的整个基因富含H3K9me2,并在脂肪分化过程中该基因上的H3K9me2水平和G9a水平明显下降[65]。在棕色前脂肪细胞和3T3-L1细胞中敲低G9a,引发H3K9me2整体水平下降,可以促进PPARγ的表达和脂肪细胞分化[65]。与之对应的是,在3T3-L1细胞中过表达G9a可以抑制脂肪细胞分化。有趣的是,在脂肪分化过程中,G9a对PPARγ的调控依赖其HMT活性,而对Wnt10a的表达调控不依赖其HMT活性[65]。更重要的是,在正常食物喂养条件下,脂肪组织特异性G9a敲除小鼠的脂肪重量明显增加[65], 进一步证实了G9a抑制脂肪分化和累积。
5 结语
传统上认为,G9a的主要作用是对常染色体上的组蛋白进行甲基化,进而抑制相关基因转录。但最新研究表明G9a也可以作为脚手架蛋白来募集转录激活子,从而激活特定基因转录。近些年越来越多的科学家致力于研究G9a在基因转录调控中的双重作用,并阐明其生物学意义。因此,进一步筛选出与G9a有相互作用的蛋白,阐明G9a调控基因转录的具体机制,以及G9a在生理和病理条件下对信号通路的影响,都将有望更加全面地了解G9a的功能,并为相关疾病的预防和治疗提供新思路、新靶点。
[1] Pinney S E, Simmons R A. Epigenetic mechanisms in the development of type 2 diabetes. Trends Endocrinol Metab, 2010,21(4): 223-229.
[2] Portela A, Esteller M. Epigenetic modi fi cations and human disease.Nat Biotechnol, 2010, 28(10): 1057-1068.
[3] Greer E L, Shi Y. Histone methylation: a dynamic mark in health,disease and inheritance . Nat Rev Genet, 2012, 13(5): 343-57.
[4] Albert M, Helin K. Histone methyltransferases in cancer. Semin Cell Dev Biol, 2010, 21(2): 209-220.
[5] Wood A, Shilatifard A. Posttranslational modi fi cations of histones by methylation. Adv Protein Chem, 2004, 67: 201-222.
[6] Wilson J R, Jing C, Walker P A,et al.Crystal structure and functional analysis of the histone methyltransferase SET7/9. Cell,2002, 111(1): 105-15.
[7] Trievel R C, Beach B M, Dirk L M,et al.Structure and catalytic mechanism of a SET domain protein methyltransferase. Cell, 2002,111(1): 91-103.
[8] Trievel R C, Flynn E M, Houtz R L,et al.Mechanism of multiple lysine methylation by the SET domain enzyme Rubisco LSMT. Nat Struct Biol, 2003, 10(7): 545-552.
[9] Martin C, Zhang Y. The diverse functions of histone lysine methylation. Nat Rev Mol Cell Biol, 2005, 6(11): 838-849.
[10] Min J, Feng Q, Li Z,et al.Structure of the catalytic domain of human DOT1L, a non-SET domain nucleosomal histone methyltransferase. Cell, 2003, 112(5): 711-723.
[11] Kouskouti A, Scheer E, Staub A,et al.Gene-speci fi c modulation of TAF10 function by SET9-mediated methylation. Mol Cell, 2004,14(2): 175-182.
[12] Huang J, Perez B L, Placek B J,et al.Repression of p53 activity by Smyd2-mediated methylation. Nature, 2006, 444(7119): 629-632.
[13] Min J, Zhang X, Cheng X,et al.Structure of the SET domain histone lysine methyltransferase Clr4. Nat Struct Biol, 2002, 9(11):828-832.
[14] Tachibana M, Sugimoto K, Nozaki M,et al.G9a histone methyltransferase plays a dominant role in euchromatic histone H3 lysine 9 methylation and is essential for early embryogenesis. Genes Dev, 2002, 16(14): 1779-1791.
[15] Dunham I, Sargent C A, Kendall E,et al.Characterization of the class III region in different MHC haplotypes by pulsed-field gel electrophoresis. Immunogenetics, 1990, 32(3): 175-182.
[16] Tachibana M, Sugimoto K, Fukushima T,et al.Set domaincontaining protein, G9a, is a novel lysine-preferring mammalian histone methyltransferase with hyperactivity and speci fi c selectivity to lysines 9 and 27 of histone H3. J Biol Chem, 2001, 276(27):25309-25317.
[17] Brown S E, Campbel R D, Sanderson C M. Novel NG36/G9a gene products encoded within the human and mouse MHC class III regions. Mamm Genome, 2001, 12(12): 916-924.
[18] Rathert P, Dhayalan A, Murakami M,et al.Protein lysine methyltransferase G9a acts on non-histone targets. Nat Chem Biol,2008, 4(6): 344-346.
[19] Collins R E, Northrop J P, Horton J R,et al.The ankyrin repeats of G9a and GLP histone methyltransferases are mono- and dimethyllysine binding modules. Nat Struct Mol Biol, 2008, 15(3):245-250.
[20] Tachibana M, Ueda J, Fukuda M,et al.Histone methyltransferases G9a and GLP form heteromeric complexes and are both crucial for methylation of euchromatin at H3-K9. Genes Dev, 2005, 19(7): 815-826.
[21] Zhang X, Tamaru H, Khan S I,et al.Structure of the Neurospora SET domain protein DIM-5, a histone H3 lysine methyltransferase.Cell, 2002, 111(1): 117-127.
[22] Shankar S R, Bahirvani A G, Rao V K,et al.G9a, a multipotent regulator of gene expression. Epigenetics, 2013, 8(1): 16-22.
[23] Collins R E, Tachibana M, Tamaru H,et al.In vitro and in vivo analyses of a Phe/Tyr switch controlling product specificity of histone lysine methyltransferases. J Biol Chem, 2005, 280(7): 5563-5570.
[24] Kubicek S, O'sulliban R J, August E M,et al.Reversal of H3K9me2 by a small-molecule inhibitor for the G9a histone methyltransferase.Mol Cell, 2007, 25(3): 473-481.
[25] Peters A H, Kubicek S, Mechtler K,et al.Partitioning and plasticity of repressive histone methylation states in mammalian chromatin.Mol Cell, 2003, 12(6): 1577-1589.
[26] Rice J C, Briggs S D, Ueberheide B,et al.Histone methyltransferases direct different degrees of methylation to de fi ne distinct chromatin domains. Mol Cell, 2003, 12(6): 1591-1598.
[27] Dillon S C, Zhang X, Trievel R C,et al.The SET-domain protein superfamily: protein lysine methyltransferases. Genome Biol, 2005,6(8): 227.
[28] Ogawa M, Ohkuma K. Good laboratory practice (GLP) and ISO9002 Registration of Quality Management System in the fi eld of food hygiene. Shokuhin eiseigaku zasshi Journal of the Food Hygienic Society of Japan, 2002, 43(1): J16-21.
[29] Ogawa H, Ishiguro K, Gaubatz S,et al.A complex with chromatin modi fi ers that occupies E2F- and Myc-responsive genes in G0 cells[J]. Science, 2002, 296(5570): 1132-1136.
[30] Chang Y, Ganesh T, Horton J R,et al.Adding a lysine mimic in the design of potent inhibitors of histone lysine methyltransferases. J Mol Biol, 2010, 400(1): 1-7.
[31] Fu L, Yan F X, An X R,et al.Effects of the histone methyltransferase inhibitor UNC0638 on histone H3K9 dimethylation of cultured ovine somatic cells and development of resulting early cloned embryos. Reprod Domest Anim, 2014, 49(2):e21-25.
[32] Vedadi M, Barsyte L Y D, Liu F,et al.A chemical probe selectively inhibits G9a and GLP methyltransferase activity in cells. Nat Chem Biol, 2011, 7(8): 566-574.
[33] Fang T C, Schaefer U, Mecklenbrauker I,et al.Histone H3 lysine 9 di-methylation as an epigenetic signature of the interferon response.J Exp Med, 2012, 209(4): 661-669.
[34] Son H J, Kim J Y, Hahn Y,et al.Negative regulation of JAK2 by H3K9 methyltransferase G9a in leukemia. Mol Cell Biol, 2012,32(18): 3681-3694.
[35] Yokochi T, Poduch K, Ryba T,et al.G9a selectively represses a class of late-replicating genes at the nuclear periphery. Proc Natl Acad Sci U S A, 2009, 106(46): 19363-19368.
[36] Smith Z D, Meissner A. DNA methylation: roles in mammalian development. Nat Rev Genet, 2013, 14(3): 204-220.
[37] Margot J B, Cardoso M C, Leonhardt H. Mammalian DNA methyltransferases show different subnuclear distributions. J Cell Biochem, 2001, 83(3): 373-379.
[38] Robertson K D. DNA methylation and human disease. Nat Rev Genet, 2005, 6(8): 597-610.
[39] Tachibana M, Matsumura Y, Fukuda M,et al.G9a/GLP complexes independently mediate H3K9 and DNA methylation to silence transcription. EMBO J, 2008, 27(20): 2681-2690.
[40] Dong K B, Maksakova I A, Mohn F,et al.DNA methylation in ES cells requires the lysine methyltransferase G9a but not its catalytic activity. EMBO J, 2008, 27(20): 2691-2701.
[41] Ikegami K, Iwatani M, Suzuki M, et al. Genome-wide and locusspeci fi c DNA hypomethylation in G9a de fi cient mouse embryonic stem cells. Genes Cells, 2007, 12(1): 1-11.
[42] Jackson J P, Lindroth A M, Cao X,et al.Control of CpNpG DNA methylation by the KRYPTONITE histone H3 methyltransferase.Nature, 2002, 416(6880): 556-560.
[43] Tamaru H, Selker E U. A histone H3 methyltransferase controls DNA methylation in Neurospora crassa. Nature, 2001, 414(6861):277-283.
[44] Liu X, Gao Q, Li P,et al.UHRF1 targets DNMT1 for DNA methylation through cooperative binding of hemi-methylated DNA and methylated H3K9. Nat Commun, 2013, 4: 1563.
[45] Esteve P O, Chin H G, Smallwood A,et al.Direct interaction between DNMT1 and G9a coordinates DNA and histone methylation during replication. Genes Dev, 2006, 20(22): 3089-3103.
[46] Epsztejn L S, Feldman N, Abu R M,et al.De novo DNA methylation promoted by G9a prevents reprogramming of embryonically silenced genes. Nat Struct Mol Biol, 2008, 15(11):1176-1183.
[47] Bittencourt D, Wu D Y, Jeong K W,et al.G9a functions as a molecular scaffold for assembly of transcriptional coactivators on a subset of glucocorticoid receptor target genes. Proc Natl Acad Sci U S A, 2012, 109(48): 19673-19678.
[48] Purcel D J, Jeong K W, Bittencourt D,et al.A distinct mechanism for coactivator versus corepressor function by histone methyltransferase G9a in transcriptional regulation. J Biol Chem,2011, 286(49): 41963-41971.
[49] Lee D Y, Northrop J P, Kuo M H,et al.Histone H3 lysine 9 methyltransferase G9a is a transcriptional coactivator for nuclear receptors. J Biol Chem, 2006, 281(13): 8476-8475.
[50] Tachibana M, Nozaki M, Takeda N,et al.Functional dynamics of H3K9 methylation during meiotic prophase progressio. EMBO J,2007, 26(14): 3346-3359.
[51] Kim Y, Kim Y S, Kim D E,et al.BIX-01294 induces autophagyassociated cell death via EHMT2/G9a dysfunction and intracellular reactive oxygen species production. Autophagy, 2013, 9(12):212621-212639.
[52] Ding J, Li T, Wang X,et al.The histone H3 methyltransferase G9A epigenetically activates the serine-glycine synthesis pathway to sustain cancer cell survival and proliferation. Cell Metab, 2013,18(6): 896-907.
[53] Kim J, Daniel J, Espejo A,et al.Tudor, MBT and chromo domains gauge the degree of lysine methylation. EMBO Rep, 2006, 7(4):397-403.
[54] Huang J, Dorsey J, Chuikov S,et al.G9a and Glp methylate lysine 373 in the tumor suppressor p53. J Biol Chem, 2010, 285(13): 9636-9641.
[55] Mund C, Lyko F. Epigenetic cancer therapy: Proof of concept and remaining challenges. Bioessays, 2010, 32(11): 949-957.
[56] Chen M W, Hua K T, Kao H J, et al. H3K9 histone methyltransferase G9a promotes lung cancer invasion and metastasis by silencing the cell adhesion molecule Ep-CAM. Cancer Res, 2010, 70(20): 7830-7840.
[57] Kondo Y, Shen L, Ahmed S,et al.Downregulation of histone H3 lysine 9 methyltransferase G9a induces centrosome disruption and chromosome instability in cancer cells. PLoS One, 2008, 3(4):e2037.
[58] Schaefer A, Sampath S C, Inerator A,et al.Control of cognition and adaptive behavior by the GLP/G9a epigenetic suppressor complex.Neuron, 2009, 64(5): 678-691.
[59] Davis R J, Shen W, Sandler Y I,et al.Characterization of mouse Dach2, a homologue of Drosophila dachshund. Mechanisms of development, 2001, 102(1-2): 169-179.
[60] Kramer J M, Kochinke K, Ooortveld M A,et al.Epigenetic regulation of learning and memory by Drosophila EHMT/G9a.PLoS Biol, 2011, 9(1): e1000569.
[61] Maze I, Covington H E, Dietz D M,et al.Essential role of the histone methyltransferase G9a in cocaine-induced plasticity.Science, 2010, 327(5962): 213-216.
[62] Rosen E D, Spiegelman B M. PPARgamma : a nuclear regulator of metabolism, differentiation, and cell growth. J Biol Chem, 2001,276(41): 37731-37734.
[63] Rosen E D, Macdougald O A. Adipocyte differentiation from the inside out. Nat Rev Mol Cell Biol, 2006, 7(12): 885-896.
[64] Prestwich T C, Macdougald O A. Wnt/beta-catenin signaling in adipogenesis and metabolism. Curr Opin Cell Biol, 2007, 19(6):612-617.
[65] Wang L, Xu S, Lee J E,et al.Histone H3K9 methyltransferase G9a represses PPARgamma expression and adipogenesi. EMBO J, 2013,32(1): 45-59.

