甘肃红豆草无色花青素还原酶LAR基因的克隆和表达分析
陈春艳,马晖玲,董文科
(1.甘肃农业大学草业学院,甘肃 兰州 730070;2.河南科技大学农学院,河南 洛阳 471003;3.草业生态系统教育部重点实验室,甘肃 兰州 730070;4.中-美草地畜牧业可持续发展研究中心,甘肃 兰州 730070)
甘肃红豆草无色花青素还原酶LAR基因的克隆和表达分析
陈春艳1,2,3,4,马晖玲1,3,4*,董文科1,3,4
(1.甘肃农业大学草业学院,甘肃 兰州 730070;2.河南科技大学农学院,河南 洛阳 471003;3.草业生态系统教育部重点实验室,甘肃 兰州 730070;4.中-美草地畜牧业可持续发展研究中心,甘肃 兰州 730070)
以甘肃红豆草为材料,采用RT-PCR法克隆无色花青素还原酶LAR基因,分析该功能基因在甘肃红豆草不同器官表达的差异性。结果表明,克隆的甘肃红豆草LAR基因,与GenBank已报道LAR基因(登录号:HM152981)序列相似性达到98.34%,其ORF长为1089 bp,编码362个氨基酸残基;其编码的氨基酸序列与不同属豆科物种的相似性存在较大的差异。基因序列已注册到GenBank,序列登录号为KP013623。LAR基因在甘肃红豆草不同器官表达差异较大;其中叶中表达量最高,其次是花和果,茎中表达量最低,这与器官间缩合单宁含量的变化规律一致。推测其表达与甘肃红豆草缩合单宁器官间的积累差异有关。这些研究结果也为探索缩合单宁积累和表达调控的机制提供基础。
甘肃红豆草;RT-PCR;无色花青素还原酶;克隆与表达
红豆草(Onobrychisviciaefolia)为多年生草本植物,红豆草地上部分粗蛋白质及氨基酸含量丰富,含有一定数量的粗脂肪、多种维生素和矿物质及其他化学物质。红豆草的种子产量和产草量高,结实性能较好,营养物质全面而丰富,适口性优良。而且它能显著增加土壤含氮量,也是一种花蜜液数量多的植物[1]。目前,红豆草作为优良的牧草在甘肃、新疆、内蒙古、陕西、宁夏、青海等23个省区广泛栽培[2]。甘肃红豆草(Onobrychisviciifoliacv.Gansu)是由普通红豆草和高加索红豆草自由杂交多次混合授粉的植株中单株选择培育成的[3]。适合我国干旱半干旱地区建立人工草地,其产草量接近或超过早熟、速生、抗病虫害、高产、耐旱的紫花苜蓿品种。
可溶性蛋白含量高是苜蓿引起家畜臌胀病的主要原因[4]。缩合单宁能够与可溶性蛋白质作用生成沉淀,避免引起臌胀病,然而紫花苜蓿叶片不含缩合单宁,只在种皮中少量存在。红豆草在各个生长时期叶片都能生产很高浓度的缩合单宁[5]。家畜饲用富含缩合单宁的红豆草后不会患臌胀病,普遍认为红豆草是在遗传资源创新途径中和饲草利用方式解决紫花苜蓿引起牲畜臌胀病发生的一种理想牧草[6-7]。在遗传资源创新方向,从红豆草中鉴定和分离缩合单宁基因,进而利用基因工程技术将此基因导入到苜蓿中去,不失为一条改善苜蓿品质和适口性的有效途径。
目前,缩合单宁合成途径已经基本清楚。主要由公共苯丙烷途径、核心类黄酮-花青素途径、缩合单宁特异途径完成,其中缩合单宁特异途径最终决定其积累[8]。缩合单宁特异生成途径中有两个关键酶分别是无色花青素还原酶(leucoanthocyanidin reductase, LAR)和花青素还原酶(anthocyanidin reductase, ANR)[9-10]。LAR和ANR为单宁的生成提供了两条不同的途径和不同的前体物质,然而前者的底物无色花青素又是经花青素合成酶合成的花青素成为后者的底物,LAR在缩合单宁特异合成途径中尤为重要[11]。一些研究证实LAR基因的表达与缩合单宁的累积呈正相关[12],但是也有LAR活性与缩合单宁累积不符的报道[13]。LAR基因表达与缩合单宁合成受到种内和种间差异的影响[14]。为此,探清甘肃红豆草LAR基因结构、功能及其表达具有重要的科学意义和实践应用价值。
从植物组织中提取纯度高、完整性好的高质量RNA是进行Northern杂交、RT-PCR以及cDNA文库构建等分子生物学研究的必要前提[15-16]。然而红豆草的组织和器官中富含酚、原花青素、花青素等次生代谢物以及蛋白质、多糖等生物大分子,其RNA提取难度较大,这些物质严重影响RNA提取的质量和效果。本研究通过改良总RNA提取方法,将提取获得的总RNA粗提物纯化,提出了一种有效的红豆草总RNA的提取方法。进而利用RT-PCR法克隆甘肃红豆草LAR基因,分析其序列特征,与其他高等植物的LAR基因进行相似性比较,并分析LAR基因的组织表达特征。以验证该方法所提取的总RNA能否满足下游分子生物学实验,也为红豆草分子生物学进一步研究提供参考和借鉴。
1 材料与方法
1.1 材料和试剂
1.1.1 材料 红豆草品种为甘肃红豆草,由甘肃农业大学草业学院提供。
1.1.2 实验试剂和处理方法 M-MuLV First Strand cDNA Synthesis Kit购自上海生工生物工程公司,引物由北京赛百盛生物公司合成,Taq酶购自大连宝生物工程有限公司,SanPrep DNA胶回收试剂盒购自上海生工生物工程公司,其他药品、试剂为常规国产分析纯。
所用的试剂为新开封试剂,用灭菌的0.1%焦碳酸二乙酯(diethyl pyrocarbonate, DEPC)水配制,塑料耗材均需用0.1%DEPC dH2O处理24 h,高温灭菌,烘干后备用;研钵、试剂瓶、玻璃棒、容量瓶等物品经180℃高温烘烤5 h,避免RNase污染。
1.2 RNA提取方法
1.2.1 CTAB法粗提RNA 取红豆草幼嫩材料0.10 g,在液氮中研磨成粉,将粉末迅速转移至1.5 mL的离心管中;加入600 μL的65℃ RNA提取缓冲液 [2%β-巯基乙醇(W/V)、2%CTAB(W/V)、4%PVP(W/V)、1.4 mol/L NaCl、100 mmol/L Tris-Cl(pH 8.0)、25 mmol/L EDTA(pH 8.0)],然后振荡1 min,使材料裂解充分,在65℃水浴锅中温浴10 min,期间振荡2~3次;然后在4℃和12000 r/min下高速离心10 min;吸取上清置于另1支1.5 mL离心管中,加入600 μL体积比为25∶24∶1的水饱和酚:氯仿:异戊醇混合液,上下颠倒混匀,放置于冰上10 min;在4℃和12000 r/min下高速离心10 min;吸取上清至另一离心管中,重复上述除蛋白步骤;吸取上清,加入167 μL 10 mol/L LiCl,上下颠倒混匀,置于-20℃冰箱3 h,在4℃和12000 r/min下高速离心20 min;弃上清液,将沉淀溶解于500 μL 0.1% DEPC水中,加入50 μL的3 mol/L NaAC(pH 5.2),充分溶解后,再加入1375 μL -20℃的无水乙醇,混匀,放置于-70℃的超低温冰箱中30 min,在4℃和12000 r/min下高速离心10 min,弃去上清;用70%乙醇洗涤2次,在4℃和12000 r/min下高速离心5 min;弃去上清,倒扣在滤纸上吸光残余乙醇;沉淀于超净工作台中风干,加入50 μL 0.1% DEPC水溶解RNA,此为RNA粗提物。
1.2.2 RNA的纯化 取RNA粗提物100 μL,加入900 μL 0.1% DEPC水,使其溶解,然后加入1000 μL体积比为25∶24∶1的水饱和酚:氯仿:异戊醇混合液,颠倒混匀,在4℃和12000 r/min下高速离心20 min;取上清于另一支1.5 mL离心管中,立即加入200 μL的3 mol/L NaAC(pH 5.2)和500 μL预冷的无水乙醇,混匀,放置于-70℃的超低温冰箱中沉淀30 min,在4℃和12000 r/min下高速离心10 min,弃去上清;用70%乙醇洗涤2次,在4℃和12000 r/min下高速离心5 min;弃去上清,倒扣在滤纸上吸光残余乙醇;沉淀于超净工作台中风干,加入50 μL 0.1% DEPC水溶解RNA,将其保存于-80℃的超低温冰箱。
1.3 RNA质量检测
1.3.1 纯度和浓度测定 取纯化的RNA 1 μL,以0.1% DEPC水为空白液调零,在微量分光光度计下测定OD260和OD280值。
1.3.2 凝胶电泳检测 取纯化的RNA 10 μL,以1×TBE为缓冲液,在1%的琼脂糖凝胶上电泳,30 min后,在凝胶成像系统中观察并照相。
1.4 基因克隆
1.4.1 First-Strand cDNA合成 使用上海生工生物工程公司反转录试剂盒进行First-Strand cDNA合成反应,合成后产物于-20℃保存备用。
1.4.2 PCR LAR基因 根据GenBank数据库中公布的红豆草无色花青素还原酶(LAR)基因(登录号:HM152981)序列,利用分子生物学软件DNAMAN 6.0设计特异引物:
LP1:5′-CGCCCGGGATGGCGACTTCCCCAGCCAATATC-3′
SmalⅠ
LP2:5′-GCGAGCTCTCAACAGGAAGCTGTTATTGGGACTG-3′
SacⅠ
PCR反应体系(25 μL)包括cDNA 1 μL,10×Buffer 2.5 μL,10 mmol/L dNTP 2 μL,引物LP1和LP2(10 pmol/μL)各1 μL,5 U/μL Taq酶0.5 μL。反应程序为,预变性:94℃、4 min,变性:94℃、1 min,退火:57℃、1 min,延伸:72℃、1.5 min,38个循环,最后72℃延伸10 min。反应结束后取10 μL反应产物于1%琼脂糖凝胶电泳检测扩增产物。
1.4.3 与T载体连接转化感受态细胞及测序 按回收试剂盒说明书对PCR产物进行回收,按照1∶4将pMD18-T Vector与目的片段进行连接,16℃反应6 h。取连接产物转化大肠杆菌DH5α感受态细胞,并进行蓝白斑筛选。挑选阳性克隆于含100 mg/mL氨苄青霉素的LB培养基中振荡培养,提取质粒DNA,PCR扩增及SmalⅠ和SacⅠ双酶切鉴定阳性克隆后送于上海生工生物公司测序。
1.5 LAR基因生物信息学分析
基因开放阅读框通过NCBI提供的ORF Finder(http://www.ncbi. nlm.nih.gov/gorf/gorf.html)寻找;核苷酸序列相似性检索通过NCBI的BlastN、BlastP和DNAMAN 6.0软件进行分析;用MEGA 6.0软件进行氨基酸序列比对和LAR类蛋白质序列系统进化树分析;用http://expasy.org/tools/protparam.html网站进行蛋白质理化性质分析。通过应用ProtScale软件的Hphob. Kyte & Doolittle算法对LAR蛋白进行亲水/疏水性分析。利用NCBI(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)进行保守功能区预测。
1.6 LAR基因组织表达特性分析
采用半定量RT-PCR方法对甘肃红豆草LAR基因进行组织器官特异性表达分析。分别提取甘肃红豆草叶、茎、花、果的总RNA,以等量的RNA反转录合成cDNA,-20℃备用。运用DNAMAN 6.0软件设计引物和内参引物如下:
LARP1:5′-ACGGCACTGTCCAAGCATA-3′
LARP2:5′-TTTCCCACAAAGAAGCAAGG-3′
ActinP1:5′-GAGCGTTTCCGTTGTCCTGA-3′
ActinP2:5′-AGGTGCTGAGGGAAGCCAAA-3′
以等量的cDNA进行RT-PCR,反应程序为,预变性:94℃、1 min,变性:94℃、30 s,退火:55℃、30 s,延伸:72℃、40 s,30个循环,最后72℃延伸10 min。反应结束后取10 μL反应产物于1%琼脂糖凝胶电泳检测扩增产物。进行3次重复实验。通过目的条带的亮度来分析基因表达的差异。
1.7 缩合单宁含量的测定
分别取甘肃红豆草叶、茎、花、果各0.5 g,液氮研磨成粉末状,加入3 mL提取液[含0.5%乙酸的70%丙酮(V/V)]研磨至匀浆,于5000 r/min下离心10 min,取上清液,沉淀用3 mL提取液清洗2次,合并上清液,并定容至10 mL;吸取2 mL上清液加入2 mL水和2 mL三氯甲烷且充分摇匀后于5000 r/min下离心10 min去除叶绿素(重复3次);上清液即为提取液。采用正丁醇-盐酸法[17]测定缩合单宁含量。
2 结果与分析
2.1 RNA的提取和琼脂糖凝胶电泳检测
RNA的纯度和完整性是分子生物学实验成功与否的重要因素。利用改良CTAB法分离得到的甘肃红豆草总RNA,经微量分光光度计测定OD260/OD280值处于1.8~2.0之间(表1);不同器官RNA电泳显示28s和18s条带较清晰,且28s条带的亮度高于18s条带(图1),结果表明得到的总RNA既未发生降解,又无DNA、蛋白质等污染,质量和纯度较高。
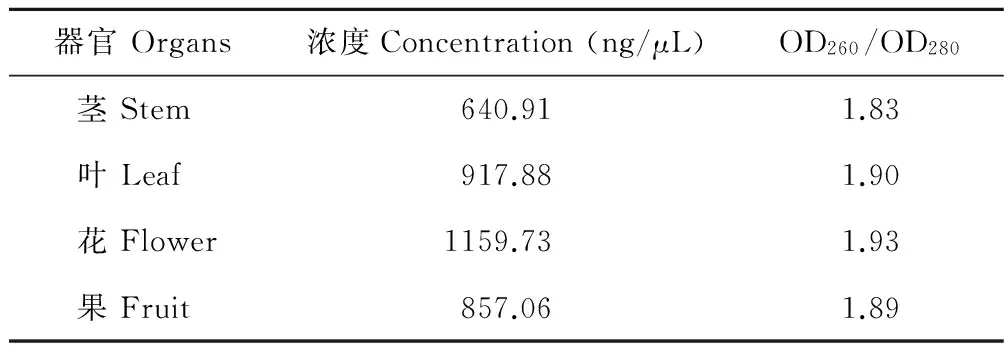
表1 甘肃红豆草不同器官总RNA的浓度及纯度
2.2 LAR基因的扩增及克隆

图1 甘肃红豆草不同器官的总RNAFig.1 Total RNA of different organs in O. viciifolia cv.Gansu 1:茎 Stem,2:叶 Leaf,3:花 Flower,4:果 Fruit.
为确定改良CTAB法提取的红豆草总RNA能否满足下游分子生物学实验。以该法提取的总RNA反转录的cDNA为模板,PCR扩增得到约1100 bp左右的片段(图2)。回收PCR产物,连接到pMD18-T Vector上,经蓝白斑筛选后,挑选阳性克隆,提取质粒进行PCR扩增及SmalⅠ和SacⅠ双酶切鉴定,得到的目的片段大小约为1100 bp(图3,图4)。表明这些克隆为阳性克隆,将其命名为:pMD-LAR,送交测序公司测序。
2.3 测序结果及序列分析
本研究克隆的甘肃红豆草LAR基因已登记在GenBank(登录号:KP013623),将核酸序列用DNAMAN 6.0软件进行比对分析,它与红豆草LAR基因(登录号:HM152981)具有98.34%的相似性。其开放阅读框架为1089 bp,包括362个氨基酸编码区和一个终止密码子(TAG)。与NCBI数据库中登录相似性较高的几种植物LAR基因核酸BLAST分析发现,该序列与豆科其他属植物百脉根、蒺藜苜蓿、大豆、豌豆和大百脉根相似性在76%~93%,与杜鹃花科兔眼蓝莓、桑科啤酒花和蔷薇科甜樱桃的相似性低于75%(表2)。说明同属间LAR基因具有很强的保守性,但不同物种间LAR基因在进化过程中发生较大变异。
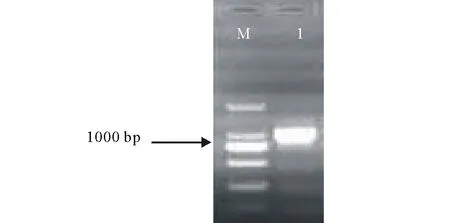
图2 LAR基因的PCR扩增Fig.2 PCR amplification of LAR gene M:DL2000 Marker;1: PCR扩增产物 PCR product.

图3 阳性克隆的PCRFig.3 PCR analysis of positive clonesM:DL1000 Marker;1,2:阳性克隆PCR产物 Positive clones PCR products.

图4 阳性克隆的双酶切鉴定Fig.4 Double digestion of positive clonesM:DL1000 Marker;1,2:阳性克隆SmalⅠ和SacⅠ双酶切Positive clones digestion by SmalⅠ and SacⅠ.
2.4 蛋白特性分析
Protparam软件分析甘肃红豆草LAR蛋白分子式为C1774H2802N470O526S17,分子量为39675.6,编码362个氨基酸,其中异亮氨酸含量最高(9.1%),色氨酸最少(0.6%)。理论等电点为5.95,原子数为5589,带正电荷氨基酸残基数(Arg+Lys)为36,带负电荷氨基酸残基数(Asp+Glu)为42。脂肪系数为92.35。不稳定系数为37.84,属于稳定蛋白。信号肽预测表明,基因编码的蛋白无信号肽结构,为非分泌性蛋白(non-secretory protein)。第20位的甘氨酸(Gly)具有最高的分值2.2,疏水性最强;第331位的赖氨酸(Lys)具有最低的分值-2.822,亲水性最强;平均亲水性(grand average of hydroelectricity,GRAVY)值为-0.064;亲水性氨基酸平均分布在整个肽链中,且多于疏水性氨基酸(图5),为亲水蛋白。利用CDD保守功能区在线分析LAR的保守区预测,发现LAR编码的氨基酸序列具有典型的苯(基)吡喃酮苄基醚还原酶(PCBER,phenylcoumaran benzylic ether reductase)的特征结构域,属于短链脱氢/还原酶(short-chain dehydrogenase/reductase,SDR)超家族成员(cd05259);此氨基酸序列含有NAD(P)结合位点[NAD(P) binding site]和赖氨酸活性位点(active site lysine)(图6)。
甘肃红豆草LAR与其他物种LAR氨基酸序列比对结果表明,其LAR氨基酸序列与豆科植物红豆草、蒺藜苜蓿、百脉根的相似性最高,分别达到96.44%,79.01%和73.28%;与蔷薇科甜樱桃、杜鹃花科兔眼蓝莓和桑科啤酒花的相似性最低,分别为62.53%,60.76%和60.66%。甘肃红豆草LAR氨基酸序列与同科植物间的相似性为67.68%~96.44%,LAR编码的氨基酸序列在豆科植物不同种间差异明显,这与核酸序列变化的特征基本一致(图7)。甘肃红豆草LAR与其他科植物比较,其相似性更低。说明在同源关系相近的植物中LAR基因的保守性很强。
2.5 系统进化分析
对NCBI下载的9个物种的LAR氨基酸序列进行系统进化树分析,发现甘肃红豆草LAR蛋白与豆科植物红豆草亲缘关系最近;它与豆科植物蒺藜苜蓿和豌豆的亲缘关系也较近,其次为豆科植物大豆、百脉根和大百脉根。LAR蛋白与杜鹃花科兔眼蓝莓、桑科啤酒花和蔷薇科甜樱桃的亲缘关系相对最远(图8)。豆科不同物种明显聚类成一组,而其他科植物聚类为另一组。说明不同物种的LAR蛋白具有明显的种属特性,LAR蛋白在种间的变化也反映了植物学分类特征。

表2 甘肃红豆草LAR基因与其他植物LAR基因cDNA序列相似性比较

图5 LAR氨基酸序列疏水性分析Fig.5 The analysis of hydrophobicity of LAR polypeptide
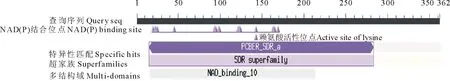
图6 预测的LAR蛋白保守结构域Fig.6 Putative conversed domains of LAR protein
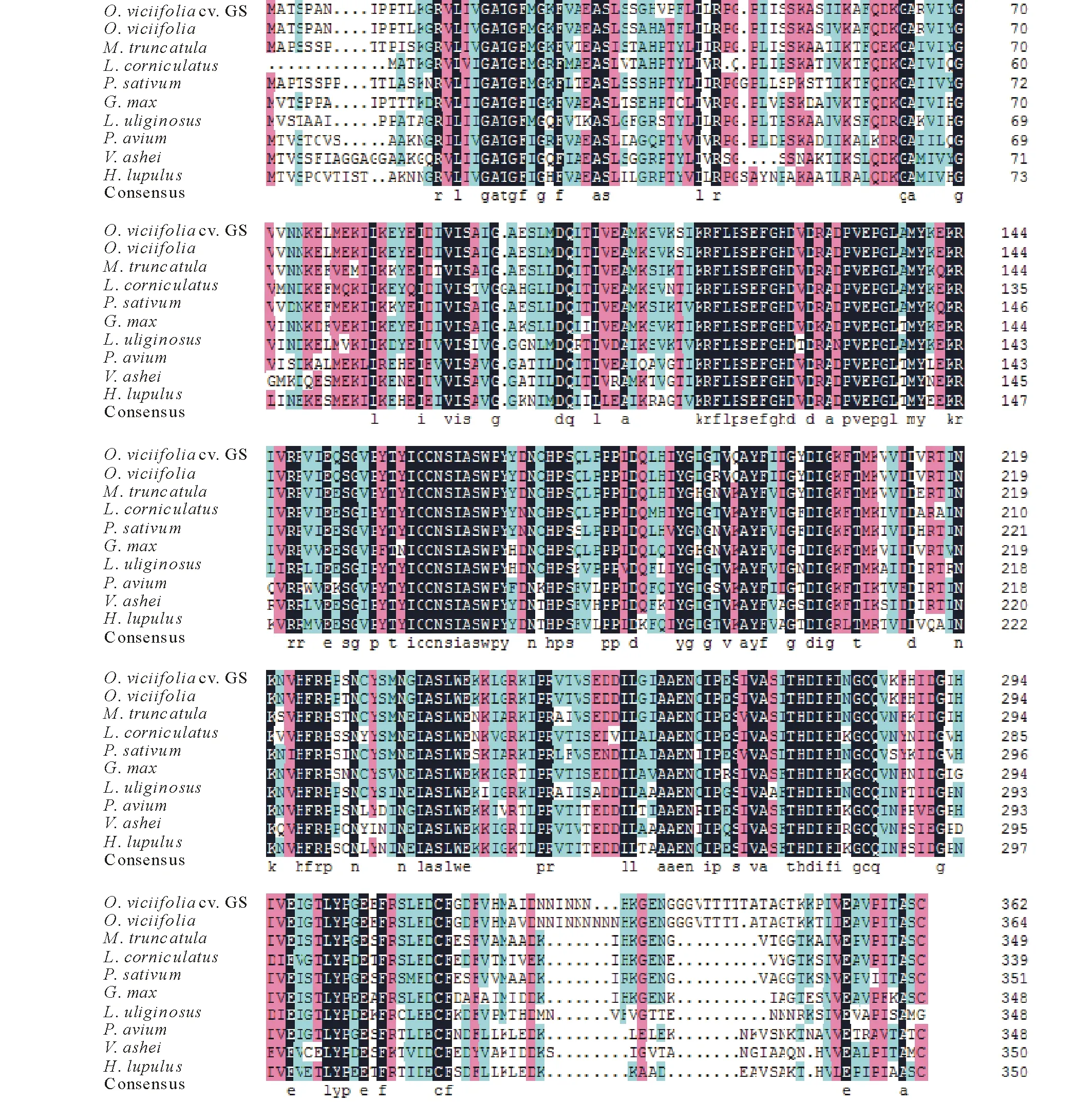
图7 不同种LAR氨基酸序列比较Fig.7 Comparison of the amino acid sequences of LAR in different species O. viciifolia cv.GS:甘肃红豆草(未公布);O. viciifolia:红豆草(AEF14422);M. truncatula:蒺藜苜蓿(XP-003591830);L. corniculatus:百脉根(ABC71331);P. sativum:豌豆(AII26024);G.max:大豆(AME23933);L. uliginosus:大百脉根(AAU45392);P. avium:甜樱桃(ADY15310);V. ashei:兔眼蓝莓(AB610768);H. lupulus:啤酒花(AEV899641);Consensus:表示一致性序列,并在序列下用小写字母列出;根据保守程度高低依次使用黑色、红色和蓝色阴影表示。括号中的编号为GenBank登录号。Consensus indicates same sequences and are listed below in lowercase letters. According to the degree of conservation, black, blue and red are indicated. The numbers in parentheses indicate accession number of GenBank.
2.6 不同器官LAR基因表达和缩合单宁含量
采用半定量RT-PCR的方法分析LAR基因在茎、叶、花、果中的表达情况,结果见图9。半定量结果表明LAR在甘肃红豆草的各个器官中均表达,在叶中表达量最高,其次是花和果,在茎中表达量最低。缩合单宁含量在器官间的高低变化特征与LAR基因表达特征一致(图10)。
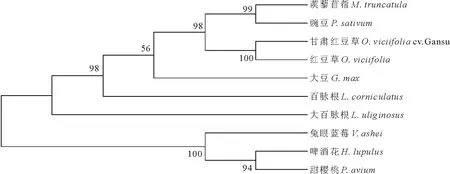
图8 LAR蛋白与其他物种LAR类蛋白构建的系统进化树Fig.8 Phylogenetic tree of LAR proteins in O. viciifolia cv.Gansu and other species GenBank登录号与图7同。Accession number of GenBank is the same as Fig.7.
3 讨论

图9 甘肃红豆草不同器官LAR表达的半定量RT-PCRFig.9 Semi-quantitative RT-PCR of LAR expression in different organs of O. viciifolia cv.Gansu
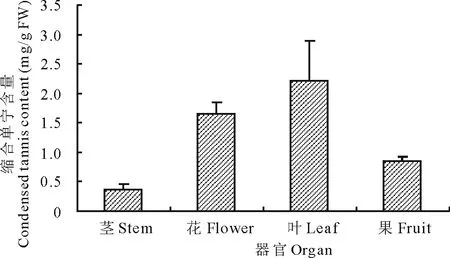
图10 甘肃红豆草不同器官缩合单宁含量Fig.10 Condensed tannins contents in different organs of O. viciifolia cv.Gansu
3.1 甘肃红豆草总RNA提取法的改良及其效果评价
目前,对不同物种植物和组织器官提取RNA的方法进行了广泛的研究,已获得一些科学高效的方法[18-20]。但是仍然不能满足实际实验过程的需要,表现出不同种类的植物提取方法和效果差异较大,同一种植物不同的组织其RNA提取方法也需要调整;即使同一种植物同一组织,因植株基因型不同,RNA提取方法也可能不相同[18-21]。因此,根据植物组织的特点,摸索出适合不同植物针对性强的高质量RNA提取方法十分必要。
红豆草在生长过程中积累了诸如缩合单宁、多糖等大量的次生代谢物。与同科植物紫花苜蓿比较,红豆草叶片积累更多的缩合单宁[5]。其组织中RNase的活性也较高,这些因素导致红豆草RNA的提取难度更大。因此,获得高质量的RNA成为进行红豆草分子生物学研究首要需解决的问题。本研究起初尝试多种商业试剂盒提取红豆草RNA,但在提取效果和稳定性方面都难以满足基因克隆和后续的实验。为此,对传统的CTAB法进行改良。实验过程中在最初的提取缓冲液中加入4%PVP以防止次生代谢物与RNA结合;加入2%β-巯基乙醇以减少多酚物质的干扰;利用水饱和酚∶氯仿∶异戊醇抽提以去除DNA、蛋白质及叶绿素、花青素等色素类物质对RNA的干扰。通过使用LiCl选择性沉淀RNA,使RNA与多糖有效分离。将2管粗提RNA合并为1管进行纯化,这样既消除了多酚、次生代谢物及色素的影响,还能弥补纯化过程中RNA的损失,保证了RNA的含量,提高其纯度。应用该方法提取的红豆草RNA,28s和18s条带较清晰,无DNA污染,同时在下游反转录实验中得到成功的应用。改良的RNA提取方法简单、有效,所使用药品为普通生化试剂。在提取的RNA效果、质量和价格方面与一般的商业试剂盒相比具有明显的优势,是一种实用的方法。另外,提取纯化后获得的RNA具有稳定性强的特点,可适用于红豆草不同器官RNA的提取和下游分子生物学实验。
3.2 甘肃红豆草LAR基因序列和表达分析
植物次生代谢物合成与积累量取决于其合成途径中的限速酶,限速酶在植物次生代谢物生物合成途径中往往位于代谢支路分叉口或途径的下游。植物缩合单宁合成代谢途径末端酶的无色花青素还原酶(LAR)基因克隆与分析,有助于解析植物次生代谢缩合单宁合成和积累的机制。本研究对RT-PCR克隆的甘肃红豆草LAR基因cDNA片段利用相关软件进行了生物信息学分析,发现其LAR基因具备一个完整的开放阅读框架,大小为1089 bp;它属于SDR超家族成员,含有NAD(P)结合位点和赖氨酸活性位点(图6)。这与依赖于无色花青素还原酶催化生成缩合单宁需要NAD(P)H参与的研究结果一致[22]。研究也发现豆科山蚂蝗属植物银叶藤[22]和蓼科植物金荞麦[23]LAR蛋白同样含有NAD(P)结合位点和赖氨酸活性位。由此推断该蛋白具有花青素还原酶的典型的结构特征和生物特性,超量表达可以提高缩合单宁的含量。
甘肃红豆草LAR基因全长序列与同属植物已知序列[24]的相似性高达98%,与不同属的几种豆科植物的相似性在76%~93%,而与其他科植物的相似性在75%以下(表2,图7,8),聚类分析表明豆科与非豆科几种植物明显聚为两类。说明在同源关系相近的植物中LAR基因的保守性强,这与其他学者对其他基因在不同物种同源性分析时的结论一致,即属内同源性高,属间或种间相对低[25-27]。可见,LAR序列在不同种植物间的差异基本符合植物学分类,在一定程度上反映不同物种的亲缘关系远近。甘肃红豆草LAR与其他几种不同属豆科植物和其他科植物在核酸序列的相似性都存在较大的差异(表2)。可见,LAR蛋白在进化上多样性,即它们是由不同祖先蛋白趋同进化的结果[26]。
缩合单宁含量高低影响牧草饲用品质和营养价值,同时缩合单宁对于植物具有抗紫外线、抗病、抗虫、清除自由基等功能。适量地增加牧草中缩合单宁的含量可以减缓反刍动物瘤胃中大量蛋白的快速分解,进而避免形成过多气体而造成胃和肠道肿大导致的臌胀病。大量研究表明LAR基因的表达与缩合单宁的累积有关[10,12,28]。本研究证实,LAR基因在甘肃红豆草的不同器官中均有表达且具有很强的组织特异性,其中在叶中表达量最高,其次是花和果,在茎中只有微量表达。研究也发现缩合单宁含量在不同器官的变化顺序依次为叶>花>果>茎。可推断甘肃红豆草LAR表达和缩合单宁的合成密切相关。毛白杨LAR表达在不同器官差异显示,PtrLAR1和PtrLAR3在根中含量最高,其次是茎、成熟叶片,在幼叶和叶柄中含量较低,这也与毛白杨器官间缩合单宁含量的差异顺序一致[7]。研究也证实葡萄编码无色花色素还原酶的VvLAR基因通过其组织及时空表达的特异性调控葡萄果实发育过程中原花色素的种类和积累[10]。进一步说明编码无色花色素还原酶的LAR基因在器官间的表达量差异较大,具有很强的组织和时空表达的特异性,而且与缩合单宁积累密切相关,也可能影响牧草的产量和品质[29]。可见,LAR基因在不同器官间表达差异的分子机制的研究也十分必要,这将为调控缩合单宁的积累提供必要的借鉴。
4 结论
以甘肃红豆草为材料,采用常规CTAB法提取总RNA后,增加总RNA粗提物的纯化步骤,得到高质量的总RNA。以获得的总RNA为模板,通过RT-PCR克隆无色花青素还原酶LAR基因。获得的LAR基因与已发表的LAR基因具有98.34%的相似性,其开放阅读框长为1089 bp,编码362个氨基酸残基;预测编码的蛋白质具有典型的苯(基)吡喃酮苄基醚还原酶的特征结构域,含有NAD(P)结合位点和赖氨酸活性位点。注册该基因到GenBank中,注册号为KP013623。表达分析发现,LAR基因在甘肃红豆草叶中表达量最高,其次是花和果,茎中表达量最低,这与器官间缩合单宁含量的变化规律一致,推测LAR表达与缩合单宁器官间的积累差异有关。这些成果的取得为红豆草种质资源改良提供理论支持和技术保障,也为利用转基因技术提高紫花苜蓿叶片缩合单宁的含量,以改善紫花苜蓿品质、培育抗臌胀病苜蓿新品质提供理论依据。
[1] Chen B S. Research of yield and nutrition in sainfoin. Grassland of China, 1983, (2): 36-38.
[2] Chen B S. Sainfoin is an excellent leguminous forage and a plant for water and soil conservation. Science of Soil and Water Conservation, 1984, 29(8): 14-18.
[3] Chen B S. Sainfoin[M]. Lanzhou: Gansu Science and Technology Press, 1992: 18.
[4] Howarth R E, Sarkar S K, Fesser A C,etal. Some properties of soluble proteins from alfalfa (Medicagosativa) herbage and their possible relation to ruminant bloat. Journal of Agricultural and Food Chemistry, 1977, 25(1): 175-179.
[5] Wu Z L, Niu J L, Zhang Y P,etal. Tannin dynamics study for sainfoin. Chinese Journal of Grassland, 1993, (5): 25-27.
[6] McMahon L R, McAllister T A, Berg B,etal. A review of the effects of forage condensed tannins on ruminal fermentation and bloat in grazing cattle. Canadian Journal of Plant Science, 2000, 80: 469-485.
[7] Wang Y P, Yang Z M, Zhang B. Identification of protein related to biosynthesis of condensed Tannin inOnobrychistanaiticaspreng. Acta Agrestia Sinica, 2000, 8(2): 126-131.
[8] Zhao W J, Zhang D, Ma L J,etal. Biosynthetic pathway, functional genes and metabolic engineering of proanthocyanidins. Plant Physiology Communications, 2009, 45(5): 509-519.
[9] Xie D Y, Sharma S B, Paiva N L,etal. Role of anthocyanidin reductase, encoded by BANYULS in plant flavonoid biosynthesis. Science, 2003, 299: 396-399.
[10] Bogs J, Downey M O, Harvey J S,etal. Proanthocyanidin synthesis and expression of genes encoding leucoanthocyanidin reductase and anthocyanidin reductase in developing grape berries and grapevine leaves. Plant Physiology, 2005, 139(2): 652-663.
[11] Yuan L, Wang L, Han Z,etal. Molecular cloning and characterization of PtrLAR3, a gene encoding leucoanthocyanidin reductase fromPopulustrichocarpa, and its constitutive expression enhances fungal resistance in transgenic plants. Journal of Experiment Botany, 2012, 63(7): 2513-2524.
[12] Paolocci F, Robbins M P, Madeo L,etal. Ectopic expression of a basic helix-loop-helix gene transactivates parallel pathways of proanthocyanidin biosyn thesis: structure, expression analysis, and genetic control of leucoanthocyanidin 4-reductase and anthocyanidin reductase genes in Lotus corniculatus. Plant Physiology, 2007, 143(1): 504-516.
[13] Wang Y, Zhang Q L, Luo Z R. Isolation and expression of gene encoding leucoanthocyanidin reductase fromDiospyroskakiduring fruit development. Biologia Plantarum, 2010, 54(4): 707-710.
[14] Pang Y, Wenger J P, Saathoff K,etal. A WD40 repeat protein fromMedicagotruncatulais necessary for tissue-specific anthocyanin and proanthocyanidin biosynthesis but not for trichome development. Plant Physiology, 2009,151(3): 1114-1129.
[15] Sambrook J, Russell D W. Molecular Cloning: A Laboratory Manual[M]. Vol.2. Beijing: Beijing Sciences Press, 1995: 343-392.
[16] Shi B S, Zhuo L H. Modification and comparation of total extraction methods fromPrunuscerasiferaleaves. Molecular Plant Breeding, 2006, 4(5): 721-725.
[17] Terrill T H, Rowan A M, Douglas G B.etal. Determination of extractable and bound condensed tannin concentrations in forage plants, prote in concentrate meals and cereal grains. Journal of the Science of Food and Agriculture, 1992, 58(3): 321-329.
[18] Li H, Wang X L. The difficulties in the isloation of RNA from plant tissues and their resolving strategies. Bio Technology Information, 1999, (1): 36-39.
[19] Gasic K, Hernandez A, Korban S S. RNA extraction from different apple tissues rich in polyphenols and polysaccharides for cDNA library construction. Plant Molecular Biology Reporter, 2004, 22(4): 437-438.
[20] Rodrigues S M, Soares V L F, de Oliveira T M,etal. Isolation and purification of RNA from tissues rich in polyphenols, polysaccharides, and pigments of annatto (BixaOrellanaL.). Molecular Biotechnology, 2007, 37(3): 220-224.
[21] Ainsworth C. Isolation of RNA from floral tissue ofRumexacetosa(sorrel). Plant Molecular Biology Reporter, 1994, 12(3): 198-203.
[22] Tanner G J, Francki K T, Abrahams S,etal. Proanthocyanidin biosynthesis in plants:purification of legume leucoanthocyanidin reductase and molecular cloning of its cDNA. Journal of Biology Chemistry, 2003, 278: 31647-31656.
[23] Ma J, Wang B, Dai Y,etal. Cloning and expression analysis of leucoanthocyanidin reductase gene inFagopyrumdibotrys. Acta Pharmaceutica Sinica, 2012, 47(7): 953-961.
[24] Thill J, Regos I, Farag M A,etal. Polyphenol metabolism provides a screening tool for beneficial effects ofOnobrychisviciifolia(sainfoin). Phytochemistry, 2012, 82: 67-80.
[25] Shen B Y, Liu Y H, Zhang J L,etal. Analysis of cloning and sequence characteristics of cDNA ending the GBSS I gene from tubers ofSloanumtuberosum. Acta Prataculturae Sinica, 2010, 19(5): 1-8.
[26] Li L Q, Huang Y B, Wang X Y. Cloning, sequence analysis and prokaryotic expression of the WRKY6 gene of potato. Acta Prataculturae Sinica, 2011, 20(2): 177-183.
[27] Lv H Y, Li Y X, Kong F J,etal. Recent progress in studies on Na+/H+antiporter in plants. Chinese Bulletin of Botany, 2003, 20(3): 363-369.
[28] Wang L, Jiang Y, Yuan L,etal.Isolation and characterization of cDNAs encoding leucoanthocyanidin reductase and anthocyanidin reductase fromPopulustrichocarpa. PLoS One, 2013, 8(5): e64664. doi: 10.1371/journal. Pone. 0064664.
[29] Daniel A H. Biomass allocation is an important determinant of the tannin concentration in growing plants. Annals of Botany, 2007, 99: 111-120.
参考文献:
[1] 陈宝书. 红豆草产量和营养成分的研究. 中国草原, 1983, (2): 36-38.
[2] 陈宝书. 红豆草是优良的豆科牧草和水土保持植物. 中国水土保持, 1984, 29(8): 14-18.
[3] 陈宝书. 红豆草[M]. 兰州: 甘肃科学技术出版社, 1992: 18.
[5] 吴自立, 牛菊兰, 张跃平, 等. 红豆草中丹宁的动态研究. 中国草地学报, 1993, (5): 25-27.
[7] 王玉萍, 杨茁萌, 张博. 顿河红豆草缩合单宁生物合成相关蛋白鉴定. 草地学报, 2000, 8(2): 126-131.
[8] 赵文军, 张迪, 马丽娟, 等. 原花青素的生物合成途径、功能基因和代谢工程. 植物生理学通讯, 2009, 45(5): 509-519.
[15] 萨姆布鲁克J, 拉塞尔D W. 分子克隆实验指南[M]. 2版. 北京: 科学出版社, 1995: 343-392.
[16] 史宝胜, 卓丽环. 紫叶李叶片总RNA提取方法的改进与比较. 分子植物育种, 2006, 4(5): 721-725.
[18] 李宏, 王新力. 植物组织RNA提取的难点及对策. 生物技术通报, 1999, (1): 36-39.
[23] 马婧, 王斌, 代银, 等. 金荞麦无色花色素还原酶基因FdLAR的克隆和表达分析. 药学学报, 2012, 47(7): 953-961.
[25] 沈宝云, 刘玉汇, 张俊莲, 等. 马铃薯块茎GBSSI基因的cDNA克隆及其序列特征分析. 草业学报, 2010, 19(5): 1-8.
[26] 李立芹, 黄玉碧, 王西瑶. 马铃薯WRKY6基因的克隆、序列分析与原核表达研究. 草业学报, 2011, 20(2): 177-183.
[27] 吕慧颖, 李银心, 孔凡江, 等. 植物Na+/H+逆向转运蛋白研究进展. 植物学通报, 2003, 20(3): 363-369.
Cloning and Expression Analysis of a Leucoanthocyanidin Reductase (LAR) Gene fromOnobrychisviciifoliacv.Gansu
CHEN Chun-Yan1,2,3,4, MA Hui-Ling1,3,4*, DONG Wen-Ke1,3,4
1.CollegeofPrataculturalScience,GansuAgriculturalUniversity,Lanzhou730070,China; 2.CollegeofAgronomy,HenanUniversityofScienceandTechnology,Luoyang471003,China; 3.KeyLaboratoryofGrasslandEcosystem,MinistryofEducation,PrataculturalEngineeringLaboratoryofGansuProvince,Lanzhou730070,China; 4.Sino-U.S.CenterforGrazinglandEcosystemSustainability,Lanzhou730070,China
A leucoanthocyanidin reductase (LAR) gene was cloned using RT-PCR fromOnobrychisviciifolia(sainfoin) cv. Gansu, and the level of LAR gene expression was analyzed in different plant organs. The LAR gene sequence fromO.viciifoliacv. Gansu shared a high level of similarity (98.34% homology) with anO.viciifoliaLAR gene in GenBank (accession No: HM152980). However, the coding sequence of amino acids is obviously different from those in other genera of the Fabaceae. The open reading frame (ORF) comprised 1089 bp, encoding 362 amino acid residues. The sequence of the LAR gene from the present study was submitted to GenBank with the accession number KP013623. LAR expression differed between plant organs ofO.viciifoliacv. Gansu. LAR expression was highest in leaves, followed by flowers and fruit, and was lowest in stem tissues, which corresponded to concentration of condensed tannins in different organs ofO.viciifoliacv. Gansu. It is inferred that LAR gene expression is in some way involved in condensed tannin accumulation inO.viciifoliacv.Gansu. These results provide fundamental information and a method for exploring the regulation mechanism of condensed tannin accumulation.
Onobrychisviciifoliacv.Gansu; RT-PCR; leucoanthocyanidin reductase; cloning and expression
10.11686/cyxb2014452
http://cyxb.lzu.edu.cn
2014-11-03;改回日期:2015-01-07
甘肃省科技厅科技支撑计划项目(1104NKCA087)和国家自然科学基金项目(31200299)资助。
陈春艳(1977-),女,甘肃兰州人,实验师,在读博士。E-mail:ccy0713@126.com *通讯作者Corresponding author. E-mail:mahl@gsau.edu.cn
陈春艳,马晖玲,董文科. 甘肃红豆草无色花青素还原酶LAR基因的克隆和表达分析. 草业学报, 2015, 24(6): 177-187.
Chen C Y, Ma H L, Dong W K. Cloning and Expression Analysis of a Leucoanthocyanidin Reductase (LAR) Gene fromOnobrychisviciifoliacv.Gansu. Acta Prataculturae Sinica, 2015, 24(6): 177-187.
——“单宁”

