基于线粒体COI序列的洪泽湖河蚬(Corbicula fluminea)遗传多样性和种群结构分析*
李大命 张彤晴 唐晟凯 钟立强 刘小维
(江苏省淡水水产研究所 江苏省内陆水域渔业资源与环境重点实验室 南京 210017)
河蚬(Corbicula fluminea),俗称“黄蚬”,是一种栖息于淡水、咸淡水的常见双壳贝类,广泛分布于世界各地水域,并成为河流湖泊等淡水生态系统的优势种,对水生生态系统的物质循环和能量流动有重要影响(Hakenkamp et al,1999;Perceval et al,2002)。同时河蚬具有很高的营养价值,国内外市场对河蚬的需求量越来越大,其经济价值亦不断上涨,河蚬养殖业已成为淡水经济贝类养殖的热点之一。湖泊富营养化、栖息环境破坏和捕捞过度等原因造成河蚬天然资源量减少,严重制约了河蚬产业的可持续发展。洪泽湖是中国第四大淡水湖泊,河蚬是其底栖动物群落中的常见优势种(韩爱民等,2002;张超文等,2012),也是洪泽湖重要的出口创汇水产品,2007年12月在洪泽湖临淮水域建立了首个国家级河蚬自然保护区。调查研究结果(袁永浒等,1994;严维辉等,2007)表明,洪泽湖河蚬产量从20世纪80年代初期的32万吨下降至90年代初的21.29万吨。近几年来洪泽湖河蚬资源量呈持续下降趋势,引起了政府相关部门的高度重视,加强洪泽湖河蚬种质资源保护已成为当务之急。国内外有关河蚬的研究主要集中在基础生物学、营养价值及环境毒理学等方面。有关洪泽湖河蚬遗传多样性的研究,仅见丁怀宇等(2011)采用微卫星标记技术分析洪泽湖河蚬野生群体的遗传结构。
种群遗传多样性是保护遗传学重要的研究内容,也是制定保护策略的重要前提和基础(Avise,1989;Waits et al,1989)。DNA分子标记是一种检测物种遗传多样性和遗传结构的常用方法。在众多的DNA分子标记中,线粒体 DNA(mtDNA)是一种重要的分子标记,在水生动物进化生物学和群体遗传学研究中被广泛采用(刘云国等,2009)。COI基因序列是mtDNA中的一段,进化速率适中,是mtDNA最常用的分子标记之一(Martínez-Navarro et al,2005),其在双壳贝类分子系统学研究中已有较多文献报道(Giribet et al,2002;Lee et al,2005)。本研究首次采用COI基因作为分子标记,调查洪泽湖河蚬种质资源遗传多样性现状及其种群遗传结构,以期为洪泽湖河蚬资源保护和可持续开发利用提供科学依据。
1 材料与方法
1.1 实验材料
2014年 5月中旬对洪泽湖河蚬资源量分布和水环境状况进行了调查分析。根据研究结果,在洪泽湖的顾勒河、临淮、蒋坝和高良涧水域各设置1个采样点),随机采集河蚬野生群体(分别用 GLH、LH、JB和GLJ表示 4个河蚬群体),采样点位置和样本量见表1和图1。河蚬用无水乙醇固定后带回实验室,将外套膜肌肉组织从壳中取出装入离心管中,加入无水乙醇,置于–20°C保存。
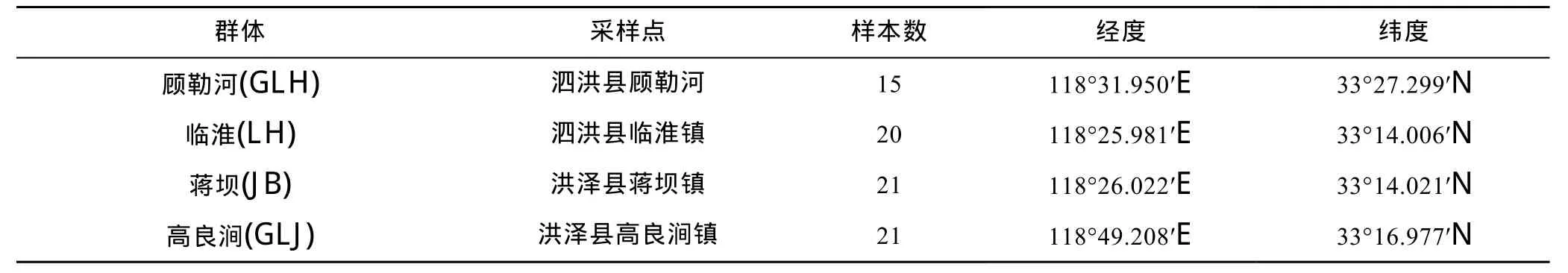
表1 采样点位置和河蚬群体样本数Tab.1 Sampling locations and the number of C.fluminea populations in Hongze Lake
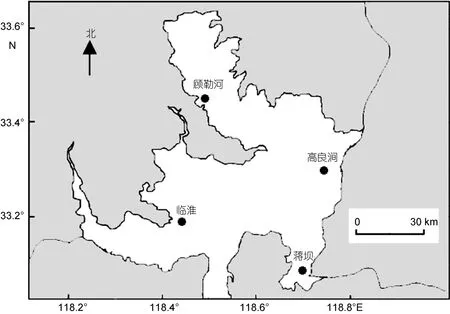
图1 洪泽湖河蚬采样点分布Fig.1 The distribution of sampling locations in Hongze Lake
1.2 DNA提取和检测
取河蚬外套膜肌肉组织作为提取DNA的材料。采用常规苯酚-氯仿法提取基因组 DNA,加入200µLTE溶液溶解。DNA完整性采用1%的琼脂糖凝胶电泳鉴定,浓度采用紫外分光光度计测定。
1.3 PCR扩增及序列测定
PCR扩增采用文献报道 COI基因的通用引物(Folmer et al,1994)。上游引物为 LCO1490: 5´-GGTC AA-CAAATCATAAAGATATTGG-3´),下 游 引 物 为HCO2198: 5´-TAAACTTCAGGGTGACCAAAAAATCA-3´。引物均由上海生工生物工程股份有限公司合成。
PCR反应总体积为 50µL,其中2×PCR mix(包含Taq酶2.5µmol,dNTPs 10µmol,MgCl20.1mmol)25µL,上下游引物(10µmol)各 2µL,DNA 模板 2.0µL,ddH2O补足体积。PCR反应条件为: 94°C预变性4min,94°C变性40s,50°C退火50s,72°C延伸1min。30个循环;72°C最后延伸10min。
取 PCR产物于 1.5%的琼脂糖凝胶电泳检测,Goldview染色,对凝胶成像进行观察和拍照。采用琼脂糖凝胶纯化试剂盒(Takara)将PCR产物纯化后送至上海生工生物工程股份有限公司进行双向测序。
1.4 数据分析
测序获得的COI序列通过Chromas1.45软件获得原始序列数据;采用用ClustalX 1.83软件对序列进行比对,并辅以人工校正。用DnaSP 5.0软件计算单倍型数、单倍型多样性(haplotype diversity,h)和核苷酸多样性(nucleotide diversity,π)等;利用 Arlequin 3.1软件中的分子变异分析(AMOVA)方法估算遗传在群体间和群体内的分布,并计算群体间遗传分化系数Fst及其显著性。用 Mega4.0软件统计COI基因序列碱基组成,并基于 Kimura 2-parameter遗传距离,用临接法(Neighbor-joining,NJ)构建单倍型系统进化树。利用Tajima’s D和Fu’Fs中性检验和核苷酸不配对分布检测河蚬群体的历史动态。
2 结果与分析
2.1 河蚬mtDNA COI基因序列特征
本研究对洪泽湖河蚬的mtDNA COI基因片段进行PCR扩增、测序和比对,获得614bp COI基因片段。经过分析,所有序列中,A、T、G和C4种碱基平均含量分别为22.6%、42.4%、21.0%和14.0%。A+T的含量为65.0%,明显高于G+C的含量35.0%(表2),表现出较为明显的碱基组成偏倚性。但4个群体间的COI基因序列碱基组成无明显差异。
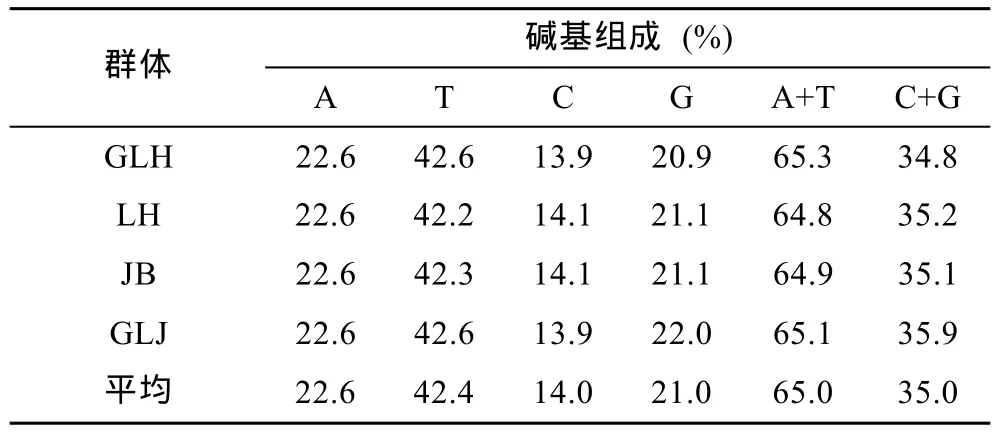
表2 河蚬群体的mtDNA COI基因序列碱基组成Tab.2 The base composition of COI gene fragments of C.fluminea populations in Hongze Lake
COI序列共包含 73个核苷酸变异位点,占分析位点总数的11.9%,其中包括66个简约信息位点和7个单一信息位点,没有插入和缺失位点。4个群体77条COI序列中共定义了22个单倍型(表3),其中单倍型 H5和 H13出现的次数较多,单倍型 H5、H13和H14为4个河蚬群体共享单倍型,单倍型H1为顾勒河群体特有的单倍型,单倍型H2、H8、H9、H17和H18为蒋坝群体特有的单倍型,单倍型H3、H7、H11、H15和H22为临淮群体特有的单倍型,单倍型H4、H19和H21为高良涧群体特有的单倍型。
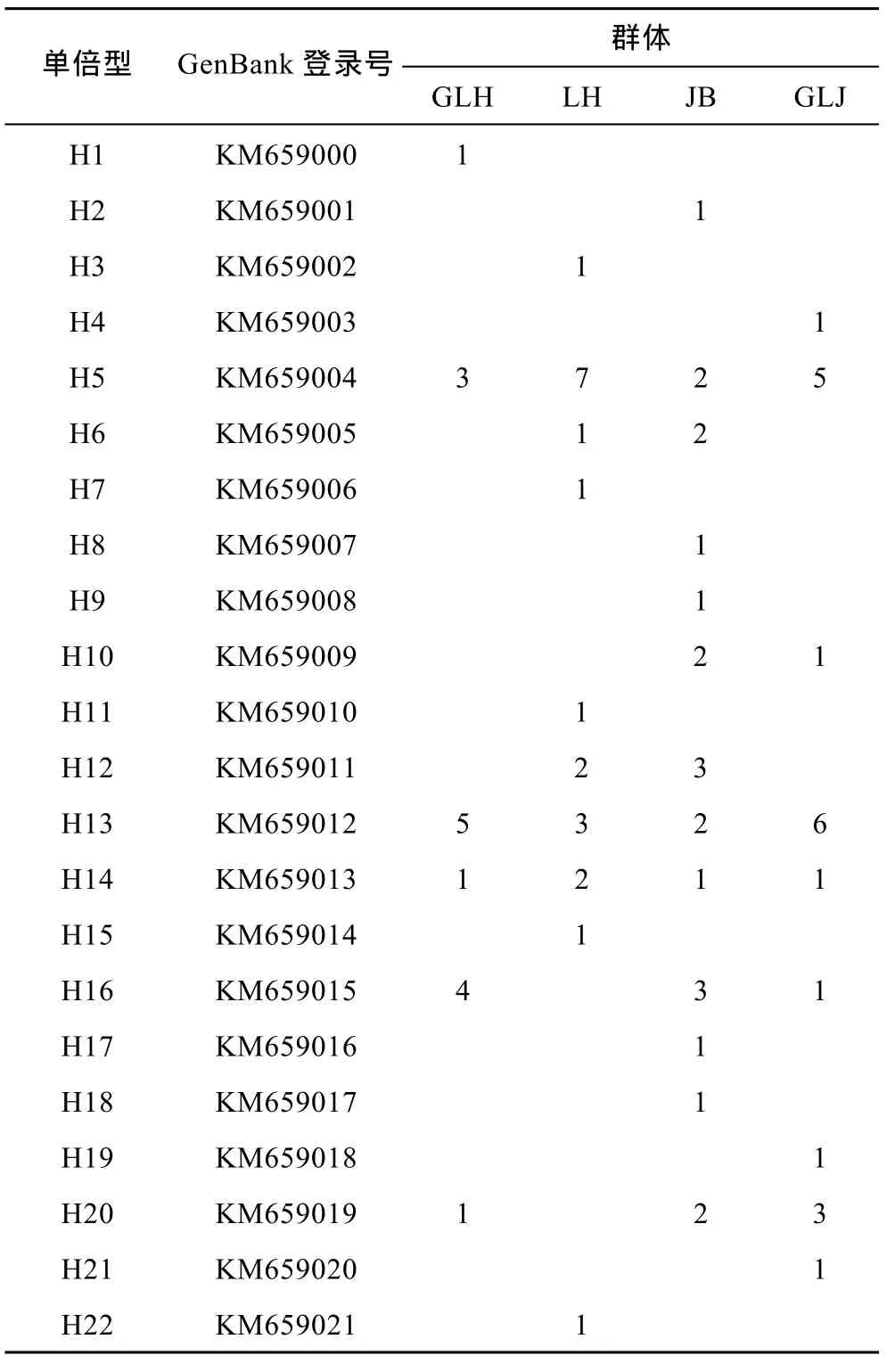
表3 河蚬COI基因单倍型(H1—H22)在4个群体中的分布Tab.3 Distribution of COI gene haplotypes in the four C.fluminea populations
2.2 河蚬种群COI基因的遗传多样性分析
基于 COI基因所有序列得到的 4个河蚬群体的单倍型数及遗传多样性参数如表4所示。从表4可以得出,4个河蚬群体的单倍型多样性指数为0.889±0.020,核苷酸多样性指数为 0.04499±0.00127,平均核苷酸差异数为27.622,这表明洪泽湖河蚬种群具有较丰富的遗传多样性。另外,4个河蚬群体的遗传多样性高低存在差异,蒋坝群体的遗传多样性最 大,顾勒河群体的遗传多样性最小。
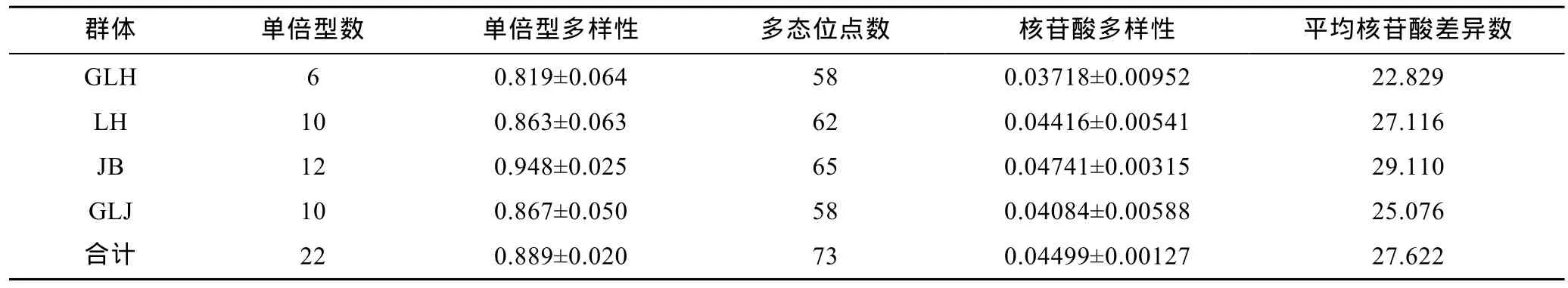
表4 洪泽湖河蚬群体的遗传多样性Tab.4 Genetic diversity of the four C.fluminea populations in Hongze Lake
2.3 河蚬群体遗传变异和遗传结构
利用MEGA4.0软件中Kumara双参数模型计算群体间的遗传距离(表 5)。由表可知,顾勒河和临淮群体之间的遗传距离最远为 0.04969,顾勒河与高良涧群体间的遗传距离最近为 0.03729。基于群体间的遗传距离,构建4个群体之间的NJ系统树(图2),结果表明,顾勒河群体和高良涧群体聚为一支,蒋坝群体和临淮群体聚为一支。
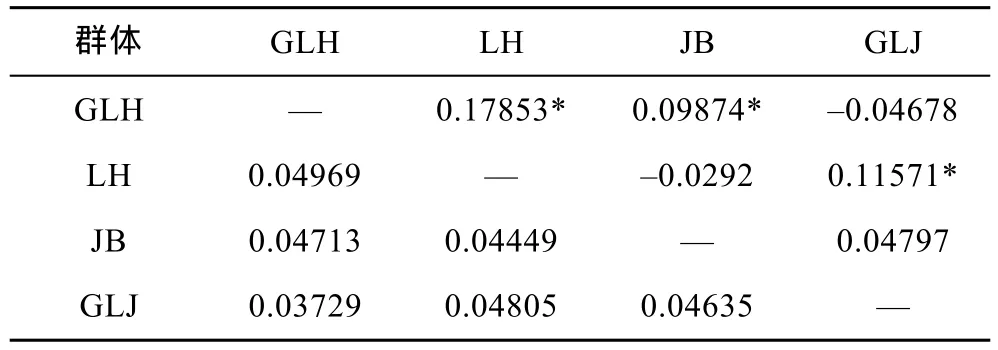
表5 洪泽湖河蚬群体间的遗传分化系数(Fst)(对角线上)和遗传距离(对角线下)Tab.5 Fixation index (above diagonal)and genetic distances(below diagonal)among C.fluminea populations in HongzeLake

图2 四个河蚬群体的NJ聚类树Fig.2 Dendrogram of the four C.fluminea populations by based on the genetic distance in NJ method
为进一步分析河蚬群体间的遗传变异组成,采用 Arlequin软件对 4个河蚬群体进行分子方差分析(AMOVA)。结果显示,93.67%的遗传变异出现在种群内,仅有 6.33%出现在种群间,群体间总的遗传分化系数Fst=0.06325(P>0.05)(表6),说明4个群体间无显著遗传分化。两两群体间的遗传分化系数如表5所示,可以得出,顾勒河群体与蒋坝和临淮群体及高良涧群体与临淮群体间的遗传分化系数具有显著性差异(P<0.05),而顾勒河与高良涧和蒋坝及蒋坝与临淮群体间的遗传分化系数为负值,表明这两组群体间无遗传分化。
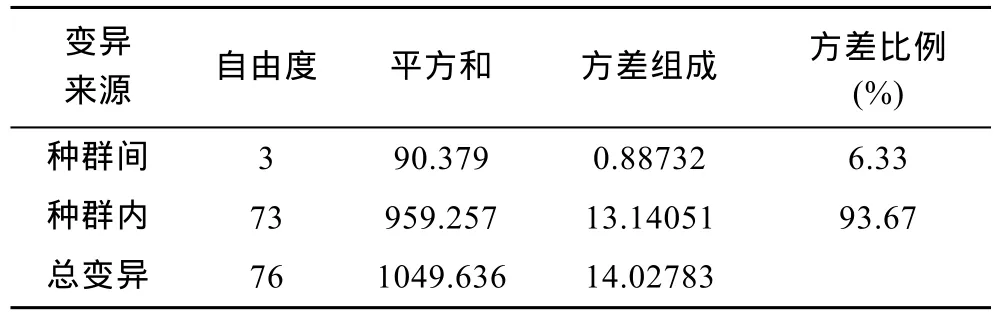
表6 河蚬4个群体间遗传差异的分子方差分析表(AMOVA)Tab.6 Analysis of molecular variance (AMOVA)in the four populations of C.fluminea
2.4 COI序列单倍型NJ系统树
基于Kumara双参数模型的单倍型间的遗传距离在 0.002—0.100,平均遗传距离为 0.051。利用MEGA4.0软构建mtDNA COI基因单倍型NJ系统树,并对所得的系统树进行自展法检验(1000次重复)(图3)。结果显示,22个COI单倍型聚为2个明显分支,且有很高的置信度支持,其中单倍型H1到H12聚为一支(I),单倍型H13到H22聚为一支(II)。分支内单倍型之间的遗传距离较小,而分支间的单倍性遗传距离较大。同时可以看出,4个河蚬群体的COI单倍型个体交错在一起,不具有明显的地理结构,说明群体间无明显的遗传分化。
2.5 河蚬种群动态分析
利用Tajima’s D和Fu’s Fs中性检验和歧点分布分析洪泽湖河蚬是否经历种群扩张。中性检验结果如表7所示,中性检验中Tajima’s D值和Fu's Fs值均为正,除顾勒河群体外,Tajima’s D检验结果均为显著或极显著;所有群体的 Fu’s Fs检验结果均为显著或极显著。所有河蚬个体的碱基歧点分布(mismatch–distribution)分布图谱呈现双峰型(图4),这些结果表明洪泽湖河蚬种群大小保持稳定,未发生明显的种群扩张。
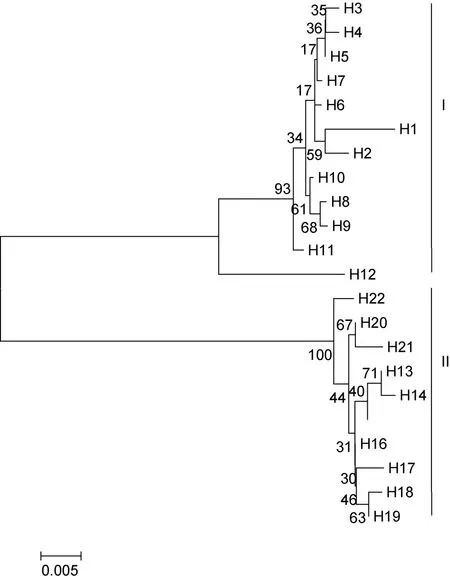
图3 河蚬COI序列22个单倍型的NJ分子系统树Fig.3 NJ tree among 22 haplotypes of COI sequences of C.fluminea
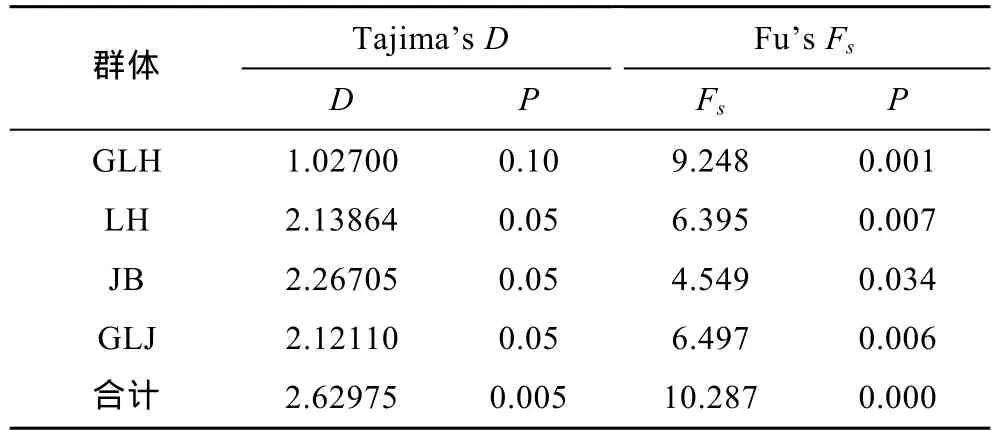
表7 洪泽湖河蚬种群的中性检验结果Tab.7 Results of neutral test for natural selection on C.fluminea populations in Hongze Lake
3 讨论
3.1 河蚬种群遗传多样性分析
mtDNA是生物系统进化和群体遗传多样性研究最常用的分子标记之一,4种核苷酸A、T、G和C在线粒体基因组中的分布呈不均匀性,是动物线粒体基因组的一个基本特征。本研究中洪泽湖河蚬mtDNA COI碱基序列中A+T含量为65.0%,明显高于 G+C含量(35.0%),这与三角帆蚌(李家乐等,2008)、褶纹冠蚌(贾名静等,2009)、厚壳贻贝(李咏梅等,2009)、紫贻贝(沈玉帮等,2011)、牡蛎(毛阳丽等,2014)等双壳贝类的 mtDNA COI基因序列碱基组成具有相似特点,表明双壳贝类中 mtDNA序列碱基组成中表现出很强的偏倚性。
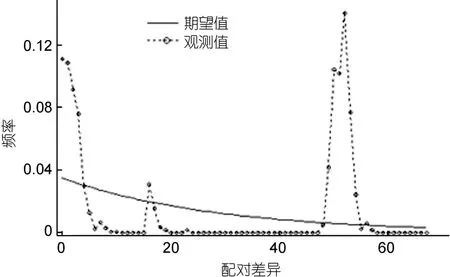
图4 洪泽湖河蚬种群的歧点分布图Fig.4 Mismatch distribution analysis of C.fluminea populations in Hongze Lake
遗传多样性是形成生物多样性的基础,与生物的适应能力、生存能力和进化潜力密切相关。单倍型多样性指数(h)及核苷酸多样性指数(π)是衡量一个种群遗传变异的2个重要指标。本研究所分析的洪泽湖4个河蚬种群 77个体间的单倍型序列显示,mtDNA COI基因单倍型序列种类比较丰富,总数达到22个,占全部序列的28.6%,平均单倍型多样性和核苷酸多样性分别为0.889±0.020和 0.04499±0.00127,表现出很高的遗传多样性,这为开展洪泽湖河蚬人工繁殖和选育提供了种质保障。有学者通过对线粒体 DNA序列的遗传变异分析,将不同核苷酸多样性和单倍型多样性间的组合分成了四种类型: 低 h(h<0.5)和低π(π<0.005)、高h和低π、低h和高π和高h和高π(Grant et al,1998)。本研究结果显示洪泽湖河蚬种群具有较高h和π,说明洪泽湖河蚬种质资源遗传多样性比较丰富。从历史进化的角度看,这种高h和高π的遗传多样性模式通常是由一个较大而稳定的有效种群经过长时间进化所产生。从生物学和生态学的角度来讲,大的有效群体、环境异质性以及适于种群快速增长的生活习性是维持自然种群较高的遗传多样性的基础(Nei,1987)。河蚬是我国的土著种类,在淡水生态系统中分布广泛。调查表明,河蚬是洪泽湖底栖动物群落中的第一优势种类,生物量比较大(张超文等,2012)。同时河蚬具有很强的环境适应能力和较快的繁殖速度,这些因素均有利于保持洪泽湖河蚬丰富的遗传多样性。
从遗传多样性参数得出(表4),4个河蚬群体间的遗传多样性存在差异,其遗传多样性大小顺序是蒋坝群体>临淮群体>高良涧群体>顾勒河群体。有研究者采用微卫星技术对洪泽湖调查了洪泽湖 4个河蚬群体(临淮、蒋坝、老子山和高良涧)的遗传多样性及其种群遗传结构,结果表明: 蒋坝、临淮群体遗传多样性高于老子山和高良涧群体(丁怀宇等,2011)。这与本研究利用 COI分子标记得到的结果一致。导致洪泽湖河蚬群体间遗传多样性差异的主要原因是 4个群体所处的地理位置及水域环境差异(丁怀宇等,2011)。研究者对洪泽湖水体富营养化水平和水环境状况进行调查分析,发现不同湖区水环境质量存在差异,临淮和蒋坝群体所在水域的水生态环境显著好于高良涧和顾勒河群体所在水域(范亚民等,2008;吴延东等,2010)。另外临淮和蒋坝群体分别位于洪泽湖上游入水口和下游出水口,河湖之间的水流交换较频繁,湖区水体流动性好于高良涧群体和顾勒河群体所在水体。而河蚬喜欢生活在水流大且开放的水体中,这种环境有利于其生长和繁殖(严维辉等,2007)。因此,加强洪泽湖生态环境保护,降低水体富营养化水平,改善水环境质量,有利于保护洪泽湖河蚬种质资源遗传多样性。
3.2 河蚬种群遗传结构分析
遗传分化系数 Fst是衡量群体间遗传分化大小的重要指标,其值在0到1之间,数值越大表明群体间的遗传分化越显著(Wright,1978)。AMOVA分析结果显示,河蚬种群间总的遗传分化指数 Fst为0.06325(P>0.05),表明4个河蚬群体间遗传分化程度较低,没有形成显著的遗传结构。在4个河蚬种群中,顾勒河群体与临淮和蒋坝群体、高良涧与临淮群体间的遗传分化显著(P<0.05),而顾勒河与高良涧群体、蒋坝与高良涧和临淮群体间的遗传分化不显著(P>0.05),这也与群体间的遗传距离和 NJ系统树聚类分析结果对应。顾勒河群体与高良涧群体间的遗传距离最小为 0.03729,顾勒河群体与临淮群体间的遗传距离最大为 0.04969,都远小于物种间遗传距离的临界值(Thorp,1982)。COI单倍型 NJ系统树显示不同群体间的单倍型呈交错分布,没有明显的地理聚群和谱系结构。群体间的AMOVA分析结果、遗传距离和COI单倍型系统进化树均表明,洪泽湖4个河蚬种群是随机交配的群体,群体间没有明显的遗传分化,这与丁怀宇等(2011)的结论一致。
基因流能减少群体间的遗传分化(Lenormand et al,1998)。当群体间的基因流大于1,能有效抑制群体间的遗传分化(Wright,1931)。本研究发现4个河蚬群体间基因流 Nm为 3.42,单倍型间的基因流 Nm为21.00,表明河蚬群体间的基因交流比较频繁。这与河蚬生活史有密切关系。河蚬受精卵发育过程中经历较长时间的浮游幼虫阶段,可以随水体交换和湖流运动而随机扩散,导致不同群体间产生基因交流。其次,洪泽湖是一个大型浅水性湖泊,4个群体同处洪泽湖湖区内,地理位置差异不大,群体间没有能够形成地理隔离的天然屏障。另外,频繁的生产捕捞活动可以有利于河蚬在湖区间的运动和迁移,从而促进不同区域的河蚬间的基因流动。整体来看,洪泽湖河蚬群体进化并未形成明显的种群遗传结构。因此,可以将洪泽湖河蚬种质资源作为一个整体进行保护和管理。
3.3 河蚬种群历史动态
通常借助于两种方法检测种群历史动态。一是采用 Tajima’s D(Tajima,1989)和 Fu’s Fs(Fu,1997)中性检验来验证,如果Tajima’s D和Fu’s Fs检验的统计值是负值并且显著性差异,可能是群体扩张引起的偏离中性进化;二是根据岐点分布曲线图来判别,如果种群呈现稳定状态,在岐点分布图谱上会呈现多峰,反之则呈单峰 (Rogers et al,1992;Rogers,1995)。本研究结果显示,将4个河蚬群体作为一个整体进行中性检测的 Tajima’s D 值和 Fu’s Fs值均为正值(P<0.01)(表 7),且岐点分布图呈现不规则的双峰型,表明河蚬种群保持相对稳定,未经历过近期的种群扩张;但河蚬种群进化过程偏离中性选择,这可能是由于基因突变、自然选择或非平衡选择所造成,进而使河蚬种群具表现出较丰富的遗传多样性水平,导致了 COI单倍性出现明显遗传分化,NJ系统树结果也表明COI单倍型分为2个明显分支。
3.4 资源保护
河蚬是洪泽湖底栖动物的优势种类,具有较高的经济价值和生态价值。由于过度捕捞、环境污染和湖泊富营养化等多种因素导致河蚬野生资源量急剧下降,资源量减少会进一步导致群体遗传多样性降低,进而危及其自身的延续能力,严重制约洪泽湖河蚬产业的可持续发展。因此,必须加强对洪泽湖河蚬种质资源的保护。我们提出以下保护措施: (1)建立严格的休渔期制度,严禁繁殖季节捕捞河蚬,严格控制捕捞量,禁止使用带有毁灭性的捕捞工具;(2)加强对河蚬繁殖生物学及早期生活史的研究,开展人工繁殖,进行人工增殖放流,努力恢复其资源量;(3)在遗传多样性水平较高的蒋坝和临淮湖区建立河蚬种质资源保护区,加强保护区水质监测和环境保护,严禁在保护区内进行人类活动,降低洪泽湖富营养化水平,提高水环境质量。
4 结论
本研究首次利用mtDNA COI分析洪泽湖河蚬群体的遗传多样性和群体遗传结构,为其种质资源的遗传多样性评估、保护利用及良种选育中亲本的选择奠定了理论基础。结果表明,洪泽湖河蚬种质资源具有较高的遗传多样性,湖区间河蚬种群的遗传多样性存在差异,湖区间河蚬种群的遗传分化程度较低,没有形成显著的遗传结构。针对洪泽湖资源现状及其发展趋势,提出了保护种质资源的措施。今后需要进一步调查分析洪泽湖河蚬资源量及其遗传多样性的时空变化,找出相关的主导环境因子,为合理开发和可持续利用河蚬资源、充分发挥其经济价值和生态价值提供参考。
丁怀宇,姜虎成,冯建彬等,2011.河蚬微卫星引物筛选及洪泽湖野生群体遗传结构分析.水产学报,35(11):1624—1632
刘云国,刘贤德,高 焕等,2009.水产生物DNA分子标记技术.北京: 科学出版社,59—62
严维辉,潘元潮,郝 忱等,2007.洪泽湖底栖生物调查报告.水利渔业,27(3): 65—66
李咏梅,陈秀荔,彭 敏等,2009.基于线粒体COI基因序列探讨广西钦州湾牡蛎的遗传分化.西北农林科技大学学报(自然科学版),37(3): 60—65
李家乐,王建军,汪桂玲等,2008.我国五大淡水湖三角帆蚌群体 mtDNA COⅠ基因片段变异分析.水生生物学报,32(5): 779—782
杨振雄,毛阳丽,宋 娜等,2014.浙江和福建沿海厚壳贻贝Mytilus coruscus群体的 COI序列比较分析.海洋湖沼通报,(2): 82—88
吴延东,刘绪庆,陈飞龙,2010.洪泽湖水质状况的主成分分析和聚类分析.淮阴工学院学报,19(1): 71—76
沈玉帮,张俊彬,冯冰冰等,2011.基于线粒体 COI序列分析对紫贻贝群体遗传多样性的研究分析.海洋通报,30(4):435—440
张超文,张堂林,朱挺兵等,2012.洪泽湖大型底栖动物群落结构及其与环境因子的关系.水生态学杂志,33(3):27—33
范亚民,何华春,崔云霞等,2008.洪泽湖水域的环境演变遥感分析.污染防治技术,21(6): 29—33,84
袁永浒,王兴元,陈安来等,1994.洪泽湖螺蚬资源调查报告.水产养殖,(6): 15—16
贾名静,李家乐,牛东红等,2009.长江中下游褶纹冠蚌10个群体COⅠ基因序列变异分析.动物学杂志,44(1): 1—8
韩爱民,杨广利,张书海等,2002.洪泽湖富营养化和生态状况调查与评价.环境监测管理与技术,14(6): 18—19,22
Avise J C,1989.A role for molecular genetics in the recognition and conservation of endangered species.Trends in Ecology&Evolution,4(9): 279—281
Folmer O,Black M,Hoeh W et al,1994.DNA primers for amplification of mitochondrial cytochrome coxidase subunit I from diverse metazoan invertebrates.Molecular Marine Biology and Biotechnology,3(5): 294—299
Fu Y X,1997.Statistical tests of neutrality of mutations against population growth,hitchhiking and background selection.Genetics,147(2): 915—925
Giribet G,Wheeler W,2002.On bivalve phylogeny: a high-level analysis of the Bivalvia (Mollusca)based on combined morphology and DNA sequence data.Invertebrate Biology,121(4): 271—324
Grant W A S,Bowen B W,1998.Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation.Journal of Heredity,89(5): 415—426
Hakenkamp C C,Palmer M A,1999.Introduced bivalves in freshwater ecosystems: the impact of Corbicula on organic matter dynamics in a sandy stream.Oecologia,19(3):445—451
Lee T,Siripattrawan S,Ituarte C F et al,2005.Invasion of the clonal clams: Corbicula lineages in the New World.American Malacological Bulletin,20(1): 113—122
Lenormand T,Guillemaud T,Bourguet D et al,1998.Evaluating gene flow using selected markers: a case study.Genetics,149(3): 1383—1392
Martínez-Navarro E M,Galián J,Serrano J,2005.Phylogeny and molecular evolution of the tribe Harpalini (Coleoptera,Carabidae)inferred from mitochondrial cytochrome-oxidase I.Molecular Phylogenetics and Evolution,35(1): 127—146 Nei M,1987.Molecular Evolutionary Genetics.New York:Columbia University Press,185
Perceval O,Pinel-Alloul B,Méthot G et al,2002.Cadmium accumulation and metallothionein synthesis in freshwater bivalves (Pyganodon grandis): relative influence of the metal exposure gradient versus limnological variability.Environmental Pollution,118(1): 5—17
Rogers A R,Harpending H,1992.Population growth makes waves in the distribution of pairwise genetic differences.Molecular Biology and Evolution,9(3): 552—569
Rogers A R,1995.Genetic evidence for a Pleistocene population explosion.Evolution,49(4): 608—615
Tajima F,1989.Statistical method for testing the neutral mutation hypothesis by DNA polymorphism.Genetics,123(3): 585—595
Thorpe J P,1982.The molecular clock hypothesis: Biochemical evolution,genetic differentiation and systematics.Annual Review of Ecology and Systematics,13(1): 139—168
Waits L P,Talbot S L,Ward R H et al,1998.Mitochondrial DNA phylogeography of the North American brown bear and implications for conservation.Conservation Biology,12(2): 408—417
Wright S,1931.Evolution in Mendelian population.Genetics,16:91—159
Wright S,1978.Evolution and the genetics of populations.Chicago: University of Chicago Press,499—506

